文章信息
- 四种长链非编码RNA在乳腺癌中的表达及与患者预后的关系
- Expression of Four Long Non-coding RNAs in Breast Cancer and Their Relation with Patient's Prognosis
- 肿瘤防治研究, 2020, 47(2): 90-96
- Cancer Research on Prevention and Treatment, 2020, 47(2): 90-96
- http://www.zlfzyj.com/CN/10.3971/j.issn.1000-8578.2020.19.0617
- 收稿日期: 2019-05-13
- 修回日期: 2019-11-07
2. 253023 德州,德州学院生命科学学院;
3. 253012 德州,德州市立医院普外科
2. College of Life Science, Dezhou University, Dezhou 253023, China;
3. Department of General Surgery, Dezhou Municipal Hospital, Dezhou 253012, China
乳腺癌是危及女性健康最常见的一种恶性疾病[1]。在接受手术、化疗等治疗方式之后仍易复发转移。而且,乳腺癌是高度异质性癌症,不同患者乳腺癌组织的免疫表型、组织形态、治疗反应和预后等均存在较大差异。乳腺癌现有的肿瘤标志物还不能满足临床需求,亟需发现新的肿瘤标志物,以实现乳腺癌的个体化精准治疗[2]。
研究发现,长链非编码RNA(LncRNA)的表达水平与乳腺癌的发生、发展甚至是诊疗、预后密切相关[3-4],这为我们治疗乳腺癌提供了新的线索。近年来由于高通量技术的快速发展,肿瘤领域产生了大量的高通量测序数据和表达谱数据,而且,研究人员已建立了诸多关于癌症的重要数据库,如:ONCOMINE[5]、TCGA[6]等。
通过数据库[7-8]检索LncRNA相关的文献,本文选取EGOT、MEG3、KCNQ1OT1和NEAT1四种与乳腺癌关系报道极少的LncRNA,通过UALCAN[9]网站挖掘TCGA数据库中的相关数据分析它们与不同乳腺癌患者的关系,通过Kaplan-Meier Plotter网站[10]分析它们与乳腺癌患者预后的关系,通过LinkedOmics网站[11]和ClueGO工具分析与它们表达相关的蛋白质的主要功能,为乳腺癌的精准治疗、靶向治疗以及预后评估提供线索。
1 资料与方法 1.1 LncRNA在乳腺癌中的表达分析通过UALCAN(http://ualcan.path.uab.edu/analysis.html)网站对四种LncRNA在乳腺癌组织与正常组织中的表达进行分析。UALCAN网站可以在线对TCGA数据库中的数据进行深入挖掘,不仅可以对基因在癌症组织和正常组织中的差异表达进行分析,还可以比较不同临床病理特征的患者之间的表达差异,比如年龄、种族、癌症分期、亚型等,并给出统计学检验结果。TCGA数据库,即肿瘤基因组图谱计划,目前共收录33种癌症的高通量测序数据和患者的性别、年龄、肿瘤分期、复发及预后情况等临床信息。
1.2 LncRNA与乳腺癌预后的关系分析通过Kaplan-Meier Plotter(http://kmplot.com/analysis/)网站对四种LncRNA与乳腺癌预后的关系进行分析,得到乳腺癌患者的无复发生存时间(relapse-free survival, RFS)与LncRNA表达量的Kaplan-Meier图,并且图中给出Log rank检验的P值。若P < 0.05,说明LncRNA表达量与乳腺癌患者的预后显著相关。
1.3 与LncRNA表达相关的蛋白质分析通过LinkedOmics网站分析与LncRNA表达具有相关性的蛋白质,并获得与LncRNA表达极显著相关(P < 0.01)的蛋白质。然后,使用Cytoscape软件中的ClueGO工具进行GO功能注释和KEGG通路富集分析,探寻LncRNA与乳腺癌发生相关的分子机制。
1.4 统计学方法不同组间LncRNA的表达差异比较使用T检验。Kaplan-Meier方法进行生存分析,生存率的比较采用Log rank检验。Pearson相关系数分析LncRNA和蛋白质的表达相关性。使用超几何富集分析方法对与LncRNA表达相关的蛋白质进行GO功能注释和KEGG通路富集分析。P < 0.05为差异有统计学意义。
2 结果 2.1 LncRNA在乳腺癌患者中的表达水平UALCAN数据库共有1 097个乳腺癌组织样本及114个正常组织样本。按患者的种族分类,白种人患者样本748个,黑种人患者样本179个,黄种人患者样本61个。按患者的性别分类,男性患者仅有12例,女性患者为1 075例。按患者的年龄段分类,21~40岁97例,41~60岁505例,61~80岁431例,81~100岁54例。
结果表明,EGOT在乳腺癌组织中的表达显著低于正常组织(P=0.0494),见图 1A。在对不同种族的乳腺癌患者进行分析时,发现除白种人外,黑种人(P=0.0001)和黄种人(P=0.0003)乳腺癌组织中EGOT的表达水平显著低于正常组织,见图 1B。女性患者癌组织中EGOT的表达水平显著低于正常组织(P=0.0045),见图 1C。EGOT在21~40岁(P=0.0391)、81~100岁患者癌组织中的表达水平显著低于正常组织(P=0.0313),而其他年龄段与正常组织没有显著差异,见图 1D。

|
| *:P < 0.05;**:P < 0.01; BRCA: Breast invasive carcinoma; Yrs: years; TCGA: The Cancer Genome Atlas. 图 1 EGOT在乳腺癌及正常组织中的表达 Figure 1 Expression of EGOT in breast cancer and normal samples |
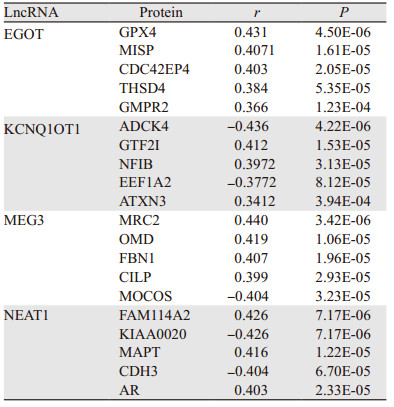
MEG3在乳腺癌组织中的表达显著低于正常组织(P < 1×10-12),见图 2A。白种人(P=1.62×10-12)、黑种人(P=2.83×10-12)和黄种人(P=1.62×10-12)乳腺癌组织中MEG3的表达水平均显著低于正常组织,见图 2B。男性(P=0.0001)与女性患者(P=1.62×10-12)中MEG3的表达水平均显著低于正常组织,但男女患者之间没有显著差异,见图 2C。MEG3在所有年龄段患者癌组织中的表达水平均显著低于正常组织(P < 0.01),见图 2D。
|
| *:P < 0.05;**:P < 0.01. 图 2 MEG3在乳腺癌及正常组织中的表达 Figure 2 Expression of MEG3 in breast cancer and normal samples |
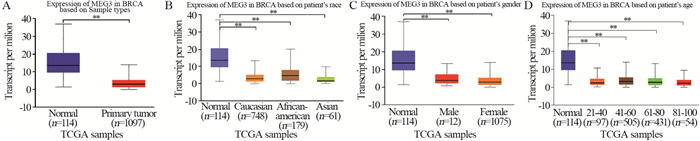
虽然KCNQ1OT1在乳腺癌组织中的表达水平与正常组织无显著差异,但是在黑种人癌组织中的表达水平显著低于正常组织(P=4.19×10-5),见图 3A。女性患者KCNQ1OT1的表达水平与正常组织比较无显著差异,但男性患者KCNQ1OT1的表达水平显著低于正常组织(P=0.0230),见图 3B。KCNQ1OT1在不同年龄段的乳腺癌患者中的表达水平与正常组织中的无显著差异。
|
| *:P < 0.05;**:P < 0.01. 图 3 KCNQ1OT1在乳腺癌及正常组织中的表达 Figure 3 Expression of KCNQ1OT1 in breast cancer and normal samples |
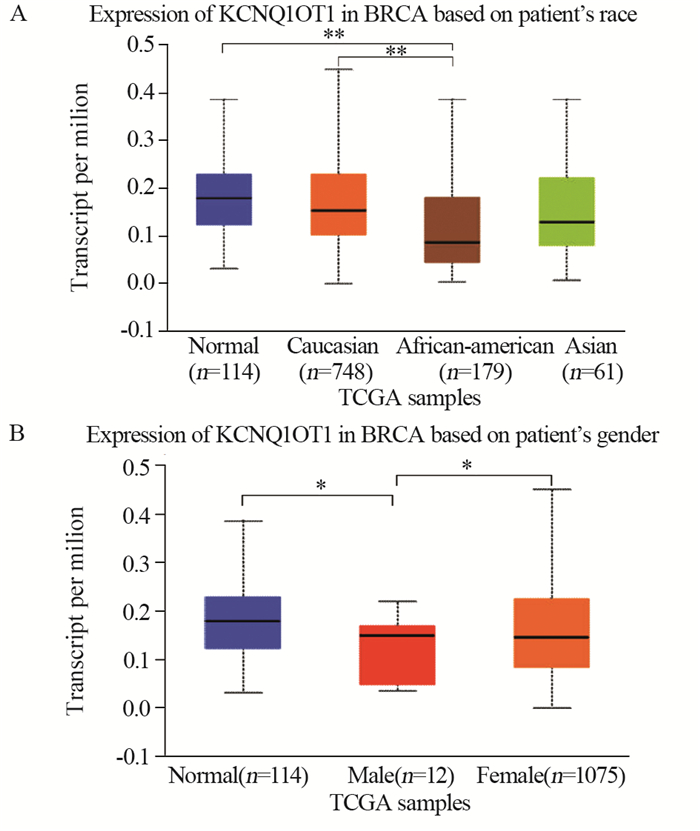
虽然NEAT1在乳腺癌组织中的表达水平与正常组织中无显著差异,但在黄种人患者中的表达水平显著低于正常组织(P=3.63×10-6),见图 4A。在21~40岁患者(P=1.98×10-10)和41~60岁患者(P=0.0024)中的表达水平显著低于正常组织,见图 4B。
|
| **:P < 0.01. 图 4 NEAT1在乳腺癌及正常组织中的表达 Figure 4 Expression of NEAT1 in breast cancer and normal samples |
由于UALCAN数据库中部分患者的信息不完整,按临床分期:Ⅰ期乳腺癌样本有183个,Ⅱ期样本有615个,Ⅲ期样本有247个,Ⅳ期样本有20个;按照分子分型:Luminal型、HER2阳性和三阴性乳腺癌分别有566、37和116个样本;按照组织学分型:浸润性导管癌有784个样本,浸润性小叶癌203个样本,黏液癌17个样本,化生性乳腺癌9个样本,髓样癌6个样本。
EGOT的表达水平在Ⅲ期(P=0.0005)和Ⅳ期乳腺癌(P=0.0035)中的表达水平显著低于正常组织,见图 5A。HER2阳性乳腺癌(P=1.62×10-12)和三阴性乳腺癌组织(P=1.02×10-11)中EGOT的表达水平显著低于正常组织,也显著低于Luminal型乳腺癌(ER、PR阳性)(P=4.20×10-12),见图 5B。此外,在不同组织学亚型中,EGOT在浸润性导管癌(P=0.0318)、黏液癌(P=2.02×10-10)、化生性乳腺癌(P=1.62×10-12)、髓样癌(P=0.0002)中的表达水平显著低于正常组织,见图 5C。
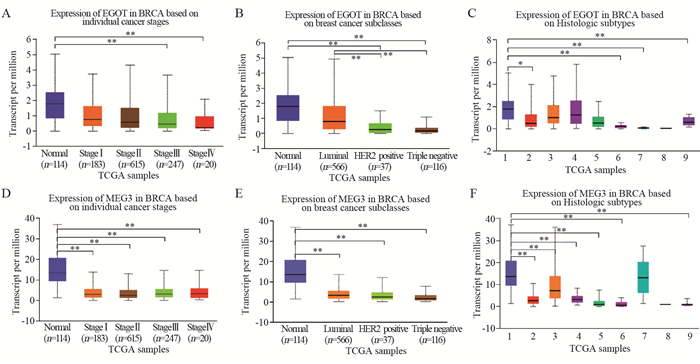
|
| *: P < 0.05; **: P < 0.01. 1: Normal(n=114); 2: IDC(n=784); 3: ILC(n=203); 4: Mixed(n=29); 5: Other(n=45); 6: Mucinous(n=17); 7: Metaplastic(n=9); 8: INOS(n=1); 9: Medullary(n=6). 图 5 EGOT和MEG3在不同临床病理特征乳腺癌患者中的表达 Figure 5 Expression of EGOT and MEG3 in breast cancer patients with different clinicopathological characteristics |
MEG3的表达水平在不同分期、不同分子分型的乳腺癌组织中均显著低于正常组织(P < 0.05),见图 5D~5E。在不同组织学亚型中,MEG3在浸润性导管癌(P < 1×10-12)、浸润性小叶癌(P=0.0002)、黏液癌(P < 1×10-12)、髓样癌(P=1.38×10-9)几种亚型中的表达水平显著低于正常组织,见图 5F。
KCNQ1OT1在不同分期乳腺癌组织中的表达水平与正常组织无显著差异。在不同分子分型中,仅HER2阳性乳腺癌中KCNQ1OT1的表达水平显著低于正常组织(P=0.0031),见图 6A。KCNQ1OT1在黏液癌(P=0.0367)和髓样癌(P=0.0091)两种组织学亚型中的表达水平显著低于正常组织(P < 0.05),见图 6B。
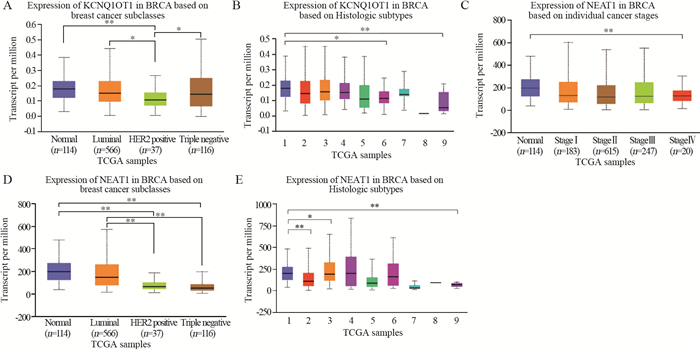
|
| *:P < 0.05;**:P < 0.01;HER2:human epidermal growth factor receptor-2;IDC:infiltrating ductal carcinoma; ILC: infiltrating lobular carcinoma; Mixed: mixed histology; Mucinous: mucinous carcinoma; Medullary: medullary carcinoma; Metaplastic: metaplastic carcinoma; INOS: infltrating carcinoma no otherwise specifed. 图 6 KCNQ1OT1和NEAT1在不同临床病理特征乳腺癌患者中的表达 Figure 6 Expression of KCNQ1OT1 and NEAT1 in breast cancer patients with different clinicopathological characteristics |
NEAT1在Ⅳ期乳腺癌患者中的表达水平显著低于正常组织(P=0.0036),见图 6C。HER2阳性(P=1.89×10-12)和三阴性乳腺癌患者(P < 1×10-12)NEAT1的表达水平显著低于正常组织,见图 6D。NEAT1在浸润性导管癌(P=0.0007)、浸润性小叶癌(P=0.0110)和髓样癌(P=3.81×10-8)三种组织学亚型中的表达水平显著低于正常组织,见图 6E。
2.3 LncRNA表达水平与乳腺癌患者预后的关系Kaplan-Meier曲线显示EGOT表达水平高的乳腺癌患者较EGOT表达水平低的患者有更好的RFS(P=0.037),即更好的预后,见图 7A。同样,MEG3(P=1.3×10-8)、KCNQ1OT1(P=8.6×10-8)、NEAT1(P < 1×10-16)的表达水平与乳腺癌患者的RFS均显著相关,而且这三种LncRNA高水平表达时,乳腺癌患者的RFS和预后相对较好,见图 7B~7D。
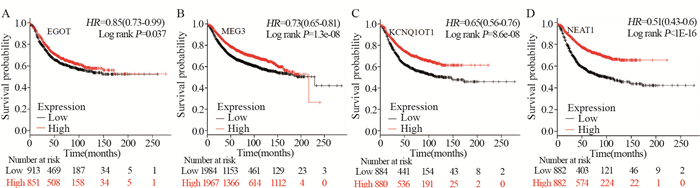
|
| 图 7 LncRNA表达水平与乳腺癌患者的生存曲线 Figure 7 LncRNA expression and Kaplan-Meier curve of breast cancer patients |
与EGOT、MEG3、KCNQ1OT1和NEAT1表达相关性最大的前五个蛋白质,见表 1。GO功能注释和KEGG通路富集分析结果见图 8。
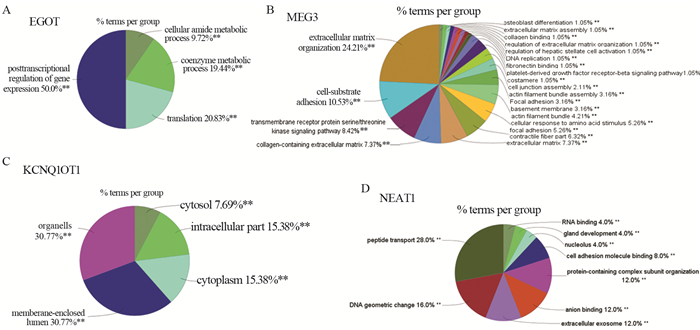
|
| *:P < 0.05;**:P < 0.01. 图 8 与LncRNA表达相关蛋白质的GO和KEGG功能富集分析结果 Figure 8 GO and KEGG function enrichment analysis results of proteins related to expression of four lncRNAs |
与EGOT表达相关的蛋白质经过功能富集分析后,共获得71项极显著(P < 0.01)的GO term,其中50%与基因表达的转录后调控有关,20.83%与翻译有关,见图 8A;获得1条极显著(P=0.009)的KEGG通路,与RNA运输相关的通路。与MEG3表达相关的蛋白质经过功能富集分析后,共获得90项极显著(P < 0.01)的GO term,其中24.21%与细胞外基质组织有关,其次10.53%与细胞基质黏附相关,见图 8B;获得5条极显著(P < 0.01)的KEGG通路,分别是与黏着斑、细胞外基质与受体相互作用、DNA复制等相关的通路。与KCNQ1OT1表达相关的蛋白质经过功能富集分析后,共获得13项极显著(P < 0.01)的GO term,其中30.77%与细胞器有关,30.77%的与膜包围的内腔相关,见图 8C。与NEAT1表达相关的蛋白质经过功能富集分析后,共获得25项极显著(P < 0.01)的GO term,其中28%与肽运输有关,其次16%与DNA几何变化相关,见图 8D。
3 讨论乳腺癌是一种异质性癌症,同样的治疗方案用在不同患者身上,往往有不同的治疗效果与预后,因此需要针对每种乳腺癌亚型开发特异的肿瘤标志物,以便更精准地个性化治疗与预后评估。
本研究发现EGOT在乳腺癌组织中的表达显著低于正常组织。同时,EGOT的表达水平在Ⅲ期和Ⅳ期、HER2阳性和三阴性乳腺癌、浸润性导管癌、黏液癌、化生性乳腺癌、髓样癌等乳腺癌亚型中显著降低。Kaplan-Meier曲线显示EGOT表达水平与患者预后显著相关,表达水平高的乳腺癌患者较EGOT表达水平低的患者有更好的预后。有报道发现EGOT在乳腺癌中表达下调[12]。与乳腺癌中低表达类似,有研究发现在肾细胞瘤中EGOT表达下调并起到肿瘤抑制因子的作用[13]。通过功能富集分析,发现与EGOT表达相关的蛋白质主要与基因表达的转录后调控、翻译和RNA运输等相关,说明EGOT可能通过与相关蛋白质相互作用来调控某些基因的表达。EGOT表达受抑制可能在乳腺癌的后期发展中发挥重要作用,并且EGOT可作为潜在的乳腺癌抑癌因子和预后标志物。
本研究发现MEG3在不同种族、性别、年龄、分期、分子分型的乳腺癌组织中均显著低于正常组织的,表明MEG3的低表达可能与乳腺癌的发生和发展相关。同样,在食管癌中,MEG3表达水平也显著降低,并且可以通过竞争性结合miR-9抑制肿瘤转移[14]。一些学者通过研究发现,在乳腺癌细胞等多种癌细胞中,MEG3是肿瘤抑制因子,其表达量很低甚至几乎不表达,造成这种情况的原因可能是其启动子区高甲基化[15]。本研究还发现当MEG3表达水平较低时,乳腺癌患者的预后也较差。通过功能富集分析,发现与MEG3表达相关的蛋白质主要与细胞基质黏附、细胞外基质与受体相互作用等相关,而这些通路主要影响细胞间的相互作用、突触形成、血脑屏障完整性等[16],说明MEG3的异常表达可能会对以上生物过程产生影响。以上结果表明,MEG3在乳腺癌中发挥抑癌作用,其低表达影响乳腺癌的发生和发展,并有望成为判断乳腺癌患者预后的生物学指标。
经分析,男性乳腺癌患者KCNQ1OT1的表达水平显著低于正常组织和女性患者。此外,KCNQ1OT1仅在HER2阳性乳腺癌中的表达水平显著降低。同样,KCNQ1OT1表达水平较低时,乳腺癌患者的预后也较差。有研究发现,KCNQ1OT1可以吸附miR-145,而miR-145抑制CCNE2基因的表达,所以KCNQ1OT1通过调控下游miR-145/CCNE2基因的表达发挥作用[17]。通过功能富集分析,发现与KCNQ1OT1表达相关的蛋白质主要与细胞器和膜包被的内腔等细胞组分方面的功能相关,表明KCNQ1OT1的异常表达可能会影响细胞的细胞器。KCNQ1OT1的低表达可能作为男性乳腺癌患者和HER2阳性乳腺癌特异性指标和不良预后指标。
本研究还发现,NEAT1亚洲患者中的表达水平显著低于正常组织,同时20~60岁患者中表达水平显著降低,表明NEAT1是潜在的黄种人乳腺癌患者特异性诊断指标,还可能作为年轻乳腺癌患者的生物学指标。此外,Ⅳ期、HER2阳性和三阴性等预后较差的乳腺癌患者中的NEAT1表达水平较低,生存分析表明NEAT1表达水平降低的患者预后较差。但是,有研究发现三阴性乳腺癌中NEAT1表达上调[18],可能是分析样本不同的原因,该文献所使用的为黄种人样本,而本文所使用的数据样本多为白种人,分析结果也表明NEAT1在黄种人和白种人之间的表达水平具有显著差异。通过功能富集分析,发现与NEAT1表达相关的蛋白质主要与肽运输、DNA几何变化等生物过程相关,表明NEAT1异常表达可能会调控以上过程,进而调控相关基因的表达。以上结果表明NEAT1在不同乳腺癌患者中具有不同的表达和功能,可以作为潜在的特异性乳腺癌诊断指标和预后判断标志物。
综上,四种LncRNA在乳腺癌或部分亚型中表达下调,可能是乳腺癌中潜在的抑癌因子,且可能成为判断乳腺癌患者预后的生物学指标。本文还通过功能富集分析方法探讨了与四种LncRNA表达相关的蛋白质的主要功能,但它们影响乳腺癌发生和发展的作用机制还需要进行更深入的研究。
作者贡献
苏嘉庆:实验设计、实施及论文撰写
李智伟:实验设计、实验指导
万召娟、王芳华:实验实施
汪梦、张恒阳、张慧欣:实验数据分析
周百灵:实验设计、论文审校
| [1] |
Miller KD, Nogueira L, Mariotto AB, et al. Cancer treatment and survivorship statistics, 2019[J]. CA Cancer J Clin, 2019, 69(5): 363-385. DOI:10.3322/caac.21565 |
| [2] |
卢振东, 孙晓晓, 谢龙祥, 等. 乳腺癌分子分型研究进展[J]. 河南大学学报(医学版), 2018, 37(4): 229-233. [Lu ZD, Sun XX, Xie LX, et al. The development of molecular subtyping in breast cancer[J]. Henan Da Xue Xue Bao (Yi Xue Ban), 2018, 37(4): 229-233.] |
| [3] |
Yuan CL, Jiang XM, Yi Y, et al. Identification of differentially expressed lncRNAs and mRNAs in luminal-B breast cancer by RNA-sequencing[J]. BMC cancer, 2019, 19(1): 1-12. DOI:10.1186/s12885-018-5219-3 |
| [4] |
皮亚平, 刘黎明. BCYRN1对乳腺癌细胞MCF7和小鼠移植瘤增殖和转移的影响[J]. 肿瘤防治研究, 2018(4): 205-209. [Pi YP, Liu LM. Influence of BCYRN1 on proliferation and migration of breast cancer cell line MCF7 and tumor-bearing mice[J]. Zhong Liu Fang Zhi Yan Jiu, 2018(4): 205-209. DOI:10.3971/j.issn.1000-8578.2018.17.0691] |
| [5] |
Rhodes DR, Kalyana-Sundaram S, Mahavisno V, et al. Oncomine 3.0: genes, pathways, and networks in a collection of 18, 000 cancer gene expression profiles[J]. Neoplasia, 2007, 9(2): 166-180. DOI:10.1593/neo.07112 |
| [6] |
Tomczak K, Czerwinska P, Wiznerowicz M. The Cancer Genome Atlas (TCGA): an immeasurable source of knowledge[J]. Contemp Oncol (Pozn), 2015, 19(1A): A68-A77. |
| [7] |
Zhou B, Zhao H, Yu J, et al. EVLncRNAs: a manually curated database for long non-coding RNAs validated by low-throughput experiments[J]. Nucleic Acids Res, 2018, 46(D1): D100-D105. DOI:10.1093/nar/gkx677 |
| [8] |
Gao Y, Wang P, Wang Y, et al. Lnc2Cancer v2.0: updated database of experimentally supported long non-coding RNAs in human cancers[J]. Nucleic Acids Res, 2019, 47(D1): D1028-D1033. DOI:10.1093/nar/gky1096 |
| [9] |
Chandrashekar DS, Bashel B, Balasubramanya SAH, et al. UALCAN: A Portal for Facilitating Tumor Subgroup Gene Expression and Survival Analyses[J]. Neoplasia, 2017, 19(8): 649-658. DOI:10.1016/j.neo.2017.05.002 |
| [10] |
Lánczky A, Nagy Á, Bottai G, et al. miRpower: a web-tool to validate survival-associated miRNAs utilizing expression data from 2178 breast cancer patients[J]. Breast Cancer Res Treat , 2016, 160(3): 439-446. |
| [11] |
Vasaikar SV, Straub P, Wang J, et al. LinkedOmics: analyzing multi-omics data within and across 32 cancer types[J]. Nucleic Acids Res, 2018, 46(D1): D956-D963. DOI:10.1093/nar/gkx1090 |
| [12] |
Xu SP, Zhang JF, Sui SY, et al. Downregulation of the long noncoding RNA EGOT correlates with malignant status and poor prognosis in breast cancer[J]. Tumor Biol, 2015, 36(12): 9807-9812. DOI:10.1007/s13277-015-3746-y |
| [13] |
Jin L, Quan J, Pan X, et al. Identification of lncRNA EGOT as a tumor suppressor in renal cell carcinoma[J]. Mol Med Rep, 2017, 16(5): 7072-7079. DOI:10.3892/mmr.2017.7470 |
| [14] |
陈梦雪, 李学良. 长链非编码RNA在食管癌中的研究进展[J]. 肿瘤防治研究, 2018, 45(11): 932-935. [Chen MX, Li XL. Research Progress of Long Non-coding RNA in Esophageal Cancer[J]. Zhong Liu Fang Zhi Yan Ju, 2018, 45(11): 932-935. DOI:10.3971/j.issn.1000-8578.2018.18.0399] |
| [15] |
Ghafouri-Fard S, Taheri M. Maternally expressed gene 3 (MEG3): A tumor suppressor long non coding RNA[J]. Biomed Pharmacother, 2019, 118: 109129. DOI:10.1016/j.biopha.2019.109129 |
| [16] |
Moradifard S, Hoseinbeyki M, Ganji SM, et al. Analysis of microRNA and gene expression profiles in Alzheimer's disease: a meta-analysis approach[J]. Sci Rep, 2018, 8(1): 4767. |
| [17] |
Feng W, Wang C, Liang C, et al. The dysregulated expression of KCNQ1OT1 and its interaction with downstream factors miR-145/CCNE2 in breast cancer cells[J]. Cell Physiol Biochem, 2018, 49: 432-446. DOI:10.1159/000492978 |
| [18] |
Shin VY, Chen J, Cheuk IW, et al. Long non-coding RNA NEAT1 confers oncogenic role in triple-negative breast cancer through modulating chemoresistance and cancer stemness[J]. Cell Death Dis, 2019, 10(4): 270. DOI:10.1038/s41419-019-1513-5 |
 2020, Vol. 47
2020, Vol. 47