文章信息
- 非小细胞肺癌患者外周血中WNT7A甲基化检测的诊断价值
- Detection of WNT7A Methylation in Peripheral Blood of NSCLC Patients and Its Diagnostic Value
- 肿瘤防治研究, 2019, 46(12): 1097-1100
- Cancer Research on Prevention and Treatment, 2019, 46(12): 1097-1100
- http://www.zlfzyj.com/CN/10.3971/j.issn.1000-8578.2019.19.0484
- 收稿日期: 2019-04-15
- 修回日期: 2019-08-08
非小细胞肺癌(non-small cell lung cancer, NSCLC)具有发病率、死亡率高及预后差等特点。尽管已有多种综合治疗NSCLC的方法,但患者术后的生存率依然十分不理想,因此预测预后尤为重要。众多研究证明,基因的甲基化异常能有效预测癌症的发生,并可作为预后评估的标志物[1-3]。Wnt信号通路对机体的正常发育至关重要,但其参与肺肿瘤发生的机制尚不明确。WNT7A及其受体FZD9是维持上皮细胞分化所必需的。有研究发现,WNT7A因甲基化的发生常在胰腺癌[4]、肾细胞癌[5]等患者的癌组织或外周血中,导致对应的WNT7A基因表达降低甚至不表达。研究表明,临床应用脱甲基剂可干预肿瘤的发生和发展[6-7]。
本文以甲基化特异性聚合酶链反应(methylation-specific polymerase chain reaction, MSP)的方法比较肺癌患者外周血和健康人群外周血中的WNT7A基因甲基化状态,探讨WNT7A基因在外周血中甲基化程度对NSCLC的临床诊断意义。
1 资料与方法 1.1 临床资料和样本收集选取湖北省第三人民医院2016年3月—2018年3月的120例NSCLC患者为NSCLC组,男66例,女54例。年龄为34~68岁。纳入标准为:(1)通过症状体征和肺部增强CT以及病理组织学检查确诊为NSCLC;(2)无既往肺癌病史,无合并其他肿瘤;(3)功能状态KPS评分 > 70分;排除标准为:(1)术前进行过手术、放疗、化疗等治疗手段;(2)对化疗相关药物过敏者及有手术禁忌证者;(3)妊娠或哺乳期女性。另外选择120位健康者为对照组,均来自于本院体检人群。所有NSCLC患者和对照组健康者均已签署知情同意书。
1.2 检测方法 1.2.1 DNA的提取取两组中每例患者的血清标本200 μl,按照EasyPure基因组DNA试剂盒(购于北京全式金生物技术有限公司,编号:EE101-02)说明和步骤,分别提取NSCLC组和对照组血清中的DNA,并采用紫外分光光度计检测提取的DNA含量和纯度。
1.2.2 亚硫酸氢钠处理取纯化的DNA样品20 μl,以DNA重亚硫酸盐转化试剂盒(购于德国Qiagen公司)对组织和血清样本DNA进行亚硫酸盐修饰。将DNA置于95℃水浴中加热20 min,再冰浴20 min,置于42℃、浓度为3 mol/L的NaOH溶液中温浴20 min。先加入NaHSO3处理液,50℃恒温16 h。再添加1 ml的37℃树脂,将混匀后的液体真空抽滤后,以80%的异丙醇洗涤,析出DNA,以NaOH、冰乙醇、醋酸铰沉淀后,以80%的乙醇洗涤,低温保存备用。
1.2.3 甲基化特异性聚合酶链反应甲基化引物由上海生工生物工程设计并合成。WNT7A甲基化引物:正义:5’-TTAGGGAGTTTAGTTTTTGGAGTTC-3’;反义:5’AACGACAATTTATATTTTTACCAACG-3’。非甲基化引物:正义:5’-TTAGGGAGTTTAGTTTTTGGAGTTT-3’;反义:5’-AACAATTTATATTTTTACCAACAAA-3’。反应体系为25 μl,甲基化的反应条件为:95℃预变性10 min,95℃变性60 s,62℃退火,72℃延伸40 s,35个循环,最后一个循环72℃延伸8 min。非甲基化的反应条件为:95℃预变性10 min,95℃变性60 s,58℃退火,72℃延伸40 s,35个循环,最后一个循环72℃延伸8 min。
根据结果计算外周血WNT7A期甲基化诊断的敏感度和特异性。敏感度=真阳性人数/(真阳性人数+假阴性人数)×100%。特异性=真阴性人数/(真阴性人数+假阳性人数)×100%。
1.3 统计学方法采用SPSS19.0进行统计学分析。NSCLC患者WNT7A基因甲基化和临床病理因素的相关性以及NSCLC组和对照组WNT7A甲基化检出率采用t或χ2检验。P < 0.05为差异具有统计学意义。
2 结果 2.1 一般临床特征对NSCLC组和对照组的性别、年龄、吸烟史、KPS评分进行统计。NSCLC组男66例,女54例,平均年龄为(55.3±3.4)岁;对照组男62例,女58例,平均年龄为(53.1±2.6)岁;NSCLC组有吸烟史70人,无吸烟史50人;对照组有吸烟史64人,无吸烟史56人;NSCLC组和对照组的KPS评分分别为(90.2±3.7)和(90.8±2.5)。两组患者在性别、年龄、吸烟史、KPS评分上差异均无统计学意义(均P > 0.05)。
2.2 WNT7A在NSCLC患者和健康者外周血中的甲基化情况NSCLC组患者的外周血中,甲基化率达51.67%(62/120),而对照组甲基化率仅为14.17%(17/120),差异有统计学意义(P=0.000),见图 1。敏感度为51.67%,特异性为85.83%。
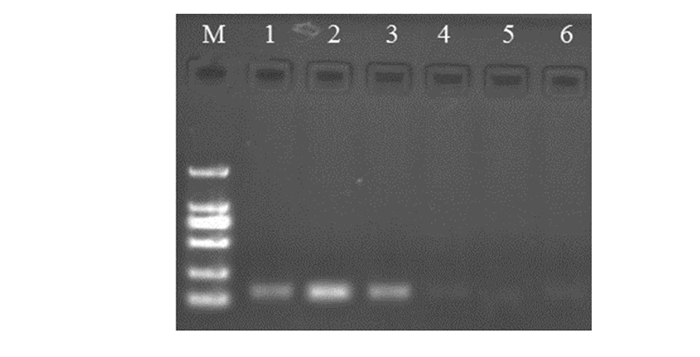
|
| M: the marker of DL2000; 1, 2, 3: the amplifcation results of WNT7A gene target fragment in NSCLC group; 4, 5, 6: the amplifcation results of WNT7A gene target fragment in control group 图 1 WNT7A基因目的片段PCR扩增产物电泳图 Figure 1 Electrophoresis image of PCR amplification products of WNT7A gene target fragment |
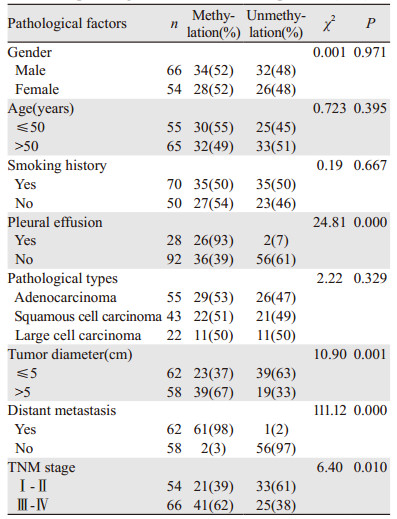
有胸腔积液的NSCLC患者外周血中WNT7A基因甲基化检出率显著高于无胸腔积液患者(P=0.000);肿瘤直径 > 5 cm的NSCLC患者外周血中WNT7A基因甲基化检出率显著高于≤5 cm的NSCLC患者(P=0.001);TNM分期为Ⅲ~Ⅳ的NSCLC患者外周血中WNT7A基因甲基化检出率显著高于分期为Ⅰ~Ⅱ的NSCLC患者(P=0.010)。WNT7A基因甲基化与患者肿瘤大小、远处转移情况、TNM分期、胸腔积液相关(均P < 0.05),而与患者的性别、年龄、吸烟史、病理类型无相关性(均P > 0.05),见表 1。
|
遗传学和表观遗传学的改变是目前被普遍认可的恶性肿瘤主要发生机制之一。随着人们对基因研究的不断深入,表观遗传在肿瘤发生过程中的重要作用日益凸显,DNA的甲基化就是肿瘤发生机制中的表观遗传形式之一[8-9]。甲基化的机制不同于基因突变,甲基化过程没有碱基序列的突变,而仅有甲基基团的改变。基因中有一个明显甲基化的区域被称为CpG岛[10],5’甲基化的mCpG结构在结合甲基结合蛋白后,能够阻止启动子和转录因子的结合过程,导致基因低表达甚至不表达,异常的甲基化活动会导致机体基因表达异常,高甲基化和低甲基化都可能使基因表达异常或者导致癌症的发生[11-13]。
越来越多的研究证实WNT7A在肿瘤的发生和发展中具有重要的调控作用,并具有一定的预后评估意义[14-15]。和传统的肿瘤标志物检测比较,基因甲基化的预后具有更高的敏感度和特异性。未来随着基因研究的发展,以高通量检测基因水平测定肿瘤标志物的诊断方法更符合精准医学的要求[16]。Ohira等[17]研究表明,WNT7A通过介导上皮细胞钙黏蛋白的表达,影响着肺癌细胞株的生长和代谢活动。当WNT7A的表达缺失时,肺癌细胞生长速度显著增加,并认为WNT7A为抑癌基因。Bikkavilli等[18]研究发现WNT7A是一种新的引起肺癌细胞衰老的诱导因子,在肺癌中具有重要的治疗意义。刺激WNT7A的表达或增加与其表达产物功能相似的化合物,能够促进肺癌细胞衰老,并使细胞减少癌变。而基因启动子甲基化通常可直接终止转录因子和启动子的结合,致使该基因低表达或不表达。且Senchenko等研究发现,WNT7A在NSCLC患者中被检测出高甲基化率[19]。这些研究结果表明,WNT7A的甲基化有望成为预测NSCLC的新标志物。
在本研究中,对照组的外周血WNT7A基因甲基化检出率极显著低于NSCLC组,提示WNT7A基因甲基化可作为NSCLC有效的诊断依据。且在NSCLC组患者外周血中,WNT7A基因甲基化和肿瘤大小、远处转移情况、TNM分期、胸腔积液情况相关,提示WNT7A的甲基化可作为NSCLC的诊断依据。简单方便的采血检测方式比较容易让患者接受,外周血WNT7A期甲基化有望成为临床上NSCLC的诊断标准,但其敏感度和特异性还有待进一步提高。
作者贡献
徐 瑶:完善文章思路,执行实验部分,提供临床数据信息并整理分析,撰写文章
张 畅:主导构思思路,提供临床数据信息并整理分析,审阅文章并修改
彭黎、张军霞:提供临床数据信息并整理分析,审核并把控主要内容和方向
| [1] |
Shou F, Xu F, Li G, et al. RASSF1A promoter methylation is associated with increased risk of thyroid cancer: a meta-analysis[J]. Onco Targets Ther, 2017, 10: 247-257. DOI:10.2147/OTT.S124417 |
| [2] |
Hao X, Luo H, Krawczyk M, et al. DNA methylation markers for diagnosis and prognosis of common cancers[J]. Proc Natl Acad Sci USA, 2017, 114(28): 7414-7419. DOI:10.1073/pnas.1703577114 |
| [3] |
Nian J, Sun X, Ming SY, et al. Diagnostic accuracy of methylated SEPT9 for blood-based colorectal cancer detection: a systematic review and meta-analysis[J]. Clin Transl Gastroenterol, 2017, 8(1): e216. |
| [4] |
Lindberg D, Akerström G, Westin G. Mutational analyses of WNT7A and HDAC11 as candidate tumour suppressor genes in sporadic malignant pancreatic endocrine tumours[J]. Clin Endocrinol (Oxf), 2007, 66(1): 110-114. |
| [5] |
Kondratov AG, Kvasha SM, Stoliar LA, et al. Alterations of the WNT7A gene in clear cell renal cell carcinomas[J]. PLoS One, 2012, 7(10): e47012. DOI:10.1371/journal.pone.0047012 |
| [6] |
Zhou J, Yao Y, Shen Q, et al. Demethylating agent decitabine disrupts tumor-induced immune tolerance by depleting myeloid-derived suppressor cells[J]. J Cancer Res Clin Oncol, 2017, 143(8): 1371-1380. DOI:10.1007/s00432-017-2394-6 |
| [7] |
Li X, Zhang Y, Chen M, et al. Increased IFN-γ+ T cells are responsible for the clinical responses of low-dose DNA demethylating agent decitabine anti-tumor therapy[J]. Clin Cancer Res, 2017, 23(20): 6031-6034. DOI:10.1158/1078-0432.CCR-17-1201 |
| [8] |
Yang Y, Liu R, Qiu R, et al. CRL4B promotes tumorigenesis by coordinating with SUV39H1/HP1/DNMT3A in DNA methylation-based epigenetic silencing[J]. Oncogene, 2015, 34(1): 104-118. DOI:10.1038/onc.2013.522 |
| [9] |
Iida T, Iwanami A, Sanosaka T, et al. Whole-genome DNA Methylation Analyses Revealed Epigenetic Instability in Tumorigenic Human iPS Cell-derived Neural Stem/progenitor Cells[J]. Stem Cells, 2017, 35(5): 1316-1327. DOI:10.1002/stem.2581 |
| [10] |
Kim SH, Park KH, Shin SJ, et al. CpG island methylator phenotype and methylation of Wnt pathway genes together predict survival in patients with colorectal cancer[J]. Yonsei Med J, 2018, 59(5): 588-594. DOI:10.3349/ymj.2018.59.5.588 |
| [11] |
Yin Y, Morgunova E, Jolma A, et al. Impact of cytosine methylation on DNA binding specificities of human transcription factors[J]. Science, 2017, 356(6337): pii: eaaj2239. DOI:10.1126/science.aaj2239 |
| [12] |
Zuo Z, Roy B, Chang YK, et al. Measuring quantitative effects of methylation on transcription factor–DNA binding affinity[J]. Sci Adv, 2017, 3(11): eaao1799. DOI:10.1126/sciadv.aao1799 |
| [13] |
Nikolova E, Stanfield RL, Dyson HJ, et al. CH···O hydrogen bonds mediate highly specific recognition of methylated CpG sites by the Zinc finger protein kaiso[J]. Biochemistry, 2018, 57(14): 2109-2120. DOI:10.1021/acs.biochem.8b00065 |
| [14] |
Hu TM, Ping LY, Hsu H, et al. Mutation analysis of the WNT7A gene in patients with schizophrenia[J]. Psychiatry Res, 2018, 246-248. |
| [15] |
Huang X, Zhu H, Gao Z, et al. Wnt7a activates canonical Wnt signaling, promotes bladder cancer cell invasion, and is suppressed by miR-370-3p[J]. J Biol Chem, 2018, 293(18): 6693-6706. DOI:10.1074/jbc.RA118.001689 |
| [16] |
Lapek JD Jr, Greninger P, Morris R, et al. Detection of dysregulated protein-association networks by high-throughput proteomics predicts cancer vulnerabilities[J]. Nat Biotechnol, 2017, 35(10): 983-989. DOI:10.1038/nbt.3955 |
| [17] |
Ohira T, Gemmill RM, Ferguson K, et al. WNT7a induces E-cadherin in lung cancer cells[J]. Proc Natl Acad Sci U S A, 2003, 100(18): 10429-10434. DOI:10.1073/pnas.1734137100 |
| [18] |
Bikkavilli RK, Avasarala S, Van Scoyk M, et al. Wnt7a is a novel inducer of β-catenin-independent tumor-suppressive cellular senescence in lung cancer[J]. Oncogene, 2015, 34(42): 5406. DOI:10.1038/onc.2015.165 |
| [19] |
Senchenko VN, Kisseljova NP, Ivanova TA, et al. Novel tumor suppressor candidates on chromosome 3 revealed by NotI-microarrays in cervical cancer[J]. Epigenetics, 2013, 8(4): 409-420. DOI:10.4161/epi.24233 |
 2019, Vol. 46
2019, Vol. 46


