文章信息
- 免疫沉淀——质谱联用揭示RCC2在乳腺癌MDA-MB-468细胞系中的新功能
- Immunoprecipitation-mass Spectrometry Reveals New Functions of RCC2 in Breast Cancer Cell Line MDA-MB-468
- 肿瘤防治研究, 2019, 46(9): 773-777
- Cancer Research on Prevention and Treatment, 2019, 46(9): 773-777
- http://www.zlfzyj.com/CN/10.3971/j.issn.1000-8578.2019.19.0183
- 收稿日期: 2019-02-18
- 修回日期: 2019-05-30
乳腺癌是全球女性最常见恶性肿瘤之一,近年来占据女性新发肿瘤第一位,病死率第四位[1-2]。尽管众多的诊断手段和治疗方式已被陆续研发,但寻找更加有效的肿瘤指示因子仍然迫切需要。RCC2于1991年被首次发现,由于其特殊的细胞定位,被视为是一种伴随蛋白[3]。RCC2主要在细胞G2期和M期表达,通过结合并激活Aurora B激酶,促使染色体伴随复合体(chromosomal passenger complex, CPC)定位在着丝粒上形成纺锤体,牵引姐妹染色单体的正常分离[4-5]。
近年来,RCC2与肿瘤之间的联系被逐渐阐明。研究表明,RCC2在众多肿瘤中异常表达,基底细胞癌[6]、胃癌[7]、肺癌[8]及卵巢癌[9]患者RCC2高表达,并预示不良的预后;微卫星稳定性结直肠癌患者RCC2低表达,预示着患者差的预后,而同样低表达的微卫星不稳定性肠癌患者却有着更好的预后[10]。然而,RCC2与乳腺癌致病发展之间的关系仍尚未明确。
本研究首次在乳腺癌MDA-MA-468细胞系中构建了RCC2稳定过表达细胞株,免疫共沉淀富集RCC2相互作用蛋白后,利用质谱鉴定互作蛋白图谱,生物信息学分析揭示RCC2在乳腺癌细胞中的新功能,为进一步治疗乳腺癌提供依据并为研究RCC2功能和调控机制指明新的道路。
1 材料与方法 1.1 材料与试剂表达载体pLVX-DsRed-Monomer-N1购自美国Clontech公司(货号:632152);慢病毒包装质粒载体psPAX2、pMD2.G购自美国Addgene(货号:12260;12259);高保真PrimeSTAR HS DNA聚合酶购自宝日医(北京)生物公司;限制性内切酶XhoⅠ、BamHⅠ、T4 DNA ligase、质粒小量抽提试剂盒和胶纯化回收试剂盒均购自美国Thermo公司;兔抗人RCC2单克隆抗体购自美国Cell Signaling Technology公司;GAPDH购自Santa Cruz公司(TX, USA)。RCC2基因扩增上游引物序列为5’-CCGCTCGAGACCATGCCCAGGAAGAA-3’,下游引物序列(含Flag标签)为5’-CGCGGATCCCTACTTGTCATCGTCGTCCTTGTAGTCTCAGAGGGTTCGGGGG-3’。RCC2 qPCR上游引物序列为5’-TTTTCTCAGAGCAGGTCGCC-3’, 下游引物序列为5’-TTCGGGGGTTGTATTCTGGC-3’。
1.2 方法 1.2.1 细胞培养乳腺癌MDA-MB-468细胞株由中山大学逸仙纪念医院医学研究中心实验室提供,用含有10%FBS,100 u/ml青霉素和100 μg/ml链霉素的DMEM培养基放置于37℃、5%CO2的培养箱中培养。
1.2.2 PCR扩增RCC2基因以U2OS全基因组cDNA为模板,高保真酶扩增目的基因。反应条件为:98℃ 3 min;98℃ 10 s;60℃ 5 s;72℃ 100 s;72℃ 10 min(35个循环)。取PCR产物电泳检测。
1.2.3 表达载体构建将电泳正确位置PCR产物胶带割下回收,纯化产物经过XhoⅠ、BamHI双酶切,并将其连接到经过同样酶切的表达载体中,转化大肠杆菌DH5α,PCR筛选重组子,并进行测序测定,扩增测序正确菌落,提取质粒为PLVX-Flag-RCC2。
1.2.4 稳转细胞株构建利用慢病毒包装系统获得携带有表达载体的病毒感染颗粒,将对照载体与目的载体病毒分别感染MDA-MB-468细胞株,24 h后更换新鲜培养基,48 h后添加嘌呤霉素,连续筛选5天获得稳定表达外源质粒细胞株。Western blot与qPCR验证稳转细胞株中目的基因表达情况。
1.2.5 免疫沉淀与质谱鉴定采用10 cm细胞培养皿扩增Flag-RCC2稳定过表达的MDA-MB-468细胞与阴性对照细胞,每组细胞设定2个重复。待细胞长满后,去除上清液,收集细胞,加入含蛋白抑制剂的RIPA裂解液(50 mmol/L Tris-HCl pH8.0, 5 mmol/L EDTA, 150 mmol/L NaCl, 0.5%Nonidet P-40)600 ml,冰上裂解细胞30 min,每5 min振荡混匀。4℃ 12 000 g高速离心30 min去除细胞沉淀,收集上清液。将用裂解液清洗三次的含Flag标签琼脂糖珠与上清液4℃孵育过夜。次日收集、清洗琼脂糖珠,获得互作蛋白。取少量样品加入蛋白上样缓冲液,95℃高温煮10 min,跑10% SDS-PAGE胶后银染,剩余样品进行置换,除盐、酶解处理,Orbitrap Tribrid(Thermo)高分辨率质谱仪检测。
1.2.6 蛋白质组学数据分析采用Protein Discovery(Thermo)软件搜索数据库,鉴定出蛋白。根据软件评分,选取大于4分的蛋白质分子。进一步对高可信度蛋白进行聚类分析,包括GO分析,通过生物学过程、细胞组分、分子功能三个层面对蛋白进行聚类;KEGG信号通路富集分析,预测该基因参与的调控通路;蛋白与蛋白互作网络(protein-protein interaction network, PPIN)的可视化构建,获取蛋白在细胞中互作的关系网络。
1.3 统计学方法用SPSS20.0软件进行统计学分析, 两组间比较用Student t检验分析。P < 0.05为差异有统计学意义。
2 结果 2.1 Flag-RCC2表达载体与MDA-MB-468稳转细胞株的构建及鉴定实验设计及分析思路,见图 1。首先需要构建RCC2稳定过表达乳腺癌MDA-MA-468细胞株。RCC2基因位于染色体1p36位点,编码区全长1 569 bp,以U2OS细胞cDNA为模板,设计含Flag标签及酶切位点的扩增引物,利用高保真酶扩增该编码区,PCR产物跑胶,结果显示获得与预期大小一致的片段,见图 2A,切下该位置胶带回收。同时对该产物与表达载体(结构图,见图 2B)进行双酶切,两份酶切产物连接过夜,得到含目的基因的表达载体。将其转化大肠杆菌DH5α,挑选菌落测序验证,阳性菌落测序结果显示RCC2基因成功插入表达载体中,序列正确(图略)。说明表达质粒PLVX-Flag-RCC2构建成功。
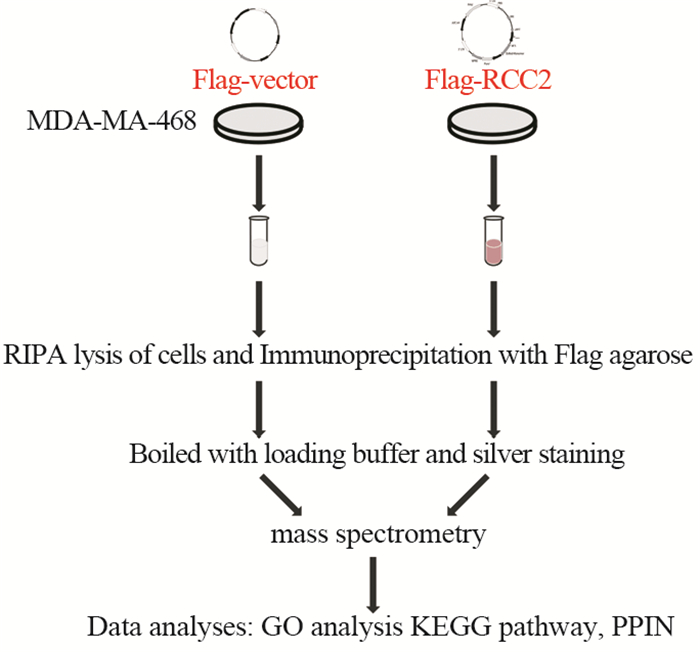
|
| 图 1 实验及分析流程示意图 Figure 1 Schematic representation of experimental and analytical workflow |

|
| A: amplification of RCC2 gene; B: map of overexpression vector 图 2 PLVX-Flag-RCC2过表达质粒载体构建 Figure 2 Construction of PLVX-Flag-RCC2 overexpression vector |
采用慢病毒包装感染技术将外源质粒导入MDA-MB-468细胞中,嘌呤霉素筛选获得稳转细胞株。Western blot和qPCR实验表明相比对照组,稳转RCC2基因过表达载体可以实现MDA-MB-468细胞内RCC2 mRNA及蛋白水平的高表达,见图 3。

|
| 图 3 qPCR(A)和Western blot法(B)验证稳转MDA-MB-468细胞株中PLVX-Flag-RCC2的表达 Figure 3 Expression of PLVX-Flag-RCC2 in stably transfected MDA-MB-468 cell line verified by qPCR(A) and Western blot(B) |
收集细胞,免疫沉淀获得同RCC2互作蛋白质,银染结果显示实验组存在大量与空白对照组相异的蛋白,见图 4A。质谱鉴定出蛋白分子,在同一组实验中去除实验组(Flag-RCC2)鉴定得到的阴性结合的角质蛋白,以及对照组(Flag-vector)中出现的假阳性结合蛋白,再对两组重复中筛选获得的蛋白取交集,最终得到高可信度的互作蛋白集合共计164个,见图 4B。
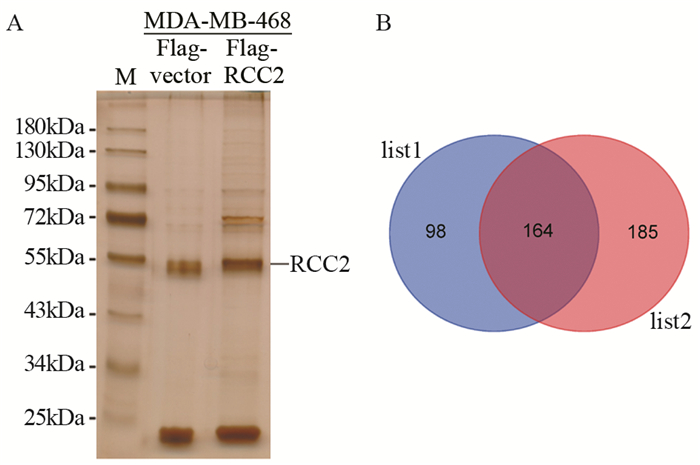
|
| 图 4 RCC2互作蛋白银染(A)与质谱鉴定(B)结果 Figure 4 Silver staining of RCC2 interacting proteins(A) and mass spectrometry identification(B) |
对164个高可信度互作蛋白进行GO分析,见图 5A,分子功能分析显示RCC2及其互作蛋白具有poly(A)RNA结合能力,并参与翻译过程中所必须的核糖体组装过程,值得注意的是,这些蛋白不仅可以结合RNA,同时可以结合核糖体核蛋白,这些信息预示RCC2可能在乳腺癌细胞系中介导了蛋白翻译过程。而GO生物学过程(BP)分析则提示了这一预测的正确性,BP分析结果表明RCC2互作蛋白组主要参与蛋白翻译,蛋白定位,蛋白折叠等细胞生理进程。细胞组分分析结果中胞质核糖体大亚基的定位也确定了蛋白翻译调控的可能性。对互作蛋白组进行KEGG富集分析发现RCC2与其相互作用蛋白主要参与核糖体、剪切体、非同源末端连接、病原微生物感染及细胞周期相关调控通路,见图 5B。进一步对互作蛋白组中丰度排名前50的蛋白构建蛋白相互作用关系网络。颜色表示蛋白互作程度,大小表示蛋白富集丰度,我们发现其中具有重要调控功能的蛋白分子包括RPL6(60S ribosomal protein L6)、RPL4(60S ribosomal protein L4)、RPS4X(40S ribosomal protein S4, X isoform)、PKM(Pyruvate kinase)、HSPA5(Endoplasmic reticulum chaperone)等,见图 5C。
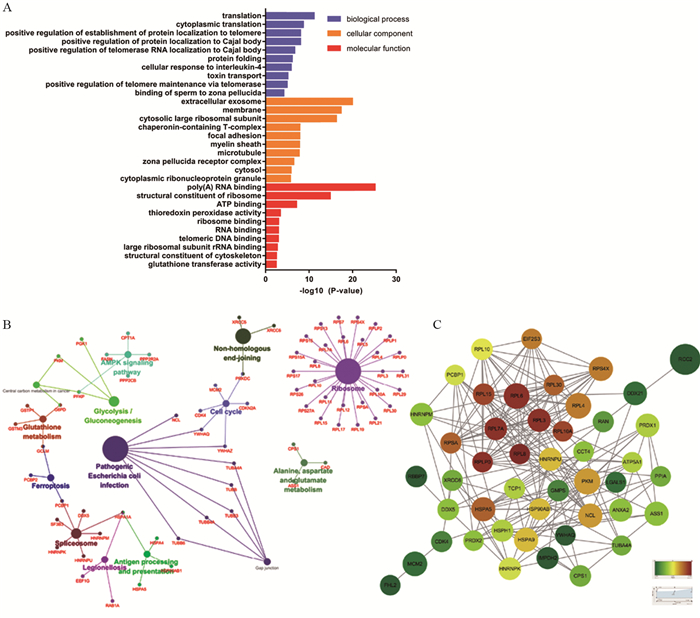
|
| A: genetic ontology annotation; B: KEGG pathway enrichment; C: protein-protein interaction network 图 5 RCC2互作蛋白组聚类分析 Figure 5 Bioinformatic clustering analyses of RCC2 interactome |
乳腺癌是女性最常见恶性肿瘤之一,根据雌激素受体(estrogen receptor, ER)、孕激素受体(progesterone receptor, PR)和人类表皮生长因子受体2(human epidermal growth factor receptor-2, HER2)表达差异,乳腺癌被划分为不同的分子亚型[11]。其中ER-/PR-/HER2-三阴性乳腺癌具有高度转移侵袭能力以及差的预后[12],MDA-MB-468细胞既属于这一类型。我们通过在MDA-MB-468细胞中构建RCC2稳定过表达细胞株,分析RCC2互作蛋白组,探讨RCC2基因在乳腺癌细胞中的功能。与以往研究结果不同,我们发现RCC2不仅与细胞周期相关[13],更重要地,RCC2与其互作蛋白富集在核糖体相关通路中,参与翻译相关的胞内活动,包括Pre-mRNA剪切,结合核糖体核蛋白,调控蛋白翻译及成熟。核糖体是由大小不同两个亚基组成,在蛋白作用网络中,我们发现RPL6、RPL4、RPS4X蛋白与RCC2有着密切的互作关系,其中,RPL6,RPL4是核糖体大亚基组成蛋白[14],而RPS4X是核糖体小亚基组成蛋白[15],RCC2同核糖体蛋白的互作揭示其在乳腺癌MDA-MB-468细胞中的翻译调控功能。
综上所述,分析乳腺癌细胞系中RCC2与其相互作用蛋白组调控网络为揭示RCC2在乳腺癌中的具体功能和作用机制研究指明方向。RCC2对蛋白翻译进程的调控机制仍需进一步探索。
作者贡献
陈震:实验设计与实施、数据分析及论文撰写
陈恒星:数据分析
胡开顺:实验设计、论文审核
| [1] | DeSantis CE, Ma J, Goding Sauer A, et al. Breast cancer statistics, 2017, racial disparity in mortality by state[J]. CA Cancer J Clin, 2017, 67(6): 439–448. DOI:10.3322/caac.21412 |
| [2] | Siegel RL, Miller KD, Jemal A. Cancer statistics, 2018[J]. CA Cancer J Clin, 2018, 68(1): 7–30. DOI:10.3322/caac.21442 |
| [3] | Andreassen PR, Palmer DK, Wener MH, et al. Telophase disc: a new mammalian mitotic organelle that bisects telophase cells with a possible function in cytokinesis[J]. J Cell Sci, 1991, 99(Pt 3): 523–534. |
| [4] | Rosasco-Nitcher SE, Lan W, Khorasanizadeh S, et al. Centromeric Aurora-B activation requires TD-60, microtubules, and substrate priming phosphorylation[J]. Science, 2008, 319(5862): 469–472. DOI:10.1126/science.1148980 |
| [5] | Papini D, Langemeyer L, Abad MA, et al. TD-60 links RalA GTPase function to the CPC in mitosis[J]. Nat Commun, 2015, 6: 7678. DOI:10.1038/ncomms8678 |
| [6] | Stacey SN, Gudbjartsson DF, Sulem P, et al. Common variants on 1p36 and 1q42 are associated with cutaneous basal cell carcinoma but not with melanoma or pigmentation traits[J]. Nat Genet, 2008, 40(11): 1313–1318. DOI:10.1038/ng.234 |
| [7] | Matsuo M, Nakada C, Tsukamoto Y, et al. MiR-29c is downregulated in gastric carcinomas and regulates cell proliferation by targeting RCC2[J]. Mol Cancer, 2013, 12: 15. DOI:10.1186/1476-4598-12-15 |
| [8] | Pang B, Wu N, Guan R, et al. Overexpression of RCC2 Enhances Cell Motility and Promotes Tumor Metastasis in Lung Adenocarcinoma by Inducing Epithelial-Mesenchymal Transition[J]. Clin Cancer Res, 2017, 23(18): 5598–5610. DOI:10.1158/1078-0432.CCR-16-2909 |
| [9] | Lin H, Zhang X, Feng N, et al. LncRNA LCPAT1 Mediates Smoking/ Particulate Matter 2.5-Induced Cell Autophagy and Epithelial-Mesenchymal Transition in Lung Cancer Cells via RCC2[J]. Cell Physiol Biochem, 2018, 47(3): 1244–1258. |
| [10] | Bruun J, Kolberg M, Ahlquist TC, et al. Regulator of Chromosome Condensation 2 Identifies High-Risk Patients within Both Major Phenotypes of Colorectal Cancer[J]. Clin Cancer Res, 2015, 21(16): 3759–3770. DOI:10.1158/1078-0432.CCR-14-3294 |
| [11] | Harbeck N, Gnant M. Breast cancer[J]. Lancet, 2017, 389(10074): 1134–1150. DOI:10.1016/S0140-6736(16)31891-8 |
| [12] | Denkert C, Liedtke C, Tutt A, et al. Molecular alterations in triple-negative breast cancer-the road to new treatment strategies[J]. Lancet, 2017, 389(10087): 2430–2442. DOI:10.1016/S0140-6736(16)32454-0 |
| [13] | Yenjerla M, Panopoulos A, Reynaud C, et al. TD-60 is required for interphase cell cycle progression[J]. Cell Cycle, 2013, 12(5): 837–841. DOI:10.4161/cc.23821 |
| [14] | Cautain B, Hill R, de Pedro N, et al. Components and regulation of nuclear transport processes[J]. FEBS J, 2015, 282(3): 445–462. DOI:10.1111/febs.13163 |
| [15] | Kuang J, Li QY, Fan F, et al. Overexpression of the X-linked ribosomal protein S4 predicts poor prognosis in patients with intrahepatic cholangiocarcinoma[J]. Oncol Lett, 2017, 14(1): 41–46. DOI:10.3892/ol.2017.6137 |
 2019, Vol. 46
2019, Vol. 46


