文章信息
- 基于TCGA数据库的胶质母细胞瘤LncRNA风险预测模型的建立
- Establishment of LncRNA Risk Prediction Model for Glioblastoma Based on TCGA Database
- 肿瘤防治研究, 2019, 46(5): 417-420
- Cancer Research on Prevention and Treatment, 2019, 46(5): 417-420
- http://www.zlfzyj.com/CN/10.3971/j.issn.1000-8578.2019.19.0055
- 收稿日期: 2019-01-15
- 修回日期: 2019-02-22
胶质瘤起源于神经外胚层分化而来的胶质细胞,约占颅内原发肿瘤的70%,是中枢神经系统最常见的原发性肿瘤。世界卫生组织(WHO)将胶质瘤分为Ⅰ~Ⅳ级,级别越高,恶性程度越高,其中Ⅳ级胶质母细胞瘤(Glioblastoma, GBM)占所有胶质瘤的50%,恶性程度最高,经过手术、放疗、化疗等综合治疗后,生存期仍仅有12~15月[1]。深入了解胶质母细胞瘤的分子机制是改善患者预后和识别新的预后生物标志物的关键。
长非编码RNA(LncRNA)是长度超过200个核苷酸的转录物,这些核苷酸不翻译成蛋白质,但在基因转录和mRNA翻译中起调节作用[2-4]。越来越多的与癌症发生发展有关的LncRNA被筛选出来[5]。世界上最大的肿瘤数据存储和分析网站肿瘤基因组图谱(Cancer Genome Atlas, TCGA)至今收录了26种组织类型,共计33个癌种的11 000多名患者的肿瘤基因数据,包括蛋白编码基因、LncRNA、microRNA以及表观遗传学数据[6]。
本研究利用TCGA数据库研究胶质母细胞瘤与正常组织的差异表达LncRNA,从中筛选与胶质母细胞瘤预后相关的LncRNA,建立胶质母细胞瘤预后风险评估的LncRNA模型,并在胶质母细胞瘤患者中进行验证。
1 资料与方法 1.1 数据来源2018年12月20日从TCGA数据库中下载所有胶质母细胞瘤患者的基因表达谱数据及相关的临床数据。
1.2 方法对下载的基因表达谱数据进行注释,区分其中的蛋白编码基因及LncRNA,使用R语言软件edgeR包对下载的数据进行数据标准化,并筛选在胶质母细胞瘤组织和正常组织样本中差异表达的LncRNA,筛选条件为logFC≥2或≤-2,FDR < 0.05。对差异LncRNA采用单因素Cox回归分析,依据P < 0.001筛选影响患者生存的差异LncRNA,纳入多因素Cox回归分析建立LncRNA预后风险评分模型,并计算每位患者的风险评分(risk score, RS),依据评分中位值将患者分为高、低风险组,并进一步验证风险评估模型。
1.3 统计学方法应用R3.5.2软件进行统计学分析及相应图形绘制,edgeR包筛选差异基因,Survival包进行单因素和多因素Cox比例回归模型筛选,并建立多基因预后模型。使用Survival ROC包计算受试者工作特征(ROC)曲线评价模型的有效性,并计算曲线下面积(AUC)。
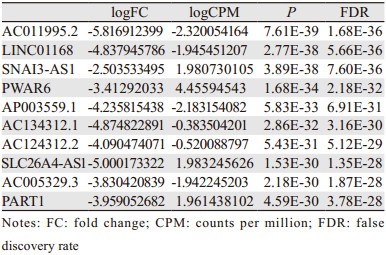
2 结果 2.1 差异LncRNA的筛选从TCGA数据库中下载得到胶质母细胞瘤表基因表达谱矩阵,包含肿瘤组织169份、正常组织标本5份,共33 800个基因的表达,其中LncRNA 14 143个。使用R语言edgeR包进行差异基因分析(logFC≥2或≤-2, FDR < 0.05)得到差异基因7 978个,其中差异LncRNA 1 643个。根据FDR值排序前10位差异表达LncRNA,见表 1。
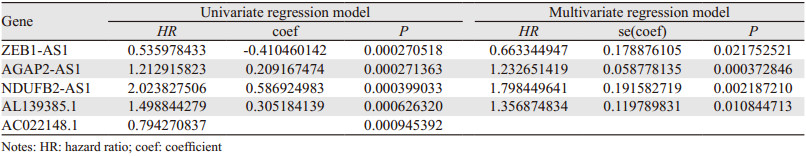
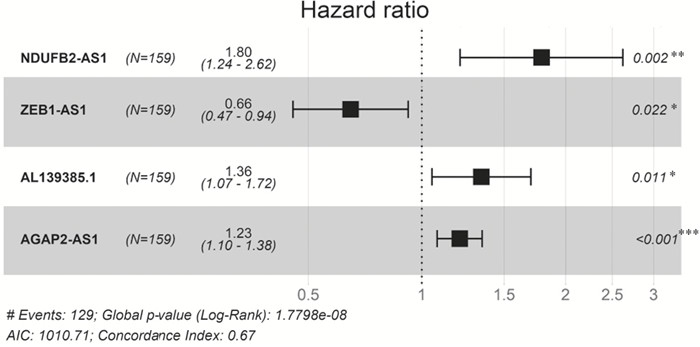
对筛选出的1 643个胶质母细胞瘤和正常组织的差异LncRNA进行单因素Cox回归分析,依据P < 0.001筛选得到影响患者生存的5个LncRNA,分别是ZEB1-AS1、AGAP2-AS1、NDUFB2-AS1、AL139385.1和AC022148.1,将上述5个基因纳入多因素Cox回归分析,构建模型得到基于4个LncRNA的多因素预后风险模型:风险得分=0.59×NDUFB2-AS1-0.41×ZEB1-AS1+0.31×AL139385.1+0.21×AGAP2-AS1,见表 2、图 1。
|
|
| 图 1 多因素Cox回归模型中4个LncRNA森林图 Figure 1 Forest maps of four LncRNA in multivariate Cox regression model |
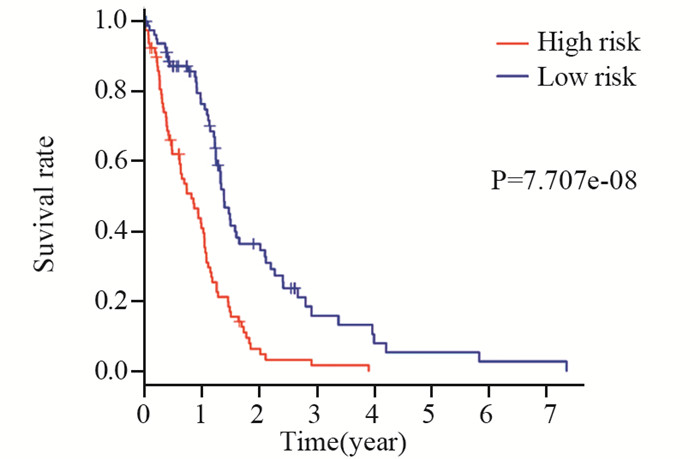
K-M生存分析显示高风险组预后显著差于低风险组(P=7.707×10-8),见图 2。绘制模型的ROC曲线,见图 3,曲线下面积AUC=0.864,模型具有较好的灵敏度和特异性。多因素Cox模型中RS评分与相关LncRNA表达热图及生存时间点图,见图 4,可见随着RS值得升高,患者生存时间呈缩短趋势,且死亡患者(图中红点显示)明显增多。
|
| 图 2 高低风险组的K-M生存分析 Figure 2 K-M survival analysis of high and low risk groups |

|
| 图 3 多因素Cox分析模型ROC曲线 Figure 3 ROC curve of multivariate Cox analysis model |
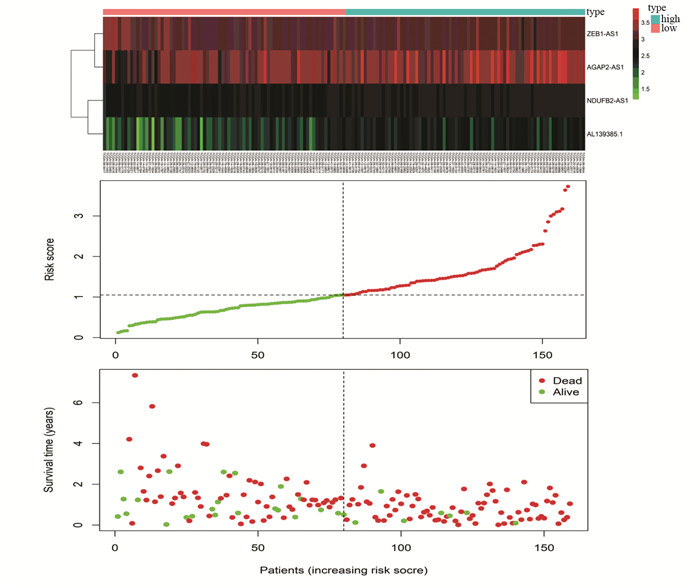
|
| 图 4 风险评分与相关LncRNA表达热图及生存时间点图 Figure 4 Risk score and heat map of related LncRNA expression and scattered plots of survival time |
近年来的研究显示,LncRNA可以通过染色质修饰、转录和翻译过程参与基因调控,在基因调控网络中具有关键性作用。其在各种生理病理过程中,例如上皮-间充质转化、组织再生和肿瘤发生等,具有广泛作用[5, 7-9]。研究证实,不受管制的LncRNA表达谱是癌症的一个新特征,其丰度与肿瘤侵袭性和患者预后有显著相关性[10-11]。
本研究从TCGA数据库中胶质母细胞瘤表达谱数据和临床生存数据进行统计分析得到基于4个LncRNA的多因素预后风险模型:风险得分RS=0.59×NDUFB2-AS1-0.41×ZEB1-AS1+0.31×AL139385.1+0.21×AGAP2-AS1。其中,ZEB1-AS1为保护性LncRNA,其余为危险性LncRNA。这4个LncRNA在其他肿瘤中的研究较少,Tian等[12]研究发现AGAP2-AS1在胶质母细胞瘤中表达高于邻近正常脑组织,且高表达者总生存期更短。对胶质母细胞瘤细胞敲降AGAP2-AS1后,细胞的增殖侵袭能力明显受到抑制。Qi等[13]在胃癌组织和细胞系中得到相似的结论,且研究显示转录因子SP1可以激活AGAP2-AS1的表达,AGAP2-AS1通过与LSD1和EZH2相互作用并抑制CDKN1A(P21)和E-钙黏蛋白转录而发挥致癌作用。Li等[14]研究显示AGAP2-AS1在非小细胞肺癌中高表达,可能通过抑制肿瘤抑制因子LATS2和KLF2转录而作为癌基因发挥作用。
本研究经过TCGA胶质母细胞瘤表达谱数据库的挖掘,筛选出差异表达LncRNA并成功构建风险预测模型,用于计算胶质母细胞瘤患者的风险评分,可以较好地反应患者的预后,且模型有较好的敏感度和特异性,为胶质母细胞瘤的临床预后判断提供帮助,并为基础研究提供更多可供选择的生物标志物。然而本研究尚存在许多不足之处,首先模型的预测能力仍需大量多中心的循证医学证据证实,其次纳入模型的LncRNA在生物体内的功能尚不明确,其在胶质母细胞瘤发生发展中起的作用尚缺乏实验证据的支持。
作者贡献
彭慧:数据分析、文章撰写
秦凯:数据收集下载
戴宇翃:数据整理
张孟贤:数据分析指导
郭秋云:数据分析
| [1] | Balca-Silva J, Matias D, Carmo AD, et al. Cellular and molecular mechanisms of glioblastoma malignancy: Implications in resistance and therapeutic strategies[J]. Semin Cancer Biol, 2018, pii: S1044-579X(18)30072-5. |
| [2] | Trimarchi T, Bilal E, Ntziachristos P, et al. Genome-wide mapping and characterization of Notch-regulated long noncoding RNAs in acute leukemia[J]. Cell, 2014, 158(3): 593–606. DOI:10.1016/j.cell.2014.05.049 |
| [3] | Wu H, Yang L, Chen LL. The Diversity of Long Noncoding RNAs and Their Generation[J]. Trends Genet, 2017, 33(8): 540–52. DOI:10.1016/j.tig.2017.05.004 |
| [4] | Rynkeviciene R, Simiene J, Strainiene E, et al. Non-Coding RNAs in Glioma[J]. Cancers (Basel), 2018, 11(1). pii: E17. |
| [5] | Lin C, Yang L. Long Noncoding RNA in Cancer: Wiring Signaling Circuitry[J]. Trends Cell Biol, 2018, 28(4): 287–301. DOI:10.1016/j.tcb.2017.11.008 |
| [6] | Deng M, Brägelmann J, Schultze JL, et al. Web-TCGA: an online platform for integrated analysis of molecular cancer data sets[J]. BMC Bioinformatics, 2016, 17: 72. DOI:10.1186/s12859-016-0917-9 |
| [7] | Qian X, Zhao J, Yeung PY, et al. Revealing lncRNA Structures and Interactions by Sequencing-Based Approaches[J]. Trends Biochem Sci, 2019, 44(1): 33–52. DOI:10.1016/j.tibs.2018.09.012 |
| [8] | Kopp F, Mendell JT. Functional Classification and Experimental Dissection of Long Noncoding RNAs[J]. Cell, 2018, 172(3): 393–407. DOI:10.1016/j.cell.2018.01.011 |
| [9] | Adams BD, Parsons C, Walker L, et al. Targeting noncoding RNAs in disease[J]. J Clin Invest, 2017, 127(3): 761–71. DOI:10.1172/JCI84424 |
| [10] | Sahu A, Singhal U, Chinnaiyan AM. Long noncoding RNAs in cancer: from function to translation[J]. Trends Cancer, 2015, 1(2): 93–109. DOI:10.1016/j.trecan.2015.08.010 |
| [11] | Huarte M. The emerging role of lncRNAs in cancer[J]. Nat Med, 2015, 21(11): 1253–61. DOI:10.1038/nm.3981 |
| [12] | Tian Y, Zheng Y, Dong X. AGAP2-AS1 serves as an oncogenic lncRNA and prognostic biomarker in glioblastoma multiforme[J]. J Cell Biochem, 2019, 120(6): 9056–62. DOI:10.1002/jcb.v120.6 |
| [13] | Qi F, Liu X, Wu H, et al. Long noncoding AGAP2-AS1 is activated by SP1 and promotes cell proliferation and invasion in gastric cancer[J]. J Hematol Oncol, 2017, 10(1): 48. DOI:10.1186/s13045-017-0420-4 |
| [14] | Li T, Xie J, Shen C, et al. Upregulation of long noncoding RNA ZEB1-AS1 promotes tumor metastasis and predicts poor prognosis in hepatocellular carcinoma[J]. Oncogene, 2016, 35(12): 1575–84. DOI:10.1038/onc.2015.223 |
 2019, Vol. 46
2019, Vol. 46