文章信息
- 肿瘤纯度——肿瘤研究中一个被忽视的潜在混杂因素
- Tumor Purity: A Potential Ignored Confounder in Tumor Research
- 肿瘤防治研究, 2019, 46(6): 570-574
- Cancer Research on Prevention and Treatment, 2019, 46(6): 570-574
- http://www.zlfzyj.com/CN/10.3971/j.issn.1000-8578.2019.19.0018
- 收稿日期: 2019-01-07
- 修回日期: 2019-04-09
2. 430079 武汉,湖北省肿瘤医院超声科
2. Department of Ultrasound, Hubei Cancer Hospital, Wuhan 430079, China
免疫治疗发展迅速,在多种肿瘤类型中都取得了重大突破,是一种前景广阔的治疗手段。但是,其在大多数实体瘤中有效率仅为20~30%[1],而对于有效人群的筛选目前尚缺乏特异性手段。最常用的疗效预测标志物有细胞程序性死亡配体-1(programmed cell death ligand-1, PD-L1)表达水平、肿瘤突变负荷(tumor mutational burden, TMB)、高度卫星不稳定性或错配修复缺陷等[2-5]。肿瘤组织结构复杂,除了肿瘤细胞,也有基质细胞、炎性细胞、脉管系统和细胞外基质等,共同构成肿瘤微环境[6]。研究显示PD-L1表达量与免疫检查点抑制剂疗效呈正相关,但未明确区分PD-L1表达的细胞来源,如肿瘤细胞还是免疫细胞或是肿瘤微环境中其他细胞[7]。
肿瘤纯度是指肿瘤组织中肿瘤细胞所占的比例。研究显示肿瘤纯度与肿瘤患者的临床特征、基因组表达和生物学特性均显著相关,忽视肿瘤纯度的影响可导致肿瘤基因分型、复发风险及疗效预测等过程产生系统性偏倚[8],准确评估肿瘤纯度有助于客观分析肿瘤样本。然而,目前极少有肿瘤分型、临床研究、标志物检测等研究过程考虑了肿瘤纯度的影响。非肿瘤细胞影响肿瘤纯度,在肿瘤生物学中扮演重要的角色。本文主要从概念、计算方式及对基因组分析的影响三个方面介绍肿瘤纯度。
1 肿瘤纯度概念临床工作中获得的肿瘤组织往往混合了非肿瘤细胞,其在肿瘤生长、进展或耐药等过程中具有重要作用,如基质或间质细胞可促进肿瘤生长并影响肿瘤治疗反应[9],而免疫细胞如肿瘤浸润性细胞毒性T淋巴细胞(cytotoxic T lymphocytes, CTLs)可能抑制肿瘤生长[10]。
肿瘤组织中肿瘤细胞所占的比例即为肿瘤纯度。肿瘤纯度很大程度上取决于肿瘤组织的获取方式,标准的肿瘤手术标本取样纯度通常低于70%。虽然癌症基因组图谱(The Cancer Genome Atlas, TCGA)数据库认为60%的纯度就足以区分肿瘤成分与非肿瘤成分,但是肿瘤纯度会导致基于基因组分析的肿瘤研究结果产生系统性偏倚,对肿瘤纯度进行评估可以减少分析偏倚[11]。
2 肿瘤纯度的计算细胞分选技术和单细胞测序可以用于确定肿瘤纯度,但由于成本和技术的限制,不可能用于大规模研究。传统的肿瘤纯度计算方法是根据病理图像分析来估计,往往受组织病理学、观察者和试剂仪器精确度等各方面的影响。随着基因组学、表观遗传组学、计算机及统计学等方面技术的发展进步,已经可以根据肿瘤样本的基因表达、突变、拷贝数或甲基化数据分析计算肿瘤纯度[12-14]。
2.1 经典计算方法(1)HE染色。通过苏木精和伊红染色剂载玻片的图像分析来估计肿瘤纯度。(2)白细胞去甲基化纯度(leukocytes unmethylation for purity, LUMP)。根据44个非甲基化免疫特异性CpG位点平均值计算白细胞非甲基化程度,进而估计肿瘤纯度。(3)ABSOLUTE[13]。通过SNP array(基因芯片技术)测量体细胞拷贝变异数据,利用极大似然模型估计肿瘤纯度,是一种直接测量样本中的肿瘤细胞的计算方式。大多数TCGA样本都采用ABSOLUTE方式计算肿瘤纯度,被认为是纯度估计的金标准。(4)ESTIMATE[15]。利用单个样本的基因富集分析以及免疫细胞和基质细胞的基因表达谱,推断肿瘤样本中基质细胞和免疫细胞浸润程度,根据经验累计分布函数来估计肿瘤细胞纯度。LUMP和ESTIMATE是通过测量样本中免疫成分和基质成分间接评估肿瘤纯度,这两种方法无法测量样本中非免疫和基质成分(如正常细胞)的存在,评估出的肿瘤纯度较ABSOLUTE可能有差异。有研究者利用这四种方法计算出的肿瘤纯度的中值作为肿瘤样本纯度。
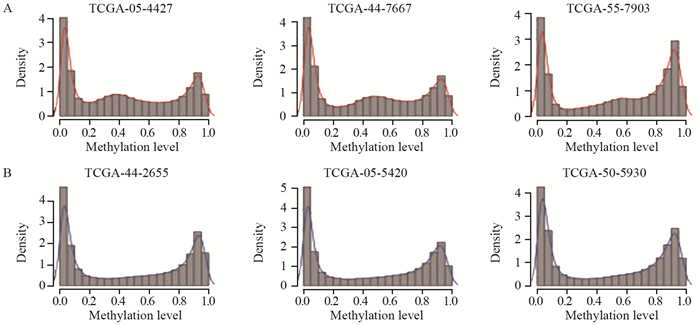
2.2 新型计算方法(1)Zheng等[14]提出的统计算法MethylPurify是利用亚硫酸氢盐来显示差异甲基化区域,从而仅从单个肿瘤样品推断肿瘤纯度,是第一种不需要正常细胞、肿瘤细胞或细胞学基因组变异信息或其他信息进行参照的方法,但是该方法需要足够高的亚硫酸氢盐的测序深度,价格昂贵,限制了其临床应用。(2)系统分析TCGA数据库中32种肿瘤类型的450k甲基化芯片数据之后发现:与正常样本相比,肿瘤样本甲基化水平除了在0/1附近富集,在中间区域也出现了一个明显的峰,肿瘤样本在中间甲基化区域位点的比例更高,而这种现象正是由于肿瘤样本“不纯”导致,见图 1。InfiniumPurify则利用肿瘤样本中与正常样本甲基化差异显著的位点,将其分为高甲基化组和低甲基化组,然后利用高斯核密度估计方法计算肿瘤样本的纯度。该方法估算出的肿瘤纯度值与ESTIMATE、ABSOLUTE、LUMP、IHC等经典方法得到的结果有较高的一致性,进一步分析发现,当正常样本个数大于或等于30时,肿瘤样本纯度估计与样本的选择无关[16-17]。在此基础上进一步开发的UiInfiniumPurify则利用肿瘤样本和正常样本通用的CpG位点估计肿瘤纯度,可运用于TCGA数据库之外肿瘤样本较少的肿瘤组织纯度估计[18]。(3)AbsCN-seq是Bao等[19]开发的一种简单稳定的可根据全基因组测序数据中评估肿瘤纯度的算法。在NCI60细胞系、dbGAP、TCGA等数据库中验证,与ABSOLUTE一致性良好,并且可与ABSOLUTE互补,用于对方不适用的情况。(4)PAMES(Purity Assessment from Clonal Methylation Sites)是Benelli等[20]开发的利用高度克隆的肿瘤类型特异性CpG位点的甲基化水平来估计肿瘤样品纯度的方法,不需要匹配正常对照,也不受肿瘤微环境的影响。在不同数据集的6 000多个样本和肿瘤细胞系进行了验证评估,与其他方式的计算结果高度一致。并且研究者将PAMES的计算能力扩展到利用CpG岛进行分析,而不仅限于特异性的CpG位点。(5)Sequenza是Favero等[21]开发的利用配对的肿瘤DNA和正常细胞DNA外显子测序数据估计肿瘤纯度的软件。利用TCGA数据库中的30个肿瘤样本的外显子数据进行验证,Sequenza分析结果与等位基因特异性拷贝数函数分析SNP array的检测结果高度一致,并且优于AbsCN-seq、ABSOLUTE。此外,除了计算肿瘤纯度,目前已构建多种校正肿瘤纯度的基因组分析统计模型,如差异表达分析、表达数量性状基因座识别、聚类分析等[22-24]。
肿瘤纯度是否代表生物学相关性和肿瘤内在特征,或是仅仅代表外在特征(如手术切除和组织准备)所决定的系统性偏倚仍存在争议。(1)Aran等[8]评估了数百个临床特征与肿瘤纯度之间的相关性,但并未发现两者之间有明确关联,故认为肿瘤纯度差异很大程度上取决于外在因素。(2)Zhang等[25]针对脑胶质瘤的分析表明纯度较低的肿瘤恶性程度更高、预后更差,将肿瘤纯度作为影响因子纳入预测预后的列线图中精确度显著提高。不同纯度的样本基因组学特征也有所不同,低纯度的胶质瘤多具有7号染色体扩增与10号染色体缺失(胶质母细胞瘤基因组标志),高纯度的胶质瘤多具有染色体1p19q共缺失(少突神经胶质瘤的基因组标志)。此外,低纯度胶质瘤免疫表型更强。(3)在结肠癌研究中也发现低肿瘤纯度是预后差的独立预测因素,纯度低的肿瘤突变负荷更高,免疫表型更强[26]。
肿瘤纯度具有重要的临床、基因组和生物学意义,是癌症基因组或转录组分析的重要混杂因素。
3.1 对肿瘤转录组分析的影响(以免疫相关基因为例)肿瘤组织混合了肿瘤细胞和非肿瘤细胞的RNA转录物,其中非肿瘤细胞RNA转录物会影响肿瘤表达谱,使肿瘤转录组分析复杂化。Aran等[8]的研究表明肿瘤组织中正常细胞特别是免疫细胞的比例,可使遗传分析或其他检测的结果产生严重偏差。免疫细胞是非肿瘤细胞主要成分之一,肿瘤组织中免疫相关基因的表达可能与其有关,因此肿瘤转录组分析尤其是侧重于免疫相关基因的分析必须考虑肿瘤纯度的问题。肿瘤基因组的TMB和免疫治疗相关靶基因的表达被认为是免疫检查点抑制剂疗效的可靠预测指标,但是只有当肿瘤组织中免疫细胞的浸润程度明确量化时,这些指标用于预测检查点抑制剂药物有效性才是精确的。
Rhee等[11]从TCGA数据库获得21种肿瘤类型共7794例肿瘤标本的RNA测序基因表达谱和肿瘤纯度,计算个体基因表达和肿瘤纯度的相关性,据此将基因分为与肿瘤纯度正相关或负相关两组,并进行基因富集分析以鉴定两组基因的分子功能。结果显示:与肿瘤纯度负相关的基因中免疫相关基因持续富集,与肿瘤纯度正相关的基因根据不同的分子功能富集。肿瘤纯度较低的肿瘤样本免疫细胞更多,突变负荷往往较高,因为免疫细胞引起的炎性反应可以增加肿瘤细胞的突变率,免疫治疗效果可能更佳[8]。有研究表示免疫相关基因的表达与突变负荷之间具有一定的相关性。粒酶A、穿孔素-1可代表肿瘤浸润性T淋巴细胞活性,计算两者表达的几何平均值作为细胞溶解活性(cytolytic activity, CYT),研究显示CYT与突变负荷显著相关,与患者总生存率一定程度上相关[27]。然而,Rhee等发现CYT和突变负荷均与肿瘤纯度显著负相关,控制肿瘤纯度之后,免疫相关基因与突变负荷的相关性降低,可能与肿瘤纯度有关。进一步研究在控制或不控制肿瘤纯度时CYT和生存率之间的关系发现,未控制肿瘤纯度时,乳腺浸润癌、低级别胶质瘤、肝细胞癌和皮肤黑色素瘤等多种肿瘤中都观察到显著相关,但在控制肿瘤纯度之后,仅膀胱尿路上皮癌显示CYT基因表达与患者存活率之间有显著关联。即CYT基因表达与生存率的相关性也可能是由肿瘤纯度引起的。不同肿瘤类型的CYT与生存关系的差异可能不能单纯用肿瘤纯度来解释,但也应将肿瘤纯度作为临床病理特征相关性分析,如患者免疫基因的表达与存活率的相关性分析中潜在的混杂因素。
免疫相关基因在与肿瘤纯度呈负相关的基因中占主要部分,且无论肿瘤纯度如何,免疫相关基因始终与肿瘤纯度负相关。从转录组数据中获得免疫治疗表达标志物时需要考虑肿瘤纯度。
3.2 对基因聚类的影响基因聚类分析是将基因分为数类,并使同一类的基因尽可能相似,而不同类的基因相似度很小。通过在分子标记的基础上对肿瘤进行分组的基因聚类可对肿瘤进行亚型分析,实现精准治疗。肿瘤纯度在多种肿瘤类型中对基因共表达和基因聚类有重要影响,大量基因对在纯度调整后失去了聚类成员一致性,并且在不同肿瘤类型中影响程度不尽相同[11]。
通过共表达基因对可以发现肿瘤相关驱动基因和与转移相关的分子通路,而有研究发现共表达基因对的出现可能仅仅归因于肿瘤纯度。膀胱癌中JAK3和CSF1R为高水平共表达基因对,提示这两个基因可能采取共同的机制促进肿瘤发展。但如果考虑肿瘤纯度,两者共表达差异很大,纯度高的肿瘤中两者表达的相关性不大,因此这两个基因在膀胱癌驱动和转移通路中是否具有联合作用还有待确定,也突出了纯度调整的必要性[8]。Zhang等[22]研究发现,在聚类分析时,具有相似纯度的肿瘤样本倾向于分为一类,如肿瘤纯度较低的样本,其甲基化谱与正常细胞甲基化谱更接近的倾向于分为一类。Rhee等[11]将基因通过聚类分析分配到六个基因簇中,基因对按照是否属于同一簇分为聚类匹配对和聚类不匹配对,然后分别在控制或不控制肿瘤纯度时检查了基因对的聚类成员。结果显示在部分肿瘤类型中,属于同一簇的基因对在控制肿瘤纯度后55%以上将被分配到不同的簇。为进一步验证校正基因纯度的必要性,分别测量控制肿瘤纯度前后聚类匹配对和聚类不匹配对的蛋白质-蛋白质相互作用(protein-protein interaction, PPI)基因对的频率。无论控制肿瘤纯度与否,聚类匹配对PPI基因对频率均高于聚类不匹配对,控制肿瘤纯度前后聚类匹配结果一致的基因对中PPI基因对频率比不一致基因对更高,纯度调整可以增加基因聚类的功能一致性。
3.3 对分子分类的影响利用特征基因作为基因分类的标志可对肿瘤进行分子分类,肿瘤纯度可能影响该过程[28]。肿瘤纯度在不同肿瘤亚型中有所不同,特征基因在不同肿瘤亚型和整体肿瘤中与肿瘤纯度的相关性也不同。如840个特征基因将TCGA多形性胶质母细胞瘤(glioblastoma multiforme, GBM)分为四个亚型(前神经元型、神经元型、经典型和间质型),代表四个GBM亚型的特征基因与肿瘤纯度显著正相关(前神经元型和经典型)或负相关(神经元型和间充质型)[11]。而GBM特征基因对正相关或负相关的单一分布正好可以解释Aran等[8]观察到的整体GBM特征基因表达与肿瘤纯度相关性的双峰分布。进一步使用与肿瘤纯度正相关或负相关的部分特征基因进行分层聚类,发现主要在神经元型和前神经元型分子分类中存在干扰。GBM的分子分类学以及特征基因的选择可能因肿瘤纯度而有偏差[11]。类似的其他研究也发现间质肿瘤亚型的代表性表达信号主要归因于基质细胞,而不是肿瘤细胞[29-30]。
3.4 对差异分析的影响基因突变导致基因表达模式发生异常,这些差异表达的基因可能与肿瘤的发生发展相关。研究发现差异分析也受肿瘤纯度的影响。在不考虑肿瘤纯度的情况下,对肿瘤免疫治疗重要靶点CTLA-4和CD86蛋白的相对表达进行差异分析的结果可能有偏倚[8]。此外,校正肿瘤纯度的差异表达分析可以发现传统差异表达分析不能检测的免疫治疗基因特征[8]。
4 展望对肿瘤进行基因组分析有助于增进对肿瘤的理解,了解其发生发展的分子机制,帮助肿瘤的早期诊断和治疗,更是肿瘤精准治疗的基础。然而,肿瘤是一种复杂的微环境,除了肿瘤细胞还有其他多种细胞类型,如正常细胞、免疫细胞、基质细胞、血管细胞等,肿瘤组织的不纯会使遗传分析及其他检测结果产生严重偏差。肿瘤纯度对基因组分析,包括基因转录组分析、基因聚类、分子分类和差异分析等各方面都有显著影响,因此,对肿瘤组织进行分析时除了肿瘤细胞,也该关注非肿瘤细胞并建立合适的统计模型计算肿瘤纯度,以校正肿瘤纯度对基因组分析带来的偏差。肿瘤纯度的影响给肿瘤精准治疗带来更多的挑战与困难,希望将来有更多针对肿瘤组织的研究将肿瘤纯度精确测量纳入分析,以校正肿瘤纯度的影响,准确阐述肿瘤生物学的基本原理,为患者的精准化个性医疗开辟新的途径。
作者贡献
李岚:确定主题、文献查阅、撰写文章
许斌:写作指导、对文章知识性内容进行审阅
查莉:写作指导
| [1] | Iwai Y, Hamanishi J, Chamoto K, et al. Cancer immunotherapies targeting the PD-1 signaling pathway[J]. J Biomed Sci, 2017, 24(1): 26. DOI:10.1186/s12929-017-0329-9 |
| [2] | Sacher AG, Gandhi L. Biomarkers for the Clinical Use of PD-1/PD-L1 Inhibitors in Non-Small-Cell Lung Cancer: A Review[J]. JAMA Oncol, 2016, 2(9): 1217. DOI:10.1001/jamaoncol.2016.0639 |
| [3] | Yarchoan M, Hopkins A, Jaffee EM. Tumor Mutational Burden and Response Rate to PD-1 Inhibition[J]. N Engl J Med, 2017, 377(25): 2500–1. DOI:10.1056/NEJMc1713444 |
| [4] | Baretti M, Le DT. DNA Mismatch Repair in Cancer[J]. Pharmacol Ther, 2018, 189: 45–62. DOI:10.1016/j.pharmthera.2018.04.004 |
| [5] | Meng X, Huang Z, Teng F, et al. Predictive biomarkers in PD-1/PD-L1 checkpoint blockade immunotherapy[J]. Cancer Treat Rev, 2015, 41(10): 868–76. DOI:10.1016/j.ctrv.2015.11.001 |
| [6] | Tang H, Qiao J, Fu YX. Immunotherapy and tumor microenvironment[J]. Cancer Lett, 2016, 370(1): 85–90. |
| [7] | Zou W, Wolchok JD, Chen L. PD-L1 (B7-H1) and PD-1 pathway blockade for cancer therapy: Mechanisms, response biomarkers, and combinations[J]. Sci Transl Med, 2016, 8(328): 328rv4. DOI:10.1126/scitranslmed.aad7118 |
| [8] | Aran D, Sirota M, Butte AJ. Systematic pan-cancer analysis of tumour purity[J]. Nat Commun, 2015, 6: 8971. DOI:10.1038/ncomms9971 |
| [9] | Junttila MR, de Sauvage FJ. Influence of tumour micro-environment heterogeneity on therapeutic response[J]. Nature, 2013, 501(7467): 346–54. DOI:10.1038/nature12626 |
| [10] | Pagès F, Galon J, Dieu-Nosjean MC, et al. Immune infiltration in human tumors: a prognostic factor that should not be ignored[J]. Oncogene, 2010, 29(8): 1093–102. DOI:10.1038/onc.2009.416 |
| [11] | Rhee JK, Jung YC, Kim KR, et al. Impact of tumor purity on immune gene expression and clustering analyses across multiple cancer types[J]. Cancer Immunol Res, 2018, 6(1): 87–97. DOI:10.1158/2326-6066.CIR-17-0201 |
| [12] | Zhu W, Kuziora M, Creasy T, et al. BubbleTree: an intuitive visualization to elucidate tumoral aneuploidy and clonality using next generation sequencing data[J]. Nucleic Acids Res, 2016, 44(4): e38. DOI:10.1093/nar/gkv1102 |
| [13] | Carter SL, Cibulskis K, Helman E, et al. Absolute quantification of somatic DNA alterations in human cancer[J]. Nat Biotechnol, 2012, 30(5): 413–21. DOI:10.1038/nbt.2203 |
| [14] | Zheng X, Zhao Q, Wu HJ, et al. MethylPurify: tumor purity deconvolution and differential methylation detection from single tumor DNA methylomes[J]. Genome Biol, 2014, 15(8): 419. |
| [15] | Yoshihara K, Shahmoradgoli M, Martínez E, et al. Inferring tumour purity and stromal and immune cell admixture from expression data[J]. Nat Commun, 2013, 4: 2612. DOI:10.1038/ncomms3612 |
| [16] | Zhang N, Wu HJ, Zhang W, et al. Predicting tumor purity from methylation microarray data[J]. Bioinformatics, 2015, 31(21): 3401–5. DOI:10.1093/bioinformatics/btv370 |
| [17] | Zheng X, Zhang N, Wu HJ, et al. Estimating and accounting for tumor purity in the analysis of DNA methylation data from cancer studies[J]. Genome Biol, 2017, 18(1): 17. |
| [18] | Dou H, Fang Y, Zheng X. Universal informative CpG sites for inferring tumor purity from DNA methylation microarray data[J]. J Bioinform Comput Biol, 2018, 16(30): 1750030. |
| [19] | Bao L, Pu M, Messer K. AbsCN-seq: a statistical method to estimate tumor purity, ploidy and absolute copy numbers from next-generation sequencing data[J]. Bioinformatics, 2014, 30(8): 1056–63. DOI:10.1093/bioinformatics/btt759 |
| [20] | Benelli M, Romagnoli D, Demichelis F. Tumor purity quantification by clonal DNA methylation signatures[J]. Bioinformatics, 2018, 34(10): 1642–9. DOI:10.1093/bioinformatics/bty011 |
| [21] | Favero F, Joshi T, Marquard AM, et al. Sequenza: allele-specific copy number and mutation profiles from tumor sequencing data[J]. Ann Oncol, 2015, 26(1): 64–70. DOI:10.1093/annonc/mdu479 |
| [22] | Zhang W, Feng H, Wu H, et al. Accounting for tumor purity improves cancer subtype classification from DNA methylation data[J]. Bioinformatics, 2017, 33(17): 2651–7. DOI:10.1093/bioinformatics/btx303 |
| [23] | Zhang W, Long H, He B, et al. DECtp: Calling Differential Gene Expression Between Cancer and Normal Samples by Integrating Tumor Purity Information[J]. Front Genet, 2018, 9: 321. DOI:10.3389/fgene.2018.00321 |
| [24] | Geeleher P, Nath A, Wang F, et al. Cancer expression quantitative trait loci (eQTLs) can be determined from heterogeneous tumor gene expression data by modeling variation in tumor purity[J]. Genome Biol, 2018, 19(1): 130. DOI:10.1186/s13059-018-1507-0 |
| [25] | Zhang C, Cheng W, Ren X, et al. Tumor Purity As an Underlying Key Factor in Glioma[J]. Clin Cancer Res, 2017, 23(20): 6279–91. DOI:10.1158/1078-0432.CCR-16-2598 |
| [26] | Mao Y, Feng Q, Zheng P, et al. Low tumor purity is associated with poor prognosis, heavy mutation burden, and intense immune phenotype in colon cancer[J]. Cancer Manag Res, 2018, 10: 3569–77. DOI:10.2147/CMAR |
| [27] | Rooney MS, Shukla SA, Wu CJ, et al. Article: Molecular and Genetic Properties of Tumors Associated with Local Immune Cytolytic Activity[J]. Cell, 2015, 160(1-2): 48–61. DOI:10.1016/j.cell.2014.12.033 |
| [28] | Elloumi F, Hu Z, Li Y, et al. Systematic Bias in Genomic Classification Due to Contaminating Non-neoplastic Tissue in Breast Tumor Samples[J]. BMC Med Genomics, 2011, 4: 54. DOI:10.1186/1755-8794-4-54 |
| [29] | Isella C, Terrasi A, Bellomo SE, et al. Stromal contribution to the colorectal cancer transcriptome[J]. Nat Genet, 2015, 47(4): 312–9. DOI:10.1038/ng.3224 |
| [30] | Moffitt RA, Marayati R, Flate EL, et al. Virtual microdissection identifies distinct tumor- and stroma-specific subtypes of pancreatic ductal adenocarcinoma[J]. Nat Genet, 2015, 47(10): 1168–78. DOI:10.1038/ng.3398 |
 2019, Vol. 46
2019, Vol. 46