文章信息
- lncRNA MEG3通过调控miR-543/PTEN分子轴抑制胰腺癌细胞增殖及转移的机制
- LncRNA MEG3 Suppresses Proliferation and Metastasis of Pancreatic Cancer Cells via Regulating miR-543/PTEN Axis
- 肿瘤防治研究, 2019, 46(7): 588-593
- Cancer Research on Prevention and Treatment, 2019, 46(7): 588-593
- http://www.zlfzyj.com/CN/10.3971/j.issn.1000-8578.2019.18.1876
- 收稿日期: 2019-01-24
- 修回日期: 2019-03-26
虽然胰腺癌的诊断和治疗已经取得了明显的进步,但胰腺癌高侵袭性和易转移性,造成患者高死亡率和极差的预后。为此,寻找可用于胰腺癌早期诊断及预后判断的生物标志物具有重要的临床意义。近年来,长链非编码RNA(long noncoding RNA, lncRNA)已被证实在多种肿瘤的增殖和转移过程中发挥重要调节作用[1-2]。母系表达基因3(maternally expressed gene 3, MEG3)作为肿瘤的抑癌基因[3]参与调控宫颈癌[4]、结直肠癌[5]、慢性髓系白血病[6]、胰腺癌[7]等多种恶性肿瘤细胞增殖和侵袭。同时,多项研究发现,lncRNAs与miRNAs的相互作用在癌症发生过程中起重要作用,而miRNAs能够通过调控其下游蛋白介导肿瘤发展进程[8]。这预示lncRNA-miRNA-mRNA调控网络有望成为胰腺癌诊断和治疗的分子标志物。本研究通过检测MEG3在胰腺癌临床组织样本及细胞系中的表达,再经细胞实验进一步验证MEG3通过调控miR-543/PTEN分子轴介导胰腺癌PANC-1细胞增殖和转移的作用机制。
1 材料与方法 1.1 样本收集、细胞和主要试剂收集2012年4月至2017年12月间青岛大学附属医院收治的资料完整的胰腺癌30例,所有研究对象对本研究均签署知情同意书。本研究经青岛大学附属医院伦理委员会批准。人胰腺癌细胞株(PANC-1, Capan-1, BaxPC-3和SW1990)和人胰腺导管上皮细胞株(HPDE6-C7)均购自中科院上海细胞研究所。DMEM和胎牛血清购自美国Gibco公司;Lipofectamine 2000和反转录试剂盒购自日本TaKaRa公司;CCK-8试剂盒购自南京建成生物工程研究所;Transwell小室购自美国康宁公司;高纯总RNA快速提取试剂盒购自北京百泰克生物技术有限公司;蛋白提取试剂盒和SDS-PAGE凝胶快速制备试剂盒购自美国Bio-Rad公司;双荧光素酶报告基因试剂盒和报告基因载体购自索莱宝科技公司;免疫印迹一抗(抗PTEN抗体、抗E-cadherin抗体、抗N-cadherin抗体和抗Vimentin抗体)和二抗(IgG(H+L))均购自美国CST公司。
1.2 细胞培养和转染人胰腺癌细胞株和人胰腺导管上皮细胞采用含10%胎牛血清的DMEM培养液在37℃、5%CO2培养箱中常规培养。
选取对数生长期的胰腺癌(PANC-1)细胞,胰酶消化后,利用DMEM培养基调整浓度细胞为每毫升1×105个。将细胞接种到6孔板,每孔添加2 ml细胞悬液,并于37℃、5%CO2培养箱培养24 h,进行pcDNA-MEG3、MEG3 shRNA、miR-543 mimics/inhibitor和PTEN siRNA转染,转染方法参考Lipofectamine 2000转染试剂说明书,转染48 h后于荧光显微镜下观察细胞转染效果。
1.3 实验方法 1.3.1 qRT-PCR法检测MEG3、miR-543和PTEN的表达收集临床样本及转染后48 h的胰腺癌细胞,并采用TRIzol一步法分别提取组织和培养好的细胞中总RNA。NanoDrop检测RNA的浓度及纯度,并反转录制备cDNA。取2 μl反转录产物进行PCR检测。按试剂盒说明建立终体积为20 μl的PCR反应体系:2 μl反转录产物、10 μl SYBR Green Mix、上下游引物(10 μmol/L)各0.5 μl,并采用ddH2O补足至20 μl。PCR热循环参数为:95℃ 5 min,然后3步反应:94℃变性30 s,60℃退火30 s,72℃延伸60 s,45个循环。引物序列如下,U6: F: 5’-GCTTCGGCAGCACATATACTAAAAT-3’,R: 5’-CGCTTCACGAATTTGCGTGTCAT-3’。GAPDH: F: 5’-GGTCTCCTCTGACTTCAACA-3’,R: 5’-GTGAGGGTCTCTCTCTTCCT-3’。MEG3: F: 5’-CAGCCAAGCTTCTTGAAAGG-3’,R: 5’-TTCCACGGAGTAGAGCGAGT-3’。miR-543: F: 5’-GGGGAAACATTCGCGGTGCA-3’,R: 5’-TGCGTGTCGTGGAGTC-3’。检测结果采用2-ΔΔCt法进行计算。实验重复3次。
1.3.2 Western blot检测PANC-1细胞中PTEN和EMT相关蛋白的表达收集转染48 h的细胞,使用RIPA蛋白裂解液提取各组细胞总蛋白,BCA检测蛋白浓度。使用10%SDS-PAGE进行电泳分离目的蛋白,电转法转移蛋白至PVDF膜上,封闭后加入相应一抗4℃过夜孵育,洗涤后HRP标记二抗37℃孵育1 h,使用ECL化学发光剂显色,最后在凝胶成像仪上扫描分析,采用Image J软件分析靶带的灰度水平。实验重复3次。
1.3.3 CCK-8实验检测PANC-1细胞增殖活力将处于对数生长期的胰腺癌PANC-1细胞接种于96孔板,每孔含细胞1×105个,每孔含培养基100 μl。待检测前1 h向每孔加入10 μl CCK-8溶液。将培养板在培养箱内孵育1~4 h。用酶标仪测定450 nm处的吸光度。实验重复3次。
1.3.4 Transwell实验检测PANC-1细胞侵袭能力选择转染细胞为实验组,未转染细胞为对照组。分别将各处理组细胞用胰酶消化处理后接种于Transwell小室24孔板内,上室加100 μl(密度为1×105个/毫升)细胞悬液,下室加250 μl含10%胎牛血清的培养基,37℃、5%CO2培养箱中培养48 h,取出小室,棉签擦去微孔膜上室的细胞,PBS小心冲洗小室2遍,4%多聚甲醛固定侵袭并黏附到小室微孔膜下面的细胞15 min,结晶紫染色15 min,PBS冲洗小室,干燥后置于100倍的倒置显微镜下观察。每组实验重复3次。
1.3.5 双荧光素酶报告基因验证MEG3、miR-543和PTEN的靶向关系利用生物信息分析软件预测MEG3与miR-543以及miR-543与PTEN的结合片段,PCR分别扩增MEG3和PTEN与miR-543的结合片段。扩增产物插入到pGL3-Promoter质粒载体中,构建MEG3和PTEN野生型(WT)质粒;利用基因突变技术将结合片段定点突变,构建MEG3和PTEN突变型(MUT)质粒。将重组质粒分别与miR-543、miR-NC共同转染293T细胞。转染48 h后,收集细胞。荧光素酶检测按照双荧光素酶报告基因试剂盒说明书,采用酶标仪检测萤火虫和海肾荧光值,并以海肾荧光值作为内参。
1.4 统计学方法采用SPSS20.0软件进行统计分析,计量数据以(x±s)表示,多组间采用单因素方差分析,进一步两两比较采用LSD-t检验。GraphPad Prism 7对实验数据进行相关图片的绘制。以P < 0.05为差异有统计学意义。
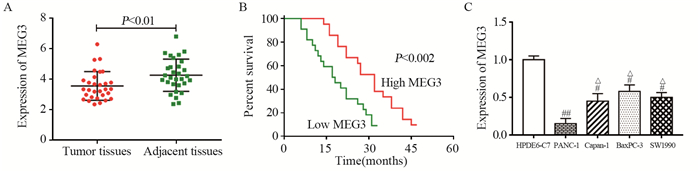
2 结果 2.1 MEG3在胰腺癌组织及细胞系中的表达qRT-PCR检测结果显示,MEG3在胰腺癌组织中的表达水平明显低于癌旁组织(t=2.585, P=0.006),见图 1A。同时,采用Kaplan-Meier生存曲线分析发现,MEG3表达下调的胰腺癌患者预后较差,生存率显著低于MEG3高表达的患者(P=0.002),见图 1B。此外,MEG3在胰腺癌细胞系(PANC-1、Capan-1、BaxPC-3和SW1990)中的表达水平显著低于人胰腺导管上皮细胞(HPDE6-C7)(F=20.717, P=0.003),且PANC-1细胞中MEG3的表达水平明显低于Capan-1、BaxPC-3和SW1990(F=7.011, P=0.045),见图 1C,故选择该PANC-1细胞进行后续实验。由此可知,MEG3的异常表达可能与胰腺癌发生发展有关联。
|
| #: P < 0.05, ##: P < 0.01, compared with HPDE6-C7 cells; △: P < 0.05, compared with PANC-1 cells 图 1 MEG3在胰腺癌组织及细胞系中的表达水平 Figure 1 Expression of MEG3 in pancreatic cancer tissues and cell lines |
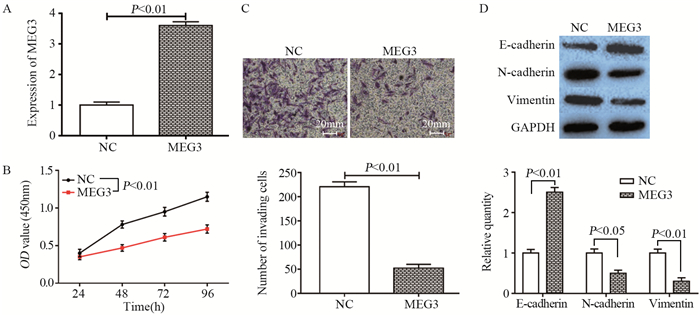
qRT-PCR检测结果证实,过表达MEG3可稳定上调PANC-1细胞中MEG3的表达水平(t=16.645, P=0.002),见图 2A。CCK-8检测结果证实,过表达MEG3可显著抑制PANC-1细胞增殖活力(t=4.530, P=0.005),见图 2B。同时,Transwell检测结果显示,过表达MEG3后PANC-1细胞侵袭能力明显受到抑制(t=13.197, P=0.003),见图 2C。Western blot检测结果表明,相比于对照组,过表达MEG3显著促进了PANC-1细胞上皮细胞标志物E-cadherin的表达(t=10.040, P=0.005),明显下调了间质细胞标志物N-cadherin和Vimentin的表达水平(t1=3.904, P=0.030, t2=5.349, P=0.017),见图 2D。由此可知,过表达MEG3显著抑制了PANC-1细胞增殖、侵袭和上皮间质转化(EMT)能力。
|
| 图 2 过表达MEG3对胰腺癌细胞增殖、侵袭和上皮间质转化的影响 Figure 2 Effect of MEG3 overexpression on proliferation, invasion and epithelial mesenchymal transition (EMT) of pancreatic cancer cells |
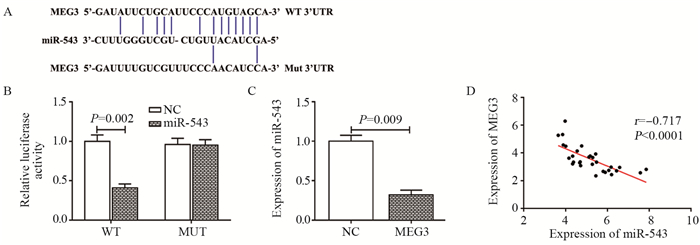
生物信息学数据库StarBase预测发现,miR-543是MEG3的靶基因,其预测序列如图 3A所示。双荧光素酶报告基因验证结果显示,过表达miR-543可以使荧光素酶活性显著下降(t=6.254, P=0.002),见图 3B,而共转染miR-543 mimics和靶向位点发生突变的pmirGLO-MEG3-mut载体,miR-543对荧光素酶活性的抑制作用丧失。进一步,qRT-PCR分析结果表明,过表达MEG3后显著抑制了miR-543的表达(t=7.080, P=0.009),见图 3C。此外,通过对30例胰腺癌组织进行检测发现,MEG3与miR-543的表达呈负相关(r=-0.717, P < 0.0001),见图 3D。由此可知,miR-543是MEG3的直接靶基因,且MEG3可负调控miR-543的表达。
|
| 图 3 MEG3对miR-543表达的调控作用 Figure 3 Effect of MEG3 on miR-543 expression |
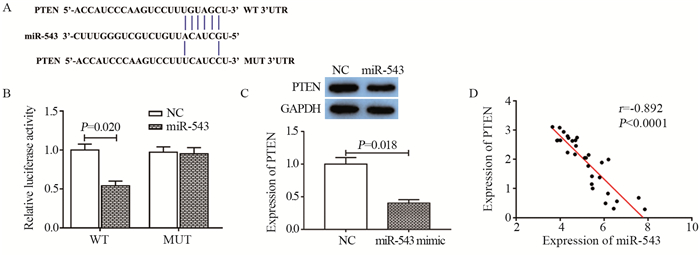
进一步,通过生物信息学数据库TargetScan对miR-543的靶基因进行预测,发现PTEN是miR-543的候选靶基因,其预测结合序列见图 4A。随后,双荧光素酶报告基因验证实验发现,相比于野生型PTEN序列质粒,过表达miR-543和野生型PTEN质粒可显著下调荧光素酶活性(t=4.789, P=0.020),转染miR-543 mimic和突变型PTEN序列质粒与单独转染突变型PTEN序列质粒相比,荧光素酶活性无明显变化(t=0.188, P=0.434),见图 4B。同时,Western blot检测结果显示,相比于对照组,miR-543过表达后可显著下调PTEN蛋白的表达水平(t=5.145, P=0.018,图 4C)。此外,通过对30例胰腺癌组织样本检测发现,miR-543与PTEN的表达呈负相关(r=-0.892, P < 0.0001,图 4D)。由此可知,PTEN是miR-543的靶基因,并且miR-543可负调控PTEN的表达。
|
| 图 4 miR-543对PTEN表达的调控作用 Figure 4 Effect of miR-543 on PTEN expression |
为了进一步明确MEG3通过靶向下调miR-543并上调PTEN对PANC-1细胞增殖、侵袭和EMT的作用机制,本实验进一步观察了细胞的生物学行为。qRT-PCR检测结果证实,相比于对照组,敲降miR-543可显著下调PANC-1细胞中miR-543的表达水平(t=9.009, P=0.006),而同时敲降MEG3或PTEN对miR-543的表达与对照组差异没有统计学意义(t1=1.546, P=0.131, t2=1.359, P=0.154),见图 5A。Western blot检测结果表明,相比于对照组,敲降miR-543显著上调了PTEN的表达水平(t=11.559, P=0.004),而同时敲降MEG3或PTEN对PTEN的表达与对照组差异无统计学意义(t1=0.198, P=0.431; t2=0.918, P=0.228),见图 5B。随后,CCK-8和Transwell检测结果共同证实,敲降miR-543后显著抑制了PANC-1细胞增殖及侵袭能力(t1=3.751, P=0.010; t2=8.150, P=0.007),而同时敲降MEG3或PTEN则减弱了敲降miR-543的抑制作用(t1=11.715, P=0.004, t2=7.695, P=0.008, F=59.744, P=0.004),见图 5C~E。此外,Western blot检测发现,敲降miR-543可显著下调PANC-1细胞中N-cadherin和Vimentin的表达(t1=7.809, P=0.008; t2=10.780, P=0.004),而E-cadherin的表达则明显上调(t=8.480, P=0.007),同时敲降MEG3或PTEN则恢复了敲降miR-543对EMT相关蛋白的调控作用(F1=52.574, P=0.005; F2=39.994, P=0.007; F3=63.480, P=0.004),见图 5F。结果表明,MEG3通过靶向下调miR-543对PTEN的抑制作用,进而抑制PANC-1细胞增殖、侵袭和上皮间质转化。
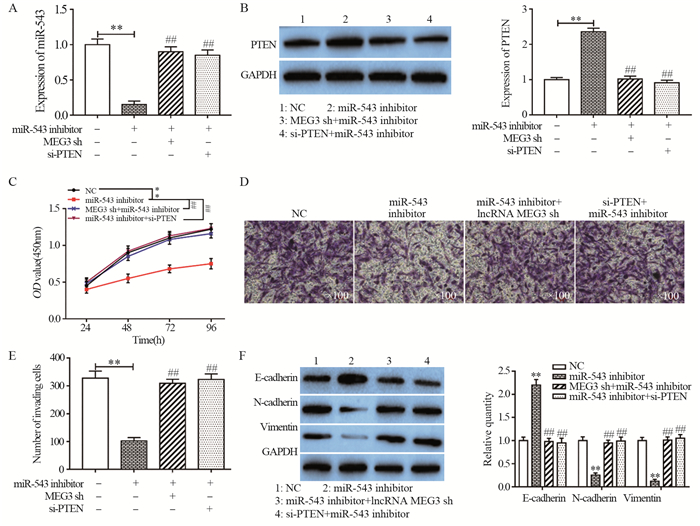
|
| **: P < 0.01, compared with NC group; ##: P < 0.01, compared with miR-543 inhibitor group 图 5 MEG3通过miR-543/PTEN分子轴对PANC-1细胞生物学行为的影响 Figure 5 MEG3 modulated biological behavior of pancreatic cancer cells via regulating miR-543/PTEN axis |
多项研究显示,非编码RNA在胰腺癌的发生发展中扮演着重要角色,其中lncRNAs生物学功能包括参与表观遗传调控、转录调控、转录后调控、miRNA调控、细胞分化及发育,其重要机制之一是通过对其周围的相关编码基因的选择性表达发挥调控作用[9]。例如,MEG3在胰腺癌细胞中低表达,过表达MEG3可抑制胰腺癌增殖和转移[10]。同时,过表达MEG3也被报道可显著抑制胆囊癌[11]、宫颈癌[12]和骨肉瘤[13]细胞增殖和侵袭。同时,本研究也发现,MEG3在胰腺癌组织和细胞系中低表达,过表达MEG3可明显抑制胰腺癌PANC-1细胞增殖、侵袭和EMT,再次证实MEG3对胰腺癌进程具有重要的调控作用。
为了进一步探索MEG3参与调控胰腺癌发生发展的作用机制而进行的功能研究表明,MEG3作为多种恶性肿瘤的抑癌基因,通过靶向结合下游miRNA并介导mRNA参与多种肿瘤细胞增殖、侵袭和迁移。例如,Li等研究发现,MEG3通过靶向下调miR-184抑制慢性髓系白血病细胞增殖和转移[14]。Long等证实,MEG3靶向下调miR-499-5p对CYLD表达起抑制作用,进而抑制黑色素瘤细胞增殖和侵袭[15]。Zhang等研究发现,MEG3通过调控miR-421/E-cadherin抑制乳腺癌细胞上皮间质转化过程[16]。Zheng等研究显示,MEG3通过阻断PKM2/β-catenin及上调PTEN的表达,进而抑制肝癌发展进程[17]。本研究通过StarBase数据库分析可能与MEG3结合的miRNA,发现miR-543是MEG3潜在的靶向作用基因。同时,大量研究证实,miR-543作为促癌基因,并在许多恶性肿瘤中高表达,比如前列腺癌[18]、非小细胞肺癌[19]、肾细胞癌[20]以及胃癌[21]。Yang等研究证实,过表达miR-543可显著促进肾透明细胞癌细胞增殖和侵袭[22]。本研究发现,miR-543在胰腺癌组织中的表达明显上调,且与胰腺癌细胞增殖、侵袭和EMT相关。同时,Zhao等研究证实,miR-543通过靶向下调PLA2G4A促进食管癌细胞增殖、侵袭和上皮间质转化[23]。进一步,生物信息学数据库TargetScan预测miR-543的靶基因可能为PTEN。大量研究证实,PTEN作为抑癌基因,其对癌细胞增殖、迁移、侵袭和凋亡具有重要调控作用,且其在肿瘤中低表达[24]。Sun等研究发现,miR-543通过靶向下调PTEN促进结直肠癌细胞增殖、侵袭和迁移[25]。本研究证实,沉默miR-543是通过上调PTEN的表达来抑制PANC-1细胞增殖、侵袭和EMT。
综上所述,本研究通过细胞实验和临床样本充分证实,过表达MEG3通过下调miR-543对PTEN的抑制作用,进而抑制胰腺癌细胞增殖、侵袭和EMT,从而下调胰腺癌发生发展进程。
作者贡献
成鉴晓: 实验设计、实施及文章执笔
江月萍: 实验设计及文章审校
任琳琳、巩芮宁: 实验实施
章程: 实验评估
| [1] | Huang X, Zhi X, Gao Y, et al. LncRNAs in pancreatic cancer[J]. Oncotarget, 2016, 7(35): 57379–57390. |
| [2] | Han T, Hu H, Zhuo M, et al. Long non-coding RNA: an emerging paradigm of pancreatic cancer[J]. Curr Mol Med, 2016, 16(8): 702–709. DOI:10.2174/1566524016666160927095812 |
| [3] | Ma L, Wang F, Du C, et al. Long non-coding RNA MEG3 functions as a tumour suppressor and has prognostic predictive value in human pancreatic cancer[J]. Oncol Rep, 2018, 39(3): 1132–1140. |
| [4] | Zhang J, Yao T, Wang Y, et al. Long noncoding RNA MEG3 is downregulated in cervical cancer and affects cell proliferation and apoptosis by regulating miR-21[J]. Cancer Biol Ther, 2016, 17(1): 104–113. DOI:10.1080/15384047.2015.1108496 |
| [5] | Yin DD, Liu ZJ, Zhang E, et al. Decreased expression of long noncoding RNA MEG3 affects cell proliferation and predicts a poor prognosis in patients with colorectal cancer[J]. Tumour Biol, 2015, 36(6): 4851–4859. DOI:10.1007/s13277-015-3139-2 |
| [6] | Li Z, Yang L, Liu X, et al. Long noncoding RNA MEG3 inhibits proliferation of chronic myeloid leukemia cells by sponging microRNA21[J]. Biomed Pharmacother, 2018, 104: 181–192. DOI:10.1016/j.biopha.2018.05.047 |
| [7] | Gu L, Zhang J, Shi M, et al. lncRNA MEG3 had anti-cancer effects to suppress pancreatic cancer activity[J]. Biomed Pharmacother, 2017, 89: 1269–1276. DOI:10.1016/j.biopha.2017.02.041 |
| [8] | Klinge CM. Non-coding RNAs: long non-coding RNAs and microRNAs in endocrine-related cancers[J]. Endocr Relat Cancer, 2018, 25(4): R259–R282. DOI:10.1530/ERC-17-0548 |
| [9] | Zhang A, Xu M, Mo YY. Role of the lncRNA-p53 regulatory network in cancer[J]. J Mol Cell Biol, 2014, 6(3): 181–191. DOI:10.1093/jmcb/mju013 |
| [10] | Zhang YY, Feng HM. MEG3 suppresses human pancreatic neuroendocrine tumor cells growth and metastasis by down-regulation of miR-183[J]. Cell Physiol Biochem, 2017, 44(1): 345–356. DOI:10.1159/000484906 |
| [11] | Jin L, Cai Q, Wang S, et al. Long noncoding RNA MEG3 regulates LATS2 by promoting the ubiquitination of EZH2 and inhibits proliferation and invasion in gallbladder cancer[J]. Cell Death Dis, 2018, 9(10): 1017. DOI:10.1038/s41419-018-1064-1 |
| [12] | Zhang J, Lin Z, Gao Y, et al. Downregulation of long noncoding RNA MEG3 is associated with poor prognosis and promoter hypermethylation in cervical cancer[J]. J Exp Clin Cancer Res, 2017, 36(1): 5. DOI:10.1186/s13046-016-0472-2 |
| [13] | Sun L, Yang C, Xu J, et al. Long noncoding RNA EWSAT1 promotes osteosarcoma cell growth and metastasis through suppression of MEG3 expression[J]. DNA Cell Biol, 2016, 35(12): 812–818. DOI:10.1089/dna.2016.3467 |
| [14] | Li J, Zi Y, Wang W, et al. Long noncoding RNA MEG3 inhibits cell proliferation and metastasis in chronic myeloid leukemia via targeting miR-184[J]. Oncol Res, 2018, 26(2): 297–305. DOI:10.3727/096504017X14980882803151 |
| [15] | Long J, Pi X. lncRNA-MEG3 suppresses the proliferation and invasion of melanoma by regulating CYLD expression mediated by sponging miR-499-5p[J]. Biomed Res Int, 2018, 2018: 2086564. |
| [16] | Zhang W, Shi S, Jiang J, et al. LncRNA MEG3 inhibits cell epithelial-mesenchymal transition by sponging miR-421 targeting E-cadherin in breast cancer[J]. Biomed Pharmacother, 2017, 91: 312–319. DOI:10.1016/j.biopha.2017.04.085 |
| [17] | Zheng Q, Lin Z, Xu J, et al. Long noncoding RNA MEG3 suppresses liver cancer cells growth through inhibiting beta-catenin by activating PKM2 and inactivating PTEN[J]. Cell Death Dis, 2018, 9(3): 253. DOI:10.1038/s41419-018-0305-7 |
| [18] | Du Y, Liu XH, Zhu HC, et al. MiR-543 promotes proliferation and epithelial-mesenchymal transition in prostate cancer via targeting RKIP[J]. Cell Physiol Biochem, 2017, 41(3): 1135–1146. DOI:10.1159/000464120 |
| [19] | Bi M, Chen W, Yu H, et al. miR-543 is up-regulated in gefitinib-resistant non-small cell lung cancer and promotes cell proliferation and invasion via phosphatase and tensin homolog[J]. Biochem Biophys Res Commun, 2016, 480(3): 369–374. DOI:10.1016/j.bbrc.2016.10.055 |
| [20] | Chen ZY, Du Y, Wang L, et al. MiR-543 promotes cell proliferation and metastasis of renal cell carcinoma by targeting Dickkopf 1 through the Wnt/beta-catenin signaling pathway[J]. J Cancer, 2018, 9(20): 3660–3668. DOI:10.7150/jca.27124 |
| [21] | Xu J, Wang F, Wang X, et al. miRNA-543 promotes cell migration and invasion by targeting SPOP in gastric cancer[J]. Onco Targets Ther, 2018, 11: 5075–5082. DOI:10.2147/OTT.S161316 |
| [22] | Yang F, Ma J, Tang Q, et al. MicroRNA-543 promotes the proliferation and invasion of clear cell renal cell carcinoma cells by targeting Kruppel-like factor 6[J]. Biomed Pharmacother, 2018, 97: 616–623. DOI:10.1016/j.biopha.2017.10.136 |
| [23] | Zhao H, Diao C, Wang X, et al. MiR-543 promotes migration, invasion and epithelial-mesenchymal transition of esophageal cancer cells by targeting phospholipase A2 group IVA[J]. Cell Physiol Biochem, 2018, 48(4): 1595–1604. DOI:10.1159/000492281 |
| [24] | Li A, Qiu M, Zhou H, et al. PTEN, insulin resistance and cancer[J]. Curr Pharm Des, 2017, 23(25): 3667–3676. |
| [25] | Sun J, Zhou J, Dong M, et al. Dysregulation of microRNA-543 expression in colorectal cancer promotes tumor migration and invasion[J]. Mol Carcinog, 2017, 56(1): 250–257. DOI:10.1002/mc.22489 |
 2019, Vol. 46
2019, Vol. 46


