文章信息
- 食管癌放射抵抗相关基因的筛选
- Microarray Analysis of Radioresistance-associated Genes in Esophageal Squamous Cell Carcinoma
- 肿瘤防治研究, 2019, 46(3): 227-232
- Cancer Research on Prevention and Treatment, 2019, 46(3): 227-232
- http://www.zlfzyj.com/CN/10.3971/j.issn.1000-8578.2019.18.1007
- 收稿日期: 2018-07-23
- 修回日期: 2018-11-23
中国北部处于被称为“食管癌地带”的食管癌高发区[1]。尤其晋冀豫交界区,比世界低发区的发病率高出近60倍,食管癌仍居该地区恶性肿瘤发病的首位[2]。放射治疗是食管癌非手术治疗的主要手段[3]。晚期不能手术的食管癌常规单纯放疗的5年生存率为10%左右。近年来虽然放疗手段和放疗技术取得巨大进步,但食管癌放疗的总体生存率仍然没有显著提高[4]。临床中经常见到临床分期相近的食管癌患者放疗后疗效存在明显差异,这可能由于其基因突变谱的差异,使得相同病理类型的肿瘤具有不同的临床表型,同时对放疗的疗效存在显著差异[5]。研究显示放射抵抗是肿瘤局部复发、放疗失败的重要原因,各种信号通路在食管癌放射抵抗形成中起到重要作用[6]。如果能在治疗前发现放射抵抗的基因类型,从而预测出放疗效果不佳的患者,逆转放射抵抗,将能进一步提高生存率。基因芯片技术可以一次性对样品大量基因序列进行检测和分析,本研究即应用基因芯片进行全基因组CNV分析,筛查食管癌放射抵抗相关基因,为选择合理的治疗方案提供有力的依据。
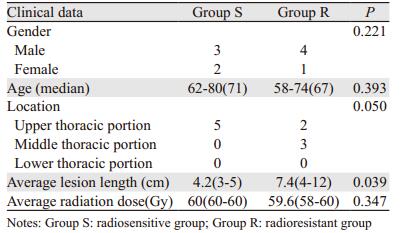
1 资料与方法 1.1 临床资料收集2013年12月—2016年8月在安阳市肿瘤医院行放射治疗的食管癌患者66例,经内镜活检证实,病理类型均为鳞状细胞癌,临床资料完整,男女不限,年龄不限,放射治疗均采取三维适形调强(IMRT)放疗技术。根据临床疗效分为两组,放射敏感组:放射治疗后达到临床完全缓解并且持续一年以上(以下称S组); 放射抗拒组:放射治疗未控,指放疗无效或放疗后半年内放射野内局部病变进展(以下称R组)。每组各33例。
1.2 实验方法 1.2.1 基因芯片CNV检测利用Affymetrix公司的OncoScan Array全基因组芯片检测全基因组SNP及拷贝数变异(copy number variation, CNV)。以提取自福尔马林固定石蜡包埋(formalin-fixed and paraffin-embedded, FFPE)样品的基因组80 ng DNA为起始,采用分子倒置探针(molecular inversion probe, MIP)技术。比较基因组杂交(comparative genomic hybridization, CGH)实验检测荧光标记。主要包括如下步骤:(1)从FFPE组织切片中提取基因组DNA、纯化和电泳质控,用Qubit方法定量,琼脂糖凝胶电泳检测其完整度。平均绝对百分偏差(MAPD)大于0.3的样品认为不合格; (2)FFPE样品的基因组80 ng DNA为起始,待检测DNA采用分子倒置探针技术在液相中进行杂交、连接、第一次酶切、扩增和第二次酶切反应。分子倒置探针由7部分序列组成:2个内切酶识别位点,可用限制性内切酶切割,2段与目的基因互补的序列、2段通用引物序列以及1段特异性标签序列; (3)酶切后DNA进行生物素标记; (4)标记DNA的特异性标签序列与芯片杂交; (5)芯片杂交、清洗、染色,染色完成后,将芯片放到扫描仪(GeneChip® Scanner 3000)扫描。
1.2.2 数据提取和分析当完成杂交和荧光标记后,芯片的荧光扫描图像被保存成DAT文件,AGCC软件(Affymetrix® GeneChip®Command Console® Software)将DAT文件从图像信号转化为数字信号即CEL文件。CEL文件记录探针水平的荧光信号强度值信息。OncoScan Console软件将CEL文件的AT/GC两通道合并,转换成OSCHP文件,进行数据提取。基于OSCHP文件,使用Nexus Express for OncoScan 3软件进行CNV和体细胞突变分析,采用TuScan算法。将获得的生物学信息与参考基因组进行覆盖度的统计分析。检测得到的变异结果与UCSC数据库、DBSNP数据库、外显子组测序计划数据库进行比对、分析。挑选出两组的基因拷贝数差异及代表性基因。
1.2.3 验证分析挑选出CNV差异显著的片段,应用Thermo Fisher 7500 qPCR仪对筛选出的CNV片段内的代表基因进行实时荧光定量PCR进行验证。进行DNA抽提、扩增,扩增阶段进行40个循环,采取相对定量的2-ΔΔCt方法进行基因表达分析。进一步筛选出可能的目标基因。
1.3 统计学方法采用SPSS16.0软件进行统计分析。计数资料的比较采用χ2检验,两独立样本资料的比较采用Mann-Whitney U非参数检验。计量资料用(x±s)表示,组间均数比较使用t检验。检验水准α=0.05。
2 结果 2.1 样本质量评价利用Affymetrix平台提取FFPE样品的基因组DNA,然后用Qubit方法定量,琼脂糖凝胶电泳检测其完整度。先进行20例DNA测序样品电泳,13份质检合格,为两组配比平衡,每组病例各挑选5例样本,共10例进行下一步基因芯片分析。纳入检测的10例样本的临床特征,见表 1。食管癌的病变部位分段方法结合食管钡餐、胃镜和CT检查,分段标准按UICC 1997公布的分段标准[7]。
|
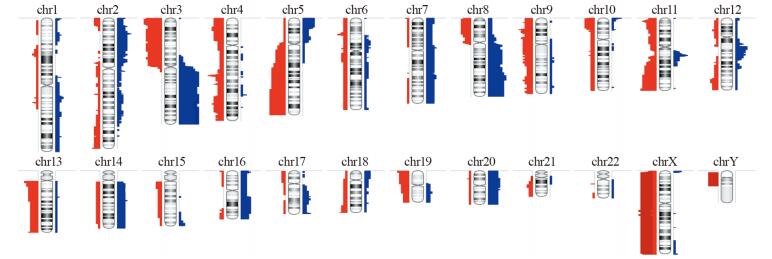
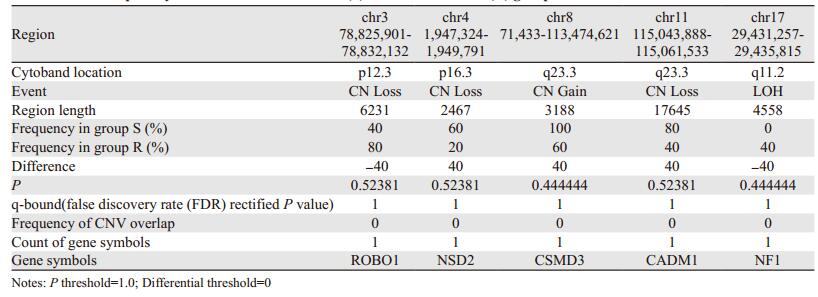
对基因芯片检测的全基因组拷贝数变异(CNV)数据进行生物信息学分析,见图 1~2。考虑样品数目较少,两组CNV差异比较的P值参考意义不大,我们挑选出两组间CNV发生频率之差≥40%,并且与已知CNV没有重叠的CNV片段做为本研究初步结果。共发现5个有意义CNV片段,分别位于3、4、8、11、17号染色体,其中拷贝数缺失的(CN Loss)3个,增加的(CN Gain)1个,杂合性缺失的(LOH)1个。3p12.3拷贝数缺失与17q11.2 LOH在放射抵抗组(R组)的发生频率高于放射敏感组(S组),4p16.3、11q23.3拷贝数缺失和8q23.3拷贝数增加在S组的发生频率高于R组,见表 2。

|
| The upper part was CNV frequency in all samples(blue: gain; red: loss). The middle part was annotation about gene, exon, CNV and miRNA in this section. The lower part was CNV in all samples 图 1 10例食管癌样本中的拷贝数变异(CNV)图示 Figure 1 Compact copy number variance(CNV) in 10 cases of ESCC samples |
|
| Blue: gain; Red: loss 图 2 10例食管癌样本中拷贝数变异(CNV)的染色体G带图 Figure 2 Ideogram of CNV in Chromosome G band in 10 cases of ESCC samples |
|
在5个CNV差异有意义的片段内,通过UCSC数据库分别挑选出5个可能的代表性基因,进行qPCR验证实验。5个代表性基因分别为ROBO1、NSD2、CSMD3、CADM1、NF1。在S组和R组每组中各选取11例,共22例PPFE标本。选取白蛋白(ALB)为内参基因,内参基因引物序列为:F: ATGCTGCACAGAATCCTTGGT; R: TCATCGACTTCCAGAGCTGAAA。内参基因和待测基因扩增曲线平滑,溶解曲线单一,实验数据真实可信,可用于后期分析。扩增及溶解曲线见图 3~8。
|
| 图 3 内参基因扩增曲线及溶解曲线 Figure 3 Amplification and dissolution curves of reference genes |

|
| 图 4 ROBO1基因扩增曲线及溶解曲线 Figure 4 Amplification and dissolution curves of ROBO1 gene |

|
| 图 5 NSD2基因扩增曲线及溶解曲线 Figure 5 Amplification and dissolution curves of NSD2 gene |
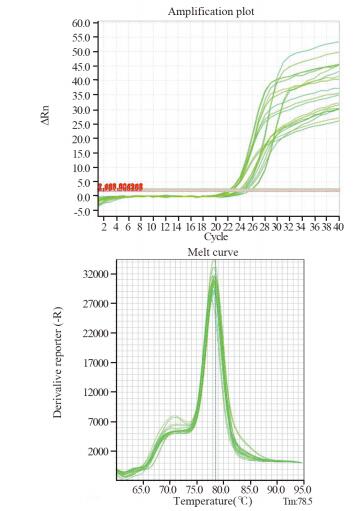
|
| 图 6 CSMD3基因扩增曲线及溶解曲线 Figure 6 Amplification and dissolution curves of CSMD3 gene |
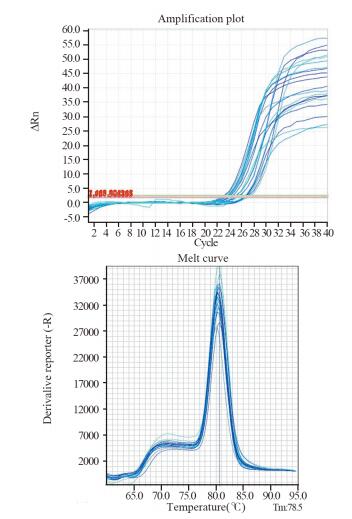
|
| 图 7 CADM1基因扩增曲线及溶解曲线 Figure 7 Amplification and dissolution curves of CADM1 gene |
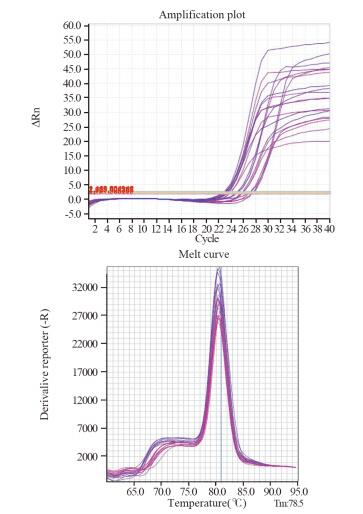
|
| 图 8 NF1基因扩增曲线及溶解曲线 Figure 8 Amplification and dissolution curves of NF1 gen |
采用2-ΔΔCt作为评价基因水平的指标,结果显示基因ROBO1、NF1在两组中的拷贝数无明显差别。CSMD3虽然在S组中的拷贝数高于R组,但两组表达均较低,没有临床意义。NSD2在R组中的拷贝数明显高于S组。CADM1在两组中的P值没有统计学意义,但是绝对值相差比较大,可能有一定意义,见表 3。
世界范围内,食管癌占癌症死亡的第六位[8]。总体来讲,食管癌预后远端好于近端。鳞癌比腺癌生存期要差[9]。我国食管癌人群中90%以上为食管鳞癌[10]。放射治疗为非手术治疗食管癌患者的根治性治疗手段,但是与手术治疗不同,颈段、胸上段食管癌患者放疗后生存优于胸中、下段食管癌患者[11]。
肿瘤放疗敏感度是一个复杂而极具个体化的问题。目前尚未得到量化指标可准确判断食管癌患者对放疗或放化疗的敏感度。基因芯片具有高通量、高效率和高自动化、高敏感度等优点,成为分析基因表达模式的一种高效方法。OncoScanTM芯片采用分子倒置探针MIP技术,能够对FFPE样本进行检测,显著提高了分辨率。
本研究采用基因芯片初步探讨了与食管癌放疗敏感度相关的分子事件,筛选出可能对食管癌放射敏感度预测价值最大的5个基因拷贝数变异(CNV)区域,并针对这5个区域内的代表性基因进行qPCR验证。发现4p16.39(代表性基因NSD2)和11q23.3(代表性基因CADM1)的拷贝数缺失(Loss)发生率在放射抵抗组(R组)中明显高于放射敏感组(S组)。我们认为NSD2和CADM1可能是与食管癌放射抵抗的相关位点,其机制有待更多的研究进一步验证。
NSD2位于染色体4p16.3上,主要功能是参与组蛋白的甲基化修饰。沉默NSD2会导致细胞对DNA损伤剂如羟基脲,喜树碱和丝裂霉素C以及电离辐射的敏感度增加。NSD2调节DNA修复基因的表达[12-14]。本研究发现在放疗抗拒组中有高水平的NSD2表达,与文献报道的一致。进一步研究NSD2甲基转移酶类的药物开发,将有可能逆转肿瘤的放射抵抗。
CADM1称细胞黏附分子1(cell adhesion molecule 1, CADM1),研究报道的免疫组织化学的检测结果显示,与ESCC组织相比,CADM1 mRNA表达在正常组织中高度上调,表明CADM1表达的丧失影响ESCC的发病机制,侵袭和转移以及预后[15-16]。其详细机制需进一步研究。这些研究结果提示食管癌对放射线产生放射抵抗是复杂的多基因共同作用的结果,以上2个基因涉及DNA损伤修复和细胞黏附等与治疗敏感度相关的重要分子生物学通路。只有通过对基因表观遗传的全面分析才有可能全面解释食管癌复杂的放射抵抗机制,为提高食管癌放射治疗的敏感度提供新的靶点及策略。
本研究中样本例数相对较少,统计学上筛选两组差异基因存在假阳性率大的缺点。同时本研究两组的临床特征并不完全相同,是因为本研究是根据临床无进展生存期的长短选择的病例,临床分期和病变分段没有严格限制,也可能会造成结果的偏倚。下一步一方面需要扩大样本量进行分层深入研究,另一方面需要进一步进行免疫组织化学验证蛋白层面的表达。
作者贡献
朱青山:立题、主持研究、论文撰写
李军扩:病理资料采集
刘维鹏:病例采集、参考文献搜索
马庭炜:患者资料统计
代宁涛:协助病理资料采集、联系相关公司进行基因检测
冯连杰:课题设计
魏涛:数据分析
周福有:立项及课题指导
| [1] | Torre LA, Siegel RL, Ward EM, et al. Global Cancer Incidence and Mortality Rates and Trends--An Update[J]. Cancer Epidemiol Biomarkers Prev, 2016, 25(1): 16–27. |
| [2] | 宋国慧, 李东方, 孟凡书, 等. 磁县2003-2012年上消化道癌发病率分析[J]. 肿瘤防治研究, 2016, 43(10): 887–93. [ Song GH, Li DF, Meng FS, et al. Upper Gastrointestinal Carcinomas Incidence from 2003 to 2012 in Cixian, Hebei Province[J]. Zhong Liu Fang Zhi Yan Jiu, 2016, 43(10): 887–93. DOI:10.3971/j.issn.1000-8578.2016.10.012 ] |
| [3] | Nomura M, Oze I, Kodaira T, et al. Comparison between surgery and definitive chemoradiotherapy for patients with resectable esophageal squamous cell carcinoma: a propensity score analysis[J]. Int J Clin Oncol, 2016, 21(5): 890–8. DOI:10.1007/s10147-016-0963-3 |
| [4] | 孔雁, 高红梅. 食管癌放射治疗10年生存分析及不同治疗方式的疗效比较[J]. 肿瘤防治研究, 2015, 42(1): 56–61. [ Kong Y, Gao HM. Long Outcome of Radiotherapy for Esophageal Carcinoma and Efficacy Comparison of Different Treatment Methods[J]. Zhong Liu Fang Zhi Yan Jiu, 2015, 42(1): 56–61. DOI:10.3971/j.issn.1000-8578.2015.01.014 ] |
| [5] | 王立东, 刘敏, 户彦龙, 等. 食管癌超长期和短期生存患者临床病理变化对比分析[J]. 肿瘤防治研究, 2014, 41(3): 193–8. [ Wang LD, Liu M, Hu YL, et al. Comparative Analysis of Clinicopathological Changes in Esophageal Cancer Patients with Ultra-long and Short-term Survival[J]. Zhong Liu Fang Zhi Yan Jiu, 2014, 41(3): 193–8. DOI:10.3971/j.issn.1000-8578.2014.03.001 ] |
| [6] | 张红, 吴清明. 食管癌放射抗拒的细胞信号通路研究进展[J]. 肿瘤防治研究, 2014, 41(4): 425–7. [ Zhang H, Wu QM. Research Progress of Cell Signaling Pathways of Esophageal Cancer Radioresistance[J]. Zhong Liu Fang Zhi Yan Jiu, 2014, 41(4): 425–7. DOI:10.3971/j.issn.1000-8578.2014.04.032 ] |
| [7] | 周志国, 甄婵军, 梁军利, 等. 食管癌AJCC/UICC第6、7版TNM分期分段方法在放疗患者中的比较[J]. 肿瘤防治研究, 2015, 42(8): 772–6. [ Zhou ZG, Zhen CJ, Liang JL, et al. Comparison of 6th and 7th Editions of AJCC/UICC TNM Staging for Esophageal Cancer Patients Received Radiotherapy[J]. Zhong Liu Fang Zhi Yan Jiu, 2015, 42(8): 772–6. DOI:10.3971/j.issn.1000-8578.2015.08.005 ] |
| [8] | Torre LA, Bray F, Siegel RL, et al. Global cancer statistics, 2012[J]. CA Cancer J Clin, 2015, 65(2): 87–108. DOI:10.3322/caac.21262 |
| [9] | Rice TW, Gress DM, Patil DT, et al. Cancer of the esophagus and esophagogastric junction-Major changes in the American Joint Committee on Cancer eighth edition cancer staging manual[J]. CA Cancer J Clin, 2017, 67(4): 304–17. DOI:10.3322/caac.v67.4 |
| [10] | Chen W, Zheng R, Baade PD, et al. Cancer statistics in China, 2015[J]. CA Cancer J Clin, 2016, 66(2): 115–32. DOI:10.3322/caac.21338 |
| [11] | 朱青山, 申静, 王能超, 等. 非手术治疗食管癌的临床分期研究[J]. 肿瘤防治研究, 2013, 40(2): 168–71. [ Zhu QS, Shen J, Wang NC, et al. Study of clinical staging of esophageal cancer treated by non-surgical methods[J]. Zhong Liu Fang Zhi Yan Jiu, 2013, 40(2): 168–71. DOI:10.3971/j.issn.1000-8578.2013.02.011 ] |
| [12] | Bennett RL, Swaroop A, Troche C, et al. The Role of Nuclear Receptor-Binding SET Domain Family Histone LysineMethyltransferases in Cancer[J]. Cold Spring Harb Perspect Med, 2017, 7(6): pii: a026708. DOI:10.1101/cshperspect.a026708 |
| [13] | Morishita M, Mevius D, di Luccio E. In vitro histone lysine methylation by NSD1, NSD2/MMSET/WHSC1 and NSD3/WHSC1L[J]. BMC Struct Biol, 2014, 14: 25. |
| [14] | 雷宏维, 陶凯雄. NSD2与肿瘤关系的研究进展[J]. 世界华人消化杂志, 2017, 25(36): 3218–23. [ Lei HW, Tao KX. Relationship between NSD2 and tumors[J]. Shijie Hua Ren Xiao Hua Za Zhi, 2017, 25(36): 3218–23. ] |
| [15] | Qian JB, Liu HB, Zhu Y, et al. CADM1 mRNA expression and clinicopathological significance in esophageal squamous cell carcinoma tissue[J]. Genet Mol Res, 2017, 16(2). |
| [16] | Zeng D, Wu X, Zheng J, et al. Loss of CADM1/TSLC1 Expression Is Associated with Poor Clinical Outcome in Patients with Esophageal Squamous Cell Carcinoma[J]. Gastroenterol Res Pract, 2016, 2016: 6947623. |
 2019, Vol. 46
2019, Vol. 46



