文章信息
- 环状RNA ciRS-7在三阴性乳腺癌中的表达及其对细胞侵袭和迁移的影响
- Expression of Circular RNA ciRS-7 and Its Effect on Invasion and Migration of Triple-negative Breast Cancer Cells
- 肿瘤防治研究, 2019, 46(3): 233-238
- Cancer Research on Prevention and Treatment, 2019, 46(3): 233-238
- http://www.zlfzyj.com/CN/10.3971/j.issn.1000-8578.2019.18.0855
- 收稿日期: 2018-06-25
- 修回日期: 2018-11-28
2. 050011 石家庄,河北医科大学第四医院外三科;
3. 050011 石家庄,河北医科大学第四医院动物中心
2. The Third Department of Surgery, The Fourth Hospital of Hebei Medical University, Shijiazhuang 050011, China;
3. Animal Center, The Fourth Hospital of Hebei Medical University, Shijiazhuang 050011, China
乳腺癌是全球范围内女性发病率最高的一种恶性肿瘤,在女性癌症相关死因中居第二位[1]。中国乳腺癌的发病率占世界总发病率的12.2%,死亡率高达9.6%,严重危害我国女性的身体健康和生活质量[2]。三阴性乳腺癌(triple negative breast cancer, TNBC)是指雌激素受体(ER)、孕激素受体(PR)和人类表皮生长因子受体2(HER2)均呈阴性表达的乳腺癌,恶性程度高,容易发生转移[3-4]。虽然目前对乳腺癌的诊断及治疗已取得一定成效,但三阴性乳腺癌的患者在1~3年内极易复发,并且大多于治疗后的5年内死亡[5]。针对以上问题,快速、准确地找到TNBC更有效的生物靶标成为关键所在。
环状RNA(circRNA)是生物体内广泛存在的一类具有调节功能的非编码RNA[6]。它们一般头尾连接形成环状结构,因为不含5' 末端帽和3' 末端poly(A)尾而表达稳定[7-8]。第一个环状RNA ciRS-7(CDR1as)由Hansen等首次发现,其可作为miR-7海绵在大脑中稳定表达并发挥重要的基因调控功能[9]。最近有研究发现,ciRS-7敲除后的体内动物模型出现了miR-7和miR-671表达失调,最终影响哺乳动物的大脑发育功能[10]。ciRS-7主要通过miRNA海绵的方式调节miR-7的表达,进而影响肿瘤的发生发展过程[9]。越来越多的研究发现环状RNA可调节人类多种肿瘤的进展。三阴性乳腺癌作为一类高度恶性的肿瘤,其潜在的分子机制仍有待阐明,而且环状RNA对其发病的调控作用尚未见报道。
本研究旨在探讨环状RNA ciRS-7在三阴性乳腺癌(TNBC)中的表达及其对细胞侵袭和迁移的影响,为寻找TNBC新型肿瘤生物标志物和治疗靶标奠定基础。
1 资料与方法 1.1 临床资料选取2016年9月—2017年10月在河北医科大学第四医院接受手术治疗的乳腺癌患者132例,其中Luminal型89例、TNBC25例和HER2过表达型18例。所有患者均未接受术前化疗和放疗。所有肿瘤组织均由经验丰富的病理学专家证实。所有患者均签署了知情同意书,研究方案征得医院伦理委员会批准。
1.2 细胞株及主要试剂人乳腺癌细胞株MDA-MB-231、MDA-MB-453、BT-549和MCF-7购自美国ATCC细胞库。RPMI 1640培养基、胰蛋白酶购自美国Gibco公司,胎牛血清购自上海依科赛生物制品有限公司。TRIzol购自美国Invitrogen公司,Platinum SYBR Super Mix试剂、Fu GENE HD转染试剂及反转录试剂盒均购自美国Promega公司,PCR引物购自英潍捷基贸易有限公司。Transwell小室(孔径3.0 μm)购自美国Corning公司,Matrigel基质胶购自美国BD公司。ciRS-7 siRNA购自广州锐博生物科技公司。
1.3 qRT-PCR法检测ciRS-7在乳腺癌组织和细胞中的表达乳腺癌组织和细胞用TRIzol裂解后提取总RNA,按照Promega反转录试剂盒说明书制备cDNA,然后进行实时荧光定量PCR扩增,以GAPDH为内参,环状RNA ciRS-7来源于CDR1as,于是本实验设计两种引物,一种为发散引物,可扩增出环状转录本ciRS-7,另一种为聚合引物,能扩增CDR1as的线性转录本。然后以任意两例TNBC组织和两种TNBC细胞系(MDA-MB-231和BT-549)的cDNA和基因组DNA(gDNA)为模板,进行普通PCR反应。目的基因的引物序列及反应条件见表 1。利用各实验组目的基因及内参基因的Ct值,按照公式ΔCt=Ct目的基因-Ct内参基因,以2-ΔΔCt法计算目的基因的相对表达量。
如1.3提取细胞总RNA后反转录为cDNA,PCR扩增,GAPDH为内参,目的基因的引物序列及反应条件,见表 1。扩增产物经2%琼脂糖凝胶电泳分离后进行核酸显色。
1.5 细胞转染人TNBC细胞株MDA-MB-231和BT-549用含10%胎牛血清、100 u/ml青霉素和100 μg/ml链霉素的RPMI 1640培养液培养,于37℃、5%CO2体积分数的培养箱中。按Fu GENE HD转染试剂说明书方法将ciRS-7 siRNA转染至MDA-MB-231和BT-549细胞中,并在两种细胞中分别转染control siRNA作为对照。
1.6 划痕实验检测敲低ciRS-7后的细胞迁移能力收集对数期的MDA-MB-231和BT-549细胞,调整细胞密度为5×105个每毫升,取2 ml接种于6孔板,并在6孔板背面划5条平行线做标记,24 h后用200 μl枪头在细胞中划2条垂直于背面平行线的直线,PBS冲洗2次,加入2 ml无胎牛血清的RPMI 1640培养液,于0、24 h在倒置显微镜下观察细胞向划痕中间迁移的距离并拍照,每组实验设3个平行孔。
1.7 Transwell小室检测敲低ciRS-7后的细胞迁移和侵袭能力取对数期生长的细胞悬液,加入Transwell小室的上室(2×105个/小室),小室上铺胶检测侵袭能力,不铺胶者检测迁移能力; 下室加入600 μl含10%胎牛血清的RPMI 1640培养液,常规培养48 h,用棉签擦净上室细胞,PBS清洗后,结晶紫染色,倒置相差显微镜下(×200)观察并拍照,随机选取5个视野,计数穿膜细胞数。
1.8 动物实验所有动物实验均经河北医科大学第四医院动物保护委员会批准。选用4周龄BALB/c裸鼠,随机分为2组(每组8只)。用si ciRS-7或对照siRNA转染的MDA-MB-231细胞(每只小鼠5×106个细胞)分别经尾静脉注射BALB/ c裸鼠中。然后将50 OD胆固醇修饰的siciRS-7或对照siRNA经尾静脉注射到BALB/c裸鼠中(每周2次)。2月后处死裸鼠,解剖肺组织。肉眼检查肺的外观,然后进行HE染色,并在显微镜下观察每个视野肺组织切片的转移克隆数。
上述实验均重复3次。
1.9 统计学方法本实验数据应用SPSS21.0软件进行统计分析,计量资料以均数±标准(x±s)表示,两组之间均数比较采用t检验,P < 0.05为差异有统计学意义。
2 结果 2.1 环状RNA ciRS-7在TNBC组织和细胞中稳定高表达结果显示,环状转录本ciRS-7只能用发散引物在cDNA中检测到,gDNA中未见扩增产物; 但是线性形式的CDR1as在cDNA和gDNA中均出现了扩增产物,见图 1A、B。这说明本研究使用的环状RNA ciRS-7引物的有效性,并进一步验证了其在TNBC中具有一定的表达丰度。qRT-PCR检测结果显示,TNBC样本中ciRS-7的相对表达量为(6.52±0.38),显著高于Luminal型(1.56±0.17)(P < 0.001)和HER2过表达型乳腺癌组织(2.27±0.66)(P < 0.001)以及癌旁正常组织(0.83±0.09)(P < 0.001),见图 1C。ciRS-7在TNBC细胞系MDA-MB-231和BT-549中高表达,但在Lunimal型MCF-7细胞和HER2过表达型MDA-MB-453细胞中低表达,见图 1D。qRT-PCR方法进一步证实ciRS-7对RNase R的抗性,MDA-MB-231和BT-549细胞经RNase R处理后CDR1线性基因表达明显下降,而环状形式的ciRS-7无明显变化,见图 1E。综上表明,环状RNA ciRS-7在TNBC组织和细胞中稳定高表达。
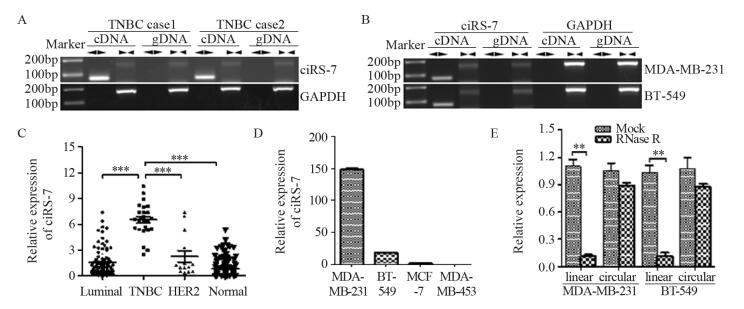
|
| A: the expression of ciRS-7 in two cases of TNBC tissues detected by RT-PCR; B: the expression of ciRS-7 in MDA-MB-231 and BT-549 cells detected by RT-PCR; C: the expression of ciRS-7 in 132 cases of breast cancer tissue and paired normal adjacent tissues detected by qRT-PCR; D: the expression of ciRS-7 in MDA-MB-231, BT-549, MCF-7 and MDA-MB-453 cells detected by qRT-PCR; E: the expression of ciRS-7 with RNase R treatment in MDA-MB-231 and BT-549 cells detected by qRT-PCR; **: P < 0.01, ***: P < 0.001 图 1 环状RNA ciRS-7在乳腺癌组织和细胞中的表达 Figure 1 Expression of circular RNA ciRS-7 in breast cancer tissues and cell lines |
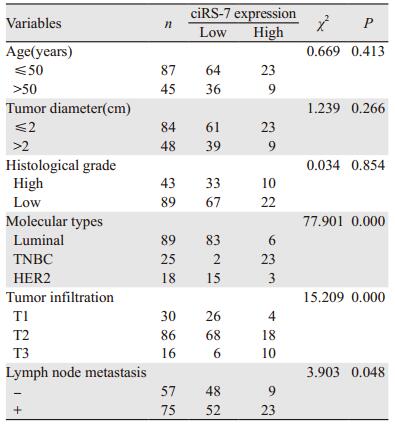
为方便分析数据,将所有乳腺癌组织分类为ciRS-7高表达组和低表达组,使用132例乳腺癌患者组织中ciRS-7的平均相对表达量(4.82)作为所有乳腺癌患者的临界阈值。132例乳腺癌患者中ciRS-7高表达组32例,低表达组100例; ciRS-7表达与患者年龄、肿瘤直径和病理分级无关(均P > 0.05),而与分子分型、肿瘤浸润和淋巴结转移明显相关(均P < 0.05),见表 2。由此推断ciRS-7可能与乳腺癌患者的病情进展密切相关,于是本实验重点研究高表达ciRS-7且恶性度高的TNBC型乳腺癌。
|
划痕实验结果显示siRNA介导的ciRS-7敲低组MDA-MB-231和BT-549细胞的迁移能力明显被抑制,见图 2A;转染ciRS-7 siRNA后两株细胞的ciRS-7表达明显降低(均P=0.000),见图 2B,证明本研究中针对环状RNA ciRS-7 siRNA干扰有效; 实验组和对照组两株细胞迁移面积变化百分率差异具有统计学意义,见图 2C(均P=0.000)。综上表明siRNA介导的ciRS-7敲低可能抑制TNBC细胞的迁移能力。
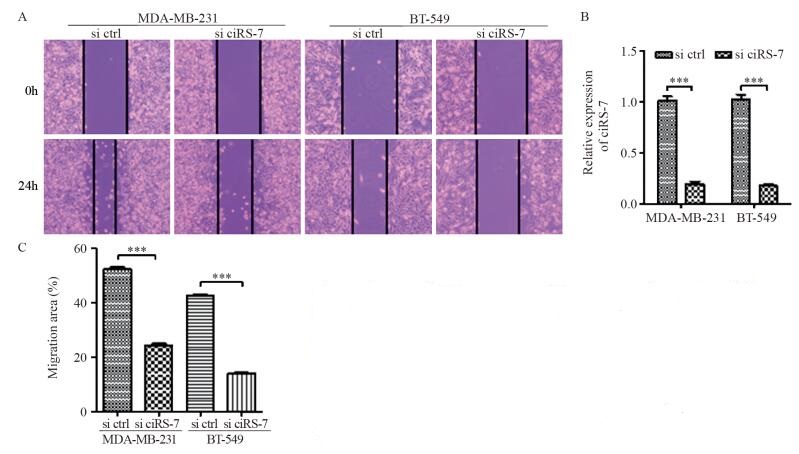
|
| A: the migration of MDA-MB-231 and BT-549 cells in each group detected by wound healing assay; B: the expression of ciRS-7 after silencing ciRS-7 in MDA-MB-231 and BT-549 cells detected by qRT-PCR; C: the migration area in each group of MDA-MB-231 and BT-549 cells; ***: P < 0.001, compared with si ctrl group 图 2 沉默ciRS-7对MDA-MB-231和BT-549细胞迁移的影响 Figure 2 Effect of silencing ciRS-7 on migration of MDA-MB-231 and BT-549 cells |
与si ctrl组相比,si ciRS-7处理组MDA-MB-231和BT-549细胞的穿膜数目明显减少,见图 3A。MDA-MB-231和BT-549中对照组和实验组的迁移和侵袭细胞数差异均有统计学意义(均P=0.000),见图 3B、C。
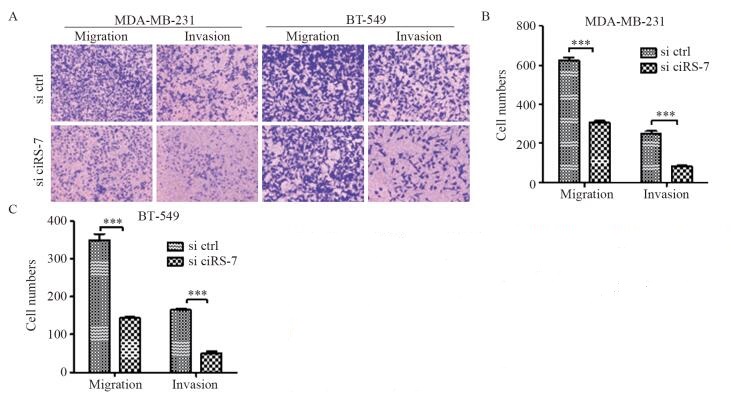
|
| A: the migration and invasion of MDA-MB-231 and BT-549 cells in each group detected by Transwell assay; B: the number of migrated and invaded MDA-MB-231 cells in each group; C: the number of migrated and invaded BT-549 cells in each group; ***: P < 0.001, compared with si ctrl group 图 3 沉默ciRS-7对MDA-MB-231和BT-549细胞迁移和侵袭的影响 Figure 3 Effect of silencing ciRS-7 on migration and invasion of MDA-MB-231 and BT-549 cells |
通过尾静脉注射建立MDA-MB-231细胞裸鼠肺转移模型。与si ctrl组相比,si ciRS-7组裸鼠的肺转移能力明显较弱,见图 4A。显微镜下观察si ciRS-7转染组的裸鼠形成的肺转移克隆数比si ctrl转染组的明显减少(P=0.000067),见图 4B。表明敲低ciRS-7可能抑制裸鼠体内TNBC的肺转移能力。
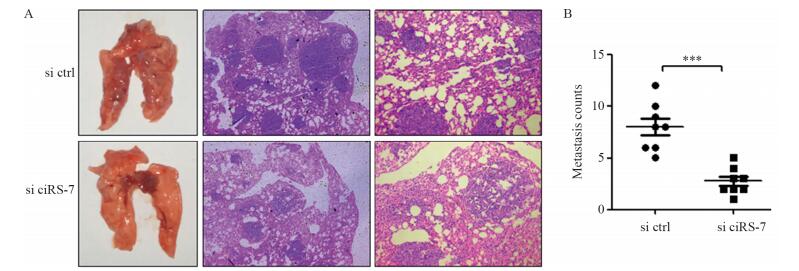
|
| A: the metastasis clones of lung tissue in nude mice; Left: the appearance of lungs by naked eyes; Middle: HE staining(40×); Right: HE staining(100×); B: the number of clones for lung metastasis in each group; ***: P < 0.001, compared with si ctrl group 图 4 沉默ciRS-7对裸鼠体内TNBC的肺转移的影响 Figure 4 Effect of silencing ciRS-7 on lung metastasis of TNBC in nude mice |
本课题组检测了132例乳腺癌患者病理组织标本及配对癌旁正常组织中ciRS-7的表达水平,分析ciRS-7与乳腺癌患者临床病理特征之间的关系。另外检测了乳腺癌细胞系中ciRS-7的表达水平,通过体外细胞功能实验检测ciRS-7对TNBC细胞迁移和侵袭能力的影响,体内动物实验探究敲低ciRS-7后TNBC发生肺转移的变化,为寻找TNBC的新型肿瘤生物标志物和治疗靶标奠定基础。
由于当时研究技术的局限性,环状RNA在发现之初被认为是错误剪接或内含子套索过程中形成的副产物[11-13]。近年来随着RNA测序(RNA-seq)技术的不断发展,大量环状RNA在小鼠、线虫及人类细胞中被检测到[14-15]。circRNA的功能包括与AGO蛋白结合调控mRNA的转录翻译过程[6],与RNA结合蛋白相互作用参与转录后调控[16]。还有一类位于细胞核内的环状RNA通过抑制内含子环化正向调控其宿主基因的表达[17]。但目前研究最为深入的还是其作为miRNA分子“海绵”发挥生物学功能。
有报道称ciRS-7可作为miR-7的分子海绵参与多种癌症相关通路影响肿瘤的发生发展[18]。胃癌中ciRS-7表达上调,进而影响miR-7下游的PTEN/PI3K/AKT信号通路,促进癌症的发生发展[19]。食管鳞状细胞癌中过表达的ciRS-7可通过靶向miR-876-5p,解除其对肿瘤相关抗原MAGE-A家族的抑制作用从而加速ESCC进程[20]。结肠癌中ciRS-7可直接结合miR-7影响其下游的EGFR-RAF1信号通路,发挥关键的促癌作用[21]。ciRS-7还可通过充当miRNA分子海绵在非小细胞肺癌和膀胱癌的进展中发挥重要作用[22-23]。
然而,ciRS-7在乳腺癌中的研究未见报道。三阴性乳腺癌作为乳腺癌中具有高度转移潜能的分子类型,其调控机制至今尚未阐明。本研究采用qRT-PCR技术检测了4种乳腺癌细胞系中ciRS-7的表达水平,发现三阴性乳腺癌细胞系中ciRS-7的表达明显高于其他类型的乳腺癌细胞系,这提示ciRS-7可能与TNBC的高侵袭转移能力相关。
为此本课题组设计了细胞迁移和侵袭试验及裸鼠转移瘤实验进一步探究ciRS-7对TNBC的侵袭和转移能力的影响。结果表明环状RNA ciRS-7在体内和体外实验中均可促进TNBC细胞的侵袭迁移水平。但其具体的分子机制尚未研究透彻,猜测其很可能通过miRNA分子海绵机制发挥调控作用,这有待在今后的研究中得到证实。
作者贡献
孟令娇:实验设计、实施及文章执笔
桑梅香:实验设计及文章审校
丁平安、巨英超、刘思桦、常胜:实验实施
韩若琦、潘烁、刘飞、谷丽娜:实验评估
| [1] | Shigemizu D, Iwase T, Yoshimoto M, et al. The prediction models for postoperative overall survival and disease-free survival in patients with breast cancer[J]. Cancer Med, 2017, 6(7): 1627–38. DOI:10.1002/cam4.2017.6.issue-7 |
| [2] | Fan L, Strasser-Weippl K, Li JJ, et al. Breast cancer in China[J]. Lancet Oncol, 2014, 15(7): e279–89. DOI:10.1016/S1470-2045(13)70567-9 |
| [3] | Brenton JD, Carey LA, Ahmed AA, et al. Molecular classification and molecular forecasting of breast cancer: ready for clinical application?[J]. J Clin Oncoly, 2005, 23(29): 7350–60. DOI:10.1200/JCO.2005.03.3845 |
| [4] | Carey LA, Dees EC, Sawyer L, et al. The Triple Negative Paradox: Primary Tumor Chemosensitivity of Breast Cancer Subtypes[J]. Clin Cancer Res, 2007, 13(8): 2329–34. DOI:10.1158/1078-0432.CCR-06-1109 |
| [5] | Dent R, Trudeau M, Pritchard KI, et al. Triple-negative breast cancer: clinical features and patterns of recurrence[J]. Clin Cancer Res, 2007, 13(15 Pt 1): 4429–34. |
| [6] | Memczak S, Jens M, Elefsinioti A, et al. Circular RNAs are a large class of animal RNAs with regulatory potency[J]. Nature, 2013, 495(7441): 333–8. DOI:10.1038/nature11928 |
| [7] | Jeck WR, Sharpless NE. Detecting and characterizing circular RNAs[J]. Nat Biotechnol, 2014, 32(5): 453–61. DOI:10.1038/nbt.2890 |
| [8] | Lasda E, Parker R. Circular RNAs: diversity of form and function[J]. RNA, 2014, 20(12): 1829–42. DOI:10.1261/rna.047126.114 |
| [9] | Hansen TB, Jensen TI, Clausen BH, et al. Natural RNA circles function as efficient microRNA sponges[J]. Nature, 2013, 495(7441): 384–8. DOI:10.1038/nature11993 |
| [10] | Piwecka M, Glažar P, Hernandez-Miranda LR, et al. Loss of a mammalian circular RNA locus causes miRNA deregulation and affects brain function[J]. Science, 2017, 357(6357): pii:eaam8526. DOI:10.1126/science.aam8526 |
| [11] | Cocquerelle C, Mascrez B, Hétuin D, et al. Mis-splicing yields circular RNA molecules[J]. FASEB J, 1993, 7(1): 155–60. DOI:10.1096/fasebj.7.1.7678559 |
| [12] | Kopczynski CC, Muskavitch MA. Introns excised from the Delta primary transcript are localized near sites of Delta transcription[J]. J Cell Biol, 1992, 119(3): 503–12. |
| [13] | Qian L, Vu MN, Carter M, et al. A spliced intron accumulates as a lariat in the nucleus of T cells[J]. Nucleic Acids Res, 1992, 20(20): 5345–50. DOI:10.1093/nar/20.20.5345 |
| [14] | Salzman J, Gawad C, Wang PL, et al. Circular RNAs Are the Predominant Transcript Isoform from Hundreds of Human Genes in Diverse Cell Types[J]. PLoS One, 2012, 7(2): e30733. |
| [15] | Jeck WR, Sorrentino JA, Wang K, et al. Circular RNAs are abundant, conserved, and associated with ALU repeats[J]. RNA, 2013, 19(2): 141–57. |
| [16] | Dang Y, Yan L, Hu B, et al. Tracing the expression of circular RNAs in human pre-implantation embryos[J]. Genome Biol, 2016, 17(1): 130. DOI:10.1186/s13059-016-0991-3 |
| [17] | Zhang XO, Wang HB, Zhang Y, et al. Complementary sequence-mediated exon circularization[J]. Cell, 2014, 159(1): 134–47. DOI:10.1016/j.cell.2014.09.001 |
| [18] | Hansen TB, Kjems J, Damgaard CK. Circular RNA and miR-7 in cancer[J]. Cancer Res, 2013, 73(18): 5609–12. DOI:10.1158/0008-5472.CAN-13-1568 |
| [19] | Pan H, Li T, Jiang Y, et al. Overexpression of Circular RNA ciRS-7 Abrogates the Tumor Suppressive Effect of miR-7 on Gastric Cancer via PTEN/PI3K/AKT Signaling Pathway[J]. J Cell Biochem, 2018, 119(1): 440–6. |
| [20] | Sang M, Meng L, Sang Y, et al. Circular RNA ciRS-7 accelerates ESCC progression through acting as a miR-876-5p sponge to enhance MAGE-A family expression[J]. Cancer Lett, 2018, 426: 37–46. DOI:10.1016/j.canlet.2018.03.049 |
| [21] | Weng W, Wei Q, Toden S, et al. Circular RNA ciRS-7-A promising prognostic biomarker and a potential therapeutic target in colorectal cancer[J]. Clin Cancer Res, 2017, 23(14): 3918–28. DOI:10.1158/1078-0432.CCR-16-2541 |
| [22] | Su C, Han Y, Zhang H, et al. CiRS-7 targeting miR-7 modulates the progression of non-small cell lung cancer in a manner dependent on NF-κB signalling[J]. J Cell Mol Med, 2018, 22(6): 3097–107. DOI:10.1111/jcmm.2018.22.issue-6 |
| [23] | Li P, Yang X, Yuan W, et al. CircRNA-Cdr1as Exerts Anti-Oncogenic Functions in Bladder Cancer by Sponging MicroRNA-135a[J]. Cell Physiol Biochem, 2018, 46(4): 1606–16. DOI:10.1159/000489208 |
 2019, Vol. 46
2019, Vol. 46



