文章信息
- 胃癌患者血清miR-181d与PDCD4的表达及意义
- Expression and Clinical Significance of miR-181d in Serum and PDCD4 in Gastric Cancer Patients
- 肿瘤防治研究, 2019, 46(2): 131-137
- Cancer Research on Prevention and Treatment, 2019, 46(2): 131-137
- http://www.zlfzyj.com/CN/10.3971/j.issn.1000-8578.2019.18.0486
- 收稿日期: 2018-04-13
- 修回日期: 2018-10-30
2. 100700 北京,中国人民解放军总医院第七医学中心消化内科
2. Department of Gastroenterology, The 7th Medical Center, Chinese PLA General, Beijing 100700, China
青海省胃癌发病率居全国首位,研究表明高海拔、高寒、缺氧、干燥的自然环境,以及高盐、含亚硝胺类化合物的饮食习惯等因素导致青海省肿瘤发病率较高[1]。然而,目前对胃癌的早期诊断及治疗方案的选择均缺少有效的客观指标,从而导致胃癌的5年生存率并无明显改善[1-2]。
前期工作获得了胃癌基因差异表达谱和miRNA差异表达谱,通过生物信息学分析,建立了microRNA(miRNA)-mRNA分子间相互作用模式[3]。近年的研究表明miRNA可通过调控细胞增殖、凋亡和分化,促进或抑制肿瘤的恶性表型[4],相比正常组织来源的细胞,肿瘤组织中miRNA存在明显异常表达,这些异常表达的miRNA在肿瘤形成中扮演重要角色,可作为肿瘤病因学、生物学特性、组织分型和临床分级分期的分子标志物[5]。由于miRNA较稳定,可在石蜡组织以及血清或血浆中检测到,因此,miRNA作为临床辅助诊断和预后判断的分子标志物具有良好的应用前景[6]。本研究前期通过胃癌miRNA表达谱分析获得关键基因miR-181d,结合胃癌基因差异表达谱分析发现其潜在靶基因PDCD4,在胃癌组织中检测发现miR-181d与PDCD4的表达具有一定的负相关[7]。本实验进一步在青海地区胃癌患者血清中进行miR-181d与PDCD4的检测,并分析其在胃癌发生发展中的作用。
1 资料与方法 1.1 资料 1.1.1 细胞系实验选用胃癌细胞系BGC823、AGS、N87、SGC7901和MGC803;1株永生化胃黏膜细胞系GES1。其中GES1、BGC823、N87、SGC7901和MGC803细胞系是国内建系,均来自北京肿瘤防治研究所,AGS细胞系购自美国ATCC公司。
1.1.2 组织标本及临床病例资料58例胃癌血清来源于2014—2016年青海大学附属医院术前未经放化疗患者,50例健康者血清标本来自青海大学附属医院检验科,并与患者签订知情同意书。
1.1.3 试剂药品胎牛血清购自美国Hyclone公司;DMEM培养基和RNA提取试剂盒购自美国Invitrogen公司;miRNeasy Serum/Plasma Kit购自德国Qiagen公司;反转录试剂盒(EasyScript First-strand cDNA Synthesis Supermix,AE301)购自北京TransGen公司;引物和Oligo(dT)15购自北京奥科生物公司;PDCD4抗体和β-action抗体购自美国Sigma公司;ELISA试剂盒购自厦门慧嘉生物科技有限公司;通用型鼠二步法(PV6001, Power VisionTM)试剂盒和DAB显色试剂盒购自北京中杉生物技术公司。ECL Plus检测试剂盒购自英国Amersham Biosciences公司, Dual-Luciferase E1910报告基因检测系统购自美国Promega公司;Lipofectamine RNAiMAX购自美国Invitrogen公司。
1.2 实验方法 1.2.1 ELISA实验用50 mmol/L的碳酸盐包被缓冲液(pH=9.6)溶解抗原,使抗原浓度为10~20 μg/ml,加100微升每孔到96孔酶标板,4℃放置过夜。第二天弃去包被液后,用PBST洗涤3次,每孔加入150 μl 1%BSA37℃封闭1 h。PBST洗涤3次后,每孔加入100 μl不同倍比稀释度的血清,并加入对照样品,37℃孵育2 h。PBST洗涤5次后,加入100 μl稀释后的HRP标记的二抗,37℃孵育1 h。PBST洗涤5次,显色剂显色20 min后,酶标仪上读取A450吸光度值。
1.2.2 Real-time PCR RNA的提取血清和血浆中的总RNA提取使用miRNeasy Serum/Plasma Kit试剂盒完成,操作步骤及注意事项按照miRNeasy Serum/Plasma Kit说明书进行。cDNA制备:采用反转录试剂盒,过程如下:取5.0 μg总RNA,加入0.5 μl Oligo(dT)或加入0.5 μl miR-181d反转录引物,2×ES反应混合物10 μl,EasyScriptRT/RI酶混合物1 μl,补Rnase-free H2O至20 μl,42℃孵育30 min。85℃加热5 min失活EasyScript RT。分装后-20℃保存备用。miR-181d成熟体Real-time PCR:根据miRNA序列设计特异性反转录引物,所使用的反转录及PCR引物见表 1。反转录反应条件设置为:16℃ 30 min;37℃ 30 min;72℃ 10 min。荧光实时定量PCR反应采用SYBR green-based Real-time PCR(Applied Biosystems AB)系统。反应程序设置为:95℃ 5 min,预变性;95℃ 30 s,40个循环;57℃ 30 s,延伸。以miR-16作为内参[8],采用2-ΔΔCT相对定量计算方法检测miRNA差异表达水平,见表 1。
在直径100 mm平皿中生长良好密度90%的贴壁培养细胞中加入200~300 μl 85℃的1×SDS凝胶加样缓冲液裂解细胞;裂解样品经煮沸、超声、离心后,取上清液,UV定量,-20℃分装保存。将50 μg样品加入2×SDS凝胶加样缓冲液,100℃变性10 min,顺序加样,行SDS-PAGE电泳。电泳结束后,经湿式电转仪(Bio-Rad)恒压冰浴转膜,5%脱脂牛奶4℃封闭过夜;孵育一抗PDCD4、β-actin,4℃过夜。加入相应种属二抗孵育1 h,化学发光、显影、定影、观察杂交信号。
1.2.4 胃癌细胞系的瞬时转染使用Lipofectamine RNAiMAX将60 nmol/L miR-181d mimics或Anti-miR-181d转染到AGS和BGC823细胞培养皿中;37℃孵育48 h后进行Real-time PCR、Western blot和双荧光报告素酶检测。
1.2.5 双荧光报告素酶检测PDCD4基因的3’-UTR荧光素酶报告PCR引物从以下人类cDNA扩增:PDCD4正向引物,5’-gcgagctc ctggttcta gaagaatgta cac-3’;PDCD4反向引物,5′-gcaagcttca ggta atattttctc atacattagg-3’。变异的miR-181d的结合位点,在PDCD4 3’-UTR互补序列(gaatgt)所取代gttaga。对PCR产物进行酶切用SacⅠ和Hind Ⅲ和插入表达载体。HEK-293细胞接种于24孔板,转染野生型或突变型细胞3’-UTR报告-质粒pRL-TK,或转染miR-NC和miR-181d。进行荧光素酶检测24 h使用双荧光素酶报告基因检测系统转染后。采用Dual-Luciferase E1910报告基因检测系统,获得miRNA是否对靶基因3' -UTR产生调控作用。LARⅡ:用Luciferase Assay BufferⅡ溶解冻干粉Luciferase Assay Substrate。将50×Stop & Glo® Substrate加入到所需要量的Stop & Glo Buffer中,使终浓度成为1倍浓度。通过监测荧光素酶的活性变化,即可获得miRNA是否对靶基因3' -UTR产生调控作用,当miRNA与3' -UTR靶标区域互补配对时,荧光素酶活力将显著降低;反之,当miRNA与报告基因(包含靶标区域突变)无法形成有效的互补配对时,荧光素酶活力将与对照无显著区别。
1.3 统计学方法采用SPSS23.0统计软件进行数据分析。miR-181d和PDCD4在胃癌血清和健康对照血清基因表达差异分析采用卡方检验,其他结果采用student t-test检验。相关性分析采用Spearman分析,临床病理资料分析采用Cox多因素生存分析。P < 0.05为差异有统计学意义。
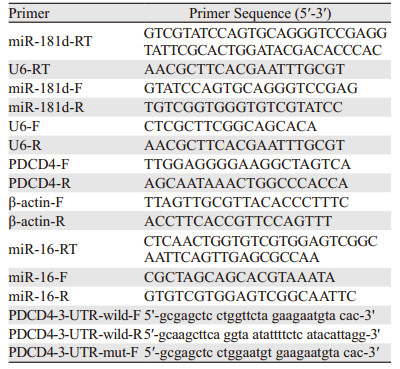
2 结果 2.1 miR-181d和PDCD4基因在胃癌细胞系及配对胃癌组织中的表达Real-time PCR检测发现miR-181d在胃癌细胞系BGC823、SGC7901、MGC803、AGS、N87中存在显著的差异表达,在永生化胃黏膜上皮细胞系GES1中表达量较低;而在胃癌细胞系BGC823、MGC803、SGC7901中表达较GES1显著增加,在AGS和N87中表达水平较高于GES1细胞中的表达,见图 1A。相反,预测的下游靶基因PDCD4在mRNA和蛋白水平均显示胃癌细胞系中的表达低于GES1,见图 1B。为了检测PDCD4蛋白是否在胃癌细胞中分泌,我们首先将胃癌细胞系BGC823、SGC7901、MGC803、AGS、N87和GES1通过双无血清培养48 h,然后分别提取上清蛋白并进行Western blot实验发现,发现在细胞上清蛋白上样浓度一致性的前提下,PDCD4在AGS、SGC7901、N87和永生化胃黏膜上皮细胞系GES1细胞上清液中均有表达,见图 1C~D。
|
| A: the expression of miR-181d in GC and GES1 cell lines; B: the expression of PDCD4 in GC and GES1 cell lines; C: the consistency of concentrated supernatant protein in GC and GES1 cell lines was showed by Coomassie Blue Staining; the detection of PDCD4 expression in double serum-free cultured GC and GES1 cell lines supernatants; D: the expression of PDCD4 protein in GC and GES1 cell lines 图 1 miR-181d与PDCD4在GES1和胃癌细胞系中的表达 Figure 1 Expression of miR-181d and PDCD4 in GES1 and gastric cancer cell lines |
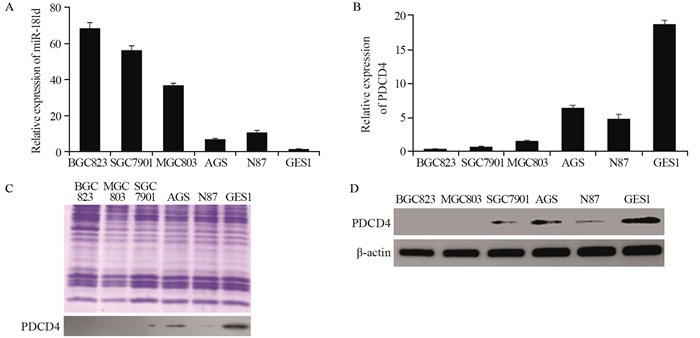
结果显示miR-181d在胃癌患者血清中的表达高于健康体检者血清(P < 0.001),而PDCD4蛋白在胃癌患者血清中的表达低于健康者血清(P < 0.05), 通过相关性分析发现胃癌患者和健康者血清miR-181d与PDCD4蛋白表达呈负相关(R2=-0.44, R2=-0.426),以上结果提示血清miR-181d与PDCD4蛋白的表达呈负相关,差异有统计学意义(P < 0.01),见图 2。
|
| 图 2 miR-181d和PDCD4在胃癌患者及健康者血清中的表达 Figure 2 Expression of miR-181d and PDCD4 in serum of gastric cancer and normal group |
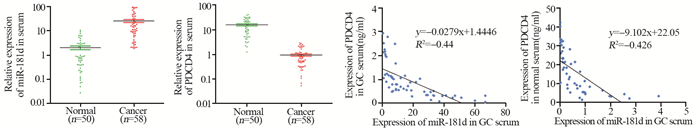
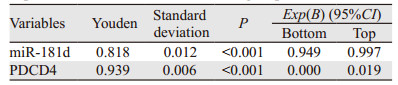
ROC曲线分析结果显示,血清miR-181d的曲线下面积为0.818,敏感度为0.879,特异性为0.939,假阳性率为6.1%,假阴性率为12.1%(P < 0.001);PDCD4基因的曲线下面积为0.939,敏感度为0.939,特异性为1.0,假阳性率为0,假阴性率为6.1%(P < 0.001)。提示二者用于体外诊断区分肿瘤和健康人群具有良好的特异性和敏感度,见图 3、表 2。
|
| 图 3 miR-181d与PDCD4的ROC曲线分析 Figure 3 ROC analysis of miR-181d and PDCD4 curves |
|
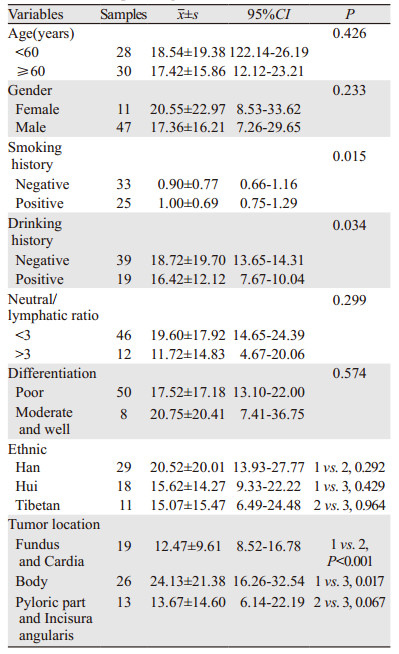
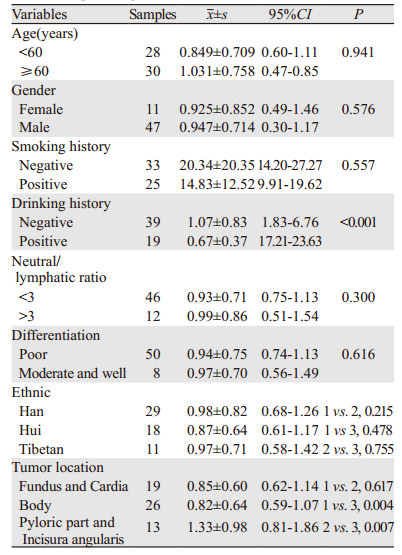
结合58例胃癌患者的临床病理资料分析发现,血清miR-181d的表达分别与吸烟喝酒史呈正相关(P=0.015, P=0.034),与肿瘤发生部位呈正相关(胃底贲门:P < 0.001,胃体:P=0.017),与年龄、性别、民族、胃癌肿瘤的分化程度、胃癌患者临床分期等无关(P > 0.05),见表 3。而PDCD4的表达与饮酒史呈正相关(P < 0.001),与年龄、性别、民族、胃癌肿瘤的分化程度、胃癌患者临床分期等无关(P > 0.05),见表 4。
|
|
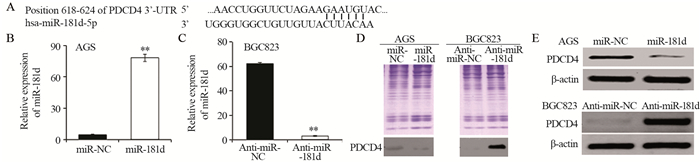
Real-time PCR和Western blot实验发现,miR-181d mimics瞬间转染AGS细胞后PDCD4的表达下降。而BGC823细胞瞬时转染反义miR-181d 48 h后PDCD4的表达水平增加。进一步实验发现,转染miR-181d mimics后PDCD4在AGS细胞上清中的表达下降,而在BGC823细胞上清中的表达水平增加,见图 4。
|
| A: the conservative binding region of PDCD4 was detected in miR-181d; B: Real-time PCR indicated that the overexpression of miR-181d in AGS cells; C: Real-time PCR indicated that the interference of miR-181d in BGC823 cells; D: the consistency of concentrated supernatant protein in gastric cell lines was showed by Coomassie Blue Staining; E: Western blot showed overexpression of miR-181d in AGS cells decreased the expression of PDCD4, and the expression of PDCD4 was increased after transfection with anti-miR-181d in BGC823 cells; NC: negative control; **: P < 0.01 图 4 在胃癌细胞中PDCD4的表达受miR-181d影响 Figure 4 miR-181d may affect PDCD4 expression in gastric cancer cell lines |
将miR-181d mimics或miR-NC一同转染AGS细胞36 h,通过双荧光报告系统检测结果发现,干扰miR-181d后,AGS细胞中的PDCD4荧光报告活性比miR-NC组高,而过表达miR-181d mimics可以降低BGC823细胞中PDCD4的荧光报告活性,提示miR-181d可以结合PDCD4的3′-UTR区,见图 5。进一步,PDCD4基因3′-UTR荧光素酶报告PCR引物从以下人类cDNA扩增:PDCD4的3′-UTR正向引物:5′-GCGAGCTC CTGGTTCTA GAAGAATGTA CAC-3′;PDCD4的3′-UTR反向引物:5′-GCAAGCTTCA GGTAATATTTTCTC ATACATTAGG-3′。将PDCD4 3′-UTR与miR-181d结合的3′-UTR的结合位点进行引物突变,以PDCD4 3′-UTR互补序列(GAATGT)取代GTTAGA。对PCR产物进行酶切用SacⅠ和Hind Ⅲ,插入表达载体。HEK-293细胞接种于24孔板,转染野生型或突变型PDCD4的3′-UTR报告质粒体系pRL-TK、miR-NC和miR-181d。转染后24 h使用双荧光素酶报告基因检测系统,结果发现野生型组荧光素酶活性低于突变型组,提示miR-181d可以结合并抑制PDCD4 3′-UTR区的荧光活性,见图 5。
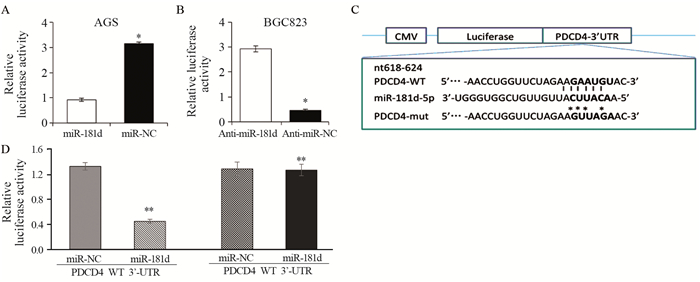
|
| A: relative fluorescence activity of PDCD4 was lower in AGS cells compared with control group; B: silence of miR-181d indecreased relative fluorescence activity of PDCD4; C: the mutated miR-181d binding site was replaced by GTTTAG in the 3'-UTR complement (GAATGT) of PDCD4; D: transfection of wild-type or mutant 3'-UTR reporter plasmids pRL-TK, miR-NC and miR-181d. The results showed that the luciferase activity of the mutant group was higher than that in wild-type group (*: P < 0.05; **: P < 0.01) 图 5 miR-181d可结合在PDCD4的3'-UTR区发挥作用 Figure 5 miR-181d could bind to 3'-UTR region of PDCD4 |
miRNA可参与肿瘤发生、发展、转移的各个阶段,并且不同类型的肿瘤具有特异性的miRNA表达谱,这些表达谱可有效反映肿瘤组织的发展与分化状态[5]。同时, 血清miRNA较稳定,可以抵抗核糖核酸酶、加热、pH变化,并能长期保存和重复冷冻、解冻,作为肿瘤的生物标志物,创造性地提供了一种非损伤性的肿瘤诊断手段[9-10]。在前期工作中通过结合胃癌基因差异表达谱和miRNA差异表达谱,发现特征基因miR-181d在胃癌中表达高于正常组织,且与预测的靶基因PDCD4的表达呈负相关[7]。本研究结果进一步发现,miR-181d在胃癌患者血清中的表达高于正常对照组,而PDCD4蛋白在胃癌患者血清中的表达低于正常对照组,且二者的表达呈负相关。通过ROC曲线分析显示,miR-181d与PDCD4对区分胃癌组织和正常组织具有良好的特异性和敏感度。目前miRNA与胃癌的研究报道认为,miRNAs可以促进或抑制胃癌的恶性表型,在胃癌进程中发挥癌基因或抑癌基因样作用,可作为胃癌早诊和预后的标志物。近年来,研究者利用小鼠模型发现弥漫型胃癌早期诊断的血清miRNA可作为生物标志物[11]。我国研究者以山东省临朐县胃癌高发现场胃癌患者为研究对象,通过miRNA芯片技术结合病例-对照和回顾前瞻性研究,确定血清中的miR-221、miR-744和miR-376c可作为一种新型的非侵入性的胃癌生物标志物[12]。
miRNA可与下游潜在靶基因mRNA的3’-UTR通过不完全配对结合降低mRNA稳定性或抑制靶基因的翻译,从而参与调控基因表达[4-5]。本研究在AGS细胞中过表达miR-181d后PDCD4的表达下降。而在BGC823细胞中转染反义miR-181d后PDCD4的表达水平增加。双荧光报告系统提示miR-181d可以结合到PDCD4的3' -UTR区。有研究发现,miR-181c和miR-181d位于同一个基因簇,在肝癌、胰腺癌、胃癌中表达均增高[13-14],并且miR-181d在小细胞肺癌化疗敏感度中起一定作用[15],在H69AR及对化疗耐药患者中呈低表达,同时伴随着BCL2高表达。上调miR-181d能降低BCL2表达水平,增加H69AR细胞对化疗药物的敏感度,miR-181d的表达与肿瘤的分期、化疗敏感度及生存时间相关。miR-181d在食管鳞癌中作为抑癌基因负调节Derlin-1的表达[16]。在乳腺导管癌中miR-181a和miR-181d的过表达促进细胞进入S期,致使细胞增殖,同时在乳腺导管癌中miR-181a和miR-181d通过调控共同目标PHLPP2和INPP4B磷酸酶升高调节Akt信号通路[17]。miR-181d/MALT1通过NF-κB通路调节胶质母细胞瘤轴衰减间质表型[18]。miR-181d在胰腺癌中高表达,下调miR-181d可负向调控KNAIN2抑制胰腺癌的恶性增殖[19]。本研究在胃癌患者的血清中检测发现miR-181d的表达水平较健康者高,而PDCD4的表达水平明显降低,二者的表达呈负相关,进一步实验发现miR-181d可能是通过作用于PDCD4的3' -UTR发挥负向调控作用。综上所述,在胃癌患者血清中联合检测miR-181d和PDCD4的表达将为胃癌血清分子诊断提供一定的理论依据。
(致谢: 感谢青海大学附属医院肿瘤外科马晓明医师和检验科杨启英医师为本课题研究提供胃癌患者及健康体验者血液标本。)
作者贡献
安娟、潘元明:设计课题、实验操作、数据分析及论文撰写 康倩、王昕:设计课题、实验操作 孙 伟:设计课题、实验操作,研究所用样本、材料的整理分析 晏宇鹏:研究所用样本、材料的整理分析 郑 伟:实验操作,研究所用样本、材料的整理分析
| [1] | Zhao JD, Geng PL, Li ZQ, et al. Associations between interleukin-1 polymorphisms and gastric cancers among three ethnicities[J]. World J Gastroenterol, 2012, 18(47): 7093–9. DOI:10.3748/wjg.v18.i47.7093 |
| [2] | Jing JJ, Liu HY, Hao JK, et al. Gastric cancer incidence and mortality in Zhuanghe, China, between 2005 and 2010[J]. World J Gastroenterol, 2012, 18(11): 1262–9. DOI:10.3748/wjg.v18.i11.1262 |
| [3] | An J, Pan Y, Yan Z, et al. MiR-23a in amplified 19p13.13 loci targets Metallothionein 2A and promotes growth in gastric cancer cells[J]. J Cell Biochem, 2013, 114(9): 2160–9. DOI:10.1002/jcb.24565 |
| [4] | Kim VN, Nam JW. Genomics of microRNA[J]. Trends Genet, 2006, 22(3): 165–73. |
| [5] | Bartel DP. MicroRNAs:genomics, biogenesis, mechanism, and function[J]. Cell, 2004, 116(2): 281–97. DOI:10.1016/S0092-8674(04)00045-5 |
| [6] | Malumbres R, Sarosiek KA, Cubedo E, et al. Differentiation stage-specific expression of microRNAs in B lymphocytes and diffuse large B-cell lymphomas[J]. Blood, 2009, 113(16): 3754–64. DOI:10.1182/blood-2008-10-184077 |
| [7] | Pan Y, Xing R, An J, et al. Amplification of the miR-181c/d cluster is inversely correlated with PDCD4 expression in gastric cancer[J]. Science Bulletin, 2014, 59(19): 2240–8. DOI:10.1007/s11434-014-0280-z |
| [8] | 李雅楠, 郝玉宾, 张志, 等. 实时荧光定量PCR相对定量法检测循环miRNA的内参选择[J]. 生物技术通讯, 2015, 26(1): 146–8. [ Li YN, Hao YB, Zhang Z, et al. Selection of Endogenous Reference Gene for RT-qPCR Analysis of Circulating microRNA Levels[J]. Sheng Wu Ji Shu Tong Xun, 2015, 26(1): 146–8. DOI:10.3969/j.issn.1009-0002.2015.01.032 ] |
| [9] | Schwarzenbach H, Nishida N, Calin GA, et al. Clinical relevance of circulating cell-free microRNAs in cancer[J]. Nat Rev Clin Oncol, 2014, 11(3): 145–56. DOI:10.1038/nrclinonc.2014.5 |
| [10] | Kroh EM, Parkin RK, Mitchell PS, et al. Analysis of circulating microRNA biomarkers in plasma and serum using quantitative reverse transcription-PCR (qRT-PCR)[J]. Methods, 2010, 50(4): 298–301. DOI:10.1016/j.ymeth.2010.01.032 |
| [11] | Rotkrua P, Shimada S, Mogushi K, et al. Circulating microRNAs as biomarkers for early detection of diffuse-type gastric cancer using a mouse model[J]. Br J Cancer, 2013, 108(4): 932–40. DOI:10.1038/bjc.2013.30 |
| [12] | Song MY, Pan KF, Su HJ, et al. Identification of serum microRNAs as novel non-invasive biomarkers for early detection of gastric cancer[J]. PLoS One, 2012, 7(3): e33608. DOI:10.1371/journal.pone.0033608 |
| [13] | Wang B, Hsu SH, Majumder S, et al. TGF beta-mediated upregulation of hepatic miR-181b promotes hepatocarcinogenesis by targeting TIMP3[J]. Oncogene, 2010, 29(12): 1787–97. DOI:10.1038/onc.2009.468 |
| [14] | Bloomston M, Frankel WL, Petrocca F, et al. MicroRNA expression patterns to differentiate pancreatic adenocarcinoma from normal pancreas and chronic pancreatitis[J]. JAMA, 2007, 297(17): 1901–8. DOI:10.1001/jama.297.17.1901 |
| [15] | Wang YT, Bai YF, Hu HT. Effect of miR181d on chemo-sensitivity in human small cell lung cancer[J]. Chin J Chin Oncol, 2015(6): 345–50. |
| [16] | Li D, Shi M, Ji H, et al. MicroRNA-181d is a tumor suppressor in human esophageal squamous cell carcinoma inversely regulating Derlin-1[J]. Oncol Rep, 2016, 36(4): 2041–8. DOI:10.3892/or.2016.5028 |
| [17] | Strotbek M, Schmid S, Sánchez-González I, et al. miR-181 elevates Akt signaling by co-targeting PHLPP2 and INPP4B phosphatases in luminal breast cancer[J]. Int J Cancer, 2017, 140(10): 2310–20. DOI:10.1002/ijc.v140.10 |
| [18] | Yang F, Liu X, Liu Y, et al. miR-181d/MALT1 regulatory axis attenuates mesenchymal phenotype through NF-κB pathways in glioblastoma[J]. Cancer Lett, 2017, 396: 1–9. DOI:10.1016/j.canlet.2017.03.002 |
| [19] | Zhang G, Liu D, Long G, et al. Downregulation of microRNA-181d had suppressive effect on pancreatic cancer development through inverse regulation of KNAIN2[J]. Tumour Biol, 2017, 39(4): 1010428317698364. |
 2019, Vol. 46
2019, Vol. 46