文章信息
- shRNA干扰半胱氨酸蛋白酶抑制剂C抑制非小细胞肺癌A549细胞侵袭和迁移
- shRNA Interferes with Cystatin C to Inhibit Invasion and Migration of Non-small Cell Lung Cancer A549 Cells
- 肿瘤防治研究, 2018, 45(12): 954-958
- Cancer Research on Prevention and Treatment, 2018, 45(12): 954-958
- http://www.zlfzyj.com/CN/10.3971/j.issn.1000-8578.2018.18.0802
- 收稿日期: 2018-06-14
- 修回日期: 2018-09-03
2. 637000 南充,南充市中心医院肿瘤科
2. Department of Oncology, Nanchong Central Hospital, Nanchong 637000, China
非小细胞肺癌(NSCLC)约占肺癌总病例的85%,5年生存率为10%~15%[1]。NSCLC患者最常见的治疗障碍是肿瘤复发和转移,因此即使手术切除依旧保持高水平的死亡率。虽然研究表明,手术切除辅助顺铂化疗可以提高NSCLC患者的生存质量,但化疗对患者造成的不良反应仍不利于患者康复[2]。因此,寻找新的NSCLC治疗靶点和药物是当前肺癌研究的方向。半胱氨酸蛋白酶抑制剂C(cystatin C, Cys C)是一种半胱氨酸蛋白酶抑制剂,又称CST3,能抑制组织蛋白酶B/H/L/V蛋白酶活性,特别能强力抑制组织蛋白酶B。作为半胱氨酸蛋白酶抑制剂超家族中的家族成员,由CST3基因编码,在炎性反应和肿瘤发生过程中起着重要的作用。现已在食管癌[3]、乳腺癌[4]、肾细胞癌[5]、前列腺癌[6]、多发性骨髓瘤[7-8]和卵巢癌[9]相关研究中证实,Cys C在这些患者体液中水平升高。Cys C被证明在结直肠癌患者中与高风险的转移和侵袭有关[10]。另有研究表明,Cys C在NSCLC患者体液中增加[11]。然而,迄今为止还未有报道证明Cys C与NSCLCs的侵袭和迁移的关系。因此,本研究主要是探索Cys C对NSCLC细胞A549侵袭和转移的影响。
1 材料与方法 1.1 主要试剂非小细胞肺癌A549细胞购自美国ATCC;培养基F-12K、胎牛血清、胰蛋白酶购自美国Gibco公司;RNA提取试剂盒Direct-zolTM RNA MiniPrep kit购自北京天漠科技有限公司;脂质体转染试剂盒Lipofectamine RNAiMAX Kit和NCodeTM miRNA第一链cDNA合成试剂盒购自美国Invitrogen公司;SYBR Green Master mix,RevertAidTM H Minus First Strand cDNA合成试剂盒购自美国赛默飞世尔;所有一抗Cys C、VEGF、MMP-9、MMP-14、GAPDH购自美国CST公司。二抗山羊抗兔IgG-HRP购自美国Santa Cruz公司;Transwell小室及人工基底膜均购自美国BD公司。
1.2 Cys C shRNA构建Cys C shRNAs的构建根据Tuschl[12]的方法来完成。Cys C mRNA的shRNA靶向序列:5’-GCTCCTGCTGGCCATCCTG-3’,scramble shRNA序列:5’-TGCGAACGACTTCTTCGCC-3’,用作阴性对照。分别将两种shRNA寡核苷酸合成并插入psil2.1-U6载体。DNA测序验证结构的完整性。
1.3 细胞培养和转染A549细胞F-12K培养于含10%胎牛血清(FBS)和1%双抗(青-链霉素)的F-12K培养液中,并置于37℃、5%CO2的恒温培养箱中培养。在转染前1天,细胞在24孔板(每孔3×105个细胞)中培养。质粒在转染前由SmaⅠ线性化。当细胞覆盖80%的培养皿时,根据文献所描述的方法[13],将400 ng的线性化质粒转染到孔中。sh-Cys C和阴性对照(scramble shRNA, sh-ctrl)用于转染细胞。使用脂质体2000将sh-Cys C-pRS(转染载体为pRS载体)转染进A549细胞。在含有400 μg/ml G418筛选培养基中培养两周,将存活的细胞用胰酶消化,稀释至密度约10~40个每毫升,取细胞悬液到96孔板,挑选单个细胞培养增殖,并筛选Cys C表达水平最低的细胞株。用该细胞株进行后续实验。分别用RT-PCR和Western blot检测Cys C mRNA和蛋白表达水平。
1.4 RT-PCR检测CysC mRNA水平根据说明书,利用TRIzol试剂(TaKaRa,中国大连)从培养细胞中提取总RNA。根据PrimeScript RT试剂盒说明书,2 μg总RNA被用来合成cDNA(第一链)。使用SYBR Green Master mix和CFX 96 Real-time PCR系统(Bio-rad)扩增cDNA模板。GAPDH作为内参。数据分析根据比较Ct法,也称为2-ΔΔCT方法。Cys C正向引物:5’-GCT CTT TCCAGA TCT ACG CT-3’;反向引物:5’-AGG CAGCCG ATG CTA CTA TT-3’;β-actin正向引物:5’-TAC ATG GCT GGG GTGTTG AA-3’;反向引物:5’-AAG AGA GGC ATCCTC ACC CT-3’。
1.5 Western blot检测蛋白表达水平A549细胞被转染sh-Cys C和sh-ctrl后,收集细胞,用PBS洗涤两次,用细胞裂解液在冰上裂解细胞15 min;细胞悬液加入含有SDS的5×loading缓冲液在沸水中变性10 min。细胞总蛋白用BCA试剂盒检测。上样等量(20 μg)蛋白样品于SDS-PAGE分离胶上,并转移到PVDF膜。5%脱脂奶粉封闭后,PVDF膜孵育一抗(抗体稀释用5%脱脂奶粉溶解于TBST)4℃过夜。使用以下抗体:HIF1α(1:1 000)、GAPDH(1:1 000)。二抗用山羊抗兔IgG-HRP(1:5 000)。用ECL系统(德国Millipore)检测目标蛋白质。用Image J软件统计灰度值计算相对表达量。GAPDH作为上样量一致的参照,至少进行三个独立的重复实验。
1.6 CCK-8检测细胞增长情况三组待测细胞:不转染(A549)、A549细胞转染sh-Cys C、A549细胞转染sh-ctrl。培养三组细胞24 h后,细胞被胰蛋白酶消化,并接种到96孔板,然后在37℃恒温培养箱中分别培养0、24、48、96 h,向每孔加入10 μl CCK-8溶液,将培养板在培养箱内孵育1~4 h。最后用酶标仪检测各个时间段三组细胞在450 nm(OD450)的吸光度,计算细胞增殖倍数。
1.7 Transwell检测细胞侵袭将小室放入培养板中,上室加入300 μl预温的无血清培养基,使基质胶再水化,再吸去剩余培养液。首先,用含BSA的无血清培养基将各组待测的细胞制成1×106个每毫升的悬液。然后,取细胞悬液100~200 μl加入Transwell上室中,并在下室中加入含500 μl含FBS的培养基。最后,在37℃下培养12~24 h,用0.5%的结晶紫对下室底部细胞进行染色,并用棉签除去下室内侧的细胞。最后显微镜下观察细胞形态并统计细胞数量。
1.8 划痕检测细胞迁移将细胞密度为每毫升1×106的细胞悬液加入到6孔板中,过夜培养至形成单层细胞。在单层细胞上用10 μl的枪头划横线,磷酸缓冲液(PBS)洗去脱落的细胞,重复2次。此时显微镜下拍照作为0 h的样,加入无血清培养基继续培养24 h后再取出拍照作为24 h的样,伤痕愈合率=24 h细胞间距/0 h细胞间距×100%。
1.9 统计学方法采用SPSS17.0软件对实验数据进行统计学分析,两两比较用独立的t检验,分析比较处理组与对照组之间增殖倍数、迁移与侵袭能力以及蛋白表达的差异。以上各组检验标准取P < 0.01为差异有统计学意义。
2 结果 2.1 sh-Cys C抑制Cys C转录和翻译水平的表达A549细胞转染sh-Cys C后,与对照组相比,Cys C表达在阴性对照sh-ctrl中表达无明显差异,sh-Cys C干扰后,Cys C表达明显被抑制包括mRNA水平和蛋白水平分别下调77%(P=0.003)和79%(P=0.003),见图 1。
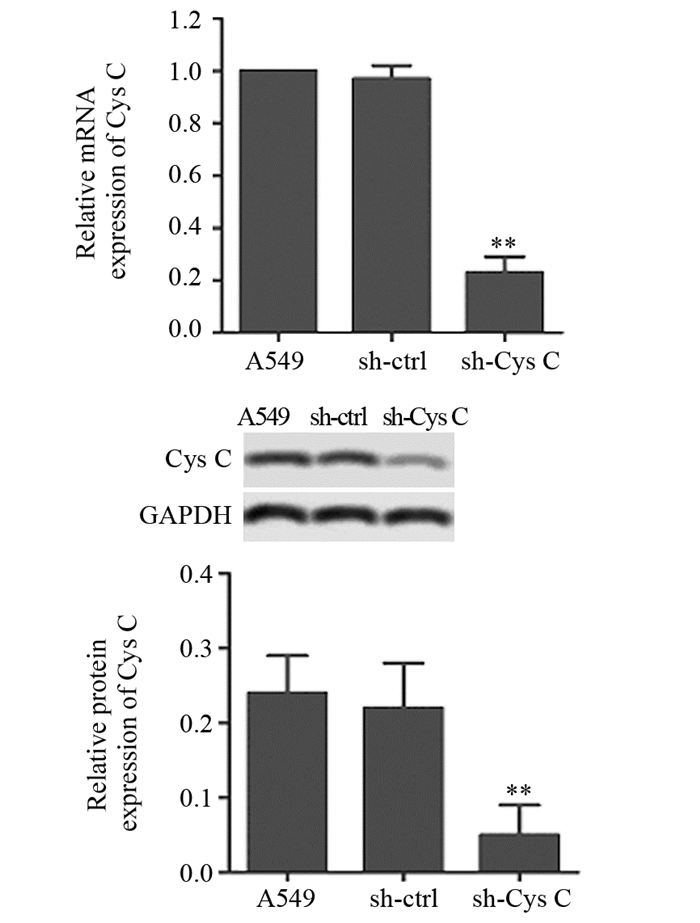
|
| **: P=0.003, compared with A549 group 图 1 Cys C shRNA导致A549细胞mRNA水平和蛋白水平降低 Figure 1 Cystatin C (Cys C) shRNA inhibited mRNA and protein expression levels of A549 cells |
为了验证Cys C对A549细胞生长的影响,本研究转染sh-Cys C到A549细胞,采用CCK-8检测细胞增殖能力的变化。sh-Cys C抑制A549细胞增殖,尤其在培养48、72、96 h后增殖抑制明显,与对照组相比增殖倍数降低,分别为1.7(P=0.005)、2.9(P=0.005)和3.3(P=0.003);而sh-ctrl对A549细胞增殖无明显影响。说明Cys C shRNA可抑制A549细胞生长,见图 2。
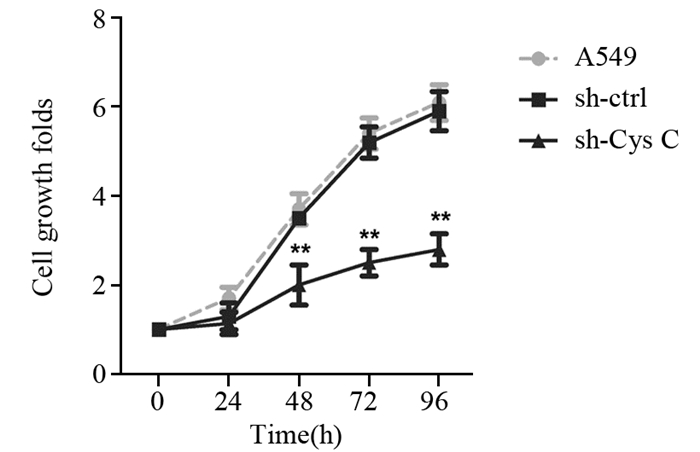
|
| **:P < 0.01, compared with A549 group 图 2 Cys C shRNA抑制A549细胞增殖 Figure 2 Cys C shRNA inhibited growth of A549 cells |
本研究采用Transwell法检测sh-Cys C转染A549细胞后侵袭能力的变化,与对照组相比,在阴性对照sh-ctrl中侵袭细胞数稍微下降,但差异无统计学意义;sh-Cys C组侵袭细胞数减少109个(P=0.003),见图 3。说明Cys C shRNA能抑制A549细胞侵袭。
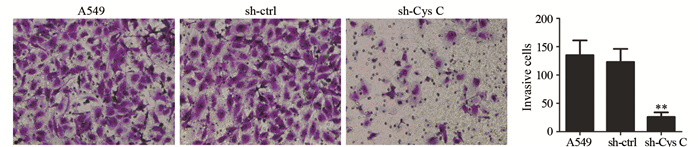
|
| **:P=0.003, compared with A549 group 图 3 Cys C shRNA抑制A549细胞侵袭 Figure 3 Cys C shRNA inhibited invasion of A549 cells |
为了探究sh-Cys C转染A549细胞后迁移能力的变化,本研究采用划痕实验检测A549细胞的迁移能力。划痕24 h后,sh-Cys C组的伤痕愈合率降低50%(P=0.007),见图 4,说明Cys C shRNA抑制A549细胞迁移。
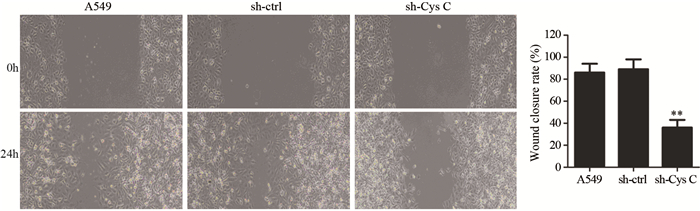
|
| **:P=0.007, compared with A549 group 图 4 Cys C shRNA抑制A549细胞迁移 Figure 4 Cys C shRNA inhibited migration of A549 cells |
为了进一步确定Cys C shRNA抑制A549细胞侵袭和迁移能力的作用,本实验检测金属蛋白酶MMP-9、MMP-14和血管内皮生长因子VEGF的表达情况,与对照组相比,在阴性对照sh-ctrl中蛋白表达无明显差异;结果显示sh-Cys C导致MMP-9、MMP-14和VEGF蛋白表达下调:MMP-9下调约5倍(P=0.004)、MMP-14下调约3.7倍(P=0.003)和VEGF下调约4倍(P=0.002),见图 5。
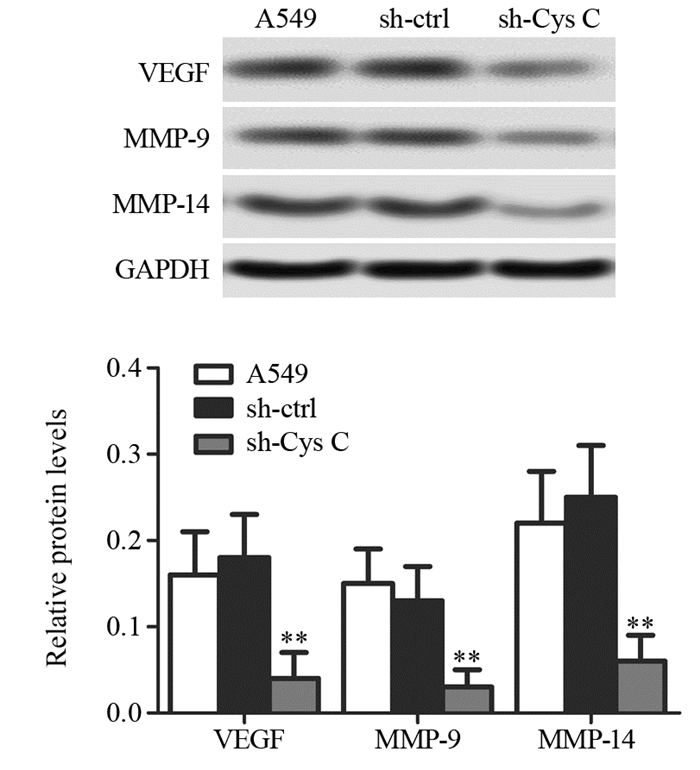
|
| **:P < 0.01, compared with A549 group 图 5 Cys C shRNA抑制A549细胞中VEGF、MMP-9和MMP-14的表达 Figure 5 Cys C shRNA downregulated expression of VEGF, MMP-9 and MMP-14 in A549 cells |
肺癌仍然是全球癌症死亡的头号杀手。2014年,估计有224 210例肺癌患者被确诊,其中大部分为非小细胞肺癌晚期[14]。因此寻找新的诊断、治疗靶点具有非常重要的意义。
在肿瘤微环境中,蛋白酶活性异常对促进肿瘤的进展、增殖、侵袭和转移具有重要作用。高水平的蛋白酶表达与乳腺癌、肺癌、头颈部、结直肠癌和许多其他癌症的预后不良有密切的关系[3-4, 10-11, 15]。Cys C能够抑制组织蛋白酶表达。研究发现,Cys C可能在肿瘤细胞中过度表达,导致循环水平升高[5]。Cys C被认为在细胞外腔内的蛋白质稳态中发挥作用,抑制由死亡细胞释放的蛋白酶引起的不适当的水解降解[16]。Cys C也可以从浸润的炎性细胞中释放出来,如巨噬细胞和嗜中性粒细胞[17]。与健康者相比,卵巢癌[15]和结直肠癌[10]患者血清中Cys C的浓度相对较高。Cys C与肿瘤转移和侵袭有关[18],其表达与结直肠癌患者死亡的高风险相关。Cys C通过灭活组织蛋白酶活性从而抑制肿瘤细胞浸润和转移[19-20]。半胱氨酸蛋白酶是Cys C的主要底物,半胱氨酸蛋白酶的活性在肿瘤转化的环境中经常出现紊乱[21]。上述研究表明,肿瘤通常伴随Cys C异常表达,Cys C可以调控组织蛋白酶活性进而调控肿瘤的侵袭和转移。基于Cys C对非小细胞肺癌发展的积极作用,本研究采用RNA干扰(RNAi)下调Cys C表达。RT-PCR和Western blot结果表明,shRNA-Cys C转染细胞株中Cys C显著下调,Cys C的转录水平和翻译水平表达都被抑制。说明本研究shRNA Cys C设计良好,干扰效率较高,将该细胞株用于后续实验能充分表明Cys C沉默对非小细胞癌细胞A549的影响。shRNA已成为成熟的RNA干扰技术,广泛应用于生物学和医学生物学的研究中。CCK-8检测细胞增殖结果表明,Cys C沉默明显抑制NSCLC细胞A549的增殖,与前人研究一致,即Cys C沉默一定程度抑制食管癌细胞EC9706增殖[3]。本文Transwell实验和划痕实验验证了Cys C沉默参与侵袭和转移抑制,与上述文献结果一致。其中,癌细胞的侵袭需要基质金属蛋白酶(MMPs)的激活,这些蛋白酶可以在局部降解构成基底膜和结缔组织的蛋白质,促进肿瘤细胞的转移。本研究表明,Cys C沉默可抑制MMPs和VEGF表达,从而抑制A549细胞转移。Cys C本身作为组织蛋白酶抑制剂,已经被证明通过调控组织蛋白酶活性进而调控肿瘤的侵袭和转移。本研究结果反面证明了Cys C的致癌作用,但其机制有待发掘。Cys C调控MMPs和VEGF表达可能是调控转移和侵袭的机制之一。
综上所述,Cys C下调可抑制A549细胞的增殖、迁移和侵袭,并下调MMPs和VEGF。本研究采用shRNA下调Cys C的表达,分析Cys C与NSCLC A549细胞的关系。结果提示,抑制Cys C表达可能抑制NSCLC细胞A549的发展。
| [1] | Siegel R, Naishadham D, Jemal A. Cancer statistics, 2013[J]. CA Cancer J Clin, 2013, 63(1): 11. DOI:10.3322/caac.v63.1 |
| [2] | Williams BA, Sugimura H, Endo C, et al. Predicting postrecurrence survival among completely resected nonsmall-cell lung cancer patients[J]. Ann Thorac Surg, 2006, 81(3): 1021–7. DOI:10.1016/j.athoracsur.2005.09.020 |
| [3] | Yan Y, Zhou K, Wang L, et al. Expression of cystatin C and its effect on EC9706 cells in esophageal carcinoma[J]. Int J Clin Exp Pathol, 2015, 8(9): 10102–11. |
| [4] | Yano M, Hirai K, Naito Z, et al. Expression of cathepsin B and cystatin C in human breast cancer[J]. Surg Today, 2001, 31(5): 385–9. DOI:10.1007/s005950170126 |
| [5] | Bodnar L, Stec R, Dzierzanowska M, et al. Cystatin C as a predictor factor in patients with renal cell carcinoma treated by everolimus[J]. Cancer Chemother Pharmacol, 2016, 78(2): 295–304. DOI:10.1007/s00280-016-3084-9 |
| [6] | Wegiel B, Jiborn T, Abrahamson M, et al. Cystatin C is downregulated in prostate cancer and modulates invasion of prostate cancer cells via MAPK/Erk and androgen receptor pathways[J]. PLoS One, 2009, 4(11): e7953. DOI:10.1371/journal.pone.0007953 |
| [7] | Nückel H, Langer C, Herget-Rosenthal S, et al. Prognostic significance of serum cystatin C in multiple myeloma[J]. Int J Hematol, 2012, 95(5): 545–50. DOI:10.1007/s12185-012-1049-2 |
| [8] | Mori J, Tanikawa C, Funauchi Y, et al. Cystatin C as a p53-inducible apoptotic mediator that regulates cathepsin L activity[J]. Cancer Sci, 2016, 107(3): 298–306. DOI:10.1111/cas.12881 |
| [9] | Bodnar L, Wcislo GB, Smoter M, et al. Cystatin C as a parameter of glomerular filtration rate in patients with ovarian cancer[J]. Kidney Blood Press Res, 2010, 33(5): 360–7. DOI:10.1159/000319097 |
| [10] | Zhang J, He P, Zhong Q, et al. Increasing Cystatin C and Cathepsin B in Serum of Colorectal Cancer Patients[J]. Clin Lab, 2017, 63(2): 365–71. |
| [11] | Naumnik W, Niklińska W, Ossolińska M, et al. Serum cathepsin K and cystatin C concentration in patients with advanced non-small-cell lung cancer during chemotherapy[J]. Folia Histochem Cytobiol, 2009, 47(2): 207–13. |
| [12] | Fischer SE. RNA Interference and MicroRNA-Mediated Silencing[J]. Curr Protoc Mol Biol, 2015, 112: 26. |
| [13] | Salazar N, Muñoz D, Hoy J, et al. Use of shRNA for stable suppression of chemokine receptor expression and function in human cancer cell lines[J]. Methods Mol Biol, 2014, 1172: 209–18. DOI:10.1007/978-1-4939-0928-5 |
| [14] | Howlader N, Mariotto AB, Woloshin S, et al. Providing clinicians and patients with actual prognosis: cancer in the context of competing causes of death[J]. J Natl Cancer Inst Monogr, 2014, 2014(49): 255–64. DOI:10.1093/jncimonographs/lgu022 |
| [15] | Zhang W, Wang S, Wang Q, et al. Overexpression of cysteine cathepsin L is a marker of invasion and metastasis in ovarian cancer[J]. Oncol Rep, 2014, 31(3): 1334–42. DOI:10.3892/or.2014.2967 |
| [16] | Strålberg F, Henning P, Gjertsson I, et al. Cysteine proteinase inhibitors regulate human and mouse osteoclastogenesis by interfering with RANK signaling[J]. FASEB J, 2013, 27(7): 2687–701. DOI:10.1096/fj.12-211748 |
| [17] | Decock J, Obermajer N, Vozelj S, et al. Cathepsin B, cathepsin H, cathepsin X and cystatin C in sera of patients with early-stage and inflammatory breast cancer[J]. Int J Biol Markers, 2008, 23(3): 161–8. DOI:10.1177/172460080802300305 |
| [18] | Kim JT, Lee SJ, Kang MA, et al. Cystatin SN neutralizes the inhibitory effect of cystatin C on cathepsin B activity[J]. Cell Death Dis, 2013, 4: e974. DOI:10.1038/cddis.2013.485 |
| [19] | Oc MA, Demir H, Cekmen MB, et al. Correlation of Cystatin-C and radionuclidic measurement method of glomerular filtration rate in patients with lung cancer receiving cisplatin treatment[J]. Ren Fail, 2014, 36(7): 1043–50. DOI:10.3109/0886022X.2014.918813 |
| [20] | Guo S, Xue Y, He Q, et al. Preoperative serum cystatin-C as a potential biomarker for prognosis of renal cell carcinoma[J]. PLoS One, 2017, 12(6): e0178823. DOI:10.1371/journal.pone.0178823 |
| [21] | Olson OC, Joyce JA. Cysteine cathepsin proteases: regulators of cancer progression and therapeutic response[J]. Nat Rev Cancer, 2015, 15(12): 712–29. DOI:10.1038/nrc4027 |
 2018, Vol. 45
2018, Vol. 45
