文章信息
- 长链非编码RNA在食管癌中的研究进展
- Research Progress of Long Non-coding RNA in Esophageal Cancer
- 肿瘤防治研究, 2018, 45(11): 932-935
- Cancer Research on Prevention and Treatment, 2018, 45(11): 932-935
- http://www.zlfzyj.com/CN/10.3971/j.issn.1000-8578.2018.18.0399
- 收稿日期: 2018-06-07
- 修回日期: 2018-08-20
食管癌(esophageal cancer, EC)居于全球癌症死亡的第六位, 也是全球排名第八的癌症类型。食管癌有两种主要类型:食管鳞状细胞癌(esophageal squamous cell carcinoma, ESCC)和食管腺癌(esophageal adenocarcinoma, EA)。中国以ESCC为主[1]。由于缺乏特异性症状和有效的早期诊断方法, ESCC确诊往往在疾病晚期[2]。
长链非编码RNA(long non-coding RNA, lncRNA)是一组包含超过200个核苷酸的RNA, 占据至少80%的人类基因组, 不具有蛋白质编码潜力[3]。越来越多的证据表明, lncRNA参与许多细胞过程, 包括增殖、转移、细胞周期进程、细胞生长和凋亡。在基因修饰过程中作为转录调节因子、转录后加工因子、染色质重塑因子和剪接调控因子[4]。lncRNA似乎也能促进或抑制癌症的发展, 在肿瘤的诊断和治疗中发挥作用[5]。微小RNA(microRNA, miRNA)是小的非编码RNA, 长约22个核苷酸。可与信使RNA(messenger RNA, mRNA)3UTR区互补结合, 抑制靶mRNA的翻译或降解[6]。
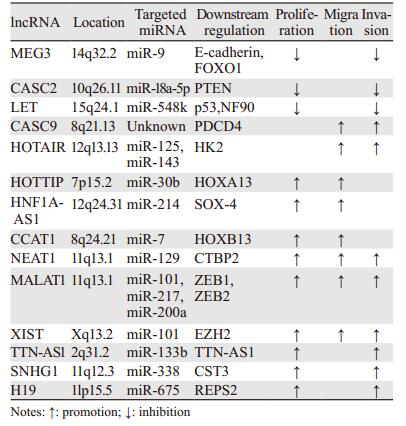
由于mRNA和lncRNA之间结构相似, lncRNAs也可以作为miRNA的靶点, 如肝细胞癌中miR-34a靶向lncRNA UFC1[7]。近几年, 越来越多的研究集中在lncRNA-miRNA-mRNA调控机制上。哈佛大学的研究人员在2011年提出竞争性内源RNA(competing endogenous RNA, ceRNA)假说。ceRNA假说指出, lncRNA或环形RNA(circular RNA, circRNA)通过竞争共享的miRNA来调节其他RNA转录物, 通常是编码蛋白质的转录物, 从而影响肿瘤发生和发展[8]。尽管lncRNA在肿瘤发展过程中受到了相当多的关注, 但其在ESCC发展中的异常表达和功能尚未完全阐明。近2年涉及食管癌的lncRNA及其调控机制, 尤其是lncRNA作为竞争性内源RNA(ceRNA)的调控机制研究见表 1。
母系表达基因3(maternally expressed gene 3, MEG3)在ESCC中的表达水平显著降低, 且MEG3的上调抑制EC细胞增殖和侵袭[9]。Dong等发现MEG3通过竞争性结合miR-9下调E-cadherin和FOXO1的表达, 抑制上皮间质转化(epithelial-mesenchymal transition, EMT), 从而抑制肿瘤转移[10]。
1.2 CASC2Zhang等发现在EC中癌症易感性候选基因2(cancer susceptibility candidate 2, CASC2)的表达显著下调, CASC2的上调显著抑制EC增殖以及侵袭。PTEN是位于10q23位点的重要肿瘤抑制基因。Zhang等表明, CASC2作为miR-18a-5p的ceRNA, 从而调节PTEN表达[11]。
1.3 LETNPTN内含子转录物1(NPTN intronic transcript 1, NPTN-IT1 or LET)的过度表达抑制了ESCC细胞的增殖和迁移。miR-548k直接靶向抑制LET, 进一步下调p53并上调NF90的表达[12], miR-548k-LET调节轴可能是ESCC有希望的预后生物标记和治疗靶标。
2 促进肿瘤细胞迁移和侵袭的lncRNA 2.1 CASC9Pan等发现癌症易感性候选基因9(cancer susceptibility candidate 9, CASC9)敲低显著抑制ESCC细胞迁移和侵袭。研究证明CASC9通过招募EZH2负性调控PDCD4表达, 从而改变H3K27me3的水平, 从而起到致癌基因的作用[13]。
2.2 HOTAIRHOX转录本反义RNA(HOX transcript antisense RNA, HOTAIR)上调促进ESCC转移, 提示预后不良。糖激酶2(hexokinase 2, HK2)是在肿瘤中过表达的主要同功酶, 现被提议作为癌症治疗的代谢靶点。Ma等发现HOTAIR通过竞争miR-125和miR-143来正向调节HK2的表达, 从而在ESCC中发挥致癌作用[14]。Xu等也发现HOTAIR作为miR-148a的ceRNA, 正向调控锌指转录因子2(Snail2)表达, 促进EC的EMT[15]。Ren等[8]证实HOTAIR作为miR-1的ceRNA上调细胞周期蛋白D1(cyclin D1, CCND1)的表达, 从而促进ESCC的发生。
基于循环中的lncRNAs已经被证实在血清中显著稳定, 并能在人类外周血中检测到的理论基础。Wang等首次证实血清HOTAIR可作为诊断ESCC的潜在生物标志物[16]。
3 促进肿瘤细胞增殖和迁移的lncRNA 3.1 CCAT1Zhang等发现结肠癌相关转录因子1(colon cancer associated transcript1, CCAT1)敲低显著抑制肿瘤细胞增殖和迁移[17]。研究发现CCAT1在细胞核和细胞质中可表现出不同的调控机制。SPRY4可以抑制肿瘤细胞增殖和迁移。CCAT1与SPRY4表达呈负相关。在细胞核中, CCAT1影响ESCC细胞的生长和迁移至少部分通过SPRY4的表观遗传修饰。在细胞质中, CCAT1通过竞争miR-7促进同源异形盒基因B13(homeobox B13, HOXB13)的表达, 从而促进细胞生长和迁移[17]。
3.2 HOTTIPChen等发现HOXA远端转录本反义RNA(homeobox A transcript at the distal tip, HOTTIP)在ESCC中上调[18]。实验证实, HOTTIP作为miR-30b的ceRNA上调同源盒基因A13(homeobox A13, HOXA13)的表达, 从而促进ESCC转移[19]。另外, HOTTIP在ESCC细胞的转录和转录后水平调节HOXA13。
3.3 HNF1A-AS12014年Yang等发现EA中的反义肝细胞核因子1(hepatocyte nuclear factor-antisense 1, HNF1A-AS1/HAS1)显著上调[20]。Wang等研究表明, HNF1A-AS1通过与miR-214竞争性结合, 上调SOX-4的表达, 促进食管鳞状细胞癌的生长和转移[21]。
4 促进肿瘤细胞增殖、迁移及侵袭的lncRNA 4.1 NEAT1Chen等报道核散斑组装转录物1(nuclear paraspeckle assembly transcript 1, NEAT1)表达增强可促进ESCC的增殖, 并且可以增强迁移和侵袭的能力[22]。CTBP2在多种癌细胞中起着致瘤过程的转录核心调节剂的作用。Li等提示NEAT1通过竞争性结合miR-129, 上调CTBP2, 促进细胞增殖和侵袭, 进而促进ESCC的发生[23]。
4.2 MALAT1Huang等指出EC中肺腺癌转移相关转录子1(metastasis associated lung adenocarcinoma transcript 1, MALAT1)的表达水平显著升高[24]。Li等发现miR-101和miR-217可以通过下调MALAT1的表达来抑制ESCC细胞[25]。Zhang等[26]指出MALAT1可能作为ceRNA, 竞争性结合miR-200a, 促进EMT进程, 导致EC的进展。
4.3 XIST据报道, X染色体非活性特异性转录物(X-inactive specific transcript, XIST)在多种癌症中呈高表达, 与癌症预后不良密切相关[27]。Wu等发现敲低XIST能抑制ESCC细胞的增殖、迁移和侵袭。Wu等还发现XIST作为miR-101的ceRNA上调EZH2的表达, 从而促进ESCC的进展[28]。
5 促进肿瘤细胞增殖和侵袭的lncRNA 5.1 TTN-AS1Lin等证明高水平的TTN反义RNA 1(TTN antisense RNA 1, TTN-AS1)促进细胞增殖和侵袭。研究表明TTN-AS1通过与miR-133b竞争性结合上调锌指转录因子1(Snail1)及FSCN1, 导致ESCC转移侵袭[29]。
5.2 SNHG1Zhang等发现ESCC组织中小核仁RNA宿主基因1(small nucleolar RNA host gene 1, SNHG1)表达显着高于癌旁组织。实验证明通过调控Notch信号通路, SNHG1敲低能抑制细胞增殖、侵袭能力及EMT[30]。Yan等证实SNHG1通过竞争性结合miR-338, 促进原癌基因胱抑素C(cystatin C, CysC)在EC中的表达[31]。
5.3 H19H19过表达可以促进细胞增殖、侵袭并诱导EMT。H19已被证明是人和小鼠中miR-675的主要miRNA前体[32]。H19在ESCC组织中过表达, Zhou等证实, REPS2是miR-675-5p的直接靶基因, miR-675-5p通过抑制REPS2促进RalBP1/RAC1/CDC42信号通路可能是ESCC发生的重要机制之一[33]。
6 应用前景编码RNA在转录翻译过程中会受到各种调控, 不能准确反映蛋白质的表达水平。而IncRNA所受调控较少, 相对准确, 并且具有更高的癌症相关度, 可以作为更有效的诊断工具。
6.1 作为诊断标志物肿瘤生物标志物, 应该具备能够简单快速检测以及与肿瘤紧密相关的特点。已证实, lncRNA在体液中具有相对稳定二级结构, 因此可以在人血浆或尿液中检测出。美国食品和药物管理局已经批准了尿液前列腺癌基因3(prostate cancer antigen 3, PCA3)用于临床检测, 代替之前的前列腺特异性抗原分析, 快速方便地诊断前列腺癌[34]。基于此, Tong和他的同事发现血浆中POU3F3对ESCC诊断的可靠性, 有助于提高早期ESCC筛查的有效率[35]。Hu等也同样发现血浆中的Linc00152、CFLAR-AS1和POU3F3可以被用作ESCC的新型快速早期诊断生物标志物[36]。因此, lncRNA极有可能成为ESCC早期诊断的肿瘤标志物。
6.2 作为预后标志物由于缺乏特异性症状和有效的早期诊断方法, EC确诊往往在疾病晚期, 食管癌在我国的高发生率, 使预后情况也成为一个不可忽视的问题。如Huang已证实, MALAT1表达高的患者总生存期较短, 表明MALAT1表达上调预示食管癌患者预后不良[24]。根据lncRNA表达水平, 用来有效预测肿瘤的增殖、迁移及侵袭情况, 为EC的诊治提供更为便捷、有效的方法。
6.3 作为治疗靶标通常新药研发的靶标为具有调节功能的蛋白或蛋白复合体, 或者药物本身即为蛋白质。RNA作为靶标进行药物开发的研究现在正逐渐增加, lncRNA会成为人们抵御疾病的有力武器之一。lncRNA表达量及功能的调控源于许多机制, lncRNA的表达及其信号通路的异常均可诱发ESCC, 近年来, lncRNA作为ceRNA来调控ESCC的进展成为研究热点, lncRNA可成为潜在的癌基因或者抑癌基因参与ESCC的生物学进程。面对不同功能的lncRNA, 许多实验已经证实过表达或者反义RNA技术纠正异常的lncRNA的表达量, 可影响ESCC细胞的增殖、迁移及侵袭。研究或开发针对这些致癌性lncRNA的沉默或调控技术将会对ESCC的治疗带来极大的影响。当然, lncRNA作为ceRNA存在许多信号通路, 对其深入探索调节或者逆转其通路, 极有潜力作为ESCC治疗的靶标。
许多lncRNA在ESCC中均有异常表达。随着对lncRNA的深入研究, 许多学者发现mRNA的表达与lncRNA的表达密切相关, mRNA的表达可能受lncRNA调控。ESCC组织中lncRNA和mRNA之间是否存在基因共表达网络值得进一步探索。
lncRNA的发现使人们对于染色体上的非编码区域有了新的认识, 也是对RNA功能的补充。当然, lncRNA并非只调控一种类型癌症, lncRNA如HOTAIR、CCAT1等至少在3种消化系统癌症(包括ESCC)中受到调节, 目前发生机制尚不明确。本文对近2年有关食管癌lncRNA的研究进展作了简单介绍, 随着研究的深入, 相信lncRNA的其他功能也会相继被挖掘。然而, 与ESCC相关的lncRNA的累积数量相比, 只有很少的lncRNA得到了充分的研究。ESCC的发生机制尚未研究透彻, 随着lncRNA在肿瘤中作用机制的深入研究, 其必将成为ESCC早期诊断、预后评估的分子标志物和ESCC治疗靶点。
| [1] | Chen W, Zheng R, Baade PD, et al. Cancer statistics in China, 2015[J]. CA Cancer J Clin, 2016, 66(2): 115–32. DOI:10.3322/caac.21338 |
| [2] | Ohashi S, Miyamoto S, Kikuchi O, et al. Recent Advances From Basic and Clinical Studies of Esophageal Squamous Cell Carcinoma[J]. Gastroenterology, 2015, 149(7): 1700–15. DOI:10.1053/j.gastro.2015.08.054 |
| [3] | Fatica A, Bozzoni I. Long non-coding RNAs:new players in cell differentiation and development[J]. Nat Rev Genet, 2014, 15(1): 7–21. |
| [4] | Zou Y, Li J, Chen Y, et al. BANCR:a novel oncogenic long non-coding RNA in human cancers[J]. Oncotarget, 2017, 8(55): 94997–5004. |
| [5] | Evans JR, Feng FY, Chinnaiyan AM. The bright side of dark matter:lncRNAs in cancer[J]. J Clin Invest, 2016, 126(8): 2775–82. DOI:10.1172/JCI84421 |
| [6] | Goodall EF, Heath PR, Bandmann O, et al. Neuronal dark matter:the emerging role of microRNAs in neurodegeneration[J]. Front Cell Neurosci, 2013, 7(40): 178–93. |
| [7] | Cao C, Sun J, Zhang D, et al. The Long Intergenic Noncoding RNA UFC1, a Target of MicroRNA 34a, Interacts with the mRNA Stabilizing Protein HuR to Increase Levels of β-Catenin in HCC Cells[J]. Gastroenterology, 2015, 148(2): 415–26. DOI:10.1053/j.gastro.2014.10.012 |
| [8] | Ren K, Li Y, Lu H, et al. Long Noncoding RNA HOTAIR Controls Cell Cycle by Functioning as a Competing Endogenous RNA in Esophageal Squamous Cell Carcinoma[J]. Transl Oncol, 2016, 9(6): 489–97. DOI:10.1016/j.tranon.2016.09.005 |
| [9] | Lv D, Sun R, Yu Q, et al. The long non-coding RNA maternally expressed gene 3 activates p53 and is downregulated in esophageal squamous cell cancer[J]. Tumor Biol, 2016, 37(12): 16259–67. DOI:10.1007/s13277-016-5426-y |
| [10] | Dong Z, Zhang A, Liu S, et al. Aberrant Methylation-Mediated Silencing of lncRNA MEG3 Functions as a ceRNA in Esophageal Cancer[J]. Mol Cancer Res, 2017, 15(7): 800–10. DOI:10.1158/1541-7786.MCR-16-0385 |
| [11] | Zhang W, He W, Gao J, et al. The long noncoding RNA CASC2 inhibits tumorigenesis through modulating the expression of PTEN by targeting miR-18a-5p in esophageal carcinoma[J]. Exp Cell Res, 2017, 361(1): 30–8. DOI:10.1016/j.yexcr.2017.09.037 |
| [12] | Chen Z, Lin J, Wu S, et al. Up-regulated miR-548k promotes esophageal squamous cell carcinoma progression via targeting long noncoding RNA-LET[J]. Exp Cell Res, 2018, 362(1): 90–101. DOI:10.1016/j.yexcr.2017.11.006 |
| [13] | Pan Z, Mao W, Bao Y, et al. The long noncoding RNA CASC9 regulates migration and invasion in esophageal cancer[J]. Cancer Med, 2016, 5(9): 2442–7. DOI:10.1002/cam4.2016.5.issue-9 |
| [14] | Ma J, Fan Y, Feng T, et al. HOTAIR regulates HK2 expression by binding endogenous miR-125 and miR-143 in oesophageal squamous cell carcinoma progression[J]. Oncotarget, 2017, 8(49): 86410–22. |
| [15] | Xu F, Zhang J. Long non-coding RNA HOTAIR functions as miRNA sponge to promote the epithelial to mesenchymal transition in esophageal cancer[J]. Biomed Pharmacother, 2017, 90: 888–96. DOI:10.1016/j.biopha.2017.03.103 |
| [16] | Wang W, He X, Zheng Z, et al. Serum HOTAIR as a novel diagnostic biomarker for esophageal squamous cell carcinoma[J]. Mol Cancer, 2017, 16(1): 75–9. DOI:10.1186/s12943-017-0643-6 |
| [17] | Zhang E, Han L, Yin D, et al. H3K27 acetylation activated-long non-coding RNA CCAT1 affects cell proliferation and migration by regulating SPRY4 and HOXB13 expression in esophageal squamous cell carcinoma[J]. Nucleic Acids Res, 2017, 45(6): 3086–101. DOI:10.1093/nar/gkw1247 |
| [18] | Chen X, Han H, Li Y, et al. Upregulation of long noncoding RNA HOTTIP promotes metastasis of esophageal squamous cell carcinoma via induction of EMT[J]. Oncotarget, 2016, 7(51): 84480–5. |
| [19] | Lin C, Wang Y, Wang Y, et al. Transcriptional and posttranscriptional regulation of HOXA13 by lncRNA HOTTIP facilitates tumorigenesis and metastasis in esophageal squamous carcinoma cells[J]. Oncogene, 2017, 36(38): 5392–406. DOI:10.1038/onc.2017.133 |
| [20] | Yang X, Song JH, Cheng Y, et al. Long non-coding RNA HNF1A-AS1 regulates proliferation and migration in oesophageal adenocarcinoma cells[J]. Gut, 2014, 63(6): 881–90. DOI:10.1136/gutjnl-2013-305266 |
| [21] | Wang G, Zhao W, Gao X, et al. HNF1A-AS1 promotes growth and metastasis of esophageal squamous cell carcinoma by sponging miR-214 to upregulate the expression of SOX-4[J]. Inter J Oncol, 2017, 51(2): 657. DOI:10.3892/ijo.2017.4034 |
| [22] | Chen X, Kong J, Ma Z, et al. Up regulation of the long non-coding RNA NEAT1 promotes esophageal squamous cell carcinoma cell progression and correlates with poor prognosis[J]. Am J Cancer Res, 2015, 5(9): 2808–15. |
| [23] | Li Y, Chen D, Gao X, et al. LncRNA NEAT1 Regulates Cell Viability and Invasion in Esophageal Squamous Cell Carcinoma through the miR-129/CTBP2 Axis[J]. Dis Markers, 2017, 2017(9): 5314649–59. |
| [24] | Huang C, Yu Z, Yang H, et al. Increased MALAT1 expression predicts poor prognosis in esophageal cancer patients[J]. Biomed Pharmacother, 2016, 83: 8–13. DOI:10.1016/j.biopha.2016.05.044 |
| [25] | Li RQ, Ren Y, Liu W, et al. MicroRNA-mediated silence of onco-lncRNA MALAT1 in different ESCC cells via ligand-functionalized hydroxyl-rich nanovectors[J]. Nanoscale, 2017, 9(7): 2521–30. DOI:10.1039/C6NR09668A |
| [26] | 张清琴, 崔艳慧, 王颖, 等. 长链非编码RNA—肺腺癌转移相关转录子1调控食管癌EC-109细胞侵袭转移的机制[J]. 中华肿瘤杂志, 2017, 39(6): 405–11. [ Zhang QQ, Cui YH, Wang Y, et al. Mechanism of long non-coding RNA-metastasis associated lung adenocarcinoma transcript 1 induced invasion and metastasis of esophageal cancer cell EC-109[J]. Zhonghua Zhong Liu Za Zhi, 2017, 39(6): 405–11. DOI:10.3760/cma.j.issn.0253-3766.2017.06.002 ] |
| [27] | Schouten PC, Vollebergh MA, Opdam M, et al. High XIST and Low 53BP1 Expression Predict Poor Outcome after High-Dose Alkylating Chemotherapy in Patients with a BRCA1-like Breast Cancer[J]. Mol Cancer Ther, 2016, 15(1): 190–8. DOI:10.1158/1535-7163.MCT-15-0470 |
| [28] | Wu X, Dinglin X, Wang X, et al. Long noncoding RNA XIST promotes malignancies of esophageal squamous cell carcinoma via regulation of miR-101/EZH2[J]. Oncotarget, 2017, 8(44): 76015–28. |
| [29] | Lin C, Zhang S, Wang Y, et al. Functional Role of a Novel Long Noncoding RNATTN-AS1 in Esophageal Squamous Cell Carcinoma Progression and Metastasis[J]. Clin Cancer Res, 2018, 24(2): 486–98. DOI:10.1158/1078-0432.CCR-17-1851 |
| [30] | Zhang Y, Jin X, Wang Z, et al. Downregulation of SNHG1 suppresses cell proliferation and invasion by regulating Notch signaling pathway in esophageal squamous cell cancer[J]. Cancer Biomarker, 2017, 21(1): 89–96. DOI:10.3233/CBM-170286 |
| [31] | Yan Y, Fan Q, Wang L, et al. LncRNA Snhg1, a non-degradable sponge for miR-338, promotes expression of proto-oncogene CST3 in primary esophageal cancer cells[J]. Oncotarget, 2017, 8(22): 35750–60. |
| [32] | Cai X, Cullen BR. The imprinted H19 noncoding RNA is a primary microRNA precursor[J]. RNA, 2007, 13(3): 313–6. DOI:10.1261/rna.351707 |
| [33] | Zhou YW, Zhang H, Duan CJ, et al. miR-675-5p enhances tumorigenesis and metastasis of esophageal squamous cell carcinoma by targeting REPS2[J]. Oncotarget, 2016, 7(21): 30730–47. |
| [34] | Walsh AL, Tuzova AV, Bolton EM, et al. Long noncoding RNAs and prostate carcinogenesis:the missing 'linc'?[J]. Trends Mol Med, 2014, 20(8): 428–36. DOI:10.1016/j.molmed.2014.03.005 |
| [35] | Tong YS, Wang XW, Zhou XL, et al. Identification of the long non-coding RNA POU3F3 in plasma as a novel biomarker for diagnosis of esophageal squamous cell carcinoma[J]. Mol Cancer, 2015, 14(1): 3–15. DOI:10.1186/1476-4598-14-3 |
| [36] | Hu HB, Jie HY, Zheng XX. Three Circulating LncRNA Predict Early Progress of Esophageal Squamous Cell Carcinoma[J]. Cell Physiol Biochem, 2016, 40(1-2): 117–25. DOI:10.1159/000452529 |
 2018, Vol. 45
2018, Vol. 45