文章信息
- 脑脊液MGMT启动子甲基化在胶质瘤诊断中的应用
- Application of MGMT Promoter Methylation of Cerebrospinal Fluid in Diagnosis of Glioma
- 肿瘤防治研究, 2018, 45(10): 796-799
- Cancer Research on Prevention and Treatment, 2018, 45(10): 796-799
- http://www.zlfzyj.com/CN/10.3971/j.issn.1000-8578.2018.18.0126
- 收稿日期: 2018-01-26
- 修回日期: 2018-05-17
胶质瘤诊断通过术前影像和术后显微手术切除或者立体定向活检获得病理组织的方式进行[1-3]。但是病理特征的不确定性、磁共振成像局限性、立体活检高风险性使得早期诊断很难实施[4],因此寻找一种无创,且具有较高敏感度和特异性的生物标志物是实现胶质瘤早期诊断方向之一。
当前,利用基因多态性与肿瘤特有相关性[5],多种分子标志物,作为一种无创、高效的肿瘤检测方式,已经应用于肿瘤的诊断或治疗[6-8]。因此,血液衍生生物标志物作为微创标志物,将在临床支持诊断和监测肿瘤发展和治疗反应上起到非常关键的作用[4]。
Balanae和Lavon等研究证实胶质瘤肿瘤患者外周血中游离DNA MGMT基因(O6-甲基鸟嘌呤-DNA-甲基转移酶)启动子区甲基化与瘤体组织高度统一,这为MGMT启动子甲基化成为胶质瘤早期检测提供了可能[9-10]。有研究[11]显示,MGMT基因启动子甲基化与生存率呈正相关,提示可以用来评估预后。
考虑到血脑屏障存在,脑脊液中肿瘤循环DNA含量高于外周血,本研究利用甲基化特异PCR(methylation-specific PCR, MSP)技术,比较肿瘤组织、脑脊液和外周血中MGMT启动子甲基化的特异性和敏感度,探讨脑脊液中MGMT启动子甲基化在胶质瘤临床诊断中的敏感度和特异性,为分子无创技术提供更加坚实的理论依据。
1 资料与方法 1.1 研究对象采集武汉大学人民医院神经外科于2016年1月—2017年1月收治的283例胶质瘤患者的瘤体组织,放疗前采集患者外周血及脑脊液标本。所有与患者肿瘤标本配对的外周血及脑脊液标本均于术后到接受放疗之前的时间段内采集,采集后立即处理并冻存。与组织标本配对的脑脊液采集全部通过腰穿完成,并且要求于采集脑脊液标本的当日采集相应配对外周血标本。男148例、女135例,年龄(51.54±6.91)岁。依照世界卫生组织神经系统肿瘤分级标准,对胶质瘤患者组织标本进行分级,其中WHOⅡ级91例,WHOⅢ级102例,WHO Ⅳ级90例。
阴性对照组采集于同期10例因颅脑外伤行去骨瓣减压术患者其无法还纳的挫伤坏死脑组织(且收集后确认患者无肿瘤病史和家族史),及其对应外周血和脑脊液。本研究经过本院伦理委员会批准后实施,已签署相关知情同意书。
胶质瘤及正常脑组织样本离体后一部分送往病理科确诊,剩余部分立即放入液氮罐中送往-80℃冰箱保存,外周血及脑脊液被抽取后提取血清亦置于-80℃冰箱保存。
1.2 检测方法 1.2.1 病例组与对照组组织、外周血和脑脊液中DNA的提取切取10 mg的组织标本,碎片后放入冰水浴中预冷,研磨成匀浆。取血清标本200 μl;按AxyPrep基因组DNA试剂盒说明,分别对组织、血清和脑脊液三个标本的DNA进行提取,并采用紫外分光光度计分析提取的DNA含量和纯度。
1.2.2 样本的亚硫酸氢钠处理DNA样品20 μl,总量约500 ng,用DNA重亚硫酸盐转化试剂盒(Qiagen公司)对各样本DNA进行亚硫酸盐修饰。
1.2.3 亚硫酸氢钠处理后的样本DNA PCR扩增引物由上海Invitrogen公司设计,甲基化(正义):5'-GCACTCTTCCGAAAACGAAACG-3';甲基化(反义):5'-TTTCGACGTTCGTAGGTTTTTCGC-3'。病例组和对照组电泳图见图 1。
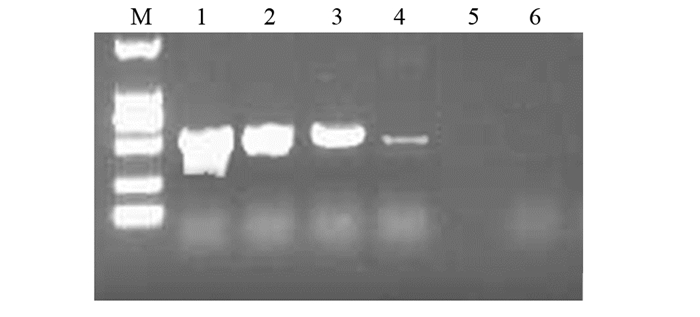
|
| 1-3: brain tissues of glioma patients and corresponding blood plasma, cerebrospinal fluid; 4-6: brain tissues of negative subjects and corresponding blood plasma, cerebrospinal fluid 图 1 MGMT基因目的片段PCR扩增产物电泳图 Figure 1 Electrophoretic map of PCR amplification products of MGMT gene target fragment |
采用SPSS16软件进行统计学分析,组间比较采用χ2检验或校正χ2检验,P < 0.05为差异有统计学意义。
2 结果 2.1 不同级别肿瘤组织MGMT启动甲基化阳性率283例患者中,总计有190例(67.14 %)患者肿瘤脑组织MGMT启动子甲基化。进一步比较不同等级胶质瘤石蜡标本中MGMT启动甲基化状态,结果显示,不同等级胶质瘤之间差异无统计学意义(P > 0.05)。卡方检验结果显示术前患者肿瘤组织,外周血和脑脊液中MGMT启动甲基化检测与肿瘤分级无关,见表 1。
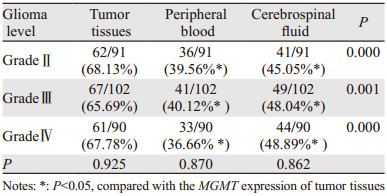
|
283例肿瘤患者的脑脊液及外周血标本中,有187例(66.08%)脑脊液及127例(44.87%)外周血标本中检测出MGMT启动子甲基化。所有阴性对照(脑外伤患者)的挫伤组织及其术前血浆或者脑脊液中均未检出MGMT启动子甲基化,病例组与对照组之间差异有统计学意义(P < 0.05),见表 1。
2.3 脑脊液和外周血甲基化状态敏感度和特异性比较对187例石蜡阳性标本用MGMT启动子甲基化的方法进行检测。经计算后得出全组患者脑脊液及外周血MGMT启动子甲基化检测的敏感度分别为79.66%和58.82%,两者之间差异有统计学(P=0.000),阴性预测值分别为46.2%和61.2%,(P=0.000)。同时,经分析石蜡标本中MGMT启动子甲基化检测为阴性的患者其对应的脑脊液和血清样本都没有出现阳性,并且健康对照组的脑脊液和外周血的标本MGMT启动子检测结果都为阴性,特异性为100%。这一结果进一步证明了MGMT启动子甲基化检测的特异性和敏感度,且脑脊液的敏感度高于外周血。
3 讨论目前已经有研究证实肿瘤患者循环DNA可以检测到与原发肿瘤一致的一些分子遗传学病变[12-13],这一技术已经成功应用于多种肿瘤(乳腺癌、前列腺癌、肝癌、肺癌)的诊断与治疗当中[6, 14-15]。但迄今为止在神经系统肿瘤领域,相关报道并不多。由于MGMT在DNA修复过程中所扮演的重要角色,因此已有大量的研究探寻MGMT在正常和肿瘤组织中的表达情况以及二者之间的关系,大胆推测MGMT表达的缺失可能是一些肿瘤的早期遗传。Balanae等[9]首次指出GBM患者肿瘤组织与血清中游离DNA MGMT基因启动子区甲基化检测结果具有显著的一致性,并且同样可以作为化疗敏感度的预测指标。近年来Lavon等[10]应用甲基化免疫沉淀方法成功针对70例不同级别胶质瘤患者的肿瘤及配对外周血标本进行了MGMT及其他肿瘤相关基因的启动子甲基化检测。最终结果同样显示外周血中循环DNA具有与原发肿瘤高度一致的遗传学改变。虽然目前已经成功的对血清中游离DNA MGMT启动子甲基化状态进行了检测,并且也显示出了与肿瘤标本高度的一致性,但是不难看出几乎所有针对神经系统肿瘤的研究结果其外周血循环DNA MGMT启动子甲基化检测敏感度均仅在50%左右。这与本研究结果也不谋而合。本研究通过分析脑脊液和外周血的MGMT启动子甲基化率,为更加准确的检测胶质瘤提供一个途径。
由于一些显著的肿瘤特征,大多数晚期胶质瘤可通过照影技术进行有效诊断和检测[16],但该技术在胶质瘤的早期诊断收效甚微。最近研究者证实血液中肿瘤细胞衍生物可以作为晚期肿瘤诊断物[5]。MGMT作为一种DNA修复酶,其基因启动子被认为可以独立预测胶质瘤对替莫唑胺(temozolomide, TMZ)治疗敏感度[17]。而Balanae和Lavon研究发现MGMT启动子甲基化存在于在胶质瘤的各个阶段[9-10],本研究进一步证实MGMT启动子甲基化在胶质瘤早期诊断中的作用,同时第一次分析比较患者脑脊液和外周血在MGMT启动子甲基化检测中的特异性。
MSP实验结果显示在肿瘤和脑挫伤组织中MGMT启动子甲基化率分别为67.14%(190/283)、0(0/10),再次证明MGMT启动子甲基化作为生物标志物的可行性。同时,单因素分析揭示不同级别胶质瘤,无论是瘤体组织,脑脊液还是外周血,其对应的MGMT启动子甲基化敏感度没有显著性差异,说明MGMT启动子甲基化检测与胶质瘤的级别没有一定的相关性,MGMT启动子检测可以应用在不同级别胶质瘤的初级诊断。
本研究比较胶质瘤患者外周血和脑脊液MGMT启动子甲基化阳性率:在胶质瘤患者脑组织甲基化阳性的研究对象中,外周血和脑脊液中MGMT启动子甲基化阳性率分别为58.82%和79.66%,两者差异有统计学意义(P < 0.05)。脑脊液中的MGMT启动子甲基化明显增加主要原因可能是与神经系统的特殊生理结构即血脑屏障的存在有关。血脑屏障导致肿瘤相关循环DNA进入到外周血的过程相对困难,而且肝肾对于循环DNA的清除作用也导致脑脊液循环与血循环不同[18],所以理论上对于神经系统肿瘤患者来说其脑脊液所包含的循环DNA数量理应高于外周血。脑脊液MGMT启动子甲基化检测敏感度显著高于外周血,同时也显示出了脑脊液标本作为神经系统肿瘤循环DNA检测对象的良好应用前景。同时为无法通过手术或活检取得组织标本的胶质瘤患者提供了变通的方案。
综上所述,本研究再次肯定了MGMT启动子甲基化有助于不同级别胶质瘤中的初级检测。相对于外周血,脑脊液中的MGMT启动子甲基化检测具有更好的特异性和敏感度。本研究结果为进一步探明MGMT启动子甲基化作用提供了新的实验依据,并为后续探讨针对胶质瘤初级检测的应用和开发奠定了基础。
| [1] | Skogen K, Schulz A, Dormagen JB, et al. Diagnostic performance of texture analysis on MRI in grading cerebral gliomas[J]. Eur J Radiol, 2016, 85(4): 824–9. DOI:10.1016/j.ejrad.2016.01.013 |
| [2] | Shioyama T, Muragaki Y, Maruyama T, et al. Intraoperative flow cytometry analysis of glioma tissue for rapid determination of tumor presence and its histopathological grade: clinical article[J]. J Neurosurg, 2013, 118(6): 1232–8. DOI:10.3171/2013.1.JNS12681 |
| [3] | Yang Y, He MZ, Li T, et al. MRI combined with PET-CT of different tracers to improve the accuracy of glioma diagnosis: a systematic review and meta-analysis[J]. Neurosurg Rev, 2017. [Epub ahead of print] |
| [4] | Keles GE, Lamborn KR, Berger MS. Low-grade hemispheric gliomas in adults: a critical review of extent of resection as a factor influencing outcome[J]. J Neurosurg, 2001, 95(5): 735–45. DOI:10.3171/jns.2001.95.5.0735 |
| [5] | Cui Y, Li G, Yan M, et al. The effects of gene polymorphisms on gliomas prognosis[J]. J Gene Med, 2017, 19(11): 345–52. |
| [6] | Schröck A, Leisse A, de Vos L, et al. Free-Circulating Methylated DNA in Blood for Diagnosis, Staging, Prognosis, and Monitoring of Head and Neck Squamous Cell Carcinoma Patients: An Observational Prospective Cohort Study[J]. Clin Chem, 2017, 63(7): 1288–96. DOI:10.1373/clinchem.2016.270207 |
| [7] | He WS, Bishop KS. The potential use of cell-free-circulating-tumor DNA as a biomarker for prostate cancer[J]. Expert Rev Mol Diagn, 2016, 16(8): 839–52. DOI:10.1080/14737159.2016.1197121 |
| [8] | 李赛, 左建宏. MicroRNA在肺癌早期诊断及预后中的应用探讨[J]. 世界最新医学信息文摘, 2017, 17(2): 17–8. [ Li S, Zuo JH. Application discussion of microRNA in early diagnosis and prognosis of lung cancer[J]. Shi Jie Zui Xin Yi Xue Xin Xi Wen Zhai, 2017, 17(2): 17–8. ] |
| [9] | Balana C, Ramirez JL, Taron M, et al. O6-methyl-guanine-DNA methyltransferase methylation in serum and tumor DNA predicts response to 1, 3-bis(2-chloroethyl)-1-nitrosourea but not to temozolamide plus cisplatin in glioblastoma multiforme[J]. Clin Cancer Res, 2003, 9(4): 1461–8. |
| [10] | Lavon I, Refael M, Zelikovitch B, et al. Serum DNA can define tumor-specific genetic and epigenetic markers in gliomas of various grades[J]. Neuro Oncol, 2010, 12(2): 173–80. DOI:10.1093/neuonc/nop041 |
| [11] | Schwaederle M, Husain H, Fanta PT. Detection rate of actionable mutations in diverse cancers using a biopsy-free (blood) circulating tumor cell DNA assay[J]. Oncotarget, 2016, 7(9): 9707–17. |
| [12] | Diehl F, Schmidt K, Choti MA, et al. Circulating mutant DNA to assess tumor dynamics[J]. Nat Med, 2008, 14(9): 985–90. DOI:10.1038/nm.1789 |
| [13] | Majure M, Logan AC. What the blood knows: interrogating circulating tumor DNA to predict progression of minimal residual disease in early breast cancer[J]. Ann Transl Med, 2016, 4(24): 543. DOI:10.21037/atm |
| [14] | Yao Y, Liu J, Li L, et al. Detection of circulating tumor DNA in patients with advanced non-small cell lung cancer[J]. Oncotarget, 2017, 8(2): 2130–40. |
| [15] | Usadel H, Brabender J, Danenberg KD, et al. Quantitative adenomatous polyposis coli promoter methylation analysis in tumor tissue, serum, and plasma DNA of patients with lung cancer[J]. Cancer Res, 2002, 62(2): 371–5. |
| [16] | Lan F, Yu H, Hu M, et al. miR-144-3p exerts anti-tumor effects in glioblastoma by targeting c-Met[J]. J Neurochem, 2015, 135(2): 274–86. DOI:10.1111/jnc.13272 |
| [17] | Qiu ZK, Shen D, Chen YS, et al. Enhanced MGMT expression contributes to temozolomide resistance in glioma stem-like cells[J]. Chin J Cancer, 2014, 33(2): 115–22. DOI:10.5732/cjc.012.10236 |
| [18] | Hisazumi J, Kobayashi N, Nishikawa M, et al. Significant role of liver sinusoidal endothelial cells in hepatic uptake and degradation of naked plasmid DNA after intravenous injection[J]. Pharm Res, 2004, 21(7): 1223–8. DOI:10.1023/B:PHAM.0000033009.17594.e5 |
 2018, Vol. 45
2018, Vol. 45


