文章信息
- 肿瘤干细胞蛋白质组学研究方法的新进展
- New Advances in Proteomics Research Methods of Cancer Stem Cells
- 肿瘤防治研究, 2018, 45(3): 175-178
- Cancer Research on Prevention and Treatment, 2018, 45(3): 175-178
- http://www.zlfzyj.com/CN/10.3971/j.issn.1000-8578.2018.17.0970
- 收稿日期: 2017-08-09
- 修回日期: 2017-11-26
抗癌治疗后发生的局部和远处肿瘤复发,是因为某些肿瘤干细胞(cancer stem cell, CSC)在传统治疗方法的细胞毒性作用后仍能存活,所以肿瘤干细胞被认为是最具治疗抗性的细胞。新近研究认为这些细胞的详细分子分析可以为我们提供新的生物标志物与治疗靶。同时研究希望能找到一种有效杀伤肿瘤干细胞的药物,或开发一种对抗CSC活性的新化合物,但至今未得满意效果,所以深入研究CSC分子谱将为未来抗CSC药物的发展提供必要的支持。
以往CSC研究采用核酸(RNA、DNA)分析来定义恶性肿瘤中的分子干扰,由于mRNA表达中的差异只能反映40%蛋白质表达变化[1],而且,核酸数据并不能说明蛋白质之间的相互作用以及它们在细胞内通路的相互作用,这是因为mRNA水平并不总是对应编码蛋白的功能活性。而蛋白质组学的方法可以提供有关CSC中蛋白质表达和活性变化引起通路失调的更深入的研究[2-3]。所以,确认CSC特异性表达的蛋白质[4]有助于阐明CSC增殖[5]、分化[6]、迁移和侵袭机制以及CSC在肿瘤传统治疗方法中不敏感和肿瘤复发的原因。
1 蛋白质组学方法分离CSC由于恶性肿瘤中仅包含少量CSC,而蛋白质谱需要成千上万CSC亚群的富集,所以这是蛋白质组学方法的一个关键问题。通常采用以下几种方法从各种来源中富集和分离CSC:荧光活化细胞分类术(fluorescence-activated cell sorting, FACS)、磁激活细胞分类术(magnetic activated cell sorting, MACS)、激光细胞捕获显微切割以及三维球/类器官细胞培养。
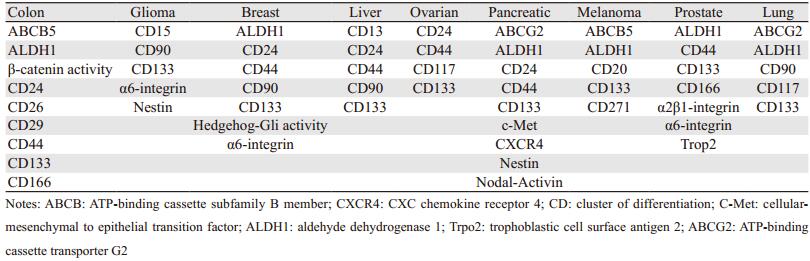
从体外培养的肿瘤组织中分离CSC,也可采用FACS、MACS细胞分类术,激光细胞捕获显微切割和三维球/类器官细胞培养从异种移植物或人肿瘤样本中富集和分离CSC。如分化群44、24、133(CD44、CD24、CD133)、上皮细胞黏附分子(EpCAM),醛脱氢酶-1(ALDH1)等[7]。这些基于抗体的FACS法和MACS法可以在无限增殖化癌细胞系、原代细胞系、异种移植物和患者肿瘤组织中简单区分出带CSC特征的纯癌细胞亚群。也可根据通常所采用的细胞系或小鼠肿瘤的某些标志物来确定肿瘤中的CSC,见表 1。但仍需要在原发性人肿瘤样品中的严格验证。因为这些类型的CSC分类中最大的局限在于:(1)目前仍未发现明确的CSC表面标志物,(2)在基于抗体的染色之后需要耗费大量时间来将肿瘤组织处理成单个细胞。这些问题都明显影响蛋白质组结果。新近研究出两种方法,即激光细胞捕获显微切割法和球形细胞培养法。与基于FACS或MACS的CSC分离相比,激光捕获显微切割法具有一个显著优势,即在处理具有特定特征的细胞时只需两步:一是对抗体染色冷冻组织或石蜡包埋肿瘤组织进行显微切割后采集CSC;二是为了区分CSC与非CSC,与已知CSC标志物抗体的细胞染色比较,可进行更为精确的激光捕获显微切割。也可以从无限增殖化癌细胞系、原发性癌细胞系、异种移植物或患者肿瘤组织中生成球形细胞培养物。这种培养物中包含大量CSC,在裸鼠中呈高度致瘤性,还能抵抗化疗药剂与辐射[8]。虽然球形培养物中富含CSC,但它们也包含非CSC的不同分化细胞。这些细胞还影响蛋白质组结果中的蛋白质,有效稀释CSC特异性蛋白质并影响检测。因此,对比球形中培养的细胞,应用FACS和MACS法更能检测出肿瘤干细胞特异性分子,并在蛋白组学分析中获得纯CSC亚群。
细胞内蛋白质从一个亚细胞结构动态转移到另一个亚细胞结构,其蛋白转位与肿瘤的治疗抗性有关。例如,表皮生长因子受体(EGFR)转位至细胞核与不同抗癌疗法的抗性密切相关。因此,核内EGFR位置与癌细胞接触DNA损伤化疗或电离辐射后DNA修复增强有关。抗乳腺癌蛋白中核EGFR诱导活化可能产生EGFR抑制剂(gefitinib)抗性。将亚细胞结构联系至特定蛋白,有助于更好地理解蛋白质信号从一个细胞内组分传输到另一个细胞内。例如,从线粒体到细胞核或细胞质的蛋白信号可以决定细胞的命运,包括细胞增殖、分化和死亡[9]。因此在某些情况下,注重特定亚细胞结构中的蛋白质含量比研究总细胞蛋白质谱更为重要[10]。亚细胞分离优先蛋白质组学分析的另一原因是它可以降低生物样本的复杂性,从而增加感兴趣的蛋白质组的覆盖范围,并检测出低分子量或低丰度的蛋白质。由于这些原因使亚细胞分离在CSC蛋白质谱的研究变得非常具有吸引力。
2.2 分泌蛋白质组学对条件培养液(分泌蛋白质组)中获得的癌细胞或新鲜分离的肿瘤样本进行蛋白质组学分析,可以增加确定血液、尿液、唾液等生物流体中CSC标志物的机会[11-12]。此外,不同肿瘤细胞亚群分泌的蛋白质的鉴定与表征分析有助于确定各种细胞类型的蛋白质特异性。CSC特异性分泌蛋白质组的知识可以提供有关CSC细胞增殖和分化调节的信息[13]。与总CSC蛋白质组类似,分析CSC分泌蛋白质组时面临的主要挑战是获得纯CSC亚群[14]。由于CSC生物标志物的定义尚不明确,所以很难获得CSC的纯亚群。在比较克隆、致瘤和转移能力或高或低的癌细胞中获得的分泌蛋白质组,将有助于检测出肿瘤内CSC形成过程中的有关分子[15]。需要在大量癌症患者的生物流体中进一步验证这些蛋白质是否能够作为CSC生物标志物或影响CSC活性的因子。
3 蛋白质组学方法鉴定CSC特异性蛋白 3.1 前列腺癌的蛋白质组学前列腺癌是最常诊断出的恶性肿瘤之一。最近的研究已经表明,前列腺癌CSC是产生治疗抗性,并导致癌症复发的原因[16]。临床体外耐辐射模型中通常伴随对化疗药物的不敏感,与亲代细胞系相比CSC数量更多。因此,研究认为应对全部亲代细胞与具有治疗抗性的耐辐射癌细胞(induced radioresistance, IRR)即IRR前列腺癌细胞的亲代细胞的蛋白质谱进行比较。采用凝胶型蛋白质组学方法,通过质谱法可以确定27种蛋白质在治疗抗性的、耐辐射细胞的前列腺癌细胞系和亲代细胞系中的表达存在差异。
3.2 肿瘤干细胞的活性与肿瘤分子特征的关系根据已知的CSC对肿瘤生成、生长和转移的作用,可以预计CSC含量较高的肿瘤与CSC含量较低的肿瘤相比,应该更大并更具侵袭性。研究认为CSC的数量与肿瘤体积呈线性相关[17]。但也有报道称,由于微环境的不同,同等数量的CSC可以表现出不同程度的侵袭性[18]。因此,可以假设侵袭性较强的前列腺癌与侵袭性较弱的前列腺癌的分子特征不同。通过激光捕获显微切割后,进行蛋白质组分析来鉴定可以调节CSC侵袭性的蛋白质。与侵袭程度较低的肿瘤相比,侵袭程度较高的前列腺癌细胞表现出明显的糖酵解途径活化。这些研究发现证实了隐藏不同代谢途径的瘤内干细胞可能是造成肿瘤异质性与不同程度侵袭性的原因[19]。因此,糖酵解途径活化的CSC表现出更具侵袭性的表型。此外,侵袭性强的肿瘤的特征是核纤层蛋白A/C的上调。核纤层蛋白属于结构蛋白的一种,可以确定核结构并参与细胞生长、迁移、侵袭和信号转导[20]。目前还不清楚核纤层蛋白A/C在CSC活性调节中的作用。不过A型核纤层蛋白可以调节属于典型CSC相关通路的Wnt[21]与Notch信号通路,以及转化生长因子ft(TGFft)信号通路。因此,需要进一步研究以确认核纤层蛋白A/C是否能被视为CSC相关蛋白。总之,肿瘤中显示的分子特征程度越高表明CSC的活性越高。
4 用蛋白质组学方法搜索新的治疗靶目前仍无特异性疗法能从恶性肿瘤中有效消除CSC。但如果可以成功破坏CSC,就可以显著改善癌症患者的临床预后。最近,研究人员致力于确定CSC特异性生物标志物和治疗靶[22],提出将Ras相关C3肉毒素底物1(Rac1)蛋白作为头颈部磷状细胞癌(HNSCC)CSC的假定生物标志物和有效治疗靶。Rac1是小GTP蛋白Ras同源物(RHO)家庭中的一员,可以作为信号转导分子调节各种细胞过程,包括细胞分裂、增殖、分化[23]、存活、运动、囊泡运输、核组装与细胞骨架控制。Rac1也被描述为蛋白质控制受体,与细胞内信号转导和ROS生成和核因子κB(NF-κB)活化[23]等有关。
最近研究表明,EMT信号转导影响癌细胞耐辐射性与化疗耐药性的发展。抑制Rac1相关信号转导就可以破坏肿瘤模型中的癌细胞增殖、克隆形成、细胞运动,还可以提高癌细胞对电离辐射和化疗药物的敏感度[24]。首次证明,Rac1可以作为可能的生物标志物和治疗靶来治疗头颈部鳞癌等CSC数量较多的恶性肿瘤。因此,最近蛋白质组初步数据表明,耐辐射乳腺癌细胞的特征伴随着CD44和Rac1表达的增强。同时还证明Rac1可以作为肺部恶性肿瘤、乳腺癌[25]、脑肿瘤和宫颈癌[26]预后的不利因素。
5 结语总之,尽管有人认为蛋白质组技术费时费力又难以实现,但这种方法可以有效阐明哪些通路参与调节CSC活化等多种瘤内过程。对临床前模型和临床样本的蛋白质图谱进行详细剖析,清楚描述并理解CSC相关通路,能查找并鉴别出单个分子作为生物标志物或治疗靶。
| [1] | Panis C, Pizzatti L, Abdelhay E, et al. How can proteomics reach cancer biomarkers?[J]. Curr Proteomics, 2013, 10(2): 136–49. DOI:10.2174/1570164611310020006 |
| [2] | 李明. 肿瘤干细胞蛋白质组学研究的新策略[J]. 中华肿瘤杂志, 2014, 36(5): 321–3. [ Li M. New strategies in the proteomic research on tumor stem cells[J]. Zhonghua Zhong Liu Za Zhi, 2014, 36(5): 321–3. ] |
| [3] | Mechref Y, Muddiman DC. Recent advances in glycomics, glycoproteomics and allied topics[J]. Anal Bioanal Chem, 2017, 409(2): 355–7. DOI:10.1007/s00216-016-0093-9 |
| [4] | Sawanobori A, Moriwaki K, Takamatsu S, et al. A glycoproteomic approach to identify novel glycomarkers for cancer stem cells[J]. Proteomics, 2016, 16(24): 3073–80. DOI:10.1002/pmic.v16.24 |
| [5] | Nie S, McDermott SP, Deol Y, et al. A quantitative proteomics analysis of MCF7 breast cancer stem and progenitor cell populations[J]. Proteomics, 2015, 15(22): 3772–83. DOI:10.1002/pmic.201500002 |
| [6] | Ragelle H, Naba A, Larson BL, et al. Comprehensive proteomic characterization of stem cell-derived extracellular matrices[J]. Biomaterials, 2017, 128: 147–59. DOI:10.1016/j.biomaterials.2017.03.008 |
| [7] | 游俊浩, 薛峰, 颜兵. 结肠肿瘤干细胞巢研究-从潘氏细胞谈起[J]. 肿瘤防治研究, 2017, 44(2): 147–52. [ You JH, Xue F, Yan B. Cancer stem cells niche in colorctal cancer: take paneth cells as an example[J]. Zhong Liu Fang Zhi Yan Jiu, 2017, 44(2): 147–52. ] |
| [8] | Kranenburg O, Emmink BL, Knol J, et al. Proteomics in studying cancer stem cell biology[J]. Expert Rev Proteomics, 2012, 9(3): 325–36. DOI:10.1586/epr.12.24 |
| [9] | Dodson M, Darley-Usmar V, Zhang J. Cellular metabolic and autophagic pathways: Traffic control by redox signaling[J]. Free Radic Biol Med, 2013, 63: 207–21. DOI:10.1016/j.freeradbiomed.2013.05.014 |
| [10] | Abbaszadegan MR, Bagheri V, Razavi MS, et al. Isolation, identification, and characterization of cancer stem cells: A review[J]. J Cell Physiol, 2017, 232(8): 2008–18. DOI:10.1002/jcp.v232.8 |
| [11] | De Wever O, De Boeck A, Maynard D, et al. Protein secretome analysis of evolving and responding tumor ecosystems[J]. Curr Proteomics, 2013, 10(2): 120–35. DOI:10.2174/15701646112099990003 |
| [12] | Schaaij-Visser TB, de Wit M, Lam SW, et al. The cancer secretome, current status and opportunities in the lung, breast and colorectal cancercontext[J]. Biochim Biophys Acta, 2013, 1834(11): 2242–58. DOI:10.1016/j.bbapap.2013.01.029 |
| [13] | Okawa S, Gagrica S, Blin C, et al. Proteome and sccretome characterization of glioblastoma-derived neural stem cells[J]. Stem Cells, 2017, 35(4): 967–80. DOI:10.1002/stem.2542 |
| [14] | Brandi J, Dalla Pozza E, Dando I, et al. Secretome protein signature of human pancreatic cancer stem-like cells[J]. J Proteomics, 2016, 136: 1–12. DOI:10.1016/j.jprot.2016.01.017 |
| [15] | Anderson JD, Johansson HJ, Graham CS, et al. Comprehensive proteomic analysis of mesenchymal stem cell exosomes reveals modulation of angiogenesis via nuclear factor-kappaB signaling[J]. Stem Cells, 2016, 34(3): 601–13. DOI:10.1002/stem.2298 |
| [16] | Wang L, Huang X, Zheng X, et al. Enrichment of prostate cancer stem-like cells from human prostate cancer cell lines by culture in serum-free medium and chemoradiotherapy[J]. Int J Biological Sci, 2013, 9(5): 472–9. DOI:10.7150/ijbs.5855 |
| [17] | Bütof R, Dubrovska A, Baumann M. Clinical perspectives of cancer stem cell research in radiation oncology[J]. Radiother Oncol, 2013, 108(3): 388–96. DOI:10.1016/j.radonc.2013.06.002 |
| [18] | Salvatori L, Caporuscio F, Verdina A, et al. Cell-to-cell signaling influences the fate of prostate cancer stem cells and their potential to generate more aggressive tumors[J]. PLoS One, 2012, 7(2). |
| [19] | Brandi J, Dando I, Pozza ED, et al. Proteomic analysis of pancreatic cancer stem cells: Functional role of fatty acid synthesis and mevalonate pathways[J]. J Proteomics, 2017, 150: 310–22. DOI:10.1016/j.jprot.2016.10.002 |
| [20] | Kong L1, Schäfer G, Bu H, et al. Lamin A/C protein is overexpressed in tissue-invading prostate cancer and promotes prostate cancer cell growth, migration and invasion through the PI3K/AKT/PTEN pathway[J]. Carcinogenesis, 2012, 33(4): 751–9. DOI:10.1093/carcin/bgs022 |
| [21] | 李明. 靶向肿瘤干细胞的新治疗策略[J]. 中国医学科学院学报, 2014, 36(5): 546–50. [ Li M. New treatment strategies targeting cancer stem cells[J]. Zhong Guo Yi Xue Ke Xue Yuan Xue Bao, 2014, 36(5): 546–50. ] |
| [22] | Eun K, Ham SW, Kim H. Cancer stem cell heterogeneity: origin and new perspectives on CSC targeting[J]. BMB Rep, 2017, 50(3): 117–25. DOI:10.5483/BMBRep.2017.50.3.222 |
| [23] | Gastonguay A, Berg T, Hauser AD, et al. The role of Rac1 in the regulation of NF-kappaB activity, cell proliferation, and cell migration in non-small cell lung carcinoma[J]. Cancer Biol Ther, 2012, 13(8): 647–56. DOI:10.4161/cbt.20082 |
| [24] | Katz E, Sims AH, Sproul D, et al. Targeting of Rac GTPases blocks the spread of intact human breast cancer[J]. Oncotarget, 2012, 3(6): 608–19. |
| [25] | Yang F, Xu J, Tang L, et al. Breast cancer stem cell: the roles and therapeutic implications[J]. Cell Mol Life Sci, 2017, 74(6): 951–66. DOI:10.1007/s00018-016-2334-7 |
| [26] | Mendoza-Catalán MA, Cristóbal-Mondragón GR, Adame-Gómez J, et al. Nuclear expression of Rac1 in cervical premalignant lesions and cervical cancer cells[J]. BMC Cancer, 2012, 12: 116. DOI:10.1186/1471-2407-12-116 |
 2018, Vol. 45
2018, Vol. 45