文章信息
- 癌症基因组图集数据库及其应用
- The Cancer Genome Atlas Database and Its Application
- 肿瘤防治研究, 2018, 45(3): 171-174
- Cancer Research on Prevention and Treatment, 2018, 45(3): 171-174
- http://www.zlfzyj.com/CN/10.3971/j.issn.1000-8578.2018.17.0814
- 收稿日期: 2017-07-06
- 修回日期: 2017-08-02
已被发现的癌症至少有200种。在基因组水平上研究DNA变异及变异间相互作用引起细胞的癌变分子机制,将有助于提高我们对癌症的预防、诊断、治疗能力。基于此,美国国家癌症研究所(National Cancer Institute, NCI)和国家人类基因组研究所(National Human Genome Research Institute, NHGRI)联合发起了癌症基因组图集计划(The Cancer Genome Atlas, TCGA)(https://cancergenome.nih.gov/)。2006年开始了试探性的工作,征集了神经胶质细胞瘤和卵巢癌,获得了海量数据,很快这些数据在全球范围内被广泛应用[1]。2009年9月宣布开启五年内完成至少20种癌症综合基因组图谱的研究计划。2013年12月结束了样品收集工作,收集到了超过30种类型的癌症。2014底这一计划宣告完成,共研究了34种类型的癌症,超过11 000个样本。这些数据已被癌症基因组图集研究组和超过1 000个独立研究者应用到他们的研究中,促进了人们对癌变分子机制的了解,开启了癌症研究的新时代。本文将对癌症基因组图谱计划的研究团队与数据产生、数据的类型与分级、数据的获取与应用、已有的贡献四个方面进行详实的介绍。旨在使癌症研究者和临床工作者了解癌症基因组图集计划及其数据,进而将这些数据运用于癌症的预防、早期诊断和治疗中。
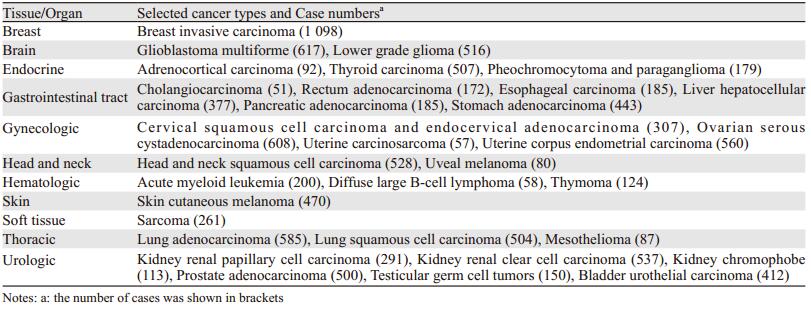
1 样品的获取与数据产生 1.1 样品获取癌症基因组研究选取的是那些发病率高且预后不良的癌症类型,当然更大程度上依赖于能够获得的样本,最终研究了34种癌症的11 000多个样本,见表 1。选择研究的样本需要有癌变组织及与其匹配的正常组织(白血病和肿瘤细胞系没有“正常组织”),并且数量和质量都要达标,对它们的分析处理也要保持一致。癌变组织及与其匹配的正常组织同时进行分析,将有助于研究者发现癌症基因组的非胚系改变;足够的数量和质量较高的组织样品以确保所提取到的DNA、RNA和蛋白质能够满足相关的组学分析。绝大多数的癌症分析数百个样品,以保证有足够的统计学效应,找出同种类型的癌症间的差异,划分为更多的亚型;每个样品的组学分析和数据处理方法基本一致,使所产生出的癌症基因组图谱完整、详实、背景误差小。
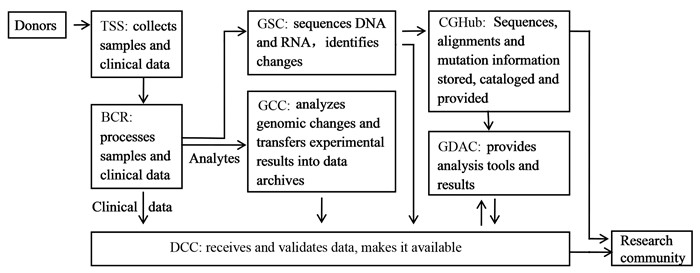
癌症基因组图集计划是一项综合的国际团队协作工作,由7个研究平台协作完成。他们使用一套高效的分析流程,获取了海量的数据,展现出了团队合作收集和分析大数据的强大力量(https://wiki.nci.nih.gov/display/TCGA/TCGA+Data+Primer)。这7个平台及其分工如下:(1)样品资源搜集点(tissue source site, TSS)搜集样品和临床原始数据,送到生物样本中心(biospecimen core resource, BCR);(2)生物样本中心对样本进行分类、条形码标记、处理、检测和保存,确保样品完全符合TCGA的标准。提取的生物大分子(DNA、RNA、蛋白质),保证足够的数量和质量,送至基因组学检测中心(genome characterization center, GCC)和基因组测序中心(genome sequencing center, GSC)。参与者的临床资料、样品的特征数据(样品的质量等)、组织学切片等分类整理后提交到数据协作中心(data coordinating center, DCC);(3)基因组学检测中心通过高通量分析技术来测定提交的生物大分子(DNA、RNA、蛋白质)的表达数据、DNA拷贝数、miRNA、表观遗传数据(DNA甲基化数据),进而确定与癌变相关的变化,然后由基因组测序中心做进一步的核实分析。把实验得到的微阵列芯片组学测定结果转化成数据档案,提交到数据协作中心;(4)基因组测序中心将提交样品DNA、RNA的高通量测定序列(外显子组序列或者所有编码基因序列、被选定的样品的全基因组序列)。鉴别癌变组织与正常组织间的差异,并确定这些变化是发生在体细胞还是性细胞。测序结果、序列比对结果、突变数据提交到癌症基因组平台(cancer genomics hub, CGHub),同时将突变结果提交到数据协作中心;(5)基因组数据分析中心(genome data analysis center, GDAC)开发相关数据管理和分析工具,并提供给使用者。获取数据协作中心及癌症基因组平台(CGHub)的数据并进行分析,分析结果提交给数据协作中心;(6)数据协作中心检查和标准化来自各个研究团队的数据,提供公共数据入口,便于资料的搜索和下载;(7)癌症基因组平台是一个安全的数据库,只存储和提供原始的实验数据及其简单的分析文件如跟踪文件、测定的序列、比对结果和突变数据等。实验数据的产生流程,见图 1。这一流程可以大规模的、高效的、有选择的分析人类基因组的变化。目前这一分析流程仍然被美国国家癌症研究中心用来研究癌症基因组,这也为其他大规模的基因组研究提供了一套高效的模板。
|
| TSS: tissue source site; BCR: biospecimen core resource; GSC: genome sequencing center; GCC: genome characterization center; CGHub: cancer genomics hub; GDAC: genome data analysis center; DCC: data coordinating center 图 1 癌症基因组图集计划实验流程与数据产生过程 Figure 1 Process of The Cancer Genome Atlas(TCGA) project and data flow |
癌症基因组图集计划产生的海量数据可归纳为如下类型:临床资料、图像资料、微卫星不稳定性、DNA序列、小RNA序列、蛋白质表达数据、信使RNA序列、总RNA序列、阵列表达数据、DNA甲基化位点、DNA拷贝数(https://portal.gdc.cancer.gov/)。
2.2 数据的访问层次癌症基因组图集的数据有些直接来自某个个体如临床数据、原始序列数据,单核苷酸多态性数据。个体的直接标识已被删除,但是仍然存在着被生物信息学方法或者第三方数据重新识别的风险。NIH和NCI视参与者的个人隐私为首要问题,为了把泄露参与者隐私的风险降至最低,癌症基因组图集的数据被分为两个访问层次,即开放数据层和限制数据层。开放数据层:来自多个样本的数据,可以自由地获取,不需要用户认证。包括去掉标识的临床和人口数据、基因表达数据、基因组部分片段拷贝数的变化数据、表观遗传学数据、专家归纳整理过的数据、匿名的单扩增子DNA序列数据。限制数据层:数据来自某个个体,删除掉了其标识,获取这些数据需要用户认证,包括原始序列数据、单核酸多态性阵列数据(一级、二级数据)、外显子阵列数据(一级、二级数据)、VCFs(描述变异的文件)、最小等位基因频率。
2.3 数据的水平癌症基因组图集的数据有的直接来源于实验的原始数据,有的是对原始数据进行整理、分析、注释而得到的衍生数据。各研究平台根据数据的来源将它们分为4个水平。水平1:单个样本的未经标准化的低水平原始数据(如序列示踪文件、芯片CEL文件、BAM文件);水平2:标准化的单个样本数据的解读(大分子表达的变化、单个样品的突变、位点的扩增与缺失、杂合性的丢失、单个样品的探针信号);水平3:整合或者分开处理后的样本数据,通过探针位点拼接成的连续片段;水平4:研究者感兴趣数据的综合分析,多个跨类样本的量化关联分析(https://wiki.nci.nih.gov/display/TCGA/TCGA+Data+Primer)。每个平台能产出多种类型的数据,每一类型的数据可以有多种水平的存在形式。不同水平的划分有利于使用者交流和查找感兴趣的数据。
3 数据的获取和应用 3.1 癌症基因组图集数据的获取癌症基因组图集计划是一个公共资源项目,其数据可以免费获取。普通使用者通常下载那些已被分析和整理的level3、level4数据。Level1和level2数据繁杂、格式多样,使用起来有很多困难。这些数据最初由癌症基因组图集的官方网站(TCGA Data Portal)来提供。2016年7月15日起,癌症基因组图集的官方网站不再提供数据服务,将所有数据转入基因组数据共享平台(Genomic Data Commons GDC, https://gdc-portal.nci.nih.gov/)。使用者通过GDC数据门户可以搜索下载癌症相关的数据。通过癌症类型、文件、病例、注释和数据来进行搜索,搜索结果可以通过其界面进行更精细的筛选。GDC提供两种数据下载方式:(1)选择的数据放入个人资料车,在个人资料车内点击download,选择cart可以直接下载其数据,此方法主要用于少量数据的下载。(2)大量的数据在个人资料车中点击download下的资料清单(manifest),通过GDC的数据传输工具(Data Transfer Tool)进行批量下载(https://wiki.nci.nih.gov/display/TCGA/TCGA+Data+Primer)。除了官网,还有一些其他的网站也提供比较完整和可靠的数据下载如GDAC(http://firebrowse.org/)、Cancer Browser(https://genome-cancer.ucsc.edu/)、cBioPortal(http://www.cbioportal.org/)等。
3.2 癌症基因组图集数据的使用分析癌症基因组图集的数据有助于我们在分子水平上洞察癌变机制。常见的数据分析有如下几种:生存分析通过对癌症患者生存时间与临床数据之间的关联分析,研究生存时间与致病因素或者治疗措施之间的关联程度;差异表达分析通过比较癌变组织与匹配的正常组织间的基因表达谱数据和蛋白质表达谱数据的差异,来探索发病机制,发现癌变相关的基因,寻找治疗的药物靶点;共表达网络分析一般通过聚类分析基因间表达值的变化来发现基因间的共表达关系,建立基因间表达调控网络,寻找癌变中的关键基因;甲基化分析通过分析甲基化数量、位置的变化来研究癌基因、抑癌基因的甲基化情况与癌变的关系,为诊断和治疗提供重要的依据;综合分析运用多种分析手段对多种类型数据进行联合分析。
4 癌症基因组图集计划的贡献癌症基因组图集计划提供了30多种癌症的基因组变化数据,由于癌症种类繁多、样品的获取有很多困难,许多病例缺乏长期生存资料和肿瘤复发等随访资料,这些资料还需要不断的完善。应用这些数据进行的相关研究文章已有几千篇,其中发表于《Cell》、《Nature》、《Science》上的文章就有几十篇,取得了很多突破性的成果,有些加强了理论认识,有些促进了现实中的应用。
在理论上加速了我们对癌变分子机制的认识。如通过对恶性胶质瘤细胞样本数据的分析发现其癌变与三个信号通路有关,分别为RTK/RAS/PI3K信号通路(88%)、TP53信号通路(87%)、RB1/CDK4信号通路(78%)[1]。恶性胶质瘤细胞中的一种小RNA miR-26a的扩增会关闭癌症抑制基因而导致发病[1]。
在应用中促进了精准医疗与个性化疗法的发展。新发现的药物靶标有助于开发新药物进行精准治疗,如研究人员对甲状腺癌数据进行了综合分析,发现了一些肿瘤的标志物如LncRNA HOTAIR、BRAF、PARP4等,这将促进对甲状腺癌的诊断和精准施药[2-5];鉴定出了更多的亚型,为癌症的个性化治疗打下了坚实的基础,如通过对乳腺癌样本的综合特征鉴定,研究人员发现存在有4种主要的亚型: HER2E、LumA、LumB、basal-like。分析发现基底样乳腺癌与浆液性卵巢癌基因组相似度比它与其他乳腺癌相似度更高,这意味着依据癌症基因组的特点来对癌症进行分析和治疗要比依据发病的器官会更有效[6]。
5 结语癌症基因组图谱计划已经完成,后续的延伸研究还在进行中。它建立了团队协作、多基因组、高效的分析流程,展现出了团队合作收集和分析大数据的强大力量。这一流程为其他大规模的基因组研究提供了高效模板。癌症基因组图集计划产生的数据在癌症分子机制的研究中发挥着巨大作用,开启了癌症研究的新时代。癌症基因组图集的样品多来自欧美,癌变机制有时会有地区差异,因此加快我国的癌症基因组的研究是十分必要的。我国是国际癌症基因组联盟(International Cancer Genome Consortium, ICGC)的创始成员,发起了胃癌、结直肠癌、食管癌、肝癌和鼻咽癌基因组的研究并取得了显著的成果[7-8]。作为普通的研究者和临床工作者参与大规模的基因组研究的机会是有限的,但是癌症基因组数据的使用为我们开启了更广阔的研究空间。由于这些数据庞杂,格式多样,分析过程需要一些特殊的分析工具和统计软件,因此普通癌症研究者和临床工作者首先要了解TCGA数据的组成与结构,熟悉常用分析工具及相应的统计软件,才能将这些资源整合到研究之中,进而探索癌症发病的分子机制,提高我们对癌症的预防、及早诊断、治疗能力。
| [1] | Tomczak K, Czerwinska P, Wiznerowicz M. The Cancer Genome Atlas (TCGA): an immeasurable source of knowledge[J]. Contemp Oncol, 2015, 19(1A): 68–77. |
| [2] | Li HM, Yang H, Wen DY, et al. Overexpression of LncRNA HOTAIR is Associated with Poor Prognosis in Thyroid Carcinoma: A Study Based on TCGA and GEO Data[J]. Horm Metab Res, 2017, 49(5): 388–99. DOI:10.1055/s-0043-103346 |
| [3] | Chai YJ, Yi JW, Jee HG, et al. Significance of theBRAFmRNA Expression Level in Papillary Thyroid Carcinoma: An Analysis of The Cancer Genome Atlas Data[J]. PLoS One, 2016, 11(7): e0159235. DOI:10.1371/journal.pone.0159235 |
| [4] | Ikeda Y, Kiyotani K, Yew PY, et al. Germline PARP4 mutations in patients with primary thyroid and breast cancers[J]. Endocr Relat Cancer, 2016, 23(3): 171–9. DOI:10.1530/ERC-15-0359 |
| [5] | Cancer Genome Atlas Research Network. Integrated Genomic Characterization of Papillary Thyroid Carcinoma[J]. Cell, 2014, 159(3): 676–90. DOI:10.1016/j.cell.2014.09.050 |
| [6] | Cancer Genome Atlas Network. Comprehensive molecular portraits of human breast tumours[J]. Nature, 2012, 490(7418): 61–70. DOI:10.1038/nature11412 |
| [7] | 胡学达, 杨焕明, 赫捷, 等. 肿瘤基因组学与全球肿瘤基因组计划[J]. 科学通报, 2015, 60(9): 792–804. [ Hu XD, Yang HM, He J, et al. The cancer genomics and global cancer genome collaboration (in Chinese)[J]. Ke Xue Tong Bao, 2015, 60(9): 792–804. ] |
| [8] | Song Y, Li L, Ou Y, et al. Identification of genomic alterations in oesophageal squamous cell cancer[J]. Nature, 2014, 509(7498): 91–5. DOI:10.1038/nature13176 |
 2018, Vol. 45
2018, Vol. 45