文章信息
- 子宫颈鳞状细胞癌组织中LncRNA HCG11和miR-590-3p的表达及其与预后的关系
- Expression of LncRNA HCG11 and miR-590-3p in Squamous Carcinoma of Cervix and Their Relationship with Prognosis
- 肿瘤防治研究, 2018, 45(3): 148-153
- Cancer Research on Prevention and Treatment, 2018, 45(3): 148-153
- http://www.zlfzyj.com/CN/10.3971/j.issn.1000-8578.2018.17.0614
- 收稿日期: 2017-06-05
- 修回日期: 2017-09-20
2. 473000 南阳,南阳市第二人民医院妇产科
2. Department of Obstetrics and Gynecology, The Second People's Hospital of Nanyang, Nanyang 473000, China
宫颈癌在全球最常见的女性恶性肿瘤中排名第二,目前仍然是女性癌症相关死亡的主要原因之一。虽然宫颈癌早期可以通过根治手术辅以放化疗得以治愈,但是一些高危因素患者的预后仍然不容乐观[1]。目前的临床数据表明,影响宫颈癌预后的因素包括肿瘤大小、浸润深度、血管的侵犯、子宫旁组织的浸润、盆腔淋巴结转移等,其中盆腔淋巴结的转移被认为是影响术后疗效最重要的危险因素[2]。作为早期诊断及预后评估的分子指标,非编码RNA(non-coding RNA, ncRNAs)已日益得到重视和研究。近年研究表明ncRNAs与宫颈鳞状细胞癌的发生发展有密切关系,参与调控宫颈鳞状细胞癌进程的分子机制和雌孕激素表达[3]。
ncRNAs主要包括microRNAs和lncRNAs[4]。miRNA可通过调控靶基因从而促进宫颈癌细胞凋亡,并能改变肿瘤细胞对药物的敏感度,且能在组织、血清和血浆等标本中稳定存在。miRNA有望成为诊断宫颈癌的新标志物和治疗的新靶点[5]。近年来,lncRNA-miRNAs的调节关系吸引了越来越多学者的注意,lncRNAs和microRNAs的交互调节是目前的研究热点[6]。本研究探讨了LncRNA HCG11和miR-590-3p在宫颈鳞状细胞癌组织中的表达,并且利用qRT-PCR结果和starBase v2.0数据库探讨两者之间的关系,结合患者的临床资料探讨LncRNA HCG11和miR-590-3p与性别、年龄、组织大小、病理分级、淋巴结转移、临床分期以及预后的关系。
1 资料与方法 1.1 临床资料 1.1.1 宫颈鳞状细胞组织样本和临床病理数据收集2013年7月—2016年3月在南阳市第二人民医院妇科接受根治性子宫切除术和盆腔淋巴结清扫的58例宫颈鳞状细胞癌患者,年龄28~72岁。根据国际妇产科联合会(FIGO,2014)的诊断标准,58例宫颈鳞状细胞癌病例中,Ⅰ~Ⅱ期12例、Ⅲ~Ⅳ46例;20例有淋巴结转移、38例无淋巴结转移;分化良好(G1)13例、中度分化(G2)32例、低分化(G3)13例。所有患者术前均未接受化疗或放疗;所有病例均有病理组织学诊断支持;所有标本直接取自手术切除物,均为配对组织。每对为肿物和癌旁正常组织距离病灶≥5 cm,经病理证实无肿瘤细胞浸润各一份。在手术切除后10 min之内获得。取材后置于无菌冻存管中,储存至-80℃。所有患者均获得随访,末次随访时间为2016年8月,随访时间5~36月。
1.1.2 试剂与仪器RNA提取试剂盒(天根生物公司,中国),RNA反转录试剂盒(诺维赞生物公司,美国),荧光定量PCR试剂盒(康为世纪生物公司,中国),Biotek Epoch微量核酸定量仪购自美国Biotek公司,7500荧光定量PCR仪购自美国ABI公司。
1.1.3 qRT-PCR检测基因LncRNA HCG11和miR-590-3p的引物序列通过PCR引物设计软件Primer Premier 5.0设计LncRNA HCG11、GAPDH、has-miR-590-3p和U6的引物序列,具体序列如下:HCG11上游引物:5′-AGGAGTGGTTGCATTTGGGA-3′,下游引物:5′-CCCACCACGCAGTGAATAGT-3′;GAPDH上游引物:5'-GGAAGGACTCATGACCACAGTCC-3',下游引物:5'-TCGCTGTI'GAAGTCAGAGGAGACC-3'。has-miR-590-3p-5p引物为5′-CCTGGCTTTTCATTCCTATGTGA-3′,U6引物为5′-GCTTCGGC AGCACATATACTAAA-3′,并由上海生工生物公司合成。引物干粉离心后用DEPC水配制成20 µmol/L,置于-20℃冰箱备用。
1.2 方法 1.2.1 总RNA的提取和反转录从子宫颈鳞状细胞癌组织和癌旁组织中抽提总RNA,按总RNA提取试剂盒说明书进行,采用定量分光光度法在A260/280处对提取RNA进行定量。按照反转录试剂盒(诺维赞公司,R111-01/02)说明书对总RNA进行反转录,反转录反应由1 µg的总RNA,与10 µl 2×RT mix,2 µl mix,1 µl Oligo dNTPs,1 µl Random hexamers,6 µl ddH2O总体系20 µl。25℃ 5 min,50℃ 15 min,85℃ 5 min,经反转录形成cDNA。
1.2.2 qRT-PCR检测LncRNA HCG11和miR-590-3p根据康为世纪生物公司实时PCR试剂盒说明书进行荧光定量PCR。实时PCR体系为20 µl : 2 µl反向转录产物,2 µl引物,2 µl反向引物,10 µl 2×mix,纯水4 µl。1个循环包括95℃ 30 s、95℃ 5 s和60℃ 30 s,总共40个循环。所有样本重复三次。以2-ΔCt表示基因的相对表达水平。
1.3 统计学方法采用SPSS 19.0软件进行统计学分析。计量资料以(x±s)表示,计数资料采用构成比(%)表示,58对配对的宫颈鳞状细胞癌和癌旁组织比较采用两配对样本t检验,与临床资料联合分析采用卡方检验,采用Kaplan-Meier法计算生存函数,差异比较采用Log rank检验,采用Cox比例风险模型用于宫颈鳞状细胞癌预后的多因素分析。以P < 0.05为差异有统计学意义。
2 结果 2.1 LncRNA HCG11和miR-590-3p的表达应用qRT-PCR检测58对配对的宫颈鳞状细胞癌组织及癌旁组织中LncRNA HCG11和miR-590-3p的表达,以癌旁为参考值,通过配对样本t检验分析2-ΔCt值。结果显示宫颈鳞状细胞癌组织中LncRNA HCG11的平均表达量为(8.23±3.77)低于宫颈鳞状细胞癌癌旁组织(9.62±3.93),miR-590-3p平均表达量为(9.61±4.17)高于宫颈鳞状细胞癌癌旁组织(8.18±4.22),差异有统计学意义(t=3.249, P=0.0019; t=3.403, P=0.0012),见图 1。
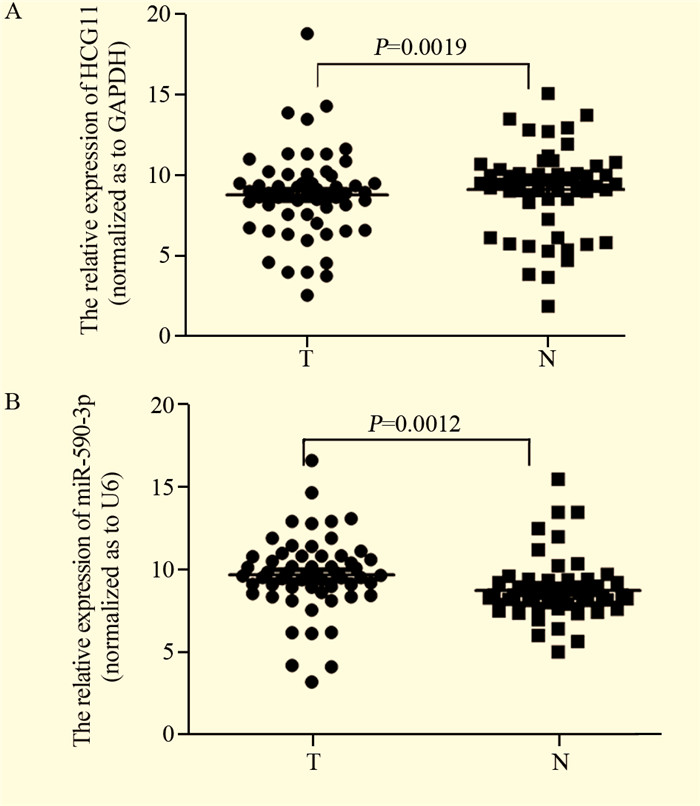
|
| T: squamous carcinoma of cervix(SCC) tissues; N: adjacent non-tumor tissues; A: the expression of LncRNA HCG11 in SCC tissues was inferior to that in non-tumor tissues; B: the expression of miR-590-3p in squamous carcinoma of cervix tissues was superior to that in non-tumor tissues 图 1 58对配对的宫颈鳞状细胞癌组织及癌旁组织中LncRNA HCG11和miR-590-3p的表达 Figure 1 LncRNA HCG11 and miR-590-3p expression in 58 pairs of squamous carcinoma of cervix and adjacent non-tumor tissues |
通过NONCODE database分析我们知道LncRNA HCG11在宫颈癌细胞系HeLa中的相对表达量为1.874,见图 2A,说明宫颈癌细胞中可以检测到该基因的表达。通过starBase v2.0数据库分析了与LncRNA HCG11可以结合miRNA,发现miR-590-3p与LncRNA HCG11有三个结合位点,见图 2B。通过qRT-PCR结果,分析了宫颈鳞状细胞癌组织中LncRNA HCG11和miR-590-3p核酸水平的相关性,见图 2C,结果呈负相关(r=-0.642, P =0.000)。
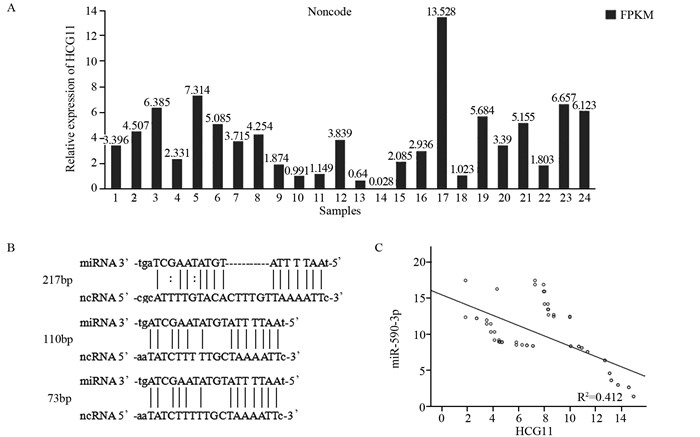
|
| 1: adipose; 2: adrenal; 3: brain; 4: brain_R; 5: breast; 6: colon; 7: fores kin; 8: heart; 9: hela_R; 10: HLF_1; 11: HLF_2; 12: kidney; 13: liver; 14: liver_R; 15: lung; 16: lymph Node; 17: ovary; 18: placenta_R; 19: prostate; 20: skeltal muscle; 21: testes; 22: testes_R; 23: thyroid; 24: white blood cell. A: NONCODE database showed that the expression of LncRNA HCG11 in HeLa cells; B: starBase v2.0 databases showed that there were three binding sites between miR-590-3p and LncRNA HCG11; C: qPCR indicated that on nucleic acid level, LncRNA HCG11 expression was negatively correlated with miR-590-3p expression in squamous carcinoma of cervix tissues 图 2 LncRNA HCG11和miR-590-3p表达相关性分析 Figure 2 Correlation analysis between LncRNA HCG11 and miR-590-3p expression |
肿瘤组织中LncRNA HCG11和miR-590-3p的表达水平与组织大小、淋巴结转移以及临床分期(P=0.027)有相关性,即癌组织大、有淋巴结转移、临床分期差的患者LncRNA HCG11表达水平越低,而miR-590-3p表达水平越高,见表 1。
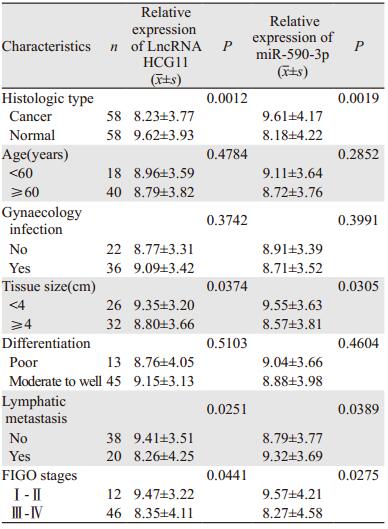
|
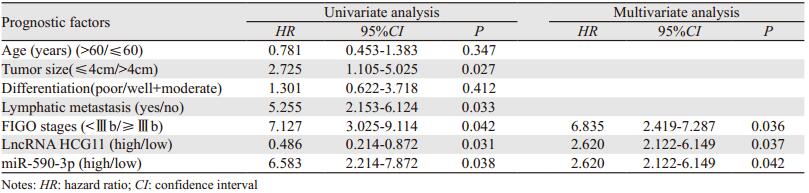
根据qRT-PCR的结果,用Kaplan-Meier方法分析LncRNA HCG11和miR-590-3p在宫颈鳞状细胞癌组织中的表达和患者生存期之间的关系。按照二者在癌组织中表达量中位数把患者分为高表达组和低表达组,其中LncRNA HCG11高表达者24例、低表达者34例。miR-590-3p高表达者35例、低表达者23例。随访时间15~36月,其中复发14例、死亡14例、13例因复发转移死亡、1例因放化疗相关并发症死亡。分析表明,在宫颈鳞状细胞癌患者中,LncRNA HCG11低表达患者的总生存期明显低于高表达患者(P=0.024);miR-590-3p高表达患者的总生存期明显低于低表达患者(P=0.047),见图 3。Cox单变量分析表明肿瘤大小、淋巴结转移、临床分期和LncRNA HCG11以及miR-590-3p的表达情况与生存相关。Cox比例风险模型多变量分析表明LncRNA HCG11和miR-590-3p表达情况分别是独立的危险因素(P=0.037, 0.042),见表 2。
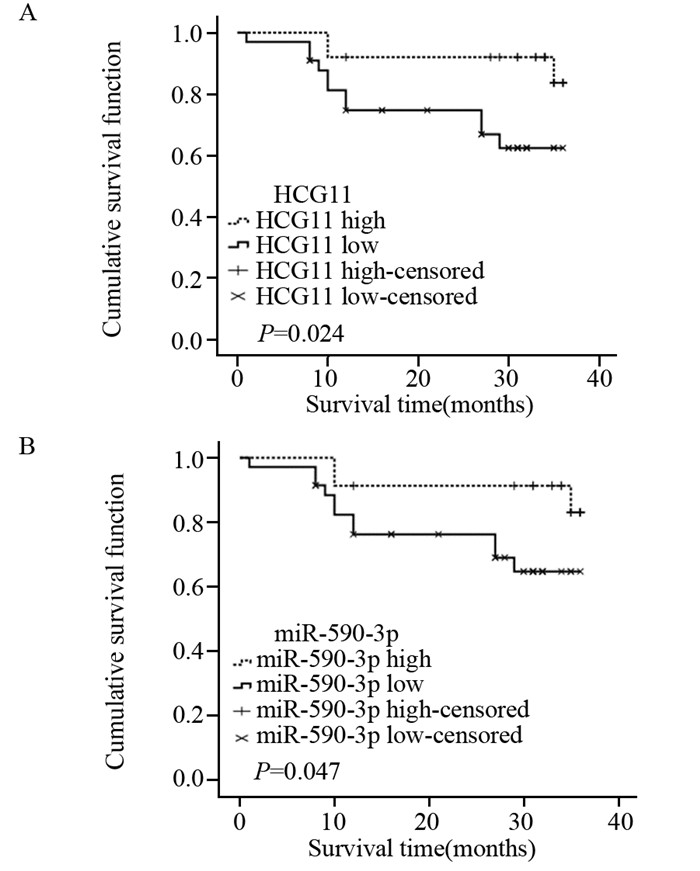
|
| A: The overall survival of patients with low LncRNA HCG11 expression was obviously shorter than that with high LncRNA HCG11 expression; B: The overall survival of patients with high miR-590-3p expression was significantly shorter than that with low miR-590-3p expression 图 3 LncRNA HCG11和miR-590-3p与患者的预后的关系 Figure 3 Relationship of LncRNA HCG11 and miR-590-3p expression with patients' prognosis |
|
为了预测HCG11的功能,首先使用TCGA构建共表达网络识别差异表达的mRNA和HCG11基因之间的相互作用。根据Pearson相关系数建立基因共表达网络。然后使用MAS 3.0(http://bioinfo.capitalbio.com/mas3/project/index)执行GO和通路分析。根据GO分析结果把前1 000个差异表达的基因进行分类。分析显示,这些基因与信号转导、转录、细胞黏附、细胞转化和蛋白质氨基酸磷酸化相关。通路分析显示HCG11的共表达基因主要参与MAPK信号通路、钙信号通路、Jak-STAT信号通路和Wnt信号通路等。
3 讨论非编码RNA占整个人类基因组的98%左右,包括lncRNAs和microRNAs[7]。miRNA能够结合到目标mRNA的3′UTR,调节mRNA的稳定性和翻译过程,最终导致mRNA的翻译被抑制或被降解[7-8]。miRNA可分为癌基因和抑癌基因,参与细胞的多个生物过程,包括增殖、凋亡、代谢、细胞分化等[9]。越来越多的证据表明,microRNA参与多种癌症的进展过程。与已经非常熟悉的microRNAs相比,LncRNAs方面的研究相对较少。长非编码RNA(LncRNAs)由超过200个核苷酸组成,是ncRNAs中重要的成员,不能被翻译成蛋白质[10]。到目前为止,绝大多数的LncRNAs已被鉴定出来[11-12]。许多研究已经证明,LncRNAs在癌症的发生发展中发挥了重要作用[13-15]。目前LncRNA的研究处于起步阶段,因此大多数LncRNA的功能仍是未知。LncRNA能不能作为肿瘤诊断和预后的生物标志物,以及在临床中的意义,人们还没有一个确切的结论。
目前,很多国家都在致力寻找并建立一些可以有效筛查宫颈鳞状细胞癌的项目,从而降低其发病率和死亡率。这些筛查基本需依赖细胞学巴氏涂片,有些联合检查可以预防多达91%的浸润性宫颈鳞状细胞癌[2]。然而,这些项目花费较高,因此,寻找客观精准而又方便检测的标志物,来提示发病和预后是宫颈鳞状细胞癌目前重要的研究方向。一些与宫颈鳞状细胞癌预后相关的分子虽然已得到初步证实,但是它们的作用机制仍不确定。
目前,国内外已发表的与宫颈鳞状细胞癌致病相关的lncRNA有HOTAIR[16]、MALAT1[17]、H19[18]、EBIC[19]、MEG3[20]、GAS5[21],XLOC_010588[22]等。这7个LncRNAs在有些文献中的报道是抑癌作用,有些文献中报道是促癌作用,比如TUG1在结肠癌中表现为上调[23],在非小细胞肺癌中表达下调[24],与其他文献报道不同,说明LncRNAs的表达存在组织特异性、个体差异性,且在组织中表达差异并不显著,均不足以成为宫颈癌的特异肿瘤标志物。
该研究发现与癌旁组织相比HCG11在宫颈鳞状细胞癌组织中显著下调,且与肿瘤组织大小、淋巴结转移以及临床分期有关。starBase v2.0数据库分析表明miR-590-3p与LncRNA HCG11有三个结合位点。随后研究发现miR-590-3p在宫颈鳞状细胞癌组织中表达明显上调,且与组织大小、淋巴结转移、临床分期以及预后有关,而且发现LncRNA HCG11和miR-590-3p核酸水平呈显著的负相关。为了进一步探讨HCG11的功能,我们进行了GO和KEGG通路分析。结果表明受其表达影响的基因参与包括信号转导、转录调控、细胞黏附、恶性转化和氨基酸磷酸化等过程。HCG11参与MAPK信号通路、钙信号通路、Jak-STAT信号通路和Wnt信号通路的调控。
目前关于LncRNA HCG11与肿瘤研究的报道较少,其中HCG11在乳腺癌与前列腺癌中表达下调的患者预后较差。miR-590-3p与肿瘤的研究比较多,Yang等[25]发现miR-590-3p通过激活PI3K-AKT信号通路下调PTEN,促进AKT1-S473的磷酸化作用,进而发挥促进肝癌细胞增殖。Sun等[26]发现,miR-590-3p通过Hippo通路促进结肠癌细胞的增殖和转移。LncRNA的作用机制之一就是通过海绵样吸附作用,竞争性与microRNA结合,抑制其进一步发挥基因调控的作用。由此推测,LncRNA HCG11有可能通过调控miR-590-3p的表达发挥其抑癌作用。可以通过进一步的体外细胞实验对该调控机制进行验证。
| [1] | Park JW, Bae JW. Prognostic significance of positive lymph node number in early cervical cancer[J]. Mol Clin Oncol, 2016, 4(6): 1052–6. DOI:10.3892/mco.2016.837 |
| [2] | Diver E, Hinchcliff E, Gockley A, et al. Minimally Invasive Radical Hysterectomy for Cervical Cancer Is Associated With Reduced Morbidity and Similar Survival Outcomes Compared With Laparotomy[J]. J Minim Invasive Gynecol, 2017, 24(3): 402–6. DOI:10.1016/j.jmig.2016.12.005 |
| [3] | Fujii T, Shimada K, Asano A, et al. MicroRNA-331-3p Suppresses Cervical Cancer Cell Proliferation and E6/E7 Expression by Targeting NRP2[J]. Int J Mol Sci, 2016, 17(8): pii: E1351. DOI:10.3390/ijms17081351 |
| [4] | Etheridge A, Lee I, Hood L, et al. Extracellular microRNA: a new source of biomarkers[J]. Mutat Res, 2011, 717(1-2): 85–90. DOI:10.1016/j.mrfmmm.2011.03.004 |
| [5] | Xin F, Liu P, Ma CF. Acirculating serum miRNA panel as early detection biomarkers of cervical intraepithelial neoplasia[J]. Eur Rev Med Pharmacol Sci, 2016, 20(23): 4846–51. |
| [6] | Deng K, Wang H, Guo X, et al. The cross talk between long, non-coding RNAs and microRNAs in gastric cancer[J]. Acta Biochimica et Biophysica Sinica, 2016, 48(2): 111–6. DOI:10.1093/abbs/gmv120 |
| [7] | Ling H. Non-coding RNAs: Therapeutic Strategies and Delivery Systems[J]. Adv Exp Med Biol, 2016, 937: 229–37. DOI:10.1007/978-3-319-42059-2 |
| [8] | Colombo N, Carinelli S, Colombo A, et al. Cervical cancer: ESMO Clinical Practice Guidelines for diagnosis, treatment and follow-up[J]. Ann Oncol, 2012, Suppl 7: vii27–32. |
| [9] | Yang J, Zhang Z, Guo W, et al. Single nucleotide polymorphisms in microRNA genes are associated with cervical cancer susceptibility in a population from Xinjiang Uygur[J]. Oncotarget, 2016, 7(44): 71447–54. |
| [10] | Kung JT, Colognori D, Lee JT. Long noncoding RNAs: past, present, and future[J]. Genetics, 2013, 193(3): 651–69. DOI:10.1534/genetics.112.146704 |
| [11] | Xie C, Yuan J, Li H, et al. NONCODEv4: exploring the world of long non-coding RNA genes[J]. Nucleic Acids Res, 2014, 42(Database issue): D98–103. |
| [12] | Volders PJ, Verheggen K, Menschaert G, et al. An update on LNCipedia: a database for annotated human lncRNA sequences[J]. Nucleic Acids Res, 2015, 43(8): 4363–4. DOI:10.1093/nar/gkv295 |
| [13] | Sugihara H, Ishimoto T, Miyake K, et al. Noncoding RNA Expression Aberration Is Associated with Cancer Progression and Is a Potential Biomarker in Esophageal Squamous Cell Carcinoma[J]. Int J Mol Sci, 2015, 16(11): 27824–34. DOI:10.3390/ijms161126060 |
| [14] | Wu Y, Yu DD, Hu Y, et al. WITHDRAWN: The long non-coding RNA, LINC00635-001, sensitizes EGFR-TKI-resistant human lung cancer cells in vitro by inhibiting Akt activation[J]. Biochem Biophys Res Commun, 2016, pii: S0006-291X(16)30057–2. |
| [15] | Jiang CY, Gao Y, Wang XJ, et al. Long non-coding RNA lnc-MX1-1 is associated with poor clinical features and promotes cellular proliferation and invasiveness in prostate cancer[J]. Biochem Biophys Res Commun, 2016, 470(3): 721–7. DOI:10.1016/j.bbrc.2016.01.056 |
| [16] | Guo L, Lu X, Zheng L, et al. Association of Long Non-Coding RNA HOTAIR Polymorphisms with Cervical Cancer Risk in a Chinese Population[J]. PLoS One, 2016, 11(7): e0160039. DOI:10.1371/journal.pone.0160039 |
| [17] | Zhang Y, Wang T, Huang HQ, et al. Human MALAT-1 long non-coding RNA is overexpressed in cervical cancer metastasis and promotes cell proliferation, invasion and migration[J]. J BUON, 2015, 20(6): 1497–503. |
| [18] | Iempridee T. Long non-coding RNA H19 enhances cell proliferation and anchorage-independent growth of cervical cancer cell lines[J]. Exp Biol Med (Maywood), 2017, 242(7): 183–94. |
| [19] | Sun NX, Ye C, Zhao Q, et al. Long noncoding RNA-EBIC promotes tumor cell invasion by binding to EZH2 and repressing E-cadherin in cervical cancer[J]. PLoS One, 2014, 9(7): e100340. DOI:10.1371/journal.pone.0100340 |
| [20] | Zhang J, Yao T, Wang Y, et al. Long noncoding RNA MEG3 is downregulated in cervical cancer and affects cell proliferation and apoptosis by regulating miR-21[J]. Cancer Biol Ther, 2016, 17(1): 104–13. DOI:10.1080/15384047.2015.1108496 |
| [21] | Cao S, Liu W, Li F, et al. Decreased expression of lncRNA GAS5 predicts a poor prognosis in cervical cancer[J]. Int J Clin Exp Pathol, 2014, 7(10): 6776–83. |
| [22] | Liao LM, Sun XY, Liu AW, et al. Low expression of long noncoding XLOC_010588 indicates a poor prognosis and promotes proliferation through upregulation of c-Myc in cervical cancer[J]. Gynecol Oncol, 2014, 133(3): 616–23. DOI:10.1016/j.ygyno.2014.03.555 |
| [23] | Zhai HY, Sui MH, Yu X, et al. Overexpression of Long Non-Coding RNA TUG1 Promotes Colon Cancer Progression[J]. Med Sci Monit, 2016, 22: 3281–7. DOI:10.12659/MSM.897072 |
| [24] | Lin PC, Huang HD, Chang CC, et al. Long noncoding RNA TUG1 is downregulated in non-small cell lung cancer and can regulate CELF1 on binding to PRC2[J]. BMC Cancer, 2016, 16: 583. DOI:10.1186/s12885-016-2569-6 |
| [25] | 杨红飞, 郑文宏, 赵文沟, 等. miR-590-5p和miR-590-3p参与肝细胞肝癌发展的机制[J]. 南方医科大学学报, 2013, 33(6): 804–11. [ Yang HF, Zheng WH, Zhao WH, et al. Roles of miR-590-5p and miR-590-3p in the development of hepatocellular carcinoma[J]. Nan Fang Yi Ke Da Xue Xue Bao, 2013, 33(6): 804–11. ] |
| [26] | Sun ZQ, Shi K, Zhou QB, et al. MiR-590-3p promotes proliferation and metastasis of colorectal cancer via Hippo pathway[J]. Oncotarget, 2017, 8(35): 5806–71. |
 2018, Vol. 45
2018, Vol. 45


