文章信息
- 宫颈癌细胞中ΔNp63α的MicroRNA表达谱及其对hsa-let-7b-3p的调控机制
- MicroRNA Expression Profile of ΔNp63α in Cervical Cancer Cells and Its Regulation Mechanism on hsa-let-7b-3p
- 肿瘤防治研究, 2018, 45(3): 138-143
- Cancer Research on Prevention and Treatment, 2018, 45(3): 138-143
- http://www.zlfzyj.com/CN/10.3971/j.issn.1000-8578.2018.17.0542
- 收稿日期: 2017-10-31
- 修回日期: 2017-11-27
2. 230001 合肥,安徽医科大学附属省立医院放疗科;
3. 230021 合肥,中国科学院天然免疫与慢性疾病重点实验室,中国科学技术大学生命科学院
2. Department of Radiation, Anhui Provincial Hospital Affiliated to Anhui Medical University, Hefei 230001, China;
3. The CAS Key Laboratory of Innate Immunity and Chronic Disease, School of Life Sciences, University of Science and Technology of China, Hefei 230021, China
转录因子p63是p53基因家族成员之一[1],相对于p53和p73基因,其结构更复杂,根据其N端转录激活区不同分为TA型和ΔN型[2]。TA型启动子位于第一个外显子上,而ΔN型启动子位于第3个外显子上。C端通过选择性剪接可以产生不同的亚型,称为α、β和γ型[3]。最近有研究表明,p63还存在第三个启动子区,即GTA亚型。p63在皮肤[4]及四肢的发育[5]中发挥重要调控作用,参与肿瘤的形成和转移过程,但其发挥抑癌还是促癌的作用尚存在争议[6]。本课题组前期结果表明,在宫颈癌细胞中p63的主要表达亚型为ΔNp63α[7],但其发挥的功能及调控机制尚不明确。
MicroRNA是一类由内源基因编码的长度约为18~22个核苷酸的非编码小RNA分子,能够在转录后水平调节基因的表达[8]。现有的研究证实其参与调控机体的许多基本生物学过程,如细胞增殖[9]、分化[10]、免疫调节[11]、凋亡[12]等。hsa-let-7b是Let-7家族的重要成员之一,其对肿瘤的调控作用与RAS、c-myc、HMGA2、Lin-28、Dicer等因子有关[13],在不同的组织中发挥不同的功能,有研究表明,在人类许多肿瘤中let-7表达量明显降低[14-17]等,但其在宫颈癌细胞中表达以及与ΔNp63α关系和作用尚不清楚。本研究旨在研究hsa-let-7b与ΔNp63α的关系及其在宫颈癌细胞系中的作用。
1 材料与方法 1.1 材料与试剂宫颈鳞癌细胞株SiHa,该细胞系建自一个日本患者的外科手术的原位组织样品,在裸鼠中,此细胞能形成低分化的表皮样癌(三级)。宫颈鳞癌细胞株ME-180,该细胞株来源于一个细胞集落不规则没有明显角质化的高侵袭性鳞状细胞癌,细胞株中含有人乳头瘤病毒(HPV)DNA,与HPV-39型的同源性高于HPV-18型。本实验的宫颈鳞癌细胞株SiHa、ME-180均购自上海细胞所。SiHa/con、SiHa/p63及ME-180/con、ME-180/Shp63稳转细胞系由本课题组前期构建。DMEM培养液、脂质体Lipofectamine2000、总RNA提取试剂TRIrizol及反转录试剂盒均购自美国Invitrogen公司。McCoy’s 5A Medium Modified、0.25%胰酶-EDTA、青霉素、链霉素购自美国Gibco公司。胎牛血清购自中国依科赛公司。miRNA反转录引物及上、下游引物由中国华大基因科技有限公司合成,引物序列见表 1。FastStart Universal SYBR Green Master(ROX)购自瑞士Roche公司。p63抗体购自美国GeneTex公司。MicroRNA hsa-let-7b-3p对照试剂及刺激剂购自广州锐博公司。PARP-1、β-actin抗体购自美国Santa Cruz公司。Western blot化学发光底物购自美国PIERCE公司。流式细胞周期检测试剂盒购自中国联科生物公司,流式细胞凋亡检测试剂PI、Annexin V购自美国BioLegend公司。
ME-180细胞培养液为McCoy’s 5A Medium Modified(美国Gibco公司)+10%胎牛血清(南美ExCell公司),SiHa细胞为DMEM高糖培养液(美国Hyclone公司)+10%胎牛血清(ExCell)。培养液均含有10 mmol/L HEPES和100 mg/L的氨苄青霉素和链霉素。37℃、5%CO2体积的饱和湿度培养箱中进行培养。用0.25%的胰蛋白酶消化并进行传代,约3天传代1次,取对数生长期的细胞进行实验。
1.2.2 细胞转染取对数生长期的SiHa细胞按照4×105个/孔的密度接种于6孔培养板中培养过夜,待细胞密度约为80%时进行转染,转染前1 h换为不含双抗的10%FBS的新鲜培养液。按照Lipofectaine2000说明书,分别转染20 µmol/L的对照试剂及20 µmol/L的hsa-let-7b-3p刺激剂。转染后8 h换为完全培养液,根据实验目的确定收样时间。
1.2.3 细胞增殖(1)计数法检测细胞增殖:取对数生长期的SiHa细胞进行传代,准确计数,将1 ml含8×104个细胞的细胞悬液接种于12孔板中,待细胞贴壁后,随机分为2组,分别转染hsa-let-7b-3p对照试剂及hsa-let-7b-3p刺激剂(100 nmol/L)。8 h后换液,分别于24、48、72、96 h收集细胞并计数,实验重复3次。(2)流式细胞仪检测细胞周期:取对数生长期的SiHa细胞接种于6孔板中,每孔含4×105个细胞,待细胞贴壁后,随机分为2组,分别转染hsa-let-7b-3p对照试剂及hsa-let-7b-3p刺激剂(100 nmol/L),8 h后换液,用无血清的培养液同步化24 h,更换完全培养液培养24 h后用胰酶消化并收集细胞,用预冷的75%乙醇在-20℃固定24 h。收集固定过夜的细胞,用预冷的1×PBS洗细胞两次,加入300 µl DNA染色液染色,充分吹匀,室温避光30 min,流式细胞仪检测DNA含量,数据用FlowJo 7.6软件进行分析。
1.2.4 实时荧光定量PCR检测miRNA-hsa-let-7b-3p的表达严格按照TRIzol说明书进行操作, 提取细胞总RNA, 取2 µgRNA用hsa-let-7b-3p的特异引物反转录合成cDNA,用SYBR Green进行实时荧光定量PCR仪扩增,以GAPDH为内参,检测hsa-let-7b-3p、miR-193a-3p、miR-34a、miR-4487、miR-4687-3p、miR-372-5p、miR-373-5p、miR-4717-5p、miR-4516的表达量。实验结果在荧光定量操作系统中进行分析对比,采用2-ΔΔCt对以上检测的miRNA进行相对定量。
1.2.5 细胞凋亡的检测(1)Western blot检测细胞凋亡:在SiHa细胞中分别转染hsa-let-7b-3p对照试剂和刺激剂(100 nmol/L),48 h后收集细胞, 加入含有蛋白酶抑制剂和磷酸酶抑制剂的RIPA裂解液,4℃冰箱放置40 min,4℃离心机12 000 r/min离心15 min,取上清液至新的1.5 ml EP管中,BCA定量并调整蛋白浓度,将各个样品浓度调整一致。加蛋白上样缓冲液(×4)和还原剂(1 mol/L DTT或b巯基乙醇),沸水煮10 min,瞬时离心后轻轻弹匀,上样并通过SDS-PAGE电泳、再电转到PVDF膜上,加入5%BSA封闭液封闭1 h后,分别孵育一抗PARP-1(1:1 000)、β-actin(1:3 000),4℃过夜,TBST洗膜后加入相应的二抗孵育1 h,再用TBST洗膜,使用化学发光检测信号显影。
(2)Annexin V/PI法检测细胞凋亡:参照说明书及相关文献,同1.2.3(2)中方法接种细胞,分别转染hsa-let-7b-3p对照试剂及hsa-let-7b-3p刺激剂(100 nmol/L),8 h后换液,转染48 h后,胰酶消化并收集细胞,用预冷的1×PBS缓冲液洗涤细胞两次,再用1×Binding Buffer缓冲液制成每毫升1×106个的悬液,在流式试管中分别加入300 µl细胞悬液,加入5 µl Annexin V染料混匀后室温避光孵育20 min,再加入5 µl PI混匀孵育10 min,流式细胞仪检测细胞凋亡,数据用FlowJo 7.6软件进行分析。
1.3 统计学方法实验数据用GraphPad Prism5进行单因素方差分析和作图,两组间比较采用t检验,以P < 0.05为差异有统计学意义。
2 结果 2.1 SiHa稳定转染ΔNp63α的细胞系中芯片筛出的差异显著基因将SiHa/con、SiHa/p63的细胞样本在上海康成公司进行芯片检测,初筛选出差异大于10倍的基因(P < 0.05),见图 1。
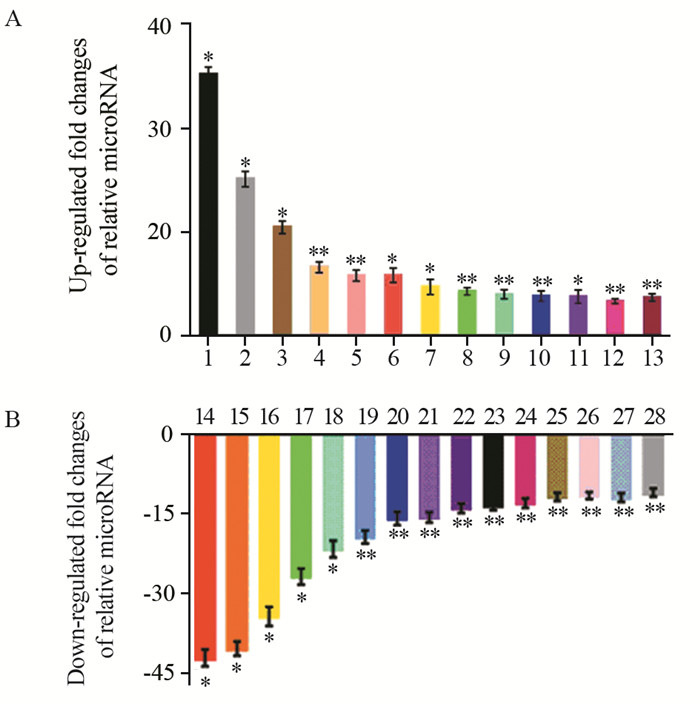
|
| *: P < 0:05, **: P < 0:01, compared with SiHa/con group; A: 1: miR-4639-3p; 2: miR-4487; 3: miR-3926; 4: miR-552-3p; 5: miRPlus-C1110; 6: miR-205-5p; 7: hsa-let-7b-3p; 8: miR-3613-3p; 9: miR-382-3p; 10: miR-3920; 11: miR-4668-5p; 12:miR-125b-5p; 13: miR-222-3p; B: 14: miR-4538; 15: miR-5191;16: miR-564; 17: miR-299-5p; 18: miR-4695-5p; 19: miRPlus-D1058; 20: miR-4717-5p; 21: miR-H4-5p; 22: miR-3126-3p; 23: miR-935; 24: miR-4683; 25: miR-4775; 26: miR-216a-5p; 27: miR-373-5p; 28: miR-490-5p 图 1 芯片结果中变化超过10倍的miRNA Figure 1 miRNAs with more than ten-fold change in microarray results |
通过分析芯片结果,设计相应的miRNA引物,同时在SiHa/con和SiHa/p63细胞系中用qRT-PCR检测相应基因的表达情况。结果发现,在细胞系验证的基因中,有10个miRNA基因的表达情况与芯片结果一致,见图 2。
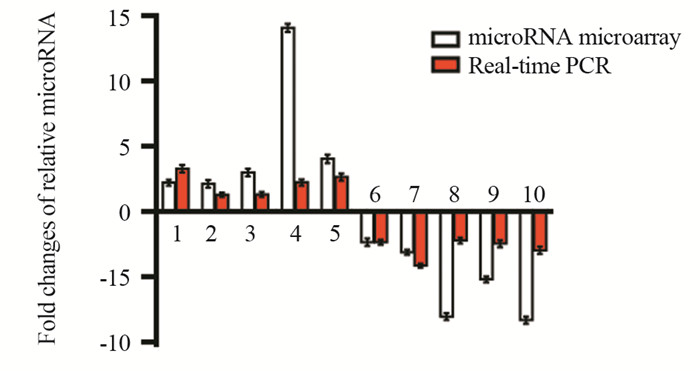
|
| 1: miR-193a-3p; 2: miR-193b-3p; 3: miR-34a; 4: miR-4487; 5: hsa-let-7b-3p; 6: miR-4687-3p; 7: miR-372-5p; 8: miR-373-5p; 9: miR4717-5p; 10: miR-4516 图 2 SiHa/con和SiHa/p63细胞系中与芯片结果一致的10个miRNA Figure 2 Ten miRNAs consistent with the trend of microarray results in SiHa/con and SiHa/p63 cell lines |
在ME-180/con和ME-180/Shp63细胞系中通过qRT-PCR检测上述10个miRNA基因表达情况,发现有5个基因的表达与p63表达变化一致,分别为miR-4717-5p、miR -34a、miR-193a-3p、miR-193b-3p、hsa-let-7b-3p,见图 3。
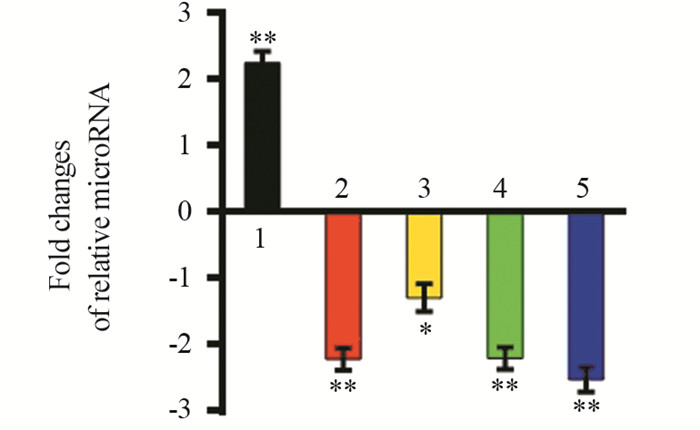
|
| *: P < 0.05, **: P < 0.01, compared with ME-180/con group; 1: miR-4717-5p; 2: miR-34a; 3: miR-193a-3p; 4: miR-193b-3p; 5: hsa-let-7b-3p 图 3 在ME-180/con和ME-180/Shp63细胞系中与芯片结果一致的5个miRNA Figure 3 Five miRNAs consistent with the trend of microarray results in ME-180/con and ME-180/Shp63 cell lines |
在筛选出的基因中,选择与p63表达显著相关的基因hsa-let-7b-3p作为研究对象,qRT-PCR结果显示,在SiHa细胞中转染hsa-let-7b-3p刺激剂(100 nmol/L)后,与对照组相比hsa-let-7b-3p的表达水平显著上调(P < 0.05),且瞬时转染效率至少能够维持72 h,见图 4。

|
| 图 4 qRT-PCR分析SiHa细胞中转染hsa-let-7b-3p刺激剂后let-7b-3p表达情况以及维持时间 Figure 4 Expression and maintenance time of let-7b-3p in SiHa cells transfected with hsa-let-7b-3p mimic analyzed by qRT-PCR |
hsa-let-7b-3p刺激剂刺激SiHa细胞后,发现与对照组相比,SiHa细胞增殖受到抑制(P=0.0247),见图 5A。流式细胞仪对其进行周期检测,结果发现与对照组相比,转染hsa-let-7b-3p刺激剂后,SiHa细胞中细胞周期处于G0/G1期的细胞数目增多(P=0.0365),见图 5B。
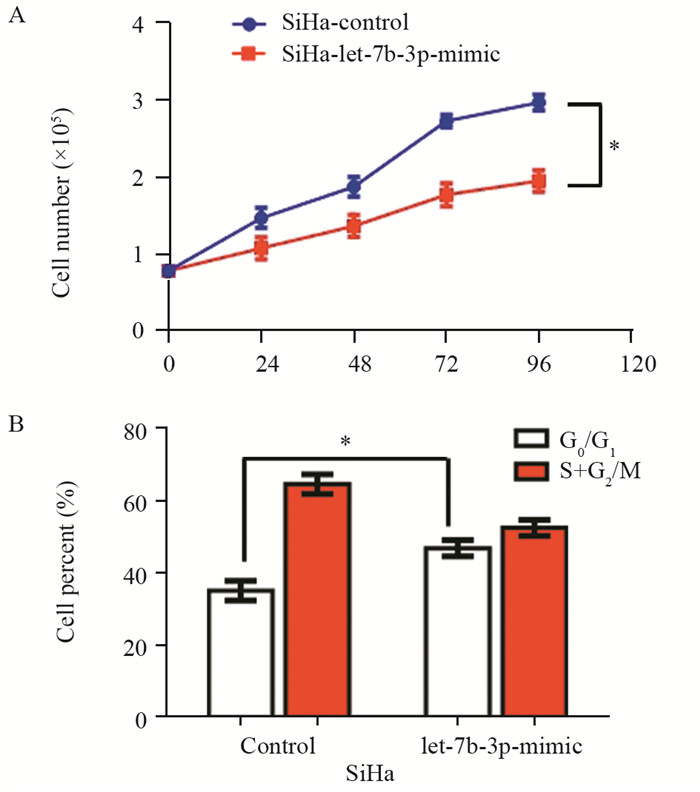
|
| *: P < 0.05, compared with SiHa-control group 图 5 hsa-let-7b-3p对SiHa细胞增殖的影响 Figure 5 Effects of hsa-let-7b-3p on proliferation of SiHa cells |
hsa-let-7b-3p刺激剂刺激SiHa细胞后,Western blot检测结果发现与对照组相比,凋亡蛋白PARP-1的表达显著增加,见图 6A;流式细胞检测结果发现与对照组相比,转染hsa-let-7b-3p刺激剂后,细胞晚期凋亡明显增加,见图 6B~C。
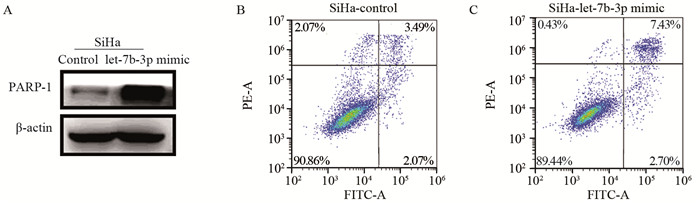
|
| 图 6 hsa-let-7b-3p对SiHa细胞凋亡的影响 Figure 6 Effects of hsa-let-7b-3p on apoptosis of SiHa cells |
Tp63和ΔNp63对真皮和上皮的发生发展至关重要。研究表明在胚胎期沉默p63基因,p63缺陷鼠会出现生长畸形,真皮和表皮组织及四肢发育不全,且在出生时就已死亡。此外,由于p63和p53之间结构的相似性,p63也可能作为DNA损伤的传感器诱导细胞凋亡,并且在用DNA损伤剂处理细胞后,p63的表达明显上调。已有研究发现ΔNp63在卵巢癌[18]、鼻咽癌、头颈癌、肺癌[19]、乳腺癌等组织中表达并未缺失反而增强,而另一方面,在食管癌、膀胱癌、前列腺癌[20]等肿瘤中,p63低表达提示患者转移率较高,预后更差。本研究发现在宫颈鳞癌细胞中,ΔNp63α可正向或负向调控许多miRNA的表达,如miR-34a、miR-193a等,提示ΔNp63α可能通过调控不同的miRNA[21-22]产生不同的功能,从而在肿瘤的发生发展中起着重要的作用。
MicroRNA在真核生物细胞中广泛存在,通过与靶mRNA的3’功能端非翻译区(3’ untranslated region,3’-UTR区)部分或完全互补配对,从而在翻译后水平调节其靶基因的表达[23]。在很多研究中发现Let-7家族在肿瘤中表达下调,近期有研究表明它在乳腺癌中表达显著上调[24]。本研究发现,过表达ΔNp63α的SiHa细胞株和ΔNp63α沉默的ME-180细胞株中,hsa-let-7b-3p的表达都发生明显变化,其中前者hsa-let-7b-3p表达上升,后者hsa-let-7b-3p表达下降,说明ΔNp63α可以调控hsa-let-7b-3p的表达且呈正性相关,但具体机制尚不清楚。下一步计划通过构建hsa-let-7b-3p的启动子报告基因质粒,来研究ΔNp63α对其启动子的调控作用。hsa-let-7b是Let-7家族中的重要成员之一,Let-7在抑制肿瘤细胞的增殖、侵袭、转移以及促进肿瘤细胞的凋亡中发挥重要作用。本研究,通过向宫颈鳞癌SiHa细胞中转染hsa-let-7b-3p刺激剂,促进hsa-let-7b-3p的表达,发现hsa-let-7b-3p能够抑制SiHa细胞的增殖,促进细胞的凋亡,提示hsa-let-7b-3p作为ΔNp63α的下游靶基因,在宫颈癌细胞中主要起抑癌作用。hsa-let-7b-3p对于宫颈癌细胞增殖抑制作用的机制有待进一步研究。有研究报道,在人胶质母细胞瘤U251和U87细胞系中,hsa-let-7b可以通过与E2F2的3’-UTR区结合来抑制细胞的增殖、转移和侵袭[25]。今后将进一步研究hsa-let-7b对宫颈癌细胞侵袭、转移的影响及其机制。在细胞凋亡过程中,hsa-let-7b-3p通过增加PARP-1的表达来促进宫颈癌细胞的凋亡。
综上,ΔNp63α可以正向调控hsa-let-7b-3p的表达,而hsa-let-7b-3p的表达上升又会抑制宫颈癌细胞的增殖,促进宫颈癌细胞的凋亡。所以hsa-let-7b-3p可以作为一个潜在的治疗靶点,为宫颈癌的诊治带来更多的临床价值,其具体机制尚需进一步完善。
| [1] | Missero C, Antonini D. Crosstalk among p53 family members in cutaneous carcinoma[J]. Exp Dermatol, 2014, 23(3): 143–6. DOI:10.1111/exd.12320 |
| [2] | Gonfloni S, Caputo V, Iannizzotto V. P63 in health and cancer[J]. Int J Dev Biol, 2015, 59(1-3): 87–93. |
| [3] | Candi E, Smirnov A, Panatta E, et al. Metabolic pathways regulated by p63[J]. Biochem Biophys Res Commun, 2017, 482(3): 440–4. DOI:10.1016/j.bbrc.2016.10.094 |
| [4] | Paris M, Rouleau M, Pucéat M, et al. Regulation of skin aging and heart development by TAp63[J]. Cell Death Differ, 2012, 19(2): 186–93. DOI:10.1038/cdd.2011.181 |
| [5] | Kawata M, Taniguchi Y, Mori D, et al. Different regulation of limb development by p63 transcript variants[J]. PLoS One, 2017, 12(3): 1–19. |
| [6] | Ferone G, Mollo MR, Missero C. Epidermal cell junctions and their regulation by p63 in health and disease[J]. Cell Tissue Res, 2015, 360(3): 513–28. DOI:10.1007/s00441-014-2108-1 |
| [7] | Coimbra EC, DA Conceição Gomes Leitão M, Júnior MR, et al. Expression Profile of MicroRNA-203 and its DeltaNp63 Target in Cervical Carcinogenesis: Prospects for Cervical Cancer Screening[J]. Anticancer Res, 2016, 36(8): 3939–46. |
| [8] | Oliveto S, Mancino M, Manfrini N, et al. Role of microRNAs in translation regulation and cancer[J]. World J Biol Chem, 2017, 8(1): 45–56. DOI:10.4331/wjbc.v8.i1.45 |
| [9] | Rodriguez RE, Schommer C, Palatnik JF. Control of cell proliferation by microRNAs in plants[J]. Curr Opin Plant Biol, 2016, 34: 68–76. DOI:10.1016/j.pbi.2016.10.003 |
| [10] | Kim DY, Sung JH. Regulatory role of microRNAs in the proliferation and differentiation of adipose-derived stem cells[J]. Histol Histopathol, 2016, 32(1): 1–10. |
| [11] | Cai Y, Shen J. Modulation of host immune responses to Toxoplasma gondii by microRNAs[J]. Parasite Immunol, 2017, 39(2): 1–13. |
| [12] | Wang J, Liew OW, Richards AM, et al. Overview of MicroRNAs in Cardiac Hypertrophy, Fibrosis, and Apoptosis[J]. Int J Mol Sci, 2016, 17(5): 1–21. |
| [13] | Bao MH, Feng X, Zhang YW, et al. Let-7 in cardiovascular diseases, heart development and cardiovascular differentiation from stem cells[J]. Int J Mol Sci, 2013, 14(11): 23086–102. DOI:10.3390/ijms141123086 |
| [14] | Joosse SA, Müller V, Steinbach B, et al. Circulating cell-free cancer-testis MAGE-A RNA, BORIS RNA, let-7b and miR-202 in the blood of patients with breast cancer and benign breast diseases[J]. Br J Cancer, 2014, 111(5): 909–17. DOI:10.1038/bjc.2014.360 |
| [15] | Yang X, Cai H, Liang Y, et al. Inhibition of c-Myc by let-7b mimic reverses mutidrug resistance in gastric cancer cells[J]. Oncol Rep, 2015, 33(4): 1723–30. DOI:10.3892/or.2015.3757 |
| [16] | Peng J, Mo R, Ma J, et al. let-7b and let-7c are determinants of intrinsic chemoresistance in renal cell carcinoma[J]. World J Surg Oncol, 2015, 13: 1–8. DOI:10.1186/1477-7819-13-1 |
| [17] | Chung YW, Bae HS, Song JY, et al. Detection of microRNA as novel biomarkers of epithelial ovarian cancer from the serum of ovarian cancer patients[J]. Int J Gynecol Cancer, 2013, 23(4): 673–9. DOI:10.1097/IGC.0b013e31828c166d |
| [18] | Kim S, Lee JJ, Heo DS. PPARγ ligands induce growth inhibition and apoptosis through p63 and p73 in human ovarian cancer cells[J]. Biochem Biophys Res Commun, 2011, 406(3): 389–95. DOI:10.1016/j.bbrc.2011.02.052 |
| [19] | 张莉萍, 候立坤, 谢惠康, 等. p63、p40、ck5/6在小细胞肺癌中的表达及其意义[J]. 中华病理学杂志, 2015, 44(9): 644–7. [ Zhang LP, Hou LK, Xie HK, et al. Expression of p63, p40 and CK5/6 in small cell lung cancer[J]. Zhonghua Bing Li Xue Za Zhi, 2015, 44(9): 644–7. ] |
| [20] | Yang K, Wu WM, Chen YC, et al. ΔNp63α Transcriptionally Regulates the Expression of CTEN That Is Associated with Prostate Cell Adhesion[J]. PLoS One, 2016, 11(1): 1–20. |
| [21] | Ng WL, Chen G, Wang M, et al. OCT4 as a target of miR-34a stimulates p63 but inhibits p53 to promote human cell transformation[J]. Cell Death Dis, 2014, 5: 1–10. |
| [22] | Ory B, Ramsey MR, Wilson C, et al. A microRNA-dependent program controls p53-independent survival and chemosensitivity in human and murine squamous cell carcinoma[J]. J Clin Invest, 2011, 121(2): 809–20. DOI:10.1172/JCI43897 |
| [23] | Rupaimoole R, Slack FJ. MicroRNA therapeutics: towards a new era for the management of cancer and other diseases[J]. Nat Rev Drug Discov, 2017, 16(3): 203–22. DOI:10.1038/nrd.2016.246 |
| [24] | Encarnacion J, Ortiz C, Vergne R, et al. High DRC Levels Are Associated with Let-7b Overexpression in Women with Breast Cancer[J]. Int J Mol Sci, 2016, 17(6): 1–14. |
| [25] | Song H, Zhang Y, Liu N, et al. Let-7b inhibits the malignant behavior of glioma cells and glioma stem-like cells via downregulation of E2F2[J]. J Physiol Biochem, 2016, 72(4): 733–44. DOI:10.1007/s13105-016-0512-6 |
 2018, Vol. 45
2018, Vol. 45



