文章信息
- 骨肉瘤miRNA分子共调控网络的构建
- Construction of Gene-coregulation-network for Osteosarcoma MicroRNA
- 肿瘤防治研究, 2017, 44(9): 601-606
- Cancer Research on Prevention and Treatment, 2017, 44(9): 601-606
- http://www.zlfzyj.com/CN/10.3971/j.issn.1000-8578.2017.17.0195
- 收稿日期: 2017-02-27
- 修回日期: 2017-07-07
骨肉瘤(osteosarcoma)是骨科最常见的恶性肿瘤[1],其发生、发展是一个复杂的、涉及多基因调控网络失衡导致的细胞异常代谢的结果。基因表达受转录水平及翻译水平的调控,转录因子(transcription factors, TFs)及microRNAs(miRNAs)在调控基因的表达方面扮演了重要的角色。TFs和miRNAs之间存在广泛的相互作用和合作调控,共同组成复杂的调控网络,对于了解细胞的生理过程、生物学功能、疾病的发生机制等都起到了重要作用。目前面临的首要难题是骨肉瘤往往不能做到早期诊断,诊断不及时会耽误治疗时机,严重影响患者预后[2-3]。因此,建立新的、有效的骨肉瘤防治措施势在必行,还需寻找并发现新的功能基因靶向治疗骨肉瘤,根据治疗效果建立分子水平预后指标,开发安全的不同于传统治疗骨肉瘤的放化疗药物,是当前科研及临床研究中的热门话题[4-5]。本研究将利用高通量基因芯片技术分析骨肉瘤中差异表达的基因和miRNAs,结合生物信息学软件预测与调控骨肉瘤相关的TFs、miRNAs及其靶基因,分析调控网络中的关键基因及关键miRNAs的功能,构建骨肉瘤发生发展的“TFs-miRNAs”共调控网络。
1 材料与方法 1.1 miRNA数据来源收集美国国家生物技术信息中心(National Center of Biotechnology Information, NCBI)维护的GEO数据库中的两组数据库(GPL8227、GPL19631)中所有骨肉瘤miRNA数据,收集数据时间截至2016年12月31日。
1.2 数据库资源用基因本体论数据库(gene ontology, GO)对所分析得到的差异表达基因进行功能注释;使用京都基因与基因组百科全书(kyoto encyclopedia of genes and genomes, KEGG)数据库对差异表达基因进行代谢通路富集分析;使用转录作用元件数据库(transcriptional regulatory element database, TRED)对差异表达基因进行转录因子筛选。
1.3 方法 1.3.1 差异表达基因筛选及注释为探究骨肉瘤细胞发生发展的分子机制,用R语言下载GEO数据库中GSE28425、GSE65071数据集的原始数据。使用MAS 5.0算法对数据集中的原始数据进行标准化预处理。使用Limma程序包中所提供的统计分析函数,计算与健康者细胞系相比,骨肉瘤患者中表达水平发生变化的基因。选取差异表达倍数≥2.0(Fold Change≥2.0)为筛选阈值,筛选获得差异表达的miRNA。通过差异表达分析获得两组平台共有的骨肉瘤细胞系表达水平发生显著性改变的miRNA,利用DIANA TOOLS数据库中收录的人类基因信息,找到具有显著差异的miRNA靶基因。利用R语言,将筛选得到的差异表达基因与GEO数据库提供的GPL平台注释信息进行匹配,对差异表达基因进行信息注释。
1.3.2 差异表达基因KEGG代谢通路富集分析为进一步探究差异表达基因参与生物代谢的代谢通路,对筛选得到的差异表达基因进行KEGG代谢通路富集分析。提取差异表达基因GPL平台注释信息中的“Entrez_Gene_ID”信息,应用R语言KEGG程序包与KEGG数据库信息,对每一个差异表达基因向已知的代谢通路进行富集映射,探究差异表达基因集中映射的细胞代谢通路,初步推测骨肉瘤所影响的主要代谢通路,并筛选出与肿瘤通路关系密切的基因,进行GO功能注释分析。
1.3.3 差异表达基因GO功能注释分析为探究差异表达且与肿瘤通路关系密切的基因所参与的生物功能,对筛选得到的与肿瘤通路关系密切的差异表达基因进行GO功能注释分析。提取差异表达基因GPL平台注释信息中的“Entrez_Gene_ID”信息,使用R语言GO程序包与GO数据库信息,对每一个差异表达基因进行功能注释,探究骨肉瘤组织与正常组织差别的基因功能,初步推测骨肉瘤作用的分子机制。
1.3.4 分子共调控网络构建为进一步挖掘骨肉瘤的分子调控模式,探究差异表达基因之间的相互调控作用关系,提取TRED数据库中所收录的所有人类转录因子数据。通过Pearson相关性分析,提取基因间分子共调控作用关系。使用Cytoscape version 3.4.0软件对得到的分子共调控作用关系进行可视化展示,构建出最终的分子共调控网络。
2 结果 2.1 差异表达基因的筛选本研究分别从GPL8227、GPL19631两组数据库中筛选出差异表达miRNA 232个(上调129个、下调103个)和265个(上调205个、下调60个)。共筛选得到两组均包含的差异表达miRNA52个,其中31个miRNA上调表达,21个miRNA下调表达见表 1。
将筛选出的52个差异表达miRNA结合DIANA TOOLS数据库中收录的人类基因信息进行分析。其中KEGG代谢通路富集分析结果见图 1,差异表达基因主要参与肿瘤相关的诸多代谢通路。差异表达的52个miRNA的整体调控模式及每个miRNA所调控的代谢通路情况见图 2。肿瘤代谢通路的分子调控关系网络见图 3。
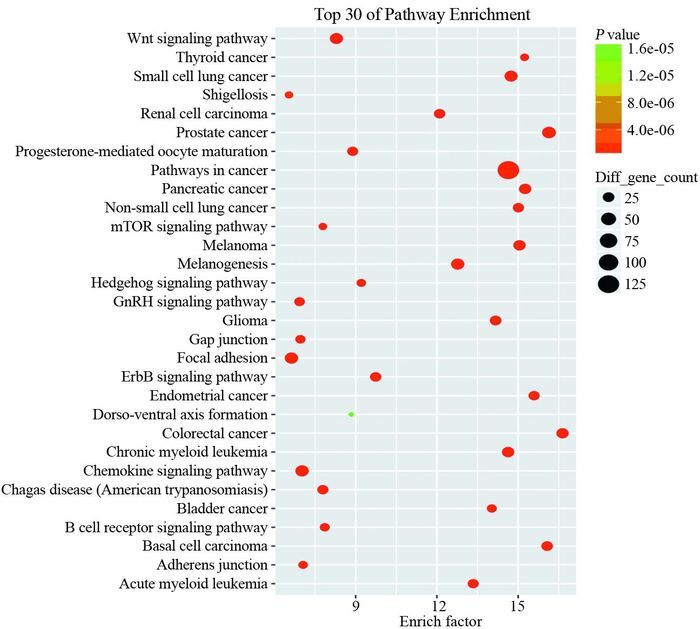
|
| 图 1 差异表达基因KEGG代谢通路富集分析 Figure 1 KEGG metabolic pathway enrichment analysis of differentially-expressed genes |
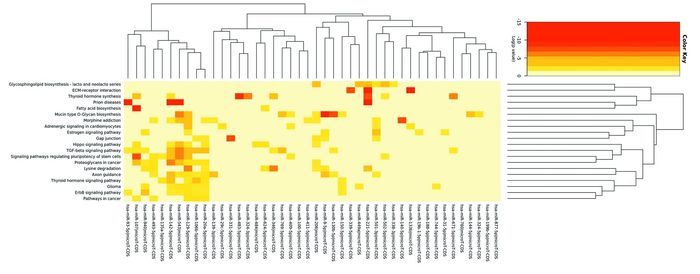
|
| 图 2 差异表达52个miRNA聚类分析热图 Figure 2 Heat map of clustering analysis of 52 differentially-expressed miRNAs |
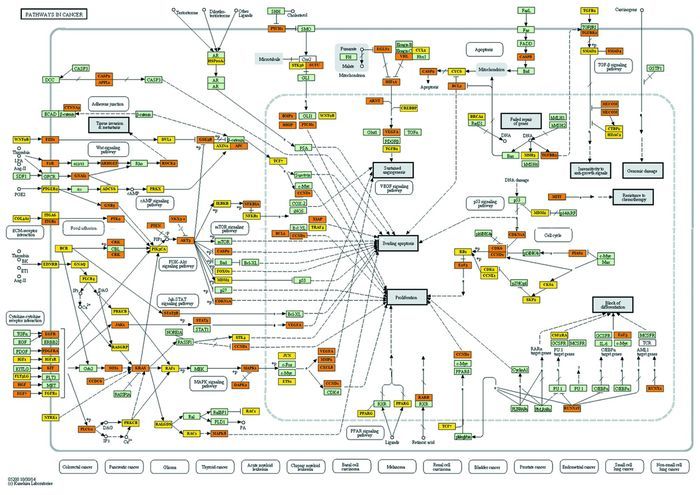
|
| 图 3 差异表达miRNA靶基因肿瘤代谢通路 Figure 3 Tumor metabolic pathway of differentially-expressed miRNA target genes |
将上述筛选出与肿瘤通路关系最紧密的5个miRNA及其靶基因进行GO功能注释分析。注释分析结果显示这些基因主要参与维生素E的代谢路径(response to vitamin E)、RNA聚合酶Ⅱ启动子的转录调控反应(regulation of transcription from RNA polymerase Ⅱ promoter in response etc)、调控神经元的成熟(regulation of neuron maturation)、受体激动剂活性(receptor agonist activity)、正调控血管内皮生长因子受体活性(positive regulation of vascular endothelial growth factor receptor activity)、通过MAPK级联非经典Wnt信号途径(non-canonical wnt signaling pathway via MAPK cascade)、间充质上皮细胞信号转导(mesenchymal-epithelial cell signaling)、Ⅳ型胶原(collagen type Ⅳ)、细胞对生长激素刺激的反应(cellular response to growth hormone stimulus)、蛋白激酶活性的激活(activation of protein kinase A activity)等生物功能,见图 4。
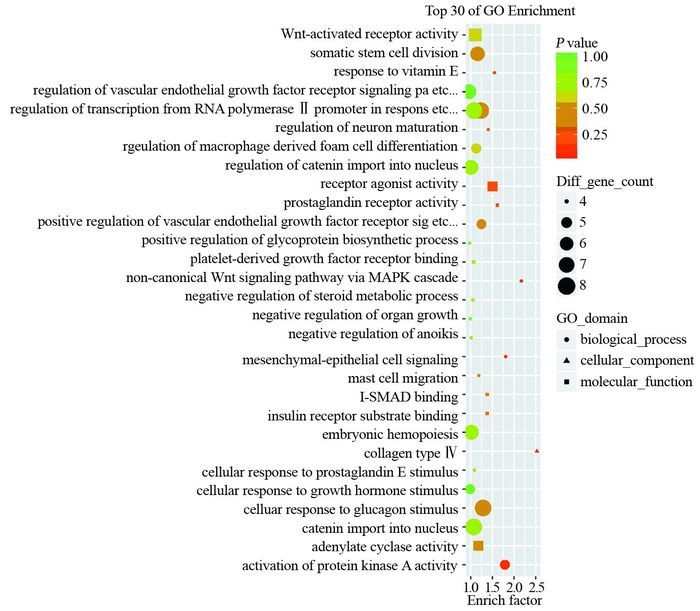
|
| 图 4 差异表达基因GO功能注释分析 Figure 4 GO functional annotation analysis of differentially-expressed genes |
最后基于与肿瘤通路密切相关的差异表达基因及TRED数据库中所收录的人类转录因子信息,对差异表达基因进行分子偶联,构建了分子共调控网络,见图 5。
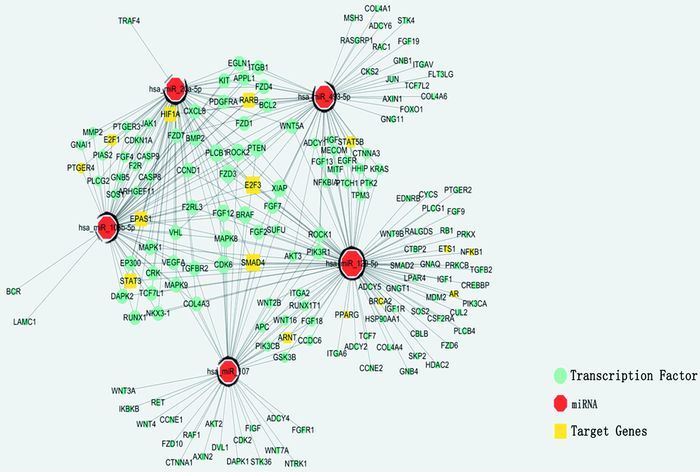
|
| 图 5 miRNA调控模式图 Figure 5 miRNA regulation pattern |
目前miRNA被广泛认为是血液学和肿瘤组织的生物标志物。miRNA有可能通过扭曲、突变、成纤维细胞生长因子(FGF)和骨形态发生蛋白(BMP)信号参与骨的形成。研究发现miRNA与多种骨科疾病有关系,比如有研究认为miRNA-140与骨性关节炎有关,miRNA-503、miRNA-3960与骨质疏松有关,miRNA-34、miRNA-146等与类风湿关节炎有关[6]。研究表明众多的miRNA在骨肉瘤发病的机制中异常表达,被miRNA决定的许多基因被上调或下调。比如Karbasy等[7]通过研究发现miR-125b和miR-300的下调与骨肉瘤的进展相关且两者都起到抑癌基因的作用。Goudarzi等[8]通过研究发现miR-185上调miR-218下调与软骨肉瘤的进展相关联,并且两者均可以作为软骨肉瘤的抑癌基因。Bahador等[9]通过研究发现在青少年、儿童以及青年人骨肉瘤患者中,如果miR-29b和miR-422a的表达下调往往表明患者的预后较差。Taheriazam等[10]通过研究发现下调microRNA-26a和微小RNA-27A的上调有助于判断人类骨肉瘤的攻击性进展。高表达的miR-130b水平和miR-218的低表达与差的临床病理特征有关。此外,有研究表明miR-130b可对预测骨肉瘤的进展起关键作用[10-11]。另有研究表明miR-145、miR-128、miR-34a在骨肉瘤中的表达均发生了改变。miR-335、miR-128、miR-340、miR-214等均与骨肉瘤的侵袭性有重要关系,表达增高后预示骨肉瘤的恶性程度较高,可以较早发生转移,患者的生存期缩短[12]。并且近期有研究表明血浆中miR-34b表达下降可能与骨肉瘤已经发生转移有密切的联系[13],如果能明确这种作用关系会对骨肉瘤的早期诊断起到非凡的作用。就目前研究现状来看骨肉瘤发生发展的分子作用机制尚不十分明确,本研究利用高通量基因芯片技术,通过生物信息学手段,系统性预测构建了一张与肿瘤通路关系密切的TF、miRNA及其靶基因共调控网络蓝图。该分子共调控网络从整体的角度鸟瞰与肿瘤通路关系密切的miRNA及其靶基因的相互作用关系,展示了差异表达基因主要参与肿瘤相关的诸多代谢通路及与肿瘤通路有关的基因主要参与的生物功能。为骨肉瘤诊断标志物簇的筛选提供可靠的选择方向;为临床诊断及预后分析分子靶点的筛选提供实用工具,同时也为其他癌症的同类研究提供新思路。
| [1] | David JP, Mehic D, Bakiri L, et al. Essential role of RSK2 in c-Fos-dependent osteosarcoma development[J]. J Clin Invest, 2005, 115(3): 664–72. DOI:10.1172/JCI200522877 |
| [2] | Weber KL. What's new in musculoskeletal oncology[J]. J Bone Joint Surg Am, 2005, 87(6): 1400–10. |
| [3] | Witlox MA, Lamfers ML, Wuisman PI, et al. Evolving gene therapy approaches for osteosarcoma using viral vectors: Review[J]. Bone, 2007, 40(4): 797–812. DOI:10.1016/j.bone.2006.10.017 |
| [4] | Patanè S, Avnet S, Coltella N, et al. MET overexpression turns human primary osteoblasts into osteosarcomas[J]. Cancer Res, 2006, 66(9): 4750–7. DOI:10.1158/0008-5472.CAN-05-4422 |
| [5] | Lee N, Smolarz AJ, Olson S, et al. A potential role for Dkk-1 in the pathogenesis of osteosarcoma predicts novel diagnostic and treatment strategies[J]. Br J Cancer, 2007, 97(11): 1552–9. DOI:10.1038/sj.bjc.6604069 |
| [6] | Bassampour SA, Abdi R, Bahador R, et al. Downregulation of miR-133b/miR-503 acts as efficient prognostic and diagnostic factors in patients with osteosarcoma and these predictor biomarkers are correlated with overall survival[J]. Tumor Biol, 2015. |
| [7] | Karbasy SH, Taheriazam A, Mirghasemi A, et al. Upregulation of miR-300 and downregulation of miR-125b act as potential predictor biomarkers in progression, metastasis, and poor prognosis of osteosarcoma[J]. Tumor Biol, 2015. |
| [8] | Goudarzi PK, Taheriazam A, Asghari S, et al. Downregulation of miR-185 and upregulation of miR-218 expression may be potential diagnostic and prognostic biomarkers of human chondrosarcoma[J]. Tumor Biol, 2016, 37(5): 5775–9. DOI:10.1007/s13277-015-4090-y |
| [9] | Bahador R, Taheriazam A, Mirghasemi A, et al. Tissue expression levels of miR-29b and miR-422a in children, adolescents, and young adults' age groups and their association with prediction of poor prognosis in human osteosarcoma[J]. Tumor Biol, 2016, 37(3): 3091–5. DOI:10.1007/s13277-015-4140-5 |
| [10] | Taheriazam A, Bahador R, Karbasy SH, et al. Down-regulation of microRNA-26a and up-regulation of microRNA-27a contributes to aggressive progression of human osteosarcoma[J]. Diagn Pathol, 2015, 10: 166. DOI:10.1186/s13000-015-0400-3 |
| [11] | Taheriazam A, Talaei AJ, Jamshidi M, et al. Up-regulation of miR-130b expression level and down-regulation of miR-218 serve as potential biomarker in the early detection of human osteosarcoma[J]. Diagn Pathol, 2015, 10: 184. DOI:10.1186/s13000-015-0422-x |
| [12] | Alexeyenko A, Alkasalias T, Pavlova T, et al. Confrontation of fibroblasts with cancer cells in vitro: gene network analysis of transcriptome changes and differential capacity to inhibit tumor growth[J]. J Exp Clin Cancer Res, 2015, 34: 62. DOI:10.1186/s13046-015-0178-x |
| [13] | Wang TY, Zhang QQ, Zhang X, et al. The effect of recombinant lentiviral vector encoding miR-145 on human esophageal cancer cells[J]. Tumour Biol, 2015, 36(12): 9733–8. DOI:10.1007/s13277-015-3743-1 |
 2017, Vol. 44
2017, Vol. 44



