文章信息
- 乳腺癌复发相关基因预测疾病的特异生存率研究
- Prediction of Disease Specific Survival Rates by Genes Associated with Relapse of Breast Cancer
- 肿瘤防治研究, 2016, 43(9): 762-766
- Cancer Research on Prevention and Treatment, 2016, 43(9): 762-766
- http://www.zlfzyj.com/CN/10.3971/j.issn.1000-8578.2016.09.007
- 收稿日期: 2015-10-12
- 修回日期: 2016-01-05
2. 545006 柳州,广西科技大学第二附属医院外三科
2. Department of Third Surgical, The Second Affiliated Hospital of Guangxi University of Science and Technology, Liuzhou 545006, China
复发转移是影响乳腺癌生存预后的关键因素,乳腺癌患者一旦出现复发,不仅大大增加治疗难度,还是导致乳腺癌患者死亡的主要原因[1]。目前乳腺癌早期诊断的主要手段为临床乳腺检查、超声成像、乳房钼靶X线和磁共振等[2]。但由于肿瘤的发生发展和转移是一个多因素、多基因参与的复杂生理过程,常规临床和病理检查很难在早期检测到复发的发生。因此,通过基因芯片技术探讨乳腺癌复发基因组水平的表达变化,找寻复发转移相关的分子生物学标记,将有利于提高乳腺癌复发的早期检测成功率,为乳腺癌的临床诊断和治疗提供更好的帮助。
1 资料与方法 1.1 芯片数据本研究采用的基因芯片表达谱数据来自美国国立生物技术信息中心(NCBI)的公共数据库GEO(gene expression omnibus),用于筛选复发相关基因的芯片数据编号分别为GSE1456(n=159)和GSE2034(n=286)。GSE1456数据中提供的临床指标只有组织分级,包含复发样本40例,其中组织学分级为Ⅰ级2例、Ⅱ级14例、Ⅲ级22例、分级缺失2例;GSE2034提供的临床指标则只有淋巴结转移状况(均为阴性),包含复发样本107例。GSE1456作为预测疾病特异生存率的芯片数据。研究中表达谱数据芯片平台均为Affymetrix Human Genome U133A Array。
1.2 乳腺癌复发相关基因的筛选芯片数据的统计学分析采用BRB-ArrayTools(Version4.2.1)软件包来进行,分别对GSE1456和GSE2034进行复发相关差异基因筛选。为获得质量可靠的信号点,采用倍值过滤和表达水平过滤等方法去除两类样本的基因中位数值小于2倍、表达值小于100的数据,合并相同基因的探针。将通过滤过标准的基因进行两样本非配对样本t检验(P<0.001,FDR<0.01),1 000次随机进行多元置换检验(Multivariate Permutations test),筛选复发和不复发两组肿瘤样本之间的差异表达基因。将GSE1456和GSE2034两组数据集中得到的复发相关差异表达基因进行交集,筛选共同的差异表达基因。
1.3 生存分析及预测将筛选得到的复发相关差异基因作为候选基因,以疾病特异生存时间作为生存时间变量,是否死亡作为生存状态,采用Cox比例风险模型和Wald Statistic,在BRB-ArrayTools中对基因表达进行单因素分析,在表达谱数据GSE1456中进一步筛选出与生存显著相关的基因(P<0.001),用Kaplan-Meler法作图分析这些基因表达对特异生存率的影响,通过这些基因的表达情况和权重计算预后指数,以判断待测样本的风险,用惩罚Cox模型对样本进行生存预测。采用100次随机对数秩检验(Log rank test),留一法(Leave-one-out)进行交叉验证,将获得的基因分类器用于预测样本的预后,将样本分为低风险和高风险两组。
1.4 基因功能注释通过在线分析工具GATHER(http://gather.genome.duke.edu/)对生存分析相关的基因进行基因本体(Gene Ontology,GO)和KEGG通路分析等方面的功能注释,找到这些基因相应的生物学功能及其对应的生物学通路,从而探讨这些基因对乳腺癌预后可能存在的影响机制。
1.5 分类器基因编码蛋白相互作用网络将筛选得到的分类器基因录入蛋白相互作用数据库分析工具STRING(http://string.embl.de),通过已知及预测的蛋白相互作用分析这些基因编码蛋白之间的相互作用,得到蛋白相互作用网络图。
2 结果 2.1 复发相关差异基因的获得通过初步数据过滤,共有757个基因符合过滤标准。然后根据乳腺癌是否复发的两组乳腺癌样本进行随机方差模型的两样本t检验,两组样本间表达倍数大于2的探针为差异表达基因,按此标准经过交集后,两组数据集中获得了76个共同差异表达基因,这些基因作为基因分类器的候选基因。
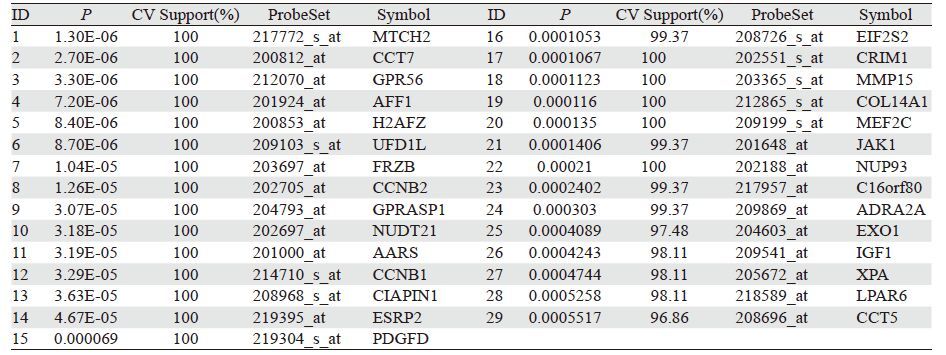
2.2 风险预测基因分类器的获得通过留一法交叉验证,差异表达基因中共有29个基因满足Cox比例风险模型,见表 1,由于在分析中只将样本分为高风险和低风险两个组,因此,构建风险分组的预后指数百分数为50,样本的预后指数大于1即为高风险,反之小于1则为低风险。风险分组的Kaplan-Meier曲线见图 1,从图中可以看出,分类器中的29个基因可以将乳腺癌样本明显分为高风险和低风险组。随时间变化的Cox曲线的时间界标为7.05,见图 2,曲线下面积为0.803,提示分类器的预测结果较为理想。如表 1所示,用于预测分类器中的基因交叉验证(cross validation,CV)准确率达均高于96%,其中有20个基因的交叉验证准确率为100%。

|
| 图 1 基因分类器预测乳腺癌样本的生存曲线 Figure 1 Survival curves of breast cancer by gene classifier |

|
| 图 2 分类预测的ROC曲线 Figure 2 ROC curve of classification |
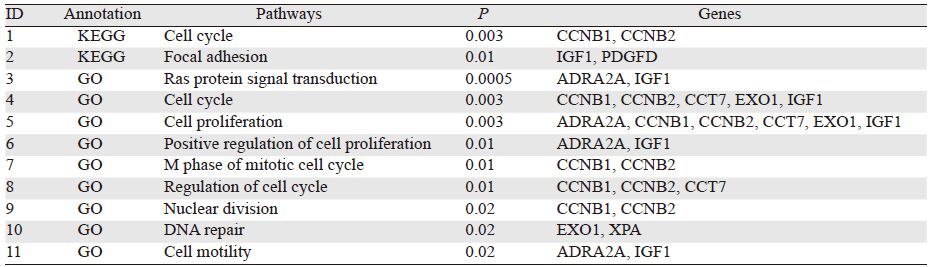
在线分析工具GATHER选择了KEGG Pathway、Gene Ontology两种通路对基因分类器中的基因进行相应的生物学功能注释和通路分析,见表 2,这些基因中富集到涉及细胞周期相关的通路共有4条,参与的基因主要有CCNB1、CCNB2、CCT7、EXO1、IGF1;这5个基因以及ADRA2A还与两条细胞增殖的通路相关。IGF1、PDGFD、ADRA2A与细胞运动和黏着密切相关。另外,EXO1、XPA与DNA修复关系密切,ADRA2A、IGF1则参与Ras蛋白信号的转导通路。
将分类器中的29个基因用STRING进行分析其编码蛋白质之间的相互作用,见图 3。从图中可以看出,这些蛋白的相互作用主要集中在AARS、CCT7、CCT5、EIF2S2、H2AFZ、CCNB2、CCNB1、EXO1和MEF2C等9个蛋白,其他蛋白之间则基本没有相互作用或者比较分散。这些基因基本都存在于P53、细胞凋亡、细胞周期等相关的通路当中,见表 2。其中,CCNB2、CCNB1和EXO1均存在于细胞周期调控的生物学通路中,CCT7、CCT5、H2AFZ、CCNB2、CCNB1、EXO1等6个基因在比较重要的节点位置。

|
| Line thickness indicated the strength of protein-protein interaction 图 3 STRING分析的蛋白相互作用 Figure 3 Protein-protein interactions analyzed by STRING |
复发与乳腺癌的预后关系密切,复发通常是造成乳腺癌预后差的主要原因。因此,与复发有关的基因的表达可能对乳腺癌的预后造成一定的影响。而且,肿瘤的发生和发展是多基因多步骤的过程,是许多癌基因表达失常或抑癌基因失活所致[3]。有研究表明仅靠单一的标志物在检测时很难同时兼顾特异性和敏感度,因而很有必要联合多个检测标记进行诊断预测[4]。基因芯片技术可以大规模平行检测不同样本的基因表达变化,从基因组水平发现全部基因在不同生理状态下的差异表达情况,已有不少研究通过整合不同的芯片数据对肿瘤进行分类预测得到较好的预测模型[5-6]。因此,通过分析基因芯片表达谱数据,应该可以探讨乳腺癌复发与特异生存率之间的关系,并能从中筛选出相关的分子标记。本研究选用了两个独立的乳腺癌表达谱数据来筛选与复发相关的差异基因,在此基础上用其中一个数据集作为预测疾病特异生存率。研究中的表达谱数据均为同一芯片平台,可以减少由于平台不同造成的误差,且GSE2034中的样本均为淋巴结阴性,提示淋巴结转移阴性同样存在复发风险,通过基因表达谱分析找到这些潜在的复发分子标记,利用GSE1456进行疾病特异生存率预测,更能体现复发相关的基因与乳腺癌预后的直接关系,从而为乳腺癌的早期诊断和预后提供更大帮助。从结果上看,通过基因芯片数据处理获得的复发相关基因用于预测乳腺癌疾病特异生存率,可以获得较高的预测准确率。通过应用曲线下面积对ROC曲线的特性进行评价,AUC可以达到0.803,由于曲线越接近左上角,AUC越大,诊断的效果越可靠[7]。因此表明用与复发相关的基因作为分类器进行预后分类预测效果较为理想。另外,本研究整合了相同芯片平台的两个完全独立的样本数据集进行,大大减少了样本预测所受的干扰,保证预测的可行性。
抵抗细胞死亡、潜力无限的复制能力、组织浸润和转移、基因组不稳定和突变等这些都是肿瘤细胞的主要特征[8]。本研究中,对分类器中的基因进行功能注释也表明,这些标记基因主要与细胞周期及调控、细胞增殖及调控、核分裂等分裂增殖相关的生物学功能关系密切;这些通路的改变与肿瘤的抗凋亡、无限增殖的特征相吻合。此外,还有部分基因与细胞运动、黏着密切相关以及DNA修复关系密切,这与肿瘤细胞的组织浸润和转移、基因组不稳定的特征相符。Ras蛋白是所有真核细胞中细胞生长的重要调控因子,研究表明多种肿瘤细胞中都出现了Ras突变[9],而且Ras通路异常也与乳腺癌关系密切[10]。这些结果充分说明筛选出来的基因具有明显的肿瘤特征,应该可以作为肿瘤筛选的标记。
其中,在蛋白相互作用网络且处于比较重要节点位置的CCT7、CCT5、H2AFZ、CCNB1、CCNB2、EXO1等6个基因中,已有研究表明CCT5与淋巴结阳性的乳腺癌转移和复发关系密切[11];CCNB1和CCNB2都是细胞周期蛋白,CCNB1在乳腺的良性瘤和恶性瘤中的表达有显著差异[12],有生物信息学分析表明用CCNB1可以很好预测乳腺癌的无病生存率、无复发生存率和无远处转移生存率[13];CCNB2表达水平与乳腺癌特异生存率显著相关(P<0.001),对预测预后差的乳腺癌是一个很有潜力的生物标记[14];EXO1为核酸外切酶1基因,参与DNA修复,其表达水平在乳腺癌中与细胞增殖、基因组稳定等方面有关,与生存率差的乳腺癌患者强烈相关[15-16]。虽然还有部分基因由于功能未知,并没有注释到相应的功能,但这些基因可能在乳腺癌的发生过程中也发挥着重要作用,值得关注和进一步探讨。
对高通量基因芯片数据进行综合分析获得乳腺癌复发相关的基因,对预测乳腺癌的特异生存率有一定的指导意义,尽管其在乳腺癌检测中的预测价值还需进一步验证,但为乳腺癌的早期临床诊断和治疗提供了新的思路。
| [1] | Jatoi I, Tsimehon A, Weiss H, et al. Hazard rates of recurrence flowing diagnosis of primary breast cancer[J]. Breast Cancer Res Treat, 2005, 89 (2) : 173–8. DOI:10.1007/s10549-004-1722-0 |
| [2] | Sun SR. Advances in early diagnosis of breast cancer[J]. Lin Chuang Wai Ke Za Zhi, 2013, 21 (7) : 500–2. [ 孙圣荣. 乳腺癌早期诊断进展[J]. 临床外科杂志, 2013, 21 (7) : 500–2. ] |
| [3] | Huang XQ, Zeng H. Correlation between the gene expression profiles of adenocarcinoma of esophagus and Barrett’s esophagus[J]. Zhong Liu Fang Zhi Yan Jiu, 2009, 36 (8) : 639–42. [ 黄绪群, 曾辉. 食管腺癌与Barrett’s食管基因表达谱的研究[J]. 肿瘤防治研究, 2009, 36 (8) : 639–42. ] |
| [4] | Han XH, Mao QX, Li XC, et al. Application of bioinformatics combining serum tumor markers in gastric carcinoma diagnosis[J]. Zhongguo Ai Zheng Za Zhi, 2010, 20 (5) : 364–8. [ 韩小宏, 毛巧霞, 李晓春, 等. 基于生物信息学方法的血清标记物模型在胃癌诊断中的应用[J]. 中国癌症杂志, 2010, 20 (5) : 364–8. ] |
| [5] | Karn T, Metzler D, Ruckh?berle E, et al. Data-driven derivation of cutoffs from a pool of 3,030 Affymetrix arrays to stratify distinct clinical types of breast cancer[J]. Breast Cancer Res Treat, 2010, 120 (3) : 567–79. DOI:10.1007/s10549-009-0416-z |
| [6] | Rody A, Karn T, Ruckh?berle E, et al. Gene expression of topoisomerase Ⅱ alpha (TOP2A) by microarray analysis is highly prognostic in estrogen receptor (ER) positive breast cancer[J]. Breast Cancer Res Treat, 2009, 113 (3) : 457–66. DOI:10.1007/s10549-008-9964-x |
| [7] | Shen Q, Song GH, Zhang JX, et al. Comprehensive evaluation of the diagnostic value of contrast-enhanced sonography for breast cancer by ROC curve and logistic regression[J]. Shi Yong Yi Xue Za Zhi, 2009, 25 (7) : 1058–60. [ 沈嫱, 宋光辉, 张建兴, 等. Logistic回归及ROC曲线综合评价超声造影对乳腺癌的诊断[J]. 实用医学杂志, 2009, 25 (7) : 1058–60. ] |
| [8] | Hanahan D, Weinberg RA. Hallmarks of cancer: the next generation[J]. cell, 2011, 144 (5) : 646–74. DOI:10.1016/j.cell.2011.02.013 |
| [9] | Malaney S, Daly RJ. The ras signaling pathway in mammary tumorigenesis and metastasis[J]. J Mammary Gland Biol Neoplasia, 2001, 6 (1) : 101–13. DOI:10.1023/A:1009572700317 |
| [10] | Yu Q, Geng Y, Sicinski P. Specific protection against breast cancers by cyclin D1 ablation[J]. Nature, 2001, 411 (6841) : 1017–21. DOI:10.1038/35082500 |
| [11] | Campone M, Campion L, Roché H, et al. Prediction of metastatic relapse in node-positive breast cancer: establishment of a clinicogenomic model after FEC100 adjuvant regimen[J]. Breast Cancer Res Treat, 2008, 109 (3) : 491–501. DOI:10.1007/s10549-007-9673-x |
| [12] | Loh SF, Cooper C, Selinger CI, et al. Cell cycle marker expression in benign and malignant intraductal papillary lesions of the breast[J]. J Clin Pathol, 2015, 68 (3) : 187–91. DOI:10.1136/jclinpath-2014-202331 |
| [13] | Ding K, Li W, Zou Z, et al. CCNB1 is a prognostic biomarker for ER+ breast cancer[J]. Med Hypotheses, 2014, 83 (3) : 359–64. DOI:10.1016/j.mehy.2014.06.013 |
| [14] | Shubbar E, Kovács A, Hajizadeh S, et al. Elevated cyclin B2 expression in invasive breast carcinoma is associated with unfavorable clinical outcome[J]. BMC Cancer, 2013, 13 : 1. DOI:10.1186/1471-2407-13-1 |
| [15] | Wang HC, Chiu CF, Tsai RY, et al. Association of genetic polymorphisms of EXO1 gene with risk of breast cancer in Taiwan[J]. Anticancer Res, 2009, 29 (10) : 3897–901. |
| [16] | Muthuswami M, Ramesh V, Banerjee S, et al. Breast tumors with elevated expression of 1q candidate genes confer poor clinical outcome and sensitivity to Ras/PI3K inhibition[J]. PLoS One, 2013, 8 (10) : e77553. DOI:10.1371/journal.pone.0077553 |
 2016, Vol. 43
2016, Vol. 43