| 单核细胞增生李斯特菌和致病性弧菌的双重肽核酸原位荧光杂交检测 |
单核细胞增生李斯特菌(简称单增李斯特菌)和致病性弧菌是生活中常见的、危害性较大的食源性致病菌。人感染单增李斯特菌往往是通过摄入被污染的食品如牛奶、奶酪、肉类和海鲜等引起的[1]。新生儿、孕妇及其他40岁以上的成人、免疫功能缺陷者都是易感人群。感染单增李斯特菌后的症状可表现为突然发热、剧烈头痛、恶心、呕吐、腹泻、败血症、脑膜炎、流产等。自20世纪90年代以来,世界卫生组织(World Health Organization, WHO)将单增李斯特菌列为4大食源性疾病的致病菌之一。霍乱弧菌、副溶血弧菌、哈维弧菌、创伤弧菌、溶藻弧菌、鳗弧菌、梅氏弧菌等都是重要的致病性弧菌,可引起食物中毒,使患者出现腹泻、恶心、呕吐、发热等症状,严重的可导致败血症,甚至死亡。其中霍乱弧菌中的O1群和O139群菌株可以引起严重的腹泻,致死率较高,被《中华人民共和国传染病防治法》列为甲类传染病。
在医学诊断及食品微生物检测中,除了通常使用的培养方法(如ISO 11290-1— 2017,ISO 11290-1—1996,ISO 11290-2—1998和ISO 11290- 1 AMD 1—2004)外,还开发了一系列的生物化学方法、免疫学方法和分子生物学方法等[1-4],如20世纪90年代以后,随着新技术的出现,国外推出了一些基于检测生理代谢的细菌自动化鉴定系统(如API、Biolog、Vitek等)及基于免疫学的酶标抗体法(ELISA)、荧光抗体染色法(免疫荧光法)、同位素标记抗体法(放射免疫)等。尽管这些方法具有操作简便、快速的特点,但也存在特异性较差、所需的耗材价格昂贵等缺点,目前很少在国内推广。以分子生物学为基础的方法主要集中在聚合酶链式反应(polymerase chain reaction, PCR)技术的应用上。PCR是一种体外酶促基因扩增技术,可在短时间内将靶DNA扩增数百万倍。该技术具有简便、快速、敏感性高和特异性强等优点,是目前国内外快速检测技术研究的重点。但PCR方法在不同的实验室或检测部门所检测的目的基因和操作流程上有一定的差异,没有形成标准,得到的检测结果也不尽相同。近年来的实践应用还表明,食品和培养基中存在的抑制剂可影响PCR反应,使检测结果呈假阴性。尽管人们对PCR方法进行了不断的改进和完善,却不能有效地阻止假阴性结果的发生。荧光原位杂交法(fluorescence in situ hybridization, FISH)是基于微生物细胞中16S或23S rRNA的高拷贝数和不易受环境影响的稳定性检测样品中的微生物的方法[5-6]。如SCHMID等[7]使用DNA探针结合FISH技术检测了李斯特菌属。肽核酸探针(peptide nucleic acid, PNA)是一种DNA的模拟物,将PNA探针和FISH技术结合可以克服DNA探针细胞穿透性差、杂交亲和力不强等弱点[5, 8]。PNA-FISH现已被用于大肠埃希菌[9]、链球菌[10]、坂歧肠杆菌[11]、白念珠菌[12]等细菌和真菌的检测。上述检测都只涉及用单重荧光标记探针进行检测。为了提高检测通量,MALIC等[13]成功地使用3个分别标记3种不同荧光的PNA探针同时检测人类皮肤伤口上的3种细菌;ALMEIDA等[14]则使用多重荧光检测技术同步检测了李斯特菌、沙门菌和大肠埃希菌。但上述的多重荧光检测对设备的要求较高,样本都需要在激光共聚焦显微镜下观察。而相比于普通荧光显微镜,激光共聚焦显微镜的普及率仍较低。因此,本研究拟尝试用普通荧光显微镜实现双重PNA- FISH检测。ZHANG等[15-16]报道了用PNA-FISH方法分别检测单增李斯特菌和弧菌的技术,在此基础上,本研究采用双色荧光分别标记单增李斯特菌和弧菌特异性的PNA探针,以实现用普通荧光显微镜同步检测单增李斯特菌和主要致病性弧菌的目的。
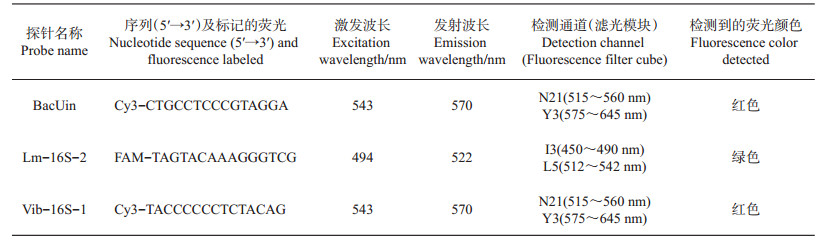
1 材料与方法 1.1 PNA探针在本研究中所使用的PNA探针如表 1所示,Lm- 16S-2[15]为单增李斯特菌特异性探针,Vib-16S-1[16]为致病性弧菌特异性探针,BacUin[17]为细菌通用探针。所有探针由Panagene公司(韩国)合成。由于所使用的探针Lm-16S-2和Vib-16S-1均来自文献[15-16],因此本研究不再对探针的特异性和灵敏度进行验证。
| 表1 PNA探针序列 Table 1 PNA probe sequences used in this study |
 |
| 点击放大 |
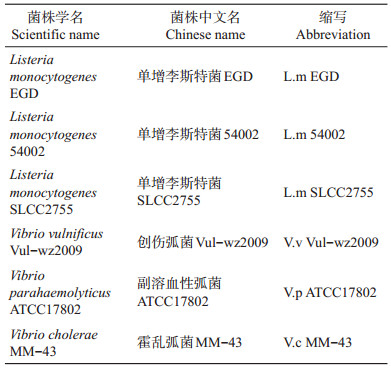
在本研究中所使用的菌株见表 2。由于本文的重点是研究使用普通荧光显微镜建立双重荧光PNA探针同步检测单增李斯特菌和致病性弧菌的方法,因此只选择了目标细菌菌株进行方法的优化,而未选择非目标菌株对探针的特异性进行验证。
| 表2 所使用的菌株 Table 2 Strains used in this study |
 |
| 点击放大 |
单增李斯特菌菌株用脑心浸出液(brain heart infusion, BHI)培养基培养,弧菌用3%氯化钠碱性蛋白胨水(3% NaCl alkaline peptone water, APW)培养基培养。上述细菌在30 ℃、130 r/min条件下振荡过夜,培养至对数生长期。
1.3 PNA-FISH操作步骤离心(2 000 g,5 min)收集对数生长期的细菌,用磷酸盐缓冲溶液(phosphate buffered solution, PBS)(pH 7.4)洗涤1次,再用PBS调整菌液浓度至D(600 nm)=1.0~1.2,取10 μL菌液(应用双重荧光同时检测2种菌时,2种菌各取5 μL)于用98%乙醇清洗过的玻片(1 cm×1 cm)上,涂匀,火焰固定,将玻片于80%乙醇中浸泡15 min,风干;取25 μL含500 pmol/mL PNA的杂交缓冲液(pH 7.5,10%硫酸葡聚糖,10 mmol/L NaCl,30%甲酰胺,0.1%焦磷酸钠,0.2%聚乙烯吡咯烷酮,0.2%聚蔗糖,5 mmol/L Na2EDTA,0.2% TritonX-100,50 mmol/L Tris/HCl)滴加于玻片上(双重荧光检测时,2种探针各加入12.5 μL),55 ℃作用30 min,杂交后将玻片于55 ℃预热的洗涤缓冲液(pH 10,5 mmol/L Tris,15 mmol/L NaCl,0.1% TritonX-100)中洗涤2次,每次10 min,将风干后的涂片滴上香柏油,同时进行阳性对照和阴性对照实验,以BacUin作为阳性对照探针,以不加任何探针的涂片作为阴性对照;在荧光显微镜(Leica DM 6000B)的100倍物镜(滴加香柏油)下观察其荧光亮度及细菌的形态。
1.4 2种菌液的比例对双重PNA探针杂交结果的影响将单增李斯特菌L.m SLCC2755和霍乱弧菌V.c MM-43的菌液浓度调节至D(600 nm)=1.0~ 1.2,将检测总量控制在10 μL的情况下,按比例添加2种菌液,以此比较菌液比例对双重PNA探针杂交结果的影响。
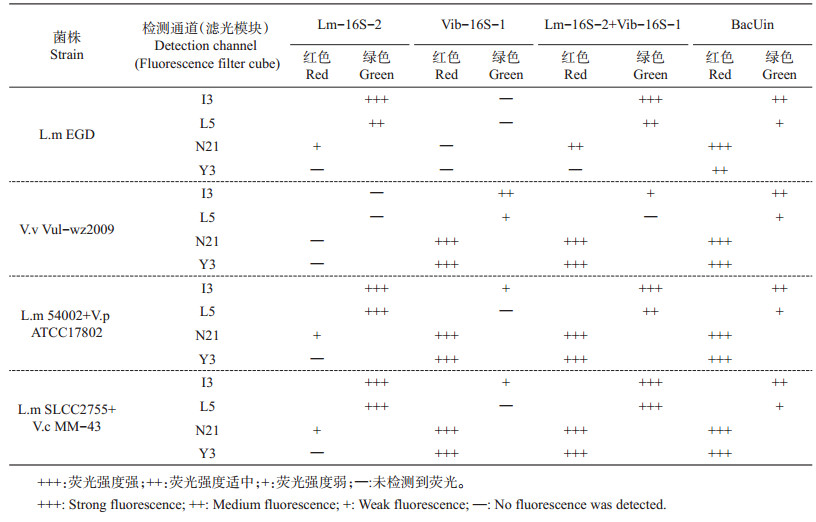
2 结果 2.1 双重PNA杂交条件在前期的研究中对单增李斯特菌和弧菌的PNA-FISH杂交程序进行了优化,且发现2种菌的最佳杂交条件相近(即1.3所述杂交条件)[15-16],因此在进行双重荧光探针杂交检测时,我们在上述优化检测条件下,对2种目标细菌浓度、相应的探针浓度及杂交条件进行了进一步优化。应用双重荧光同时检测2种细菌时,2种细菌各取5 μL,加入探针的量也变为2种探针各加入12.5 μL,检测结果如表 3及图 1~4所示。当2种细菌同时存在时,无论是加入1种探针(图 1~3)还是同时加入2种探针(图 4),都有较好的杂交结果。因此,上述检测条件可以满足2种细菌的同步双重PNA探针杂交检测。
| 表3 不同荧光标记的PNA探针在不同检测通道下对单增李斯特菌和弧菌的检测结果 Table 3 Detection result of L. monocytogenes and Vibrio with different fluorescence-labeled PNA probes at different detection channels |
 |
| 点击放大 |
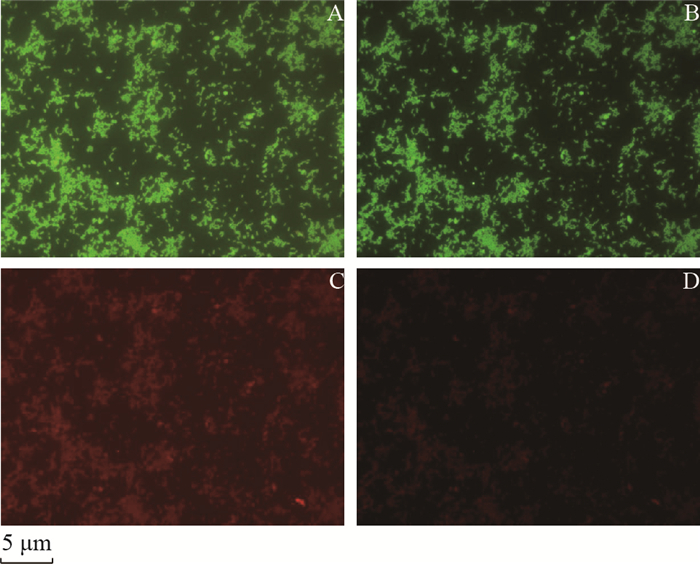 |
| A:I3检测通道;B:L5检测通道;C:N21检测通道;D:Y3检测通道。 A: I3 filter cube; B: L5 filter cube; C: N21 filter cube; D: Y3 filter cube. 图1 单增李斯特菌L.m 54002和副溶血性弧菌V.p ATCC17802的混合菌体杂交了Lm-16S-2探针后,在不同检测通道下观察到的荧光图像 Fig. 1 Fluorescence images from the hybridization results of L.m 54002 and V.p ATCC17802 with Lm-16S-2 probe under different filter cubes |
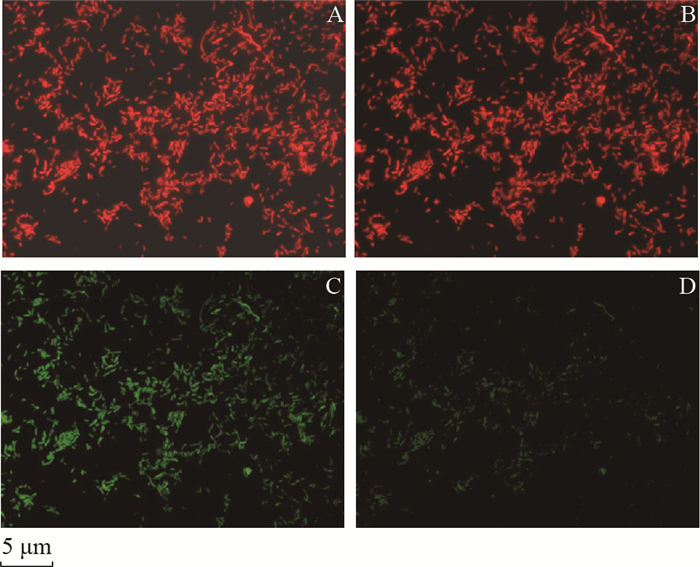 |
| A:N21检测通道;B:Y3检测通道;C:I3检测通道;D:L5检测通道。 A: N21 filter cube; B: Y3 filter cube; C: I3 filter cube; D: L5 filter cube. 图2 单增李斯特菌L.m 54002和副溶血性弧菌V.p ATCC17802的混合菌体杂交了弧菌探针Vib-16S-1后,在不同检测通道下观察到的荧光图像 Fig. 2 Fluorescence images from the hybridization results of L. m 54002 and V.p ATCC17802 with Vib-16S-1 probe under different filter cubes |
 |
| A:N21检测通道;B:Y3检测通道;C:I3检测通道;D:L5检测通道。 A: N21 filter cube; B: Y3 filter cube; C: I3 filter cube; D: L5 filter cube. 图3 单增李斯特菌L.m 54002和副溶血性弧菌V.p ATCC17802的混合菌体杂交了通用探针BacUin后,在不同检测通道下观察到的荧光图像 Fig. 3 Fluorescence images from the hybridization results of L.m 54002 and V.p ATCC17802 with BacUin probe under different filter cubes |
 |
| A:L5检测通道,即被探针Lm-16S-2杂交后的单增李斯特菌;B:Y3检测通道,即被探针Vib-16S-1杂交后的霍乱弧菌;C:A和B叠加的效果(使用Leica Application Suite V4.2软件处理)。 A: L5 filter cube (the hybridization result of L.m SLCC2755 and Lm-16S-2); B: Y3 filter cube (the hybridization result of V.c MM-43 and Vib-16S-1); C: Overlay of image A and B (processed by software Leica Application Suite V4.2). 图4 单增李斯特菌L.m SLCC2755和霍乱弧菌V.c MM- 43的混合菌体杂交了Lm-16S-2和Vib-16S-1探针后,在不同检测通道下观察到的荧光图像 Fig. 4 Fluorescence images from the hybridization results of L.m SLCC2755 and V.c MM-43 with Lm-16S-2 and Vib-16S-1 probes under different filter cubes |
Lm-16S-2是用于检测单增李斯特菌的PNA探针,用FAM荧光标记,可被I3和L5滤光模块检测到绿色荧光;Vib-16S-1和BacUin分别是用于检测弧菌和细菌的PNA探针,都是用Cy3作为荧光标记,可被N21和Y3滤光模块检测到红色荧光。比较这些滤光模块可发现,标记FAM荧光的细菌可被I3和L5滤光模块检测到绿色荧光,且在相同视野下,I3通道检测到的荧光亮度略高于L5;同样,标记Cy3荧光的细菌能被N21和Y3滤光模块检测到红色荧光,同样,在相同视野下,N21检测到的荧光亮度略高于Y3模块。如表 3所示:L.m EGD与Lm- 16S- 2探针杂交时,I3的绿色荧光强度可达到“+++”,而L5只有“++”;与BacUin探针杂交时,N21的红色荧光强度可达到“+++”,而Y3只有“++”。但同时也发现,只标记绿色荧光的细菌,也会在红色荧光检测模块下检测到红色荧光,如L.m EGD与Lm-16S-2探针杂交时,不仅能在I3、L5通道下检测到绿色荧光,也在N21模块下检测到红色荧光;同样,只标记红色荧光的细菌,也会在绿色荧光检测模块下检测到绿色荧光,如V.v Vul-wz2009与Vib- 16S-1探针杂交时,不仅能在N21、Y3下检测到红色荧光,也能在I3及L5模块下检测到绿色荧光,但绿色荧光强度明显弱于红色荧光,且L5检测到的绿光更弱于I3。这说明不同荧光检测通道间存在一定的干扰,在单色荧光检测时,该干扰可忽略,但在不同细菌的多色荧光多重标记、多通道同时检测时,则要考虑由该原因导致的假阳性问题。
在单重荧光检测时,检测通道模块I3和N21已经能满足检测要求,但在多重荧光检测时,这2个通道间的交叉干扰会导致假阳性的产生,因此引入L5和Y3检测通道模块。检测结果显示,L5和Y3较I3和N21产生的假阳性少,如L.m 54002 + V.p ATCC17802与Lm-16S-2探针杂交(图 1)时,理论上只有绿色荧光通道上能检测到绿色荧光,但N21通道也检测到了红光,相比较而言,Y3通道能成功屏蔽荧光的干扰,在该通道上未能检测到假阳性的红色荧光;同样,L.m 54002+V.p ATCC17802与Vib- 16S-1探针杂交(图 2)时,理论上只有在红色荧光通道上能检测到红色荧光,但在I3通道上检测到了较弱的绿光,而在L5通道上则未能检测到假阳性的绿光。但L5和Y3模块也未能杜绝相邻荧光间的干扰,特别是用BacUin探针杂交(图 3)时,得到的荧光亮度都较强,因此,L5通道往往也能检测到假阳性的绿色荧光。而在使用单增李斯特菌和弧菌的特异性PNA探针(分别标记FAM和Cy3荧光)进行双重荧光检测时,L5和Y3检测通道模块是可行的。
图 4是单增李斯特菌和弧菌的混合菌液同时与2种目标菌探针进行双重荧光杂交的结果,即单增李斯特菌L.m SLCC2755和霍乱弧菌V.c MM-43的混合菌体同时杂交探针Lm-16S-2和Vib-16S-1。结果表明:被探针Lm-16S-2杂交后的单增李斯特菌可在L5检测通道上观察到绿色荧光(图 4A);被探针Vib-16S-1杂交后的霍乱弧菌可在Y3检测通道上观察到红色荧光(图 4B)。
此外,本实验的阴性对照均未被检测到目标菌的荧光图像(图略)。
2.3 2种菌液的体积比例对检测结果的影响上述检测都是在2种菌液体积比接近1:1的理想条件下进行的,而在实际情况下,同步检测时可能2种菌的含量会有差异。因此,在上述检测条件不变的情况下,比较了2种菌液在不同体积比例下双色探针同步杂交检测的结果,如表 4所示:在3:7或7:3的比例下,可同步双通道检测2种菌;当比例为1:9或9:1时,量少的菌则明显在视野中减少,亮度明显减弱;当比例为1:19或19:1时,量少的菌则在视野中很难被检测到。
| 表4 L.m SLCC2755和V.c MM-43菌液体积比例对双重PNA探针杂交结果的影响 Table 4 Effect of the volume ratio of L.m SLCC2755 and V.c MM-43 on the dual PNA probe hybridization results |
 |
| 点击放大 |
我们将2种菌单独杂交时的最优化程序(2种优化后杂交程序相似度高)直接用到双重杂交上,结果显示,若2种菌含量相似,则该优化程序可直接应用。但当2种菌的含量差异较大时,量少的菌就会存在漏检,原因在于:一是由于菌液在玻片上固定后,还要经过乙醇浸泡的步骤,该步骤会使固定不牢的菌脱落而导致漏检;二是量少的菌受到量大的菌的干扰,影响探针的杂交而导致漏检。原本计划通过杂交程序的优化减少漏检的可能,但在实际检测中,某个样本很少会同时感染单增李斯特菌和弧菌。但即便同时存在,在双重检测中,样本分成两部分同时进行针对李斯特菌和弧菌的不同增菌处理,也可避免因某种菌菌量过低而导致的漏检。
在单重荧光检测时,检测通道I3和N21可完全满足检测的需求。但在多重荧光同时检测时,由于荧光通道间的干扰,会造成假阳性,因此我们引入了L5和Y3新的荧光检测通道。L5(512~542 nm)和Y3(575~645 nm)的滤光区间较I3(450~490 nm)和N21(515~560 nm)的滤光区间间隔大些,因此可更有效地屏蔽一些荧光干扰。
SHEPARD等[18]研究表明,用普通荧光显微镜及双色荧光标记PNA探针,应用PNA-FISH法可同步检测白念珠菌(Candida albicans)和光滑念珠菌(Candida glabrata)。MORGAN等[19]研究表明,用普通荧光显微镜可对大肠埃希菌(Escherchia coli)和铜绿假单胞菌(Pseudomonas aerugionsa)进行双重荧光检测。但上述实验并没有说明用的是什么型号的荧光显微镜,只显示所使用的荧光显微镜配有异硫氰酸荧光素/德克萨斯红双通带滤镜(fluorescence microscope equipped with a fluorescein isothiocyanate (FITC)/Texas Red (TXR) dual band pass filter)。由此推测,他们所标记的荧光应该是FITC和TXR,但实际并没有以这2种荧光的名字命名的滤光模块,说明他们使用的可能只是能滤过这2种荧光的滤光模块。而在结果及分析中也未提及这2种荧光间是否会存在干扰。我们发现,染料FITC(发射波长520 nm)和TXR(615 nm)的最大发射波长的差异较本研究选择的FAM(522 nm)和Cy3(570 nm)大,这也有助于减少2种荧光间的干扰,在后续研究中,可尝试用TXR替换Cy3。DELLA-LATTA等[20]使用Traffic Light PNA FISH试剂盒同步检测了3种微生物,检测结果以绿红黄三色分别表示大肠埃希菌(E. coli)、铜绿假单胞菌(P. aerugionsa)和肺炎克雷伯菌(Klebsiella pneumoniae)。可能由于使用的是试剂盒,因此DELLA-LATTA等[20]并未说明探针标记的是什么荧光,只是说明检测所使用的荧光显微镜配有异硫氰酸荧光素/德克萨斯红双通带滤镜。而在实际检测中若各种菌的形态相近,则各种标记荧光和荧光滤光模块的选择就非常重要,否则很容易由于荧光通道间的干扰造成假阳性。
总之,本实验结果表明,普通荧光显微镜也可以用作多重PNA-FISH的检测,但受荧光通道等的限制,三重检测会较困难,但可以完成双重荧光检测。
| [1] |
VOLOKHOV D, RASOOLY A, CHUMAKOV K, et al. Identification of Listeria species by microarray-based assay. Journal of Clinical Microbiology, 2002, 40(12): 4720-4728. DOI:10.1128/JCM.40.12.4720-4728.2002 |
| [2] |
ALLERBERGER F, DIERICH M, PETRANYI G, et al. Nonhemolytic strains of Listeria monocytogenes detected in milk products using VIDAS immunoassay kit. Zentralblatt für Hygiene und Umweltmedizin, 1997, 200(2/3): 189-195. |
| [3] |
INGIANNI A, FLORIS M, PALOMBA P, et al. Rapid detection of Listeria monocytogenes in foods, by a combination of PCR and DNA probe. Molecular and Cellular Probes, 2001, 15(5): 275-280. DOI:10.1006/mcpr.2001.0372 |
| [4] |
ZENG H Y, ZHANG X F, SUN Z, et al. Multiplex PCR identification of Listeria monocytogenes isolates from milk and milk-processing environments. Journal of the Science of Food and Agriculture, 2006, 86(3): 367-371. DOI:10.1002/(ISSN)1097-0010 |
| [5] |
AMANN R, FUCHS B M. Single-cell identification in microbial communities by improved fluorescence in situ hybridization techniques. Nature Reviews Microbiology, 2008, 6(5): 339-348. DOI:10.1038/nrmicro1888 |
| [6] |
ALMEIDA C, AZEVEDO N F, FERNANDES R M, et al. Fluorescence in situ hybridization method using a peptide nucleic acid probe for identification of Salmonella spp. in a broad spectrum of samples. Applied and Environmental Microbiology, 2010, 76(13): 4476-4485. DOI:10.1128/AEM.01678-09 |
| [7] |
SCHMID M, WALCHER M, BUBERT A, et al. Nucleic acid based, cultivation-independent detection of Listeria spp. and genotypes of L. monocytogenes. FEMS Immunology and Medical Microbiology, 2003, 35(3): 215-225. DOI:10.1016/S0928-8244(02)00456-X |
| [8] |
DAIMS H, WAGNER M. Quantification of uncultured microorganisms by fluorescence microscopy and digital image analysis. Applied Microbiology and Biotechnology, 2007, 75(2): 237-248. DOI:10.1007/s00253-007-0886-z |
| [9] |
STENDER H, BROOMER A J, OLIVEIRA K, et al. Rapid detection, identification, and enumeration of Escherichia coli cells in municipal water by chemiluminescent in situ hybridization. Applied Environmental Microbiology, 2001, 67(1): 142-147. DOI:10.1128/AEM.67.1.142-147.2001 |
| [10] |
MONTAGUE N S, CLEARY T J, MARTINEZ O V, et al. Detection of group B streptococci in Lim broth by use of group B Streptococcus peptide nucleic acid fluorescent in situ hybridization and selective and nonselective agars. Journal of Clinical Microbiology, 2008, 46(10): 3470-3472. DOI:10.1128/JCM.00858-08 |
| [11] |
ALMEIDA C, AZEVEDO N F, IVERSEN C, et al. Development and application of a novel peptide nucleic acid probe for the specific detection of Cronobacter genomospecies (Enterobacter sakazakii) in powdered infant formula. Applied and Environmental Microbiology, 2009, 75(9): 2925-2930. DOI:10.1128/AEM.02470-08 |
| [12] |
BISHOP J A, CHASE N, MAGILL S S, et al. Candida bracarensis detected among isolates of Candida glabrata by peptide nucleic acid fluorescence in situ hybridization: Susceptibility data and documentation of presumed infection. Journal of Clinical Microbiology, 2008, 46(2): 443-446. DOI:10.1128/JCM.01986-07 |
| [13] |
MALIC S, HILL K E, HAYES A, et al. Detection and identification of specific bacteria in wound biofilms using peptide nucleic acid fluorescent in situ hybridization (PNA FISH). Microbiology, 2009, 155: 2603-2611. DOI:10.1099/mic.0.028712-0 |
| [14] |
ALMEIDA C, AZEVEDO N F, SANTOS S, et al. Discriminating multi-species populations in biofilms with peptide nucleic acid fluorescence in situ hybridization (PNA-FISH). PLoS One, 2011, 6(3): e14786. DOI:10.1371/journal.pone.0014786 |
| [15] |
ZHANG X F, WU S, LI K, et al. Peptide nucleic acid fluorescence in situ hybridization for identification of Listeria genus, Listeria monocytogenes and Listeria ivanovii. International Journal of Food Microbiology, 2012, 157(2): 309-313. DOI:10.1016/j.ijfoodmicro.2012.05.004 |
| [16] |
ZHANG X F, LI K, WU S, et al. Peptide nucleic acid fluorescence in-situ hybridization for identification of Vibrio spp. in aquatic products and environments. International Journal of Food Microbiology, 2015, 206(3): 39-44. |
| [17] |
PERRY-O'KEEFE H, STENDER H, BROOMER A, et al. Filter-based PNA in situ hybridization for rapid detection, identification and enumeration of specific micro-organisms. Journal of Applied Microbiology, 2001, 90(2): 180-189. DOI:10.1046/j.1365-2672.2001.01230.x |
| [18] |
SHEPARD J R, ADDISON R M, ALEXANDER B D, et al. Multicenter evaluation of the Candida albicans/Candida glabrata peptide nucleic acid fluorescent in situ hybridization method for simultaneous dual-color identifi cation of C. albicans and C. glabrata directly from blood culture bottles. Journal of Clinical Microbiology, 2008, 46(1): 50-55. |
| [19] |
MORGAN M, MARLOWE E, DELLA-LATTA P, et al. Multicenter evaluation of a new shortened peptide nucleic acid fluorescence in situ hybridization procedure for species identification of select Gram-negative bacilli from blood cultures. Journal of Clinical Microbiology, 2010, 48(6): 2268-2270. DOI:10.1128/JCM.00166-10 |
| [20] |
DELLA-LATTA P, SALIMNIA H, PAINTER T, et al. Identification of Escherichia coli, Klebsiella pneumoniae, and Pseudomonas aeruginosa in blood cultures: A multicenter performance evaluation of a three-color peptide nucleic acid fluorescence in situ hybridization assay. Journal of Clinical Microbiology, 2011, 49(6): 2259-2261. DOI:10.1128/JCM.02250-10 |
 2018, Vol. 44
2018, Vol. 44


