| 向日葵黄萎病种子带菌研究 |
2. 内蒙古乌拉特前旗农业技术推广中心,内蒙古 乌拉特前旗 014400
2. Agricultural Technique Center of Wulateqianqi, Wulateqianqi 014400, Inner Mongolia, China
向日葵是一种起源于美洲的油料作物,广泛分布于欧洲、亚洲、美洲等地,目前已经成为世界第4大油料作物[1]。近年来,由于国外向日葵育种材料和杂交种的大量引入,使得我国向日葵黄萎病呈现逐年加重的态势。2009年向日葵病害普查结果表明,全国向日葵产区都有黄萎病发生,但以宁夏黄灌区发生最重,平均发病率达40%,黑龙江地区以15%的平均发病率次之,而内蒙古地区的发病率介于10%~15%之间(采自向日葵产业体系数据库)。
向日葵黄萎病(Verticillium Wilt)是由大丽轮枝菌(Verticillium dahliae Kleb.)引起的一种土传病害,该病菌可以侵染多种寄主作物,造成产量和品质的严重下降[2]。由于大丽轮枝菌存在于土壤中,因此微菌核是其在土壤中存活的主要形式,它能够通过农事操作、土壤和水进行传播[3]。由于微菌核能够在土壤中存活数年[4],因此对向日葵黄萎病的防治非常困难。目前生产中主要采用种植抗黄萎病向日葵杂交种、施用含有氨和亚硝酸的肥料以及与禾本科作物轮作等措施来控制向日葵黄萎病的发生[5-8]。裴旭等[9]研究表明,多锰锌和乙霉威等化学药剂对向日葵黄萎病菌有很好的抑菌效果,因此,可以通过药剂拌种处理来防控向日葵黄萎病。内蒙古农业大学周洪友教授的课题小组也开展了一系列有关向日葵黄萎病生防菌的筛选、鉴定,以及田间防治效果的研究,这些研究结果为后续利用生防菌进行拌种处理防控向日葵黄萎病提供了可能[10-11]。
已有的研究结果表明,常规的马铃薯葡萄糖琼脂(potato dextrose agar, PDA)培养基或吸墨纸法可以用来分离生菜和南瓜种子上携带的黄萎病菌[12-14]。但是由于上述方法分离时间较长且杂菌容易抑制黄萎病菌的生长等缺点限制了其广泛应用。基于聚合酶链式反应(polymerase chain reaction, PCR)检测种子带菌的方法具有准确、省时、特异的优点,且分离鉴定的结果快速可靠,目前已经被应用到土壤和种子中进行多种植物病原菌的快速检测[15-20]。
利用PCR方法检测向日葵种子中黄萎病菌的研究还未有相关报道。因此,本研究以向日葵种子的种皮DNA为模板,利用真菌的通用引物和内部转录间隔区(internal transcribed spacer, ITS)序列内侧黄萎菌特异引物进行巢式PCR扩增,以快速检测向日葵种子是否携带黄萎病菌。为了验证上述结果,以带有绿色荧光蛋白(green fluorescent protein, GFP)分子标记的黄萎病菌接种向日葵的根部,通过荧光显微镜观察灌浆后的向日葵种子的种皮是否带菌。这一研究结果可为明确向日葵种皮带菌进行远距离传播以及药剂拌种防控向日葵黄萎病奠定理论基础。
1 材料与方法 1.1 供试材料供试的向日葵品种来自国家向日葵产业体系育种岗位。油葵品种:S26、赤029X115R、F08-2;食葵品种:三道眉、LD5009、黑大片、3638C。实验室留存的种子也由国家向日葵产业体系提供。田间采集的种子来自内蒙古呼和浩特农业科学院试验田种植的向日葵食葵品种LD5009、三道眉、3638C。供试向日葵品种具体信息详见表 1。
| 表1 供试向日葵品种名称及来源 Table 1 Names and origins of the tested sunflower varieties |
 |
| 点击放大 |
供试接种的向日葵黄萎病菌VDGn3菌株由内蒙古农业大学农学院分子植物病理室提供,供试接种的向日葵品种食葵LD5009为商业销售杂交种,VDGn3带GFP标记的突变体来自本实验室。大丽轮枝菌的遗传转化参照DOBINSON[21]的方法进行;表达有GFP荧光突变体的鉴定参照本实验室已经发表的方法[22]进行。
1.2 向日葵种子种皮的获得和DNA提取用2% NaClO溶液浸泡向日葵种子5 min,然后将无菌水浸湿的滤纸放置在90 mm培养皿中,将去壳的向日葵种子放置在滤纸上。待向日葵种子吸水膨胀后(室温放置3 h),取下向日葵种子的种皮,将每4粒种子的种皮放入一个已灭菌的EP管中作为1份样本,-20 ℃保存用于提取DNA。
向日葵种子种皮DNA的提取参照《分子克隆》中的方法[23]。具体的操作方法如下:将收集的向日葵种子的种皮用液氮冷冻后迅速研磨成白色粉末状,加入十六烷基三甲基溴化铵(CTAB)提取液,65 ℃水浴30 min,每10 min振荡1次;随后加入等体积的氯仿-酚-异戊醇(体积比为25:24:1)混合液,振荡混匀后静置10 min,1.2万r/min离心后取上清液;再加入等体积异丙醇,颠倒混匀,在-20 ℃中放置30 min,1.2万r/min离心后弃上清液,用75%乙醇洗涤沉淀2次,去除残余液体,晾干后加入20 μL无菌水溶解。吸取2 μL DNA在1%琼脂糖凝胶上进行电泳检测,同时取2 μL DNA在Nanodrop 2000(美国Thermo Scientific公司)上测定其浓度,其余DNA于-20 ℃保存备用。
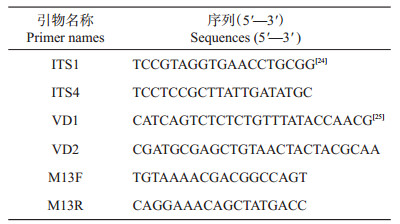
1.3 向日葵种子种皮携带大丽轮枝菌的检测首先,利用真菌rRNA基因ITS区通用引物ITS1/ITS4(表 2)对种皮DNA进行扩增。PCR扩增体系为:10×Taq缓冲液2.5 μL,2.5 mmol/L dNTP 1.25 μL,10 mmol/L引物ITS1、ITS4各0.5 μL,2.5 U/ μL Taq酶(北京天根生物公司)0.25 μL,种皮DNA 2.0 μL,最后加ddH2O补足至25 μL。反应程序为:94 ℃预变性5 min;94 ℃变性40 s,56 ℃退火40 s,72 ℃延伸40 s,32个循环;72 ℃延伸10 min。
| 表2 引物名称和序列 Table 2 Names and sequences of primers |
 |
| 点击放大 |
将上述扩增产物用ddH2O稀释5倍后,取2 μL作为模板,用大丽轮枝菌的特异性引物VD1/VD2(表 2)再次进行扩增,以ddH2O为模板作为PCR反应的阴性对照。引物VD1/VD2的扩增体系和反应程序与ITS1/ITS4扩增反应相同。取5.0 μL ITS1/ ITS4扩增产物和巢式PCR扩增产物在1.5%琼脂糖凝胶上电泳检测其PCR片段大小。
1.4 PCR产物的测序和序列分析将不同向日葵品种种皮的巢式PCR扩增产物与TA载体pCR2.1(美国Invitrogen公司)在14 ℃下连接过夜,然后将连接产物转化大肠杆菌感受态细胞Trans-T1(北京全式金公司)后涂板,进行蓝白斑筛选。从平板上挑取白斑至无菌水中,取菌液用引物M13F/M13R(表 2)进行菌液PCR鉴定。从每个品种的转化产物中挑取3个阳性克隆送往北京厚生博泰公司测序,将测序得到的序列用DNAMAN 5.0去除载体序列,然后在NCBI数据库中利用BLAST序列比对工具进行DNA序列比对,确定序列来源物种。
1.5 利用GFP标记的大丽轮枝菌接种向日葵带GFP标记转化子的菌株VDGn3经PDA平板活化5~7 d后,从菌落边缘挑取菌块接入麦麸培养基上,用于快速扩繁大量的分生孢子。25 ℃培养7~14 d后,用清水冲洗麦麸培养基,用4层纱布过滤获得分生孢子液。用血球计数板进行计数,将分生孢子液的浓度调至1×107 mL-1后备用。采用纸钵撕底蘸根法进行接种实验[26],具体操作如下:将向日葵品种LD5009的种子播种到装有灭菌土的营养钵中,每个营养钵播种3粒种子;待向日葵幼苗长出2~4片真叶时,每钵接种分生孢子液50 mL。接种后的幼苗置于22~23 ℃温室条件下等待开花灌浆后收获种子;剥去种子的种壳,取种皮置于有水滴的载玻片上,在BX51荧光显微镜(日本奥林巴斯公司)下观察是否有绿色荧光。
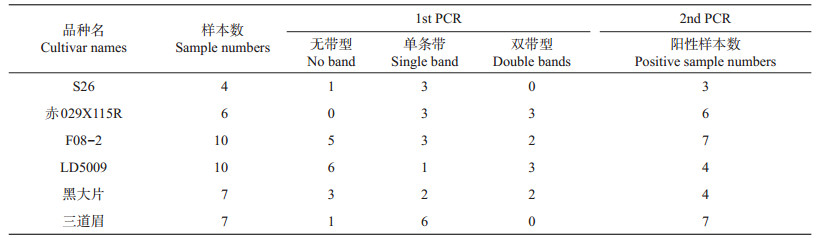
2 结果与分析 2.1 向日葵种子种皮上携带黄萎病菌的检测以真菌通用引物对ITS1/ITS4(rRNA基因)进行PCR扩增的结果如图 1和表 3所示。供试样本在541 bp附近出现2种带形,即单一带型和双带型。在油葵品种S26扩增的4份样本中,除1份样本没有扩增出目的条带外,其余3份样本均为单一带型;在杂交种赤029X115R的6份样本中,3份样本是单一带型,3份样本呈现双带型;在F08-2的10份样本中,只有5份扩增出相应的条带,其中3份样本为单一带型,2份呈现出双带型。在供试的食葵品种中,LD5009的10份样本中有4份样本扩增出条带,但是3份样本为双带型;在黑大片的7份样本中,4份样本出现条带,2份出现双带型;在三道眉的7份样本中有6份样本扩增出单一带型,1份样本没有扩增出目的条带。
 |
| M:DNA分子质量标志物;CK:阴性对照。 M: DNA marker; CK: Negative control. 图1 不同品种向日葵种皮DNA ITS1/ITS4引物的PCR扩增 Fig. 1 PCR products of seed coats from different sunflower varieties using ITS1/ITS4 primer pair |
| 表3 不同向日葵品种双重PCR扩增带型统计 Table 3 Statistics of band types by nested PCR amplification in different sunflower varieties |
 |
| 点击放大 |
为了进一步确定扩增出的条带是否为向日葵黄萎病菌,以ITS1/ITS4引物扩增出的PCR产物为模板,利用向日葵黄萎病菌的特异引物VD1/VD2(扩增rRNA基因)进行巢氏PCR扩增,结果如图 2和表 3所示。和ITS引物扩增的结果不同,利用VD1/VD2引物扩增出的条带都是单一带型,条带大小为324 bp。其中:S26的4份样本的扩增结果和ITS相似,只有3份扩增出了目的条带;F08-2有7份样本均扩增出单一目的条带,比ITS扩增出的5份阳性样本多出2份;赤029X115R有6份样本均扩增出单一条带。在供试的食葵品种中,ITS扩增呈现阳性的4份LD5009样本都相应地扩增出条带;黑大片7份样本的扩增结果和ITS相似,也只有4份样本出现目的条带;三道眉的7份样本均扩增出单一带型,而相对应的ITS扩增结果中却有1份样本没有扩增出目的条带。
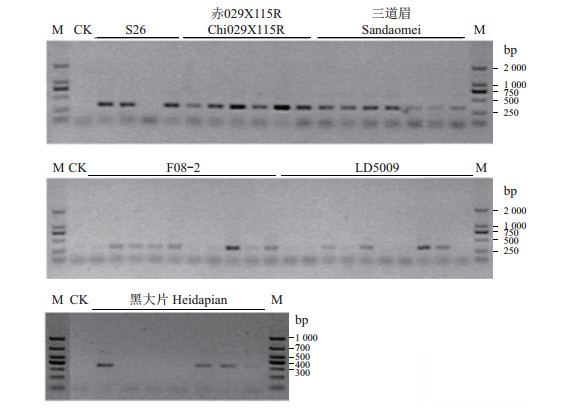 |
| M:DNA分子质量标志物;CK:阴性对照。 M: DNA marker; CK: Negative control. 图2 不同向日葵品种种皮巢式PCR产物 Fig. 2 Nested PCR products of seed coats from different sunflower varieties |
将上述巢氏PCR扩增得到的产物(单一条带)进行TA克隆后,用菌液PCR筛选阳性转化子,所得PCR产物大小为524 bp(图 3)。测序后在GenBank中进行比对,结果表明,巢氏PCR扩增得到的所有单一片段的序列(登录号: KY745897)与GenBank中已有的大丽轮枝菌ITS区序列的相似度均为100%。
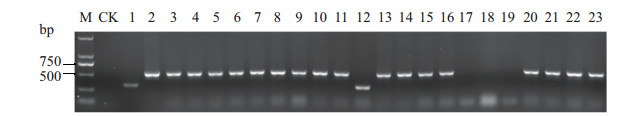 |
| M:DNA分子质量标志物;CK:阴性对照。 M: DNA marker; CK: Negative control. 图3 巢式PCR产物连接转化后阳性克隆鉴定 Fig. 3 Identification of positive clones of nested PCR products after transformation |
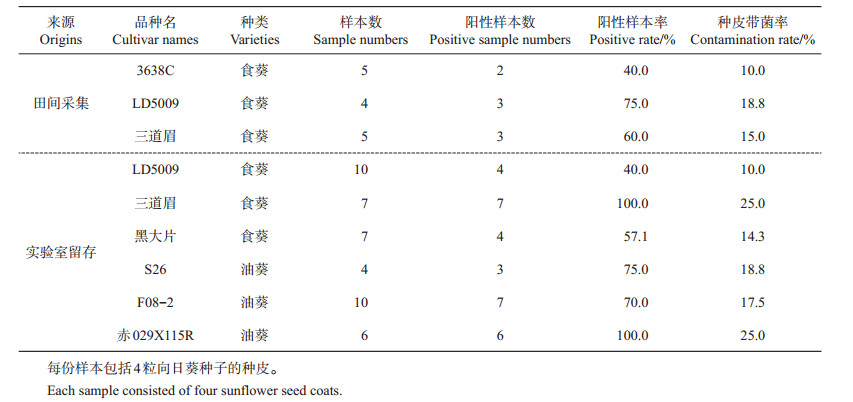
为了进一步确定不同向日葵品种携带黄萎病菌的频率,我们不仅对上述品种的带菌率进行了重复检测,而且还增加了3份当年在大田中采集的已经灌浆的不同品种的向日葵种子。PCR扩增结果(表 4)显示,在进行检测的58份不同品种的向日葵样本中,39份为阳性样本,带菌率为22.4%。其中:田间采集的新种子3638C带菌率最低,仅为10.0%;而实验室留存的赤029X115R带菌率最高,为25.0%;其余供试的向日葵品种种子带菌率介于二者之间。同时,新采集的种子和实验室留存的向日葵种子的黄萎病菌带菌率没有明显差别。
| 表4 不同向日葵品种种皮带菌率的巢氏PCR检测结果 Table 4 Contamination rate of different sunflower cultivars detected by nested PCR |
 |
| 点击放大 |
为了进一步验证巢氏PCR技术检测向日葵种皮带菌的准确性,将食葵LD5009向日葵幼苗接种标记有GFP的向日葵黄萎病菌VDGn3,75 d后收获向日葵种子。取接种株的向日葵种子种皮进行制片后在荧光显微镜下观察,结果如图 4所示。在接种后的向日葵种子的种皮上能观察到绿色荧光蛋白(GFP)信号,而对照向日葵植株的种子的种皮上未见有绿色荧光信号。这一观察结果表明种皮是向日葵黄萎病菌重要的定殖部位,同时证明了上述基于巢式PCR技术检测向日葵种皮是否携带黄萎病菌的准确性。
 |
| A:荧光通道观察;B:自然光和荧光通道观察结果的叠加;C:自然光通道观察。上排:接种后种子的种皮;下排:未接种的对照。标尺均为100 μm。 A: GFP channel; B: Overlay of GFP and natural light channel; C: Natural light channel. Upper row: Seed coats after inoculation; Lower row: No inoculation as control. Scale bars=100 μm. 图4 向日葵种子的种皮荧光观察结果 Fig. 4 Observation result of the fluorescence in sunflower seed coats |
本文利用巢式PCR技术对向日葵种子的种皮是否携带黄萎菌进行了检测。这种检测方法包括收集种子仁膜、DNA提取和用特异性引物进行巢式PCR扩增,通过快速检测有助于及早控制向日葵黄萎病的发生。基于PCR技术检测种子是否携带大丽轮枝菌的方法已经在马铃薯[27-28]和橄榄树[29]中有所报道,如DURESSA等[17]使用实时荧光定量PCR方法检测了菠菜种子中是否含有大丽轮枝菌;SOUSA等[30]采用实时荧光定量PCR方法检测了菜豆种子中是否携带有尖孢镰刀菌(Fusarium oxysporum)。以上均说明利用PCR方法能够便捷地检测种子是否带菌。尽管SACKSTON等[31]报道了可以从向日葵种子的种壳和种皮上分离到大丽轮枝菌,但是本实验室尝试了多次后,发现种皮上携带多种病原菌和杂菌,生长较慢的黄萎病菌往往容易被生长快的其他病菌或杂菌所抑制,因此没有从种子上成功分离到黄萎病菌。而本文利用巢氏PCR来检测向日葵种皮是否带有黄萎病菌的方法,将作为后续的一种可行、便捷的方法来检测向日葵种子是否携带黄萎病菌,可以在未来种子带菌检测中进行推广使用。
此外,用PCR检测种子中是否带有病原菌需要提取种子的DNA。由于向日葵种子的种壳具有厚且坚硬的特性,因此,利用普通DNA提取方法提取包括种壳在内的DNA没有可操作性,需要采用一些特殊的方法来获取用于种子带菌PCR检测所需的DNA模板[32]。因此,我们将向日葵种壳经人工剥掉后只取与种子胚紧密接触的种皮作为提取DNA的材料,从而方便了DNA的提取。由于单粒向日葵种子的种皮量非常少,因此,我们混合了4份种子的种皮样本进行DNA提取。
在本实验过程中,我们曾经尝试提取种皮DNA后直接用大丽轮枝菌的特异性引物进行扩增,但多次实验均未得到PCR条带。究其原因可能是挑选的种子的种皮上没有病菌,或者是种皮上携带的黄萎菌量很少,其DNA相对于种子的种皮DNA浓度太低。而用巢式PCR可以将种皮DNA中黄萎病菌DNA信息进行放大,从而获得目的条带进行测序分析。此外,利用真菌的通用引物对ITS1/ITS4进行PCR扩增时出现了双带型,说明在向日葵种子种皮上可能存在包括黄萎病菌在内的不同类型的其他真菌。而利用黄萎病菌的特异引物VD1/VD2进行巢氏PCR再次扩增出单一的条带,体现了黄萎病菌引物VD1/VD2的特异性。后续PCR条带的测序结果也证实了这一推测。
在利用GFP标记的黄萎病菌侵染向日葵根部的实验中,我们观察到向日葵黄萎病菌能够到达花盘进行侵染(文章待发表)。而在荧光显微镜下,我们观察到在花盘中向日葵种皮上有非常强的GFP荧光信号,而种壳上有微弱的荧光信号,籽仁上没有荧光信号,预示着种皮是黄萎病菌重要的定殖部位,而不是种壳和籽仁。这一结果和利用PCR检测的结果相一致。
利用PCR方法结合荧光显微镜观察验证了向日葵种皮是携带黄萎病菌的重要载体。而种皮带菌也为向日葵黄萎病菌的远距离传播创造了条件。因此,在后续向日葵黄萎病的防控技术中,我们认为在播前对向日葵种子进行药剂处理应该作为一项防治向日葵黄萎病菌的必要措施。至于选用哪种药剂进行处理以及药剂处理的方法还需要进行一系列的室内和田间试验研究。这些结果对今后向日葵黄萎病有效防控技术研究具有非常重要的意义。
| [1] |
王静, 张剑茹, 崔超敏, 等. 向日葵菌核病研究进展. 内蒙古农业科技, 2006(6): 25-28. WANG J, ZHAGN J R, CUI C M, et al. Review of Sclerotinia disease in sunflower. Inner Mongolia Agricultural Science and Technology, 2006(6): 25-28. (in Chinese with English abstract) |
| [2] |
张立华, 赵益平, 张颖力, 等. 内蒙古向日葵生产现状及发展对策. 内蒙古农业科技, 2007(5): 82-84. ZHAGN L H, ZHAO Y P, ZHANG Y L, et al. Production status and development counter-measures of sunflower in Inner Mongolia. Inner Mongolia Agricultural Science and Technology, 2007(5): 82-84. (in Chinese with English abstract) |
| [3] | BHAT R G, SUBBARAO K V. Host range specificity in Verticillium dahliae. Phytopathology, 1999, 89(12): 1218-1225. DOI:10.1094/PHYTO.1999.89.12.1218 |
| [4] | ZHOU L, HU Q, JOHANSSON A, et al. Verticillium longisporum and V. dahliae: Infection and disease in Brassica napus. Plant Pathology, 2006, 55(1): 137-144. DOI:10.1111/ppa.2006.55.issue-1 |
| [5] | CONN K L, TENUTA M, LAZAROVITS G. Liquid swine manure can kill Verticillium dahliae microsclerotia in soil by volatile fatty acid, nitrous acid, and ammonia toxicity. Phytopathology, 2005, 95(1): 28-35. DOI:10.1094/PHYTO-95-0028 |
| [6] | KLOSTERMAN S J, ATALLAH Z K, VALLAD G E, et al. Diversity, pathogenicity, and management of verticillium species. Annual Review of Phytopathology, 2009, 47: 39-62. DOI:10.1146/annurev-phyto-080508-081748 |
| [7] | TENUTA M, LAZAROVITS G. Ammonia and nitrous acid from nitrogenous amendments kill the microsclerotia of Verticillium dahliae. Phytopathology, 2002, 92(3): 255-264. DOI:10.1094/PHYTO.2002.92.3.255 |
| [8] | XIAO C L, SUBBARAO K V, SCHULBACH K F, et al. Effects of crop rotation and irrigation on Verticillium dahliae microsclerotia in soil and wilt in cauliflower. Phytopathology, 1998, 88(10): 1046-1055. DOI:10.1094/PHYTO.1998.88.10.1046 |
| [9] |
裴旭, 赵永新, 周洪友, 等. 8种杀菌剂对向日葵黄萎病菌的室内毒力测定. 内蒙古农业大学学报(自然科学版), 2011, 32(1): 72-75. PEI X, ZHAO Y X, ZHOU H Y, et al. Fungicide screening and toxicity against Verticillium dahliae in sunflower. Journal of Inner Mongolia Agricultural University (Natural Science Edition), 2011, 32(1): 72-75. (in Chinese with English abstract) |
| [10] |
王颖. 向日葵黄萎病生防菌的筛选及其防治. 呼和浩特: 内蒙古农业大学, 2011: 56-58. WANG Y. Screening and prevention of an antagonistic bacteria strain Y1-2 of sunflower Verticillium Wilt. Hohhot: Inner Mongolia Agricultural University, 2011: 56-58. (in Chinese with English abstract) http://cdmd.cnki.com.cn/Article/CDMD-10129-1011178657.htm |
| [11] |
徐嘉俐. 向日葵黄萎病生防木霉菌的筛选及其生防机制的研究. 呼和浩特: 内蒙古农业大学, 2016: 66-68. XU J L. Screening of antagonistic Trichoderma to sunflower yellow wilt and study on the biocontrol mechanisms. Hohhot: Inner Mongolia Agricultural University, 2016: 66-68. (in Chinese with English abstract) http://cdmd.cnki.com.cn/Article/CDMD-10129-1016249376.htm |
| [12] | KABIR Z, BHAT R G, SUBBARAO K V. Comparison of media for recovery of Verticillium dahliae from soil. Plant Disease, 2004, 88: 49-55. DOI:10.1094/PDIS.2004.88.1.49 |
| [13] | DU TOIT L J, DERIE M L, HERNANDEZ-PEREZ P. Verticillium wilt in spinach seed production. Plant Disease, 2005, 89(1): 4-11. DOI:10.1094/PD-89-0004 |
| [14] | RAMPERSAD S N. Verticillium dahliae (Kleb.) infecting pumpkin seed. Journal of Phytopathology, 2010, 158(5): 329-333. DOI:10.1111/jph.2010.158.issue-5 |
| [15] | BANNO S, SAITO H, SAKAI H, et al. Quantitative nested real-time PCR detection of Verticillium longisporum and V. dahliae in the soil of cabbage fields. Journal of General Plant Pathology, 2011, 77(5): 282-291. DOI:10.1007/s10327-011-0335-9 |
| [16] | CHILVERS M I, DU TOIT L J, AKAMATSU H, et al. A real-time, quantitative PCR seed assay for Botrytis spp. that cause neck rot of onion. Plant Disease, 2007, 91(5): 599-608. DOI:10.1094/PDIS-91-5-0599 |
| [17] | DURESSA D, RAUSCHER G, KOIKE S T, et al. A real-time PCR assay for detection and quantification of Verticillium dahliae in spinach seed. Phytopathology, 2012, 102(4): 443-451. DOI:10.1094/PHYTO-10-11-0280 |
| [18] | MBOFUNG G C Y, PRYOR B M. A PCR-based assay for detection of Fusarium oxysporum f. sp. lactucae in lettuce seed. Plant Disease, 2010, 94(7): 860-866. DOI:10.1094/PDIS-94-7-0860 |
| [19] | GLYNN N C, EDWARDS S G. Evaluation of PCR assays for quantifying seed-borne infection by Fusarium and Microdochium seedling blight pathogens. Journal of Applied Microbiology, 2010, 108(1): 81-87. DOI:10.1111/jam.2009.108.issue-1 |
| [20] | HARRIS D C, YANG J R, RIDOUT M S. The detection and estimation of Verticillium dahliae in naturally infested soil. Plant Pathology, 1993, 42(2): 238-250. DOI:10.1111/ppa.1993.42.issue-2 |
| [21] | DOBINSON K F. Genetic transformation of the vascular wilt fungus Verticillium dahliae. Canadian Journal of Botany, 1995, 73(5): 710-715. DOI:10.1139/b95-076 |
| [22] |
张键. 向日葵大丽轮枝菌T-DNA突变体库的构建及微菌核形成和致病力相关基因的研究. 呼和浩特: 内蒙古农业大学, 2016: 72-75. ZHANG J. The T-DNA mutant library construction and the function study on genes involved in the microsclerotia formation and pathogenicity. Hohhot: Inner Mongolia Agricultural University, 2016: 72-75. (in Chinese with English abstract) http://cdmd.cnki.com.cn/Article/CDMD-10129-1016250036.htm |
| [23] | SAMBROOK J, FRITSCH E F, MANIATIS T. Molecular Cloning: A Laboratory Manual. USA: Cold Spring Harbor Laboratory Press, 2001: 895-909. |
| [24] | WHITE T J, BRUNS T, LEE S, et al. Amplification and direct sequencing of fungal ribosomal RNA genes for phylogenetics//INNIS M A, GELFAND D H, SNINSKY J J, et al. PCR Protocols: A Guide to Methods and Applications. SanDiego, USA: Academic Press, 1990, 38: 315-322. |
| [25] |
朱有勇, 王云月, LYONB R. 大丽轮枝菌核糖体基因ITS区段的特异扩增. 植物病理学报, 1999, 29(3): 250-255. ZHU Y Y, WANG Y Y, LYON B R. PCR detection of Verticillium dahliae from diseased plants. Acta Phytopathologica Sinica, 1999, 29(3): 250-255. (in Chinese with English abstract) |
| [26] |
任杰, 张贵, 曹雄, 等. 向日葵黄萎病菌不同培养型产毒能力和致病力分化. 中国油料作物学报, 2014, 36(3): 393-397. REN J, ZHANG G, CAO X, et al. Crude toxin production ability and pathogenicity differentiation on sunflower Verticillium dahliae. Chinese Journal of Oil Crop Sciences, 2014, 36(3): 393-397. (in Chinese with English abstract) DOI:10.7505/j.issn.1007-9084.2014.03.016 |
| [27] | ATALLAH Z K, BAE J, JANSKY S H, et al. Multiplex realtime quantitative PCR to detect and quantify Verticillium dahliae colonization in potato lines that differ in response to Verticillium wilt. Phytopathology, 2007, 97(7): 865-872. DOI:10.1094/PHYTO-97-7-0865 |
| [28] | BOWERS J H, NAMETH S T, RIEDEL R M, et al. Infection and colonization of potato roots by Verticillium dahliae as affected by Pratylenchus penetrans and P. crenatus. Phytopathology, 1996, 86(6): 614-621. DOI:10.1094/Phyto-86-614 |
| [29] | MERCADO-BLANCO J, RODRÍGUEZ-JURADO D, PÉREZARTÉS E, et al. Detection of the defoliating pathotype of Verticillium dahliae in infected olive plants by nested PCR. Plant Pathology, 2001, 50(1): 609-619. |
| [30] | SOUSA M V, MACHADO J D C, SIMMONS H E, et al. Realtime quantitative PCR assays for the rapid detection and quantification of Fusarium oxysporum f. sp. phaseoli in Phaseolus vulgaris (common bean) seed. Plant Pathology, 2015, 64(2): 478-488. DOI:10.1111/ppa.2015.64.issue-2 |
| [31] | SACKSTON W E, MARTENS J W. Dissemination of Verticillium albo-atrum on seed of sunflower (Helianthus annuus). Canadian Journal of Botany, 1959, 37(5): 759-768. DOI:10.1139/b59-061 |
| [32] | MA Z H, MICHAILIDES T J. Approaches for eliminating PCR inhibitors and designing PCR primers for the detection of phytopathogenic fungi. Crop Protection, 2007, 26(2): 145-161. DOI:10.1016/j.cropro.2006.04.014 |
 2018, Vol. 44
2018, Vol. 44


