| 基于相关序列扩增多态性分子标记的桂花栽培品种演化分析 |
2. 浙江理工大学建筑工程学院,杭州 310018
2. School of Civil Engineering and Architecture, Zhejiang Sci-Tech University, Hangzhou 310018, China
人类早在农耕时代就已经开始了植物驯化,但对常见栽培植物的起源仍然未知;直到达尔文进化论的创立和孟德尔遗传定律的提出,人们才对植物栽培品种的形成有了科学的认识,并通过育种,使植物栽培品种的形成速度远超过去[1]。另一方面,栽培品种育种工作也为进化论和遗传学的发展提供了大量数据支持。研究表明,栽培植物演化模式包括突变、重组、选择、隔离和漂变,而转基因技术和分子标记等现代生物技术从这5个方面辅助植物育种工作,加快了育种进度。研究发现,对植物培育过程进行资料分析是研究栽培植物演化最有效的途径[1]。17—18世纪,欧洲从亚洲和美洲引进大量野生种,开展了一系列选育工作,培育出一大批栽培植物,并且详细记录了百合(Lilium brownie)[2]、郁金香(Tulipa gesneriana)[3]、报春花(Primula malacoides)[1]和四季海棠(Begonia semperflorens)[1]等观赏植物的培育过程,为栽培植物演化研究提供了重要资料。因此,研究栽培植物品种的演化对进化论、遗传学的发展以及栽培植物的培育工作都有重要意义。
桂花(Osmanthus fragrans)系木犀科(Oleaceae)木犀属(Osmanthus)木犀组(Sect. Osmanthus)植物,为中国十大名花之一[4],其品种类型丰富,近缘物种类群清楚,是研究栽培植物起源和进化的极好材料。但在长期栽培历史中,桂花人工培育资料严重缺失,只能从古人的诗词歌赋和花的性状推测四季桂品种群最为原始,秋桂品种按照花色由浅至深逐渐分化,丹桂来源于芽变[4]。但由于古人对桂花的描写往往经过艺术加工,其表型与基因型之间可能会有差异[5],因此栽培品种演化仍然存在许多疑点,无法科学指导育种工作,这也是桂花栽培品种数远少于其他观赏植物的原因之一。
系统发生树能够反映共同祖先的种或品种的演化关系,是研究选育资料稀缺的植物栽培品种演化历程的有效手段[5]。目前已使用核基因组(nDNA)、叶绿体基因组(cpDNA)和线粒体基因组(mtDNA)中单个或少数几个基因构建了中国簇毛黄耆亚属(Pogonophace)[6]、大黄(Rheum officinale Baill.)[7]、木通科(Lardizabalaceae)[8]、鸢尾属(Iris)[9]、七筋菇属(Clintonia)[10]等多个植物科、属、种系统发生树,但单一或少量基因不够全面。研究表明,分子标记能够揭示DNA序列差异,通过对整个基因组随机或选择性扫描,全面反映物种、品种和群体间的遗传差异,不受环境影响。目前,将多种分子标记结果与DNA序列研究结果进行综合分析已经成为植物演化研究趋势[5]。近年来,ISSR、RAPD、AFLP、SRAP、SSR和SCoT等多种分子标记被应用于桂花品种分类和遗传多样性研究,构建了多张桂花品种聚类分析图谱[11-16],但由于没有选择合适的外部群体,只能反映不同栽培品种的亲缘关系,无法确定演化方向。圆锥花序组在现代木犀属4组中最为原始,是包括桂花在内的木犀组植物的共同祖先;牛矢果(O. matsumuranus)作为圆锥花序组中仅有的6个种之一[17],是构建桂花栽培品种系统发生树的理想外部群体。
相关序列扩增多态性(sequence-related amplified polymorphism, SRAP)是一种新型分子标记,该标记根据外显子富含GC而启动子、内含子富含AT设计引物进行扩增,具有高效、操作简单、无需基因组信息、引物通用、成本低、有较高共显性和在基因组内均匀分布等显著优点,是演化分析的理想标记[18]。SRAP扩增产物通常使用聚丙烯酰胺凝胶电泳银染检测,存在操作复杂、低通量、影响因素较多、分辨率较低、试剂毒害大和需要人工判断带型数据等诸多缺点,而毛细管电泳技术(capillary electrophoresis,CE)能够克服以上缺点,实现无毒害、高通量、自动化、高分辨率、稳定的DNA快速检测[19]。李婷婷等[20]将SRAP分子标记技术与毛细管电泳技术相结合,获得不同种源榉树(Zelkova schneideriana)大量稳定可靠的标记数据,通过对70个榉树优良单株进行聚类分析,构建了DNA指纹图谱,表明CE-SRAP在植物进化和品种鉴定研究中具有巨大潜力。
在野生植物逐渐栽培化的过程中,作为人工选育的重要依据,观赏性状会强烈影响选择环节,导致栽培品种中优势基因型增加。花色作为桂花的重要观赏性状之一,其变化在一定程度上重演了桂花栽培品种的演变过程[21],因此有必要将花色变异与DNA分子标记数据进行综合分析,以揭示其遗传演化规律。此外,生物进化中许多基本问题都涉及交配过程,植物性别系统通过影响交配系统进而决定种群基因型频率、基因流和选择等多个进化因素[22]。桂花独特的功能型雄全异株性别系统[23]是否会影响花色等观赏性状基因型频率,从而决定栽培品种的演化历程值得进一步探讨。
本研究通过SRAP分子标记和毛细管电泳技术对45份桂花材料进行群体结构分析,使用亚群作为演化单位,以木犀属中最为原始的种牛矢果为外部群体,构建桂花品种的系统发生树,分析桂花性别系统进化,以期为桂花品种演化理论的研究提供新的方法。此外,根据现有研究推测桂花花色进化模式,为进一步揭示桂花花色进化遗传机制提供理论依据。
1 材料与方法 1.1 材料选用45份桂花(Osmanthus fragrans Lour.)材料,以木犀属华东木犀(O. cooperi)的2个品种“玉洁”和“雪桂”、柊树(O. heterophyllus)和石山桂(O. fordii)作为对照,以木犀属牛矢果(O. matsumuranus)作为外部群体(附表 1,http://www.zjujournals.com/agr/EN/article/showSupportInfo.do?id=10492)。所有材料均取自杭州市园林绿化股份有限公司青山湖基地桂花种质资源圃。采集新鲜叶片,迅速装入冰袋中,-20 ℃储存备用。
1.2 方法 1.2.1 DNA提取取0.5~1.0 g叶片于液氮中速冻后快速研磨至粉末,使用BIOTEKE植物基因组快速提取试剂盒(购自北京百泰克生物技术有限公司)提取DNA。使用1.0%琼脂糖凝胶电泳检测DNA质量,并使用NanoDropTM 2000/2000c(购自Thermo公司,美国)测定浓度,稀释至20~50 ng/μL,-20 ℃储存备用。
1.2.2 SRAP聚合酶链式反应扩增采用10 μL反应体系:20~50 ng/μL DNA模板1 μL,10 μmol/L正反引物各1 μL,2 × Power Taq PCR MasterMix(百泰克试剂盒,北京)5 μL,双蒸水2 μL。反应程序:94 ℃预变性8 min;94 ℃变性1 min,35 ℃退火1 min,72 ℃延伸90 s,5个循环;94 ℃变性1 min,59.3 ℃退火1 min,72 ℃延伸90 s,35个循环;最后72 ℃延伸8 min;4 ℃保存。
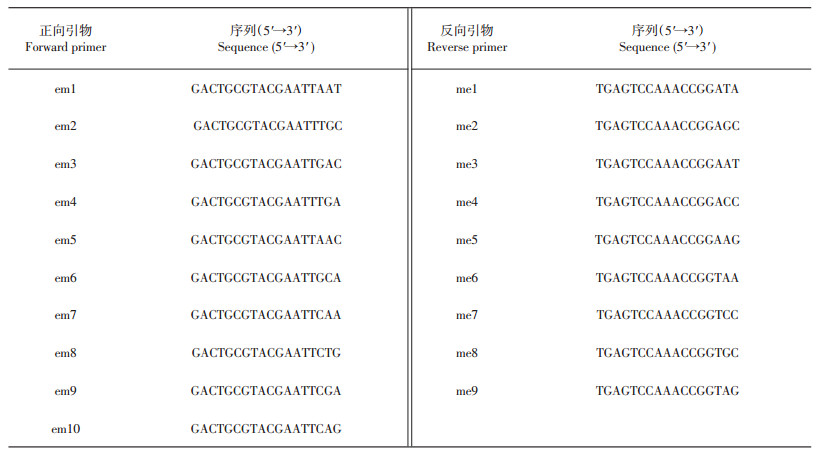
聚合酶链式反应(polymerase chain reaction, PCR)产物使用2.0%琼脂糖凝胶电泳检测以筛选引物。10个正向引物和9个反向引物(表 1)组成90对引物组合,由上海英骏生物技术有限公司合成。使用筛选出的引物组合对50个供试材料进行SRAP PCR扩增,扩增产物参照李婷婷等[20]的方法,使用全自动核酸蛋白分析系统Qsep100(购自Bioptic公司,中国台湾)进行毛细管电泳检测,以DNA标准品(5 000 bp DNA Ladder)标定扩增条带大小,使用Q Editor软件(Bioptic公司,中国台湾)记录峰值图和毛细管模拟电泳图,结合人工判断,输出标记数据,并转化为0或1。
| 表1 SRAP引物序列 Table 1 Sequences of SRAP primers |
 |
| 点击放大 |
使用POPGENE 1.32[24]分析桂花栽培品种的遗传多样性,计算观测等位基因数(Na)、有效等位基因数(Ne)、Nei遗传多样性指数(H)、Shannon多样性信息指数(I)、总群体基因多样性指数(Ht)、种群内基因多样性指数(Hs)、种群间遗传分化系数(Gst)和、Nei遗传距离。桂花群体结构分析使用Structure 2.34[25]中的混合模型(admixture model)对45份桂花材料进行群体结构分析。使用NTSYSpc v2.10e计算亚群间的、Nei遗传距离,导入MEGA 6[26],以牛矢果为外部群体,采用邻接法(neighbor-joining)构建桂花亚群有根系统发生树,并分析桂花品种、花色和性别系统的演化历程。
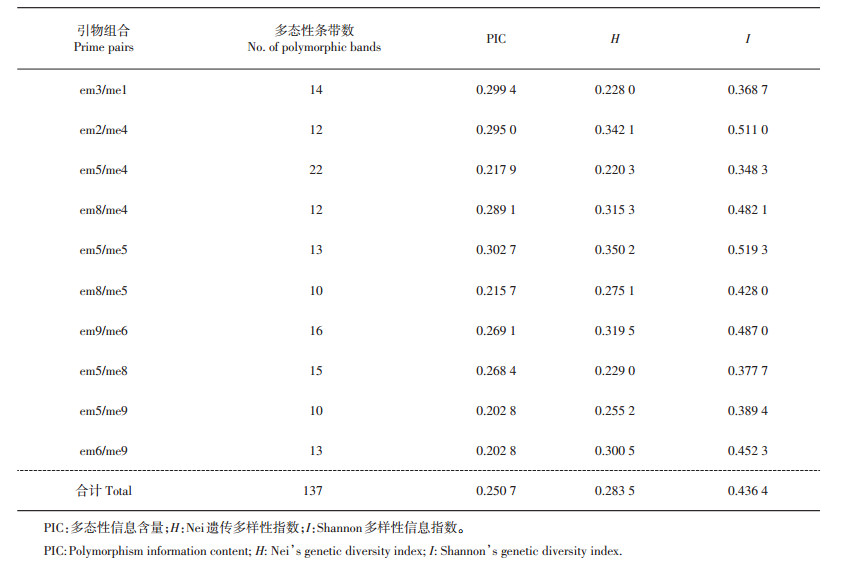
2 结果与分析 2.1 SRAP-PCR扩增结果从90对SRAP引物组合中筛选出10对扩增结果稳定、条带清晰的引物组合,对50个供试材料进行扩增。毛细管电泳检测(附图 1,http://www.zjujournals.com/agr/EN/article/showSupportInfo.do?id=10492)和统计结果(表 2)表明:总共检测到137个多态性位点,平均每对引物13.7个位点,多态性信息含量(polymorphism information content, PIC)为0.202 8~0.302 7,平均为0.250 7;、Nei遗传多样性指数(H)为0.220 3~0.350 2,平均为0.283 5;Shannon多样性信息指数(I)为0.348 3~0.519 3,平均为0.436 4。
 |
| ln P(D)为每个K值采用威尔科克森检验(Wilcoxon test)得到的对数似然值;K为估计的亚群数。图A为参数不做数迭代长度对计算结果的影响,该值越高,则计算误差越小,但计算速度越慢;图B为亚群数检验,当ln P(D)位于拐点且为极大值时,K最为接近真实值。 ln P(D) is the log-likelihood value for each K value using Wilcoxon test. K is the estimation of numbers of subgroups. Fig. A shows the effect of the parameter-length of burnin period on the calculation results. The higher the parameter is, the smaller the calculation error is, but the slower the calculation speed is. Fig. B shows the best K value test. When ln P(D) reaches the inflection point and the maximum value, the K is close to a true value. 图1 Structure软件参数估算 Fig. 1 Parameter estimate for Structure software |
| 表2 SRAP扩增结果 Table 2 Results of SRAP amplification |
 |
| 点击放大 |
Structure 2.34软件模拟结果(图 1A)表明:随着不做数迭代的长度从1万增加至10万,ln P(D)的离散程度先减少后增加;在7.5万时,离散程度最低,模拟的结果最稳定可靠。设定估计的亚群数K值为1~10,计算相对应的ln P(D)和Q值(第i品种的基因组变异源于第k群体的概率),每个K值运行5次。结果表明,在K=7时,ln P(D)最大且为拐点(图 1B)。由此判断,45份桂花材料具有明显的群体结构,可分为7个亚群体。
以K=7绘制群体结构图(图 2)。45份桂花材料中有36个(占80%)在某个亚群体中的Q值大于或等于0.5,说明这些桂花品种遗传组分相对单一。36份桂花材料可分为7个亚群:亚群1包含“绿梗籽银桂”、“籽银桂”、“宽叶籽银桂”、“潢川金桂”和“天香台阁”;亚群2包含“金球桂”、“波叶金桂”、“火炼金丹”和“火炼金丹”(跳枝);亚群3包含“青尖”、“小叶苏桂”、“玉帘银丝”、“串银球”、“大叶银桂”、“米花籽银桂”、“杭州黄”、“早籽黄”、“大花金桂”、“四季桂”、“圆叶四季桂”、“大叶佛顶珠”、“橙黄四季桂”、“日香桂”和“淡妆”,其中除“大花金桂”外,其他都为四季桂和银桂品种;亚群4包含“金秋早”、“堰虹桂”、“四季桂黄”和“墨宝香”;亚群5包含“状元红”、“朱砂丹桂”、“醉肌红”和“橙红丹桂”;亚群6包含“速生丹桂”和“莲籽丹桂”;亚群7包含“彩叶桂”和“天女散花”。“早银桂”、“庐州黄”、“丛中笑”、“硬叶丹桂”、“玉玲珑”、“矮黄”、“雄黄桂”、“白洁”和“晚银桂”9个品种在任意亚群体的Q值均小于0.5,表明这些桂花品种遗传背景复杂,划分为一个混合群体,即亚群8。
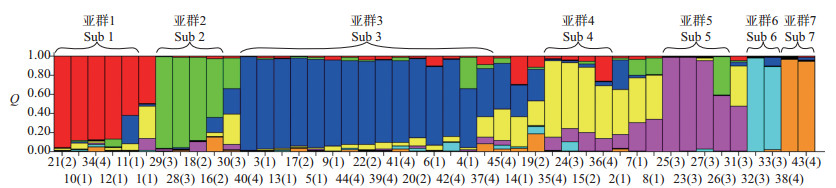 |
| Q为第i品种的基因组变异源于第K群体的概率,当Q≥0.50时,该品种属于群体K。(1~45):桂花品种(见附表 1,http://www.zjujournals.com/agr/EN/article/showSupportInfo.do?id=10492)。(1):银桂品种群;(2):金桂品种群;(3):丹桂品种群;(4):四季桂品种群。 Q is the probability that the genome variation of the i-th cultivar is from the K-th subgroup; when Q≥0.50, this cultivar belongs to subgroup K. Numbers 1-45 denote sweet osmanthus cultivars (details are shown in attached Table 1, http://www.zjujournals.com/agr/EN/article/showSupportInfo.do?id=10492). (1): Albus Group; (2): Luteus Group; (3): Aurantiacus Group; (4): Asiaticus Group. 图2 45份桂花材料群体结构图 Fig. 2 Population structure of 45 sweet osmanthus materials |
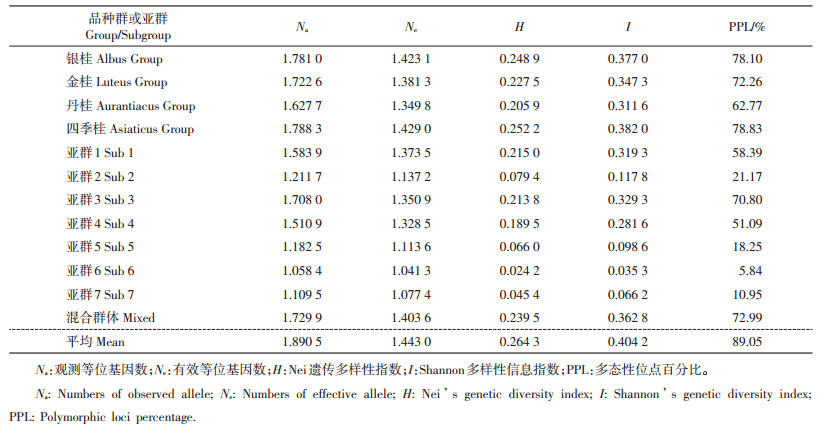
以137个SRAP多态性位点分析桂花4个品种群和8个亚群的遗传多样性和遗传分化。结果(表 3)表明:45份桂花材料总体观测等位基因数(Na)、有效等位基因数(Ne)、Nei遗传多样性指数(H)、Shannon多样性信息指数(I)和多态性位点百分比(polymorphic loci percentage, PPL)分别为1.890 5、1.443 0、0.264 3、0.404 2和89.05%;4个桂花品种群的Na与Ne分别为1.627 7~1.788 3和1.349 8~ 1.429 0,H与I分别为0.205 9~0.252 2和0.311 6~ 0.382 0,PPL为62.77%~78.83%;桂花8个亚群的Na、Ne、H、I和PPL分别为1.058 4~1.729 9、1.041 3~ 1.403 6、0.024 2~0.239 5、0.035 3~0.362 8和5.84%~ 72.99%。混合群体的遗传多样性明显高于其他亚群,这也表明混合群体中的桂花品种遗传背景相对复杂。
| 表3 桂花亚群和栽培品种群的SRAP标记遗传多样性分析 Table 3 Genetic diversity analysis of subgroups and cultivar groups of sweet osmanthus based on SRAP markers |
 |
| 点击放大 |
4个桂花品种群总群体基因多样性指数(Ht)为0.262 9,品种群内基因多样性指数(Hs)为0.233 6,种群间的遗传分化系数(Gst)为11.14%,基因流(Nm)为3.989 7,存在较广泛的基因交流。8个桂花亚群的Ht、Hs、Gst和Nm分别为0.275 4、0.134 1、51.32%和0.474 4。亚群间的遗传变异大于亚群内的变异,亚群间有较为明显的分化,且基因交流极少,而各亚群内的品种遗传背景较为单一。
2.4 桂花品种系统发生树桂花亚群的系统发生树(图 3)表明,在桂花品种的进化历程中,大致经历了10个阶段:在水平A处,木犀属演化成圆锥花序组(牛矢果作为外部群体)和木犀组(华东木犀、柊树、石山桂和桂花);在B水平处,木犀组演化成桂花;在C水平处,桂花最先形成亚群3和Ⅰ2大群体,Ⅰ类在D水平处形成Ⅱ和Ⅲ类;在水平E处,Ⅱ类分化为Ⅳ和四季桂品种“矮黄”;在水平F处,Ⅳ类演化成Ⅴ和Ⅵ 2类;在G水平处,Ⅴ类演化成Ⅶ和亚群4;在水平H处,Ⅲ类演化成Ⅹ和银桂品种“庐州黄”,Ⅶ类形成Ⅷ和Ⅸ2类;在水平Ⅰ处,形成桂花亚群6、“晚银桂”以及5个小类;在J水平处,5个小类分别演化成4个亚群和10个桂花品种。
 |
| sub 1~sub 7为7个亚群;YYDG、YLL、BJ、XHG、WY、ZY、AH、CZX、LZH依次为“硬叶丹桂”“、玉玲珑”“、白洁”“、雄黄桂”“、晚银桂”“、早银桂”“、矮黄”“、丛中笑”“、庐州黄”,属于混合群体;Osmanthus为4个木犀属物种,作为对照;outgroup为牛失果(外部群体)。 sub 1-sub 7 are seven subgroups; YYDG, YLL, BJ, XHG, WY, ZY, AH, CZX, LZH are"Yingye Dangui", "Yu Linglong", "Baijie", "Xionghuanggui", "Wan Yingui", "Zao Yingui", "Ai Huang", "Congzhong Xiao", "Luzhou Huang", respectively, which belong to a mixed group; Osmanthus are four species of Osmanthus used as controls; outgroup is O. matsumuranus. 图3 桂花亚群的系统发生树 Fig. 3 Phylogenetic tree of sweet osmanthus |
亚群3(sub 3)是最早分化出的一个亚群,为桂花中最原始的一类,57.1%的银桂品种和50%的四季桂品种聚在该亚群。这表明大部分四季桂和银桂是桂花中较为原始的品种,且亲缘关系较近。部分四季桂与银桂品种处于较晚的分支。具两性花丹桂品种与具单性雄花丹桂品种处于2个独立分支,且前者早于后者形成。金桂品种几乎均匀分布于D水平后的各分支上。丹桂品种“火炼金丹”与金桂品种“金球桂”、“波叶金桂”极其相似,并且“金球桂”能够芽变成丹桂颜色,而“火炼金丹”可以芽变成金桂颜色,这3个品种以及“火炼金丹”芽变枝构成亚群2(sub 2),与“雄黄桂”处于同一分支。
2.5 桂花性别系统进化分析两性花品种比例随着桂花演化进程逐渐下降,且较早聚在一起(表 4):45个桂花品种两性花总体比例为29%,在水平C处,两性花比例为27%,从水平D到水平F,Ⅰ类逐渐分化出Ⅱ、Ⅳ、Ⅴ,其两性花比例依次下降为21%、18%、0%;在较早的水平D和F处,Ⅰ类中大部分两性花品种聚集成Ⅲ类和Ⅵ类。此现象表明桂花的雄全异株性别系统起源于雌雄同株,是雌雄同花演化为雌雄异株的中间状态。
| 表4 桂花性别系统演化 Table 4 Sexual system evolution of Osmanthus fragrans |
 |
| 点击放大 |
植物的进化以群体为单位,其本质为群体结构的变化,即群体基因频率和基因型频率的变化[27],因此,选择合适的划分依据确定研究群体是桂花品种演化研究的关键。中性学说认为DNA或蛋白的中性突变在进化中起主导作用,是遗传多样性的来源,中性突变不引起植物的性状变异,即无选择意义[28]。而多种分子标记的桂花品种聚类分析结果都与传统分类学存在较多差异[11-16],表明少数表型性状的差异只占整个基因遗传多样性的一小部分,以此为依据划分的4大桂花栽培品群可能并不适合作为品种演化的研究单位。群体结构分析表明,桂花栽培品种具有明显的群体结构,36个桂花材料可分为7个亚群,而4个栽培品群遗传背景复杂,都出现了多个亚群遗传混杂的现象。亚群间的遗传分化系数远高于品种群间,亚群间的基因流明显小于品种群间。在本研究中,“火炼金丹”芽变枝与“火炼金丹”同属于亚群2,而“彩叶桂”与四季桂品种“天女散花”同属于亚群7,而按照传统分类则属于不同品种群,这进一步证明群体结构能较为全面地反映品种间的亲缘关系,划分的亚群比4大品种群更适合作为演化单位。以亚群为单位构建的系统发生树较为完整地还原了桂花品种的演化历程,本研究结果表明大部分四季桂品种和银桂品种最先形成,雄性丹桂品种最后形成,其他品种处于中间阶段,证明淡色花为原始性状,深色花为进化性状。此外,演化进程和性别比例的变化证明两性花为原始性状,单性花为进化性状。两性花丹桂品种远早于单性花丹桂品种形成,表明两种性别的丹桂品种可能具有不同起源。
3.2 桂花繁育系统基因突变为生物进化提供了原始物质基础,而重组则提供了进一步的变异,说明重组无论在进化还是育种过程中都具有重要作用[1]。植物繁育系统本质上就是控制重组事件的所有有性特征,其中性别系统和交配系统对后代遗传成分影响极大,一直是繁育系统的研究重点[29]。雄全异株是一种独特且稀少的性别系统,处于雌雄同花和雌雄异株的过渡状态,其起源一直存在争议[30]。目前,大部分观点认为雄全异株起源于雌雄异株[31]。郝日明等[23]根据野生桂花性别比例证明桂花性别系统为功能型雄全异株;徐沂春[15]从桂花具有不同状态退化雌蕊的现象推测桂花的雄全异株起源于雌雄同株。在本研究中两性花品种比例随着桂花栽培品种演化进程逐渐下降,在一定程度上证明了徐沂春[15]的推测,但不排除人工选择对两性花比例的影响。张洪伟[32]和李稚[33]的研究都表明,桂花为自交和异交相混合的兼性交配系统,风媒和虫媒都可以进行传粉,但访花昆虫种类较少,访花频率不高。一般认为自交来源于异交,且不可逆[34],但随着单性雄性比例增加,桂花异交比例不可避免地增加,暗示桂花的异交可能来源于自交。徐沂春[15]研究发现,野生桂花群体间遗传分化较低且存在频繁的基因交流,也表明野生桂花异交可能占据主导;这与黄蓉花属(Dalechampia)[35]相似。但随着野生桂花资源的流失,近交概率逐渐增加,导致桂花遗传多样性显著减少,群体间遗传愈加分化,而人工选择更加阻断了基因交流,加速了遗传分化。本研究栽培品种亚群间分化系数达到51.32%,表现出明显的分化。但桂花的繁育系统进化以及对野生群体和栽培品种演化的影响仍然存在许多未知,需要进一步研究。
3.3 桂花花色进化的遗传机制推测花色作为植物最重要的观赏性状之一,其进化遗传机制一直是遗传学研究的重点,花色改良也是观赏植物育种的重要目标。丰富多彩的花色主要由花瓣中的色素种类和含量决定,此外,光照、温度等环境因素和液泡pH、色素共着色和金属离子含量等内环境也对花色具有重要影响[36-38]。研究表明,植物花朵中含有的色素主要有类黄酮(flavonoid)、类胡萝卜素(carotenoid)及生物碱(alkaloid)[36]。
一般认为,桂花花色演化顺序为白色-黄色-橙红色[39],这与本研究构建的桂花品种系统发生基本一致,但对于桂花花色进化的遗传机制仍然未知。已有的研究从生理和基因层面阐述了桂花花色决定机制,即类胡萝卜素在花瓣中的种类和含量差异是导致桂花花色不同的主要原因:白色花瓣中几乎不含有类胡萝卜素,黄色花瓣中主要积累β-胡萝卜素,橙红色花瓣中大量积累α-胡萝卜素和β-胡萝卜素,而CCD4基因差异表达导致了不同品种花瓣中类胡萝卜素含量的差异[40-41]。研究表明,类胡萝卜素裂解双加氧酶(carotenoid cleavage dioxygenases, CCDs)能够降解类胡萝卜素[42],且桂花CCD4基因也可以裂解β-胡萝卜素等类胡萝卜素[43]。菊花白色花瓣的CmCCD4a转录水平显著高于黄色花瓣,通过RNA干扰使该基因的表达沉默后,花瓣变为黄色[44]。因此可以推测,CCD4基因的突变可能是桂花芽变为橙红花色的原因之一。
本文对桂花花色是否也为单个基因控制的质量性状进行了探讨。假设桂花花色由CCD4单基因控制,CCD4原有等位基因为A,基因突变产生的功能缺失型等位基因为a,则纯合体aa无法降解类胡萝卜素,花色为橙红色,纯合体AA能产生CCDs,降解类胡萝卜素,花色为白色,杂合体Aa能产生少量CCDs,花色表现为黄色。在理想群体中,应该只出现白色、黄色和橙红色3种花色的个体,且保持一定比例,但目前已发现的野生桂花群体中,花色都表现为淡黄白色,而栽培品种的花色则表现为由浅至深的连续变异。说明桂花花色可能是由多基因决定的数量性状,多基因位点的加性效应累积导致桂花花色的连续变异。在拟南芥中发现CCDs家族的9个同源基因分布于不同染色体上[45],且CCD1基因功能的缺失会导致拟南芥种子中类胡萝卜素的积累[43]。可以推测,桂花基因组中可能含有CCDs家族的多个同源基因共同调控花色。
野生桂花和栽培品种中橙红色花色个体比例差异极大是桂花花色进化的另一个疑点。研究发现,桂花野生群体规模较小,彼此之间相互隔离[46],突变产生的CCDs功能缺失型等位基因拷贝极少,单性雄性个体的比例远高于两性株[23],突变产生的新等位基因极有可能只通过花粉遗传到下一代;因此,新等位基因的遗传漂变可能是桂花野生群体中罕见橙红色花色个体的原因。而橙红色花色一直视为桂花中的珍品,长期的人工选择使具橙红色花色的栽培品种较为常见。考虑到桂花杂交的难度和较长的时间周期,很难通过构建遗传连锁图进行数量性状位点(QTLs)定位,而关联分析无需构建杂交群体,利用自然群体即可对桂花花色、花型和花香等观赏性状的遗传机制进行研究。因此,在未来的工作中,可利用关联分析进行进一步的研究。
4 结论基于SRAP分子标记的群体结构较好地反映了桂花品种间的亲缘关系,以此为单位构建的系统发生树表明:桂花的进化历程大致分为10个阶段(A~J水平),由银桂品种和四季桂品种构成的亚群3最先形成,由雄性丹桂品种构成的亚群5最晚形成,金桂品种较为均匀地分布于D水平后的各分支上,为桂花品种的演化理论提供了有益的补充。在桂花演化历程中,两性花品种比例逐渐降低,证明两性花是原始性状,其雄全异株性别系统起源于雌雄同株,是雌雄同花演化为雌雄异株的中间状态。此外,本研究推测桂花花色由CCDs家族的多个同源基因共同调控,由于较小的群体和功能型雄全异株的性别系统,遗传漂变在花色进化过程中起主导作用,使得CCDs功能缺失型的突变等位基因丧失,造成野生桂花罕见橙红色花色植株。这为桂花花色遗传机制的进一步研究奠定了理论基础。
| [1] | HORN W. The patterns of evolution and ornamental plant breeding. Acta Horticulturae, 2004(651): 19-31. |
| [2] | VAN TUYL J M, ARENS P. Lilium: Breeding history of the modern cultivar assortment. Acta Horticulturae, 2011(900): 223-230. |
| [3] | ANDERSON N O. Flower Breeding and Genetics. Netherlands: Springer, 2006: 623-641. |
| [4] |
向其柏, 刘玉莲. 中国桂花品种图志. 杭州: 浙江科学技术出版社, 2008: 2-86. XIANG Q B, LIU Y L. An Illustrated Monograph of the Sweet Osmanthuss Cultivars in China. Hangzhou: Zhejiang Science & Technology Press, 2008: 2-86. (in Chinese with English abstract) |
| [5] |
史全良, 吴均章. 植物系统学研究中常用基因分子标记特点分析. 生物学通报, 2000, 35(12): 4-6. SHI Q L, WU J Z. The characters of general gene markers in plant molecular systematics studying. Bulletin of Biology, 2000, 35(12): 4-6. (in Chinese with English abstract) DOI:10.3969/j.issn.0006-3193.2000.12.002 |
| [6] |
康云, 张明理, 陈之端. 基于ITS序列对中国簇毛黄耆亚属(黄耆属)系统学问题的初步研究. 植物学报:英文版, 2003, 45(2): 140-145. KANG Y, ZHANG M L, CHEN Z D. A preliminary phylogenetic study of the subgenus Pogonophace (Astragalus) in China based on ITS sequence data. Acta Botanica Sinica, 2003, 45(2): 140-145. (in Chinese with English abstract) |
| [7] |
姬可平, 李啸红, 李应东, 等. 应用rRNA基因间隔区碱基测序对中药(大黄)进行鉴定. 世界科学技术:中药现代化, 2002, 4(4): 44-47. JI K P, LI X H, LI Y D, et al. Identification of Rheum palmatum L. (Dahuang) by the method of measuring internal transcribed spacer regions of rRNA gene. World Science and Technology Modernization of Traditional Chinese Medicine, 2002, 4(4): 44-47. (in Chinese with English abstract) |
| [8] |
王峰, 李德铢, 杨俊波. 基于叶绿体Trn L-F序列和联合数据分析木通科的分子系统发育. 植物学报:英文版, 2002, 44(8): 971-977. WANG F, LI D Z, YANG J B. Molecular phylogeny of the Lardizabalaceae based on Trn L-F sequences and combined chloroplast data. Acta Botanica Sinica, 2002, 44(8): 971-977. (in Chinese with English abstract) |
| [9] | MAKAREVITCH I, GOLOVNINA K, SCHERBIK S, et al. Phylogenetic relationships of the Siberian iris species inferred from noncoding chloroplast DNA sequences. International Journal of Plant Sciences, 2003, 164(2): 229-237. DOI:10.1086/346160 |
| [10] | HAYASHI K, YOSHIDA S, UTECH F H, et al. Molecular systematics in the genus Clintonia and related taxa based on rbcL and matK gene sequence data. Plant Species Biology, 2001, 16(2): 119-137. DOI:10.1046/j.1442-1984.2001.00057.x |
| [11] |
段一凡, 王贤荣, 梁丽丽, 等. 古桂遗传多样性与亲缘关系的ISSR分析. 福建林学院学报, 2011, 31(4): 335-339. DUAN Y F, WANG X R, LIANG L L, et al. Analysis of genetic diversity and relationships among ancient sweet osmanthus trees using ISSR markers. Jouranl of Fujian College of Forestry, 2011, 31(4): 335-339. (in Chinese with English abstract) |
| [12] |
张维瑞, 袁王俊, 尚富德. 基于AFLP分子标记的桂花品种核心种质的构建. 西北植物学报, 2011, 32(7): 1349-1354. ZHANG W R, YUAN W J, SHANG F D. Development of core collection of Osmanthus fragrans Lour. cultivars based on AFLP molecular markers. Acta Botanica Boreali-Occidentalia Sinica, 2011, 32(7): 1349-1354. (in Chinese with English abstract) |
| [13] |
胡绍庆, 邱英雄, 吴光洪, 等. 桂花品种的ISSR-PCR分析. 南京林业大学学报(自然科学版), 2004, 9(增刊): 71-75. HU S Q, QIU Y X, WU G H, et al. A ISSR-PCR analysis of the Osmanthus fragrans cultivars. Journal of Nanjing Forestry University (Natural Sciences Edition), 2004, 9(Suppl.): 71-75. (in Chinese with English abstract) |
| [14] |
韩远记, 董美芳, 袁王俊, 等. 桂花品种资源的遗传多样性分析. 植物学报, 2008, 25(5): 559-564. HAN Y J, DONG M F, YUAN W J, et al. Study on the genetic diversity of Osmanthus fragrans cultivars. Chinese Bulletin of Botany, 2008, 25(5): 559-564. (in Chinese with English abstract) |
| [15] |
徐沂春.桂花遗传多样性及不同性别花的花芽特性.浙江, 临安:浙江农林大学, 2013:33-40. XU Y C. The genetic diversity and flower bud characteristics ofdifferent sexes in Osmanthus fragrans. Lin'an, Zhejiang: Zhejiang A & F University, 2013:33-40. (in Chinese with English abstract) http://cdmd.cnki.com.cn/Article/CDMD-10341-1014101669.htm |
| [16] |
袁王俊, 马寅峰, 董美芳, 等. 桂花SCoT标记体系的建立及其在遗传多样性分析中的应用. 园艺学报, 2015, 34(3): 569-575. YUAN W J, MA Y F, DONG M F, et al. Establishment and genetic diversity analysis of SCoT molecular marker system for Osmanthus fragrans. Acta Horticulturae Sinica, 2015, 34(3): 569-575. (in Chinese with English abstract) |
| [17] |
季春峰.木犀属(Osmanthus)的系统分类学研究.南京:南京林业大学, 2004:90. JI C F. Systematic taxonomy of the genus Osmanthus (Oleaceae). Nanjing: Nanjing Forestry University, 2004:90. (in Chinese with English abstract) http://cdmd.cnki.com.cn/Article/CDMD-10298-2004092358.htm |
| [18] |
邱帅, 丁信誉, 席梦利, 等.东方百合SRAP体系优化及引物筛选.南京林业大学学报(自然科学版), 2013, 37(3):6-10. QIU S, DING X Y, XI M L, et al. Optimization of SRAP PCR system and primers screening of oriental lily hybrids. Journal of Nanjing Forestry University (Natural Science Edition), 2013, 37(3): 6-10. (in Chinese with English abstract) http://kns.cnki.net/KCMS/detail/detail.aspx?filename=njly201303001&dbname=CJFD&dbcode=CJFQ |
| [19] |
徐娟, 孔科. 毛细管电泳在DNA分析中的应用. 生物技术, 2004, 14(1): 54-56. XU J, KONG K. Applications of capillary electrophoresis in DNA analysis. Biotechnology, 2004, 14(1): 54-56. (in Chinese with English abstract) |
| [20] |
李婷婷, 朱锦茹, 邱帅, 等. 基于CE-SRAP标记的榉树优良单株指纹图谱构建. 植物遗传资源学报, 2016, 17(1): 169-176. LI T T, ZHU J R, QIU S, et al. Fingerprinting construction for superior individuals of Zelkova schneideriana based on CE SRAP. Journal of Plant Genetic Resources, 2016, 17(1): 169-176. (in Chinese with English abstract) |
| [21] |
臧德奎.桂花品种分类研究.南京:南京林业大学, 2004:47-128. ZANG D K. Study on the cultivar classification of sweet osmanthus. Nanjing: Nanjing Forestry University, 2004:47-128. (in Chinese with English abstract) http://cdmd.cnki.com.cn/Article/CDMD-10298-2004092379.htm |
| [22] |
何田华, 葛颂.植物种群交配系统、亲本分析以及基因流动研究.植物生态学报, 2001, 25(2):144-154. HE T H, GE S. Mating system, paternity analysis and gene flow in plant populations. Acta Phytoecologica, 2001, 25(2):144-154. (in Chinese with English abstract) http://kns.cnki.net/KCMS/detail/detail.aspx?filename=zwsb200102002&dbname=CJFD&dbcode=CJFQ |
| [23] |
郝日明, 赵宏波, 王金虎, 等. 野生桂花繁育系统的观察和研究. 植物资源与环境学报, 2011, 20(1): 17-24. HAO R M, ZHAO H B, WANG J H, et al. Observation and study on breeding system of wild Osmanthus fragrans. Journal of Plant Resources and Environment, 2011, 20(1): 17-24. (in Chinese with English abstract) |
| [24] | YEH F C. Population genetic analysis of codominant and dominant markers and quantitative traits. Belgian Journal of Botany, 1997, 129: 157. |
| [25] | FALUSH D, STEPHENS M, PRITCHARD J K. Inference of population structure using multilocus genotype data: Dominant markers and null alleles. Molecular Ecology Notes, 2007, 7(4): 574-578. DOI:10.1111/j.1471-8286.2007.01758.x |
| [26] | TAMURA K, STECHER G, PETERSON D, et al. MEGA6:Molecular evolutionary genetics analysis version 6.0. Molecular Biology and Evolution, 2013, 30(12): 2725-2729. DOI:10.1093/molbev/mst197 |
| [27] | |
| [28] | KIMURA M. The neutral theory of molecular evolution. Scientific American, 1979, 241(5): 21-59. |
| [29] |
李进宇.中国北方木犀科植物繁育系统的研究.昆明:西南林业大学, 2008:2-7. LI J Y. Study on the breeding system of Oleaceae species of Northern China. Kunming: Southwest Forestry University, 2008: 2-7. (in Chinese with English abstract) http://d.wanfangdata.com.cn/Thesis/Y1592760 |
| [30] |
吕文, 刘文哲. 雄全异株植物瘿椒树(省沽油科)的传粉生物学. 植物学报, 2010, 45(6): 713-722. Lü W, LIU W Z. Pollination biology in androdioecious species Tapiscia sinensis (Staphyleaceae). Chinese Bulletin of Botany, 2010, 45(6): 713-722. (in Chinese with English abstract) |
| [31] | DORKEN M E, PANNELL J R. Hermaphroditic sex allocation evolves when mating opportunities change. Current Biology, 2009, 19(6): 514-517. DOI:10.1016/j.cub.2009.01.067 |
| [32] |
张洪伟.桂花传粉生物学研究.南京:南京林业大学, 2013:44-45. ZHANG H W. Pollination biology of Osmanthus fragrans Lour. Nanjing: Nanjing Forestry University, 2013:44-45. (in Chinese with English abstract) http://cdmd.cnki.com.cn/Article/CDMD-10298-1013045971.htm |
| [33] |
李稚.桂花传粉生物学与杂交育种研究.南京:南京林业大学, 2014:25-27. LI Z. Pollination biology and cross breeding of Osmanthus fragrans Lour. Nanjing: Nanjing Forestry University, 2014:25-27. (in Chinese with English abstract) http://cdmd.cnki.com.cn/Article/CDMD-10298-1015015942.htm |
| [34] |
何亚平, 刘建全. 植物繁育系统研究的最新进展和评述. 植物生态学报, 2003, 27(2): 151-163. HE Y P, LIU J Q. A review on recent advances in the studies of plant breeding system. Acta Phytoecologica Sinica, 2003, 27(2): 151-163. (in Chinese with English abstract) DOI:10.17521/cjpe.2003.0024 |
| [35] | ARMBRUSTER W S, BALDWIN B C. Switch from specialized to generalized pollination. Nature, 1998, 394(6694): 632. DOI:10.1038/29210 |
| [36] |
李淑娴, 侯静, 冒燕, 等. 决定植物花色的分子机制与遗传调控. 西南林业大学学报, 2012, 32(6): 92-97. LI S X, HOU J, MAO Y, et al. Molecular basis and genetic regulation of plant flower color. Journal of Southwest Forestry University, 2012, 32(6): 92-97. (in Chinese with English abstract) |
| [37] | YOSHIDA K, TOYAMAKATO Y, KAMEDA K, et al. Sepal color variation of Hydrangea macrophylla and vacuolar pH measured with a proton-selective microelectrode. Plant and Cell Physiology, 2003, 44(3): 262-268. DOI:10.1093/pcp/pcg033 |
| [38] | TOYAMA Y. Analysis of metal elements of hydrangea sepals at various growing stages by ICP-AES. Biochemical Engineering Journal, 2003, 14(3): 237-241. DOI:10.1016/S1369-703X(02)00220-6 |
| [39] |
臧德奎, 向其柏, 刘玉莲. 中国桂花品种的起源与演化//申报桂花品种国际登录权论文集(11). 长春: 吉林科学技术出版社, 2002: 1-12. ZANG D K, XIANG Q B, LIU Y L. The origins and evolution of Osmanthus cultivars in China//Collected Papers of Application to International Registration Authority of Sweet Osmanthus (Osmanthus fragrans) Cultivars (11). Changchun: Jilin Science and Technology Press, 2002: 1-12. (in Chinese with English abstract) |
| [40] |
韩远记.桂花花色变异的机理和不同花色品种花瓣的cDNA AFLP差异分析.河南, 开封:河南大学, 2014:20-25, 32-42. HAN Y J. Mechanism of flower color variation and cDNA-AFLP analysis of 2 cultivars with different flower color in Osmanthus fragrans. Kaifeng, Henan: Henan University, 2014:20-25, 32-42. (in Chinese with English abstract) http://cdmd.cnki.com.cn/Article/CDMD-10475-1014394268.htm |
| [41] |
侯丹.桂花主要品种花香和花色及其对温度变化的响应.浙江, 临安:浙江农林大学, 2014:57-67. HOU D. Analysis of floral scent and pigment constituents and the reflectance to temperature fluctuation in Osmanthus fragrans (Thunb.) Lour. Lin'an, Zhejiang: Zhejiang A & F University, 2014:57-67. (in Chinese with English abstract) |
| [42] | AULDRIDGE M E, BLOCK A, VOGEL J T, et al. Characterization of three members of the Arabidopsis carotenoid cleavage dioxygenase family demonstrates the divergent roles of this multifunctional enzyme family. The Plant Journal, 2006, 45(6): 982-993. DOI:10.1111/tpj.2006.45.issue-6 |
| [43] | HUANG F C, MOLNáR P, SCHWAB W. Cloning and functional characterization of carotenoid cleavage dioxygenase 4 genes. Journal of Experimental Botany, 2009, 60(11): 3011-3022. DOI:10.1093/jxb/erp137 |
| [44] | OHMIYA A, KISHIMOTO S, AIDA R, et al. Carotenoid cleavage dioxygenase (CmCCD4a) contributes to white color formation in chrysanthemum petals. Plant Physiology, 2006, 142(3): 1193-1201. DOI:10.1104/pp.106.087130 |
| [45] | TAN B C, JOSEPH L N, DENG W T, et al. Molecular characterization of the Arabidopsis 9-cis-epoxycarotenoid dioxygenase gene family. The Plant Journal, 2003, 35(1): 44-56. DOI:10.1046/j.1365-313X.2003.01786.x |
| [46] |
胡菀, 罗意, 阳亿, 等. 野生桂花的遗传多样性和遗传结构研究. 园艺学报, 2014, 41(7): 1427-1435. HU W, LUO Y, YANG Y, et al. Genetic diversity and population genetic structure of wild sweet osmanthus revealed by microsatellite markers. Acta Horticulturae Sinica, 2014, 41(7): 1427-1435. (in Chinese with English abstract) |
 2017, Vol. 43
2017, Vol. 43


