| 藏东南地区毛壳属真菌多样性及系统发育分析 |
2. 西北农林科技大学植物保护学院/旱区作物逆境生物学国家重点实验室,陕西 杨凌 712100;
3. 西藏高原生态研究所,西藏 林芝 860000
2. College of Plant Protection, Northwest A & F University/State Key Laboratory of Crop Stress Biology for Arid Areas, Yangling 712100, Shaanxi, China;
3. Tibet Plateau Institute of Ecology, Linzhi 860000, Xizang, China
毛壳属真菌(Chaetomium Kunze,以下简称毛壳菌)隶属于子囊菌门(Ascomycota)核菌纲(Pyrenomycetes)粪壳目(sordariales)毛壳菌科(Chaetomiaceae),主要分布在自然界各种含纤维素的基质上,包括杂食动物及鸟类和鼠类的粪便、土壤、植株残体等,能够产生大量的纤维素酶[1],同时还可以降解纤维素和木质素等大分子难降解有机物,具有拮抗土壤中某些微生物的作用。自从1817年KUNZE建立毛壳菌属以来,人们对毛壳菌的认识不断深入,分类标准也日趋一致,毛壳菌属由1881年第一部专著中报道的10个种,逐渐发展稳定到真菌字典第九版记载的81个种[2],2008年由KIRK确定为95个种。我国对毛壳菌的分类和研究始于1959年,陈庆涛[3]于1959—1965年报道了2个毛壳菌新种;随后,戴芳澜[4]在中国真菌总汇里记录了该属30个种(含异名);孙广宇等对我国部分地区的毛壳菌进行了分离和鉴定,报道了3个新种[5];谭悠久等[6]发现了我国1个新种;王雪薇[7]报道了我国毛壳菌31个种,其中包括2个新种,5个中国新记录种;刘富江[8]从我国部分省(区)采样,发现毛壳菌14个种,其中1个新记录种。目前,我国发现并记载的毛壳菌共计31种。
研究表明,微生物的生态分布与土壤和植被等环境因子密切相关[9]。藏东南地区处于我国雅鲁藏布江中下游,总体海拔高度差超过7 000 m[10],平均海拔3 100 m,地势南低北高,印度洋暖流与北方寒流汇合,形成了特殊的热带、亚热带、温带和寒带并存的特殊气候。该地区地形复杂,气候多样,资源丰富,是研究植物区系和生物多样性的理想区域。徐阿生等[11]和张惠等[12]分别对藏东南地区的大型真菌进行了调查,何建清等[9]对藏东南地区的放线菌进行了分离和鉴定。但是,目前对西藏毛壳菌的系统研究还较少,仅刘述春等[13]对来源于藏东南林芝地区的一株毛壳菌进行了生物活性筛选,发现了新聚酮类结构活性化合物;GUO等[14]从藏东南林芝地区的土壤中分离到3株毛壳菌并发现了1个新种。这些研究均表明,特殊生境是微生物新种和活性天然产物的重要来源。
自 20 世纪 60 年代以后,分子生物学方法被广泛应用于真菌分类和鉴定,其中保守的rDNA-ITS序列被推荐为真菌的通式性DNA条形码[15],但由于内部转录间隔区(internal transcribed spacer, ITS)序列提供的遗传信息有限,一些功能基因如C01,EF-a和β-tubulin等序列在一些真菌种类鉴定中成为更好的分子标记[16-18]。本文通过 rDNA-ITS 结合β-tubulin 基因序列对分离自藏东南地区的毛壳属真菌菌株进行初步鉴定,分析其多样性和系统发育情况,以期为丰富西藏毛壳属真菌资源、开发毛壳菌代谢产物提供理论依据。
1 材料与方法 1.1 材料 1.1.1 标本来源从藏东南的昌都市、工布江达县、墨脱县、察隅县、林芝县等地共采集土壤、植物残体和动物粪便等基质355份,用牛皮纸信封装好带回实验室,风干1周后,进行毛壳菌的分离。各采样区的基本情况见表 1。
| 表1 藏东南不同采样区基本情况 Table 1 Profile of different sampling areas in southeastern Tibet |
 |
| 点击放大 |
马丁培养基[19](分离培养基):蛋白胨5 g,葡萄糖10 g,KH2PO41 g,MgSO4·7H2O 0.5 g,琼脂20 g,蒸馏水1 L。培养基配制好后,加入1%的孟加拉红溶液3.3 mL,分装后用121 ℃高压蒸汽灭菌30 min。
玉米粉培养基(鉴定培养基)::玉米粉 30 g,琼脂15 g,水 1 L。将玉米粉在 70 ℃蒸馏水中加热约 1 h,用纱布过滤固体残渣后,将琼脂粉加入滤液中,加热溶化,最后用蒸馏水补足至 1 L,分装后用 121 ℃高压蒸汽灭菌30 min。
1.1.3 主要设备和试剂试剂:十六烷基三甲基溴化铵(CTAB)、Tris 饱和酚、氯仿、异戊醇、异丙醇、乙醇、超纯水、冰醋酸、氯化钠、二水乙二胺四乙酸二钠、氢氧化钠等,聚合 酶链式反应(polymerase chain reaction, PCR)扩增用 的试剂均购自大连TaKaRa宝生物工程有限公司。
设备:Nikon体式解剖镜(SMZ800-C-DS)、徕卡(Leica)系统显微镜(DM5000)、双人单面水平净化工作台(SF-CJ-20)、电热恒温培养箱(DHP-9162)、PCR 仪 、天 能 凝 胶 电 泳 成 像 分 析 系 统(Tanon-41000)、电子天平(JA10003N)、DY1002V 电泳仪、MR22低温高速离心机等。
1.2 方法 1.2.1 数据处理分离几率=分离到的菌株数/分离的样本总数×100%。
采用SPPS 13.5软件进行毛壳菌的数量统计及数据分析。
毛壳菌的多样性特征指数即Shannon-Weiner多样性指数(H)、Pielou均匀性指数(E)和丰富度指数(D)分别按以下公式计算。
| $ H = - \sum\limits_{i = 1}^s {{p_i}\ln {{p}_i};} E = H/\ln S;D = \ln S/ln{N}{.} $ |
式中:pi为物种i占采样区菌株总数的比例;S为各采样区内毛壳菌的物种数量;N为各采样区内分离到的菌株总数。
1.2.2 菌株分离与纯化[20]树枝、树叶、动物粪便等基质采用组织分离法分离:将实验材料切取成4~5 mm的小块,用60%乙醇将组织小块浸泡6~8 min,无菌水连续漂洗3次,将表面消毒过的组织小块转接至马丁氏培养基上,每个培养皿放置4~5块,28 ℃恒温培养,3~4 d后观察是否有子囊果产生,并在 Nikon体式解剖镜下挑取单个子囊果接种于玉米粉培养基上,对菌株进行纯化和观察。
采用稀释分离法分离土壤样品:称取充分混匀的土样约1.0 g置于250 mL无菌三角瓶中,加入60%乙醇2 mL使土样完全淹没,处理6~8 min,加入120 mL左右无菌水,使三角瓶中的乙醇浓度稀释至1%左右,吸取0.8~1 mL稀释液涂布于马丁氏培养基平板上,涂布均匀,28 ℃恒温培养,3~4 d后观察是否有子囊果产生,并在 Nikon体式解剖镜下挑取单个子囊果接种于玉米粉培养基,对菌株进行纯化和观察。
1.2.3 毛壳菌的初步归类以VON ARX等[21]报道的系统为主要基础和依据对毛壳菌进行初步归类。将待鉴定的菌株接种于玉米粉培养基上,28 ℃黑暗培养,记录菌落特征、子囊果成熟时间。定期观察,待子囊果成熟时,在Nikon 体式解剖镜下挑取单个子囊果,制作临时玻片,用 DM5000徕卡系统显微镜观察子囊果、附属丝和子囊的形态,以及子囊孢子的颜色、形态、萌发孔等特征,将特征一致的菌株归为一类。。
1.2.4 聚合酶链式反应和序列测定DNA的提取主要参考NAKADA等[22]的方法并加以改进。rDNA-ITS序列的聚合酶链式反应(polymerase chain reaction, PCR)引物对序列[23]如下。ITS1-F:5'-CTTGGTCATTTAGAGGAAGTAA-3',ITS4:5'-TCCTCCGCTTATTGATATGC-3'。β-tubulin基因的PCR引物对序列[24]如下。Bt2a:5'-GGTAACCAAATCGGTGCTGCTTTC-3',Bt2b:5'-ACCCTC AGTGTAGTGACCCTTGGC -3'。以上引物均由上海美吉生物科技有限公司合成。
PCR反应体系(25 μL):10×PCR缓冲液2.5 μL,DNA模板10 ng,2.5 mmol/L dNTP2.0 μL,10 μmol/L引物各0.5 μL,5 U/μL Taq酶0.2 μL。扩增内部转录间隔区(internal transcribed spacer, ITS)的PCR反应程序:95 ℃预变性3 min;95 ℃变性30 s,53 ℃退火40 s,72 ℃延伸30 s,为1个循环,扩增35个循环;72 ℃延伸10 min。扩增β-tubulin基因序列的PCR反应程序:95 ℃预变性5 min,94 ℃变性30 s,58 ℃退火30 s,72 ℃延伸1 min,为1个循环,扩增40个循环;最后72 ℃延伸10 min。PCR产物的纯化和序列测定由上海美吉生物科技有限公司进行。
1.2.5 序列分析及系统发育树的构建将分离到的36个菌株的rDNA-ITS和β-tubulin基因序列测序结果在GenBank数据库中申请接受号(表 2),利用Blast进行同源序列查找,选择15个相关种的序列(表 2)。采用Clustal X 1.8软件对所测定的核苷酸序列进行校排(alignment)后,用Bioedit软件将序列两端切割平齐,保存为fastal格式,用SequenceMatrix进行菌株rDNA-ITS和β-tubulin基因序列的拼接,并保存为nexus,扩展名为.nex。利用DnaSP v5.0软件对序列进行 DNA多态性分析,分别统计序列间每对碱基的平均差异、多态位点数目、单倍型数目基因型多样性和核苷酸差异的平均值。用 MEGA 5.0软件中的邻接法构建系统进化树(选择Sordaria macrospora为外群)。
| 表2 构建系统发育树所用的菌株 Table 2 Strains used for constructing phylogenetic tree |
 |
| 点击放大 |
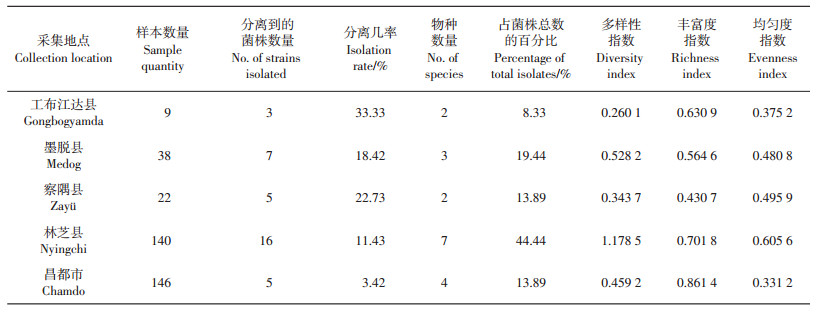
从表 3中可看出:从藏东南5个地县采集土壤、植物残体、动物粪便标本共355份,并分离到36株毛壳菌,分离几率为10.14%。其中:工布江达县的分离几率最高,为33.33%;昌都地区的标本分离几率最低,仅为3.42%;林芝县的毛壳属真菌多样性指数最高,为1.178 5,物种均匀度指数也最高,为0.605 6;工布江达的多样性指数最低,仅为0.260 1;昌都地区的毛壳属真菌丰富度指数最高,达到0.861 4;察隅县的丰富度指数最低,仅为0.430 7;藏东南各地区的物种数量存在较大差异,林芝县物种数量最多。
| 表3 不同地区的毛壳属真菌物种多样性指数特征 Table 3 Characteristics of species diversity index of Chaetomium from different regions |
 |
| 点击放大 |
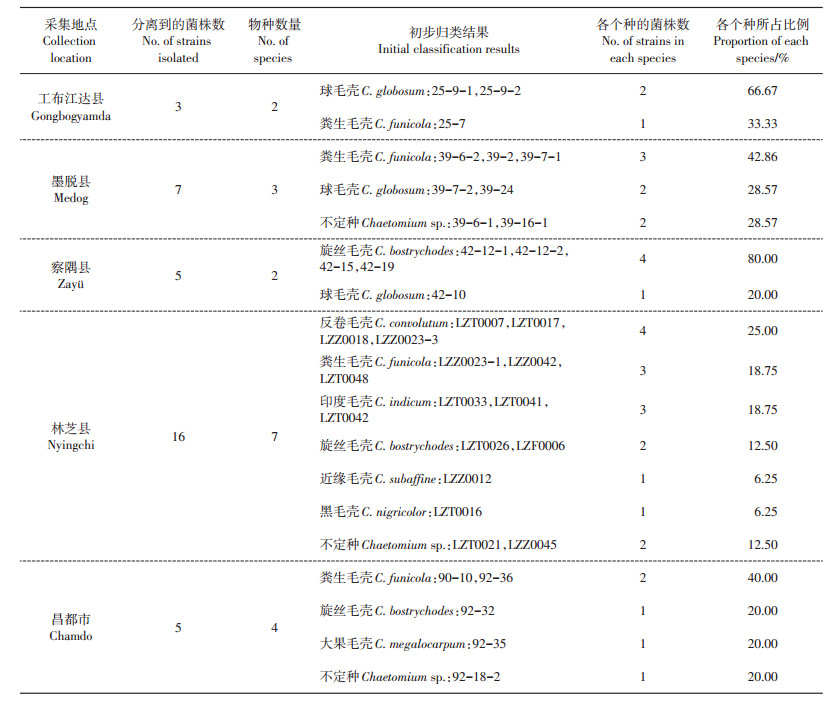
从表 4可以看出,从藏东南各地区分离到的36个菌株可归属为8个种,分别为球毛壳(C. globosum),粪生毛壳(C. funicola),旋丝毛壳(C. bostrychodes),反卷毛壳(C. convolutum),印度毛壳(C. indicum),近缘毛壳(C. subaffine),黑毛壳(C. nigricolor)和大果毛壳(C. megalocarpum)。其中近缘毛壳、黑毛壳和大果毛壳仅分离到1株,为少见种。不同地区的毛壳属真菌的优势种存在差异:工布江达县的优势种为球毛壳(C. globosum);墨脱和昌都地区的优势种为粪生毛壳(C. funicola);察隅县的优势种为旋丝毛壳(C. bostrychodes);林芝县的优势种为反卷毛壳(C. convolutum)。
| 表4 不同地区毛壳属真菌形态特征归类结果 Table 4 Results of morphological characteristics of Chaetomium from different regions |
 |
| 点击放大 |
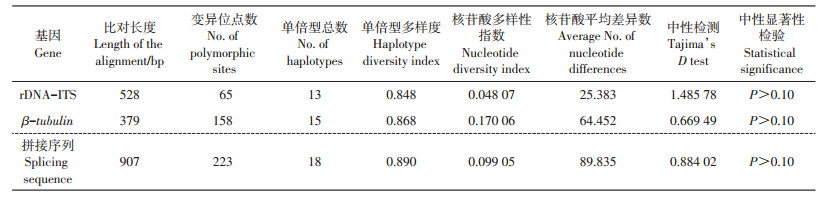
对36株毛壳菌的rDNA-ITS和β-tubulin基因序列进行扩增,结果(表 5)显示:rDNA-ITS扩增后两端切割平齐获得长度为528 bp,β-tubulin基因扩增后两端切割平齐获得长度为379 bp,拼接序列总长为907 bp;共有223个变异位点,18种单倍型,单倍型多样度为0.890,核苷酸多样性指数为0.09 905,核苷酸平均差异数为89.835;β-tubulin基因的变异位点、单倍型多样度、单倍型总数、核苷酸多样性指数和核苷酸平均差异数与rDNA-ITS的这些参数差异显著,体现出更大的碱基变异性。
| 表5 2个基因在36株毛壳属真菌中的核苷酸多样性 Table 5 Nucleotide diversity of two genes in 36 strains of Chaetomium |
 |
| 点击放大 |
利用 MEGA 5.0软件构建 rDNA-ITS和β-tubulin基因系统发育进化树,对所测定的序列及 GenBank数据库中的相关序列进行系统发育进化分析,构建藏东南地区毛壳菌的系统发育树。结果(图 1)显示,供试的 36 个菌株被分为 7 个组,各分支的支持强度均达 77%以上。其中:10个菌株与粪生毛壳(C. funicola)聚类在一个分枝上,包括形态上不能准确归类的菌株39-6-1;3个菌株与印度毛壳(C. indicum)聚类在一个分枝上;2个形态不定的菌株LZT0021、LZZ0045与C. erectum聚类在一个分枝上;菌株LZZ0012与C. subaffine聚类在一个分枝上;92-35与大果毛壳(C. megalocarpum)聚类在一起;6个菌株与球毛壳(C. globosum)聚类在一个分枝上,包括形态上不能准确归类的菌株39-16-1;此外,形态上不能准确归类的菌株92-18-2、形态上鉴定为反卷毛壳(C. convolutum)的4个菌株、形态上鉴定为旋丝毛壳(C. bostrychodes)的7个菌株、形态上归类为黑毛壳(C. nigricolor)的1个菌株,共计13个菌株聚在一起彼此分不开。
 |
| 分支处数值表示自举值;标尺表示每个核苷酸位点上的0.02替换值。 Number in each branch point denotes the percentages supported by bootstrap; scale bar represents 0.02 substitutions per nucleotide position. 图1 基于rDNA-ITS和β-tubulin基因序列构建的系统发育树 Fig. 1 Phylogenetic tree constructed based on rDNA-ITS and β-tubulin sequences |
本文从藏东南地区采集动物粪便、土壤和植物残体标本共 355 份,从中分离到毛壳菌 36 株,平均分离几率仅为 10.14%,说明毛壳菌在土壤中的分离几率较低。这与国内很多学者的研究结果一致:王雪薇[7]从 1 738份基质中仅分离到 136株毛壳菌,分离几率为 7.82%;刘富江[8]从 1 098份标本中仅分离获得 63株毛壳菌,分离几率仅为 5.74%。林芝县的毛壳菌多样性指数和均匀度指数均为最高,这与该地区植被丰富有很大关系。综合分析藏东南各地区的物种类群,旋丝毛壳为察隅县的优势种,球毛壳为工布江达县的优势种,粪生毛壳为昌都市和墨脱县的优势种,反卷毛壳为林芝县的优势种,反映出不同生态区的植被和土壤状况影响了微生物的生态分布与优势种的类型。
从碱基变异结果来看,β-tubulin基因的碱基变异位点、单倍型、单倍型多样度、核苷酸多样性指数和核苷酸平均差异数等方面较rDNA-ITS存在显著差异。β-tubulin基因既有保守的外显子又有6个可变的内含子,可作为真菌物种鉴定的标识片段[25]。SAMSON等[26]对青霉属的180株代表性菌株进行序列分析,证实了β-tubulin基因是较理想的物种标记片段,VARGA等[27]和GEISER等[28]的研究均表明β-tubulin基因适合作为曲霉属的DNA条形码,但是TANG等[29]在对粪壳菌纲(Sordariomycetes)进行系统发育研究时发现,β-tubulin对该类群真菌分辨率较低,需要结合其他基因片段才能提高物种的分辨率。本研究结合rDNA-ITS和β-tubulin基因序列进行联合分析,提高了物种鉴定的准确性。
从系统发育树上可以看出,结合rDNA-ITS和β-tubulin基因不仅可以用来区分毛壳菌属中形态差异较大的种,如印度毛壳、大果毛壳、球毛壳、粪生毛壳,还可以区分形态特征非常相似的印度毛壳和C. erectum,同时对一些形态上难以鉴定的疑难种也可以进行初步鉴定,但还不能很好地区分反卷毛壳、旋丝毛壳和黑毛壳,需要结合更多的基因片段进行区分。
目前,国内外对西藏毛壳菌的研究仅为零星报道,缺乏较系统的研究,本文选择西藏植被较为丰富的藏东南地区为标本采集地,提高了菌株的分离几率。藏东南复杂而独特的生态环境,较原始的土壤生态环境决定了其中毛壳菌的独特之处。本研究中分离到的C. erectum在国内尚无报道,大果毛壳也是比较少见的种[30]。由此可见,藏东南地区是西藏的重要菌种资源库,本研究为丰富西藏毛壳菌资源库,开发毛壳菌代谢产物奠定了基础。
| [1] |
刘守安, 李多川, 俄世瑾, 等. 嗜热毛壳菌纤维素酶(CBHⅡ) cDNA的克隆及在毕赤酵母中的表达. 生物工程学报, 2005, 21(6): 892-899. LIU S A, LI D C, E S J, et al. Cloning and expressing of cellulase gene (cbh2) from thermophilic fungi Chaetomium thermophilum CT2. Chinese Journal of Biotechnology, 2005, 21(6): 892-899. (in Chinese with English abstract) |
| [2] | KIRK P M, CANNON P F, DAVID J C, et al. Ainsworth & Bisby's Dictionary of the Fungi. 9th ed. Wallingford, UK: CAB International Publishing, 2001: 610-614. |
| [3] |
陈庆涛. 毛壳菌和壳针孢菌的新种. 微生物学报, 1973, 13(2): 124-128. CHEN Q T. Some new species of Chaetomium and Septoria. Acta Microbiologica Sinica, 1973, 13(2): 124-128. (in Chinese with English abstract) |
| [4] |
戴芳澜. 中国真菌总汇. 北京: 科学技术出版社, 1979: 157-165. DAI F L. Sylloge Fungorum Sinicorum. Beijing: Science and Technology Press, 1979: 157-165. (in Chinese with English abstract) |
| [5] |
孙广宇, 谭悠久, 张荣. 中国毛壳菌科研究Ⅰ.毛壳菌属的种. 菌物学报, 2004, 23(3): 333-337. SUN G Y, TAN Y J, ZHANG R. The family Chaetomiaceae from China Ⅰ. Species of the genus Chaetomium. Mycosystema, 2004, 23(3): 333-337. (in Chinese with English abstract) |
| [6] |
谭悠久, 张荣, 张军林, 等. 中国毛壳菌科(Chaetomiaceae)分类研究. 西北农林科技大学学报(自然科学版), 2005, 33(增刊): 267. TAN Y J, ZHANG R, ZHANG J L, et al. Classification of the family Chaetomiaceae from China. Journal of Northwest A & F University (Natural Science Edition), 2005, 33(Suppl.): 267. (in Chinese with English abstract) |
| [7] |
王雪薇. 中国毛壳属Chaetomium Kunze及其形态相似类群的系统分类研究. 北京: 中国科学院大学, 2005: 51-147. WANG X W. Systematic studies on Chaetomium Kunze and its morphologically similar taxa from China. Beijing: University of Chinese Academy of Sciences, 2005:51-147. (in Chinese with English abstract) |
| [8] |
刘富江. 中国部分省(区)毛壳属真菌的分类研究. 乌鲁木齐: 新疆农业大学, 2009: 20-67. LIU F J. Taxonomic study on the Chaetomium Kunze from some provinces (regions) in China. Urumqi: Xinjiang Agricultural University, 2009:20-67. (in Chinese with English abstract) http://cdmd.cnki.com.cn/Article/CDMD-10758-2009176020.htm |
| [9] |
何建清, 吴云锋, 张格杰. 藏东南地区土壤放线菌的生态分布及活性研究. 微生物学报, 2006, 46(5): 773-777. HE J Q, WU Y F, ZHANG G J. Activity and ecological distribution of actinomycetes from soil in the southeastern of Tibet. Acta Microbiologica Sinica, 2006, 46(5): 773-777. (in Chinese with English abstract) |
| [10] |
游金娥, 冯建孟. 藏东南地区种子植物的区系组成和物种多样性. 生态环境学报, 2013, 22(2): 207-212. YOU J E, FENG J M. Plant biodiversity and flora composition in southeast Tibet. Ecology and Environmental Sciences, 2013, 22(2): 207-212. (in Chinese with English abstract) |
| [11] |
徐阿生, 罗建. 中国盘菌属Peziza一新记录种. 菌物学报, 2007, 26(1): 148-149. XU A S, LUO J. A new record species of Peziza in China. Mycosystema, 2007, 26(1): 148-149. (in Chinese with English abstract) |
| [12] |
张惠, 范宇光, 图力古尔. 采自西藏的盔孢菌属2个中国新记录种. 东北林业大学学报, 2012, 40(5): 134-136. ZHANG H, FAN Y G, TOLGOR B. Two new records of the genus Galerina collected from Tibet, China. Journal of Northeast Forestry University, 2012, 40(5): 134-136. (in Chinese with English abstract) |
| [13] |
刘述春, 孙炳达, 旺姆, 等. 一株毛壳霉属真菌中新结构活性聚酮类化合物研究. 菌物学报, 2010, 29(5): 726-731. LIU S C, SUN B D, WANG MU, et al. Chaetomones A-E: New bioactive polyketides from Chaetomium sp. Mycosystema, 2010, 29(5): 726-731. (in Chinese with English abstract) |
| [14] | GUO Y Z, ZHU M Q, SUN G Y. The family Chaetomiaceae from China. 4. Two newly recorded species of Chaetomium. Mycotaxon, 2011, 116: 247-251. DOI:10.5248/116.247 |
| [15] | SCHOCH C L, SEIFERT K A, HUHNDORF S, et al. Nuclear ribosomal internal transcribed spacer (ITS) region as a universal DNA barcode marker for fungi. Proceedings of the National Academy of Sciences of the USA, 2012, 109(16): 6241-6246. DOI:10.1073/pnas.1117018109 |
| [16] | BUEE M, REICH M, MURAT C, et al. 454 pyrosequencing analyses of forest soils reveal an unexpectedly high fungal diversity. New Phytologist, 2009, 184(2): 449-456. DOI:10.1111/j.1469-8137.2009.03003.x |
| [17] | GREIF M D, STCHIGEL A M, MILLER A N, et al. A re evaluation of genus Chaetomidium based on molecular and morphological characters. Mycologia, 2009, 101(4): 554-564. DOI:10.3852/08-200 |
| [18] | ASGARI B, ZARE R. The genus Chaetomium in Iran, a phylogenetic study including six new species. Mycologia, 2011, 103(4): 863-882. DOI:10.3852/10-349 |
| [19] | MALLOCH D. Moulds: Their Isolation, Cultivation and Identification. Canada: University of Toronto Press, 1981: 1-59. |
| [20] |
谭悠久. 毛壳菌科(Chaetomiace)分类及分子系统发育研究. 陕西, 杨凌: 西北农林科技大学, 2005: 24-33. Classification, identification and molecular phylogenetics of the family Chaetomiaceae |
| [21] | VON ARX J A, GUARRO J, FIGUERAS M J. The Ascomycete genus Chaetomium. Nova Hedwigia, Beiheft, 1986, 84: 1-162. |
| [22] | NAKADA M, TANAKA C, TSUNEWAKI K. RFLP analysis for species separation in the genera Bipolaris and Curvularia. Mycoscience, 1994, 35(3): 271-278. DOI:10.1007/BF02268449 |
| [23] | WHITE T J, BRUNS T, LEE S, et al. Amplification and direct sequencing of fungal ribosomal RNA genes for phylogenetics// INNIS M A, GELFAND D H, SNINSKY J J, et al. PCR Protocols: A Guide to Methods and Applications. New York, USA: Academic Press, Inc., 1990:315-322. |
| [24] | GLASS N L, DONALDSON G C. Development of primer sets designed for use with the PCR to amplify conserved genes from filamentous ascomycetes. Applied and Environmental Microbiology, 1995, 61(4): 1323-1330. |
| [25] | BISCHOFF J F, REHNER S A, HUMBER R A. Metarhizium frigidum sp. nov.: A cryptic species of M. anisopliae and a member of the M. flavoviride complex. Mycologia, 2006, 98(5): 737-745. DOI:10.1080/15572536.2006.11832645 |
| [26] | SAMSON R A, SEIFERT K A, KUIJPERS A F A, et al. Phylogenetic analysis of Penicillium subgenus Penicillium using partial β-tubulin sequences. Studies in Mycology, 2004, 49: 175-200. |
| [27] | VARGA J, FRISVAD J C, KOCSUBE S, et al. New and revisited species in Aspergillus section Nigri. Studies in Myology, 2011, 69: 1-17. DOI:10.3114/sim.2011.69.01 |
| [28] | GEISER D M, KLICH M A, FRISVAD J C, et al. The current status of species recognition and identification in Aspergillus. Studies in Mycology, 2007, 59: 1-10. DOI:10.3114/sim.2007.59.01 |
| [29] | TANG A M C, JEEWON R, HYDE K D, et al. Phylogenetic utility of protein (RPB2, β -tubulin) and ribosomal (LSU, SSU) gene sequences in the systematics of Sordariomycetes (Ascomycota, fungi). Antonie Van Leeuwenhoek, 2007, 91(4): 327-349. DOI:10.1007/s10482-006-9120-8 |
| [30] |
郭云忠. 毛壳科Chaetomiaceae真菌多基因系统演化及分类鉴定研究. 陕西, 杨凌: 西北农林科技大学, 2012: 87-89. GUO Y Z. The multigene phylogeny and classification of Chaetomiaceae. Yangling, Shaanxi: Northwest A & F University, 2012:87-89. (in Chinese with English abstract) http://cdmd.cnki.com.cn/Article/CDMD-10712-1012437441.htm |
 2017, Vol. 43
2017, Vol. 43


