| 红掌2个SOC1基因的克隆、序列与表达分析 |
2. 浙江省杭州萧山技师学院,杭州 311202
2. Hangzhou Xiaoshan Technician College of Zhejiang Province, Hangzhou 311202, China
植物需要一个精准的调控系统来完成由营养生长到生殖生长的转变。植物在进化过程中已经形成了这种精准的调控体系,其中以MADS盒(MADS-box)为主的转录因子调控网络发挥了十分重要的作用。近30年来,植物学家和分子生物学家在对拟南芥(Arabidopsis thaliana)、水稻(Oryza sativa)、矮牵牛(Petunia hybrida)、金鱼草(Antirrhinum majus)等植物的研究中,逐渐总结出植物开花所涉及的4种途径:光周期途径、自主控制途径、低温春化途径和赤霉素途径[1-3]。植物在受到外界环境或内源物质等成花信号的刺激后,会通过一系列的信号转导,汇总到已经发现的FT (FLOWERING LO-CUS T)、LFY (LEAFY)、SOC1 (SUPPRESSOR OF OVEREXPRESSION OF CO1)这3种成花整合因子中,它们随后再将成花信号传递给花器官决定基因,从而诱导成花,实现由营养生长向生殖生长的转变[4]。
SOC1是MADS盒转录因子家族中的一员,其氨基酸组成较为保守,具有典型的M、I、K、C区段[5]。该转录因子最早在筛选过表达CO基因的突变体时在拟南芥中被发现[6]。SOC1功能缺失会严重抑制35S::CO植株的早花现象,说明SOC1作为CO基因的下游调控者,参与成花诱导,并且先于花原基出现,在部分营养器官如茎顶端分生组织、叶片、茎段,甚至根部中均有表达[7]。在拟南芥以外的其他物种中也存在类似的表达模式[8-10]。然而,研究发现,它也在花器官形成初期表达,并参与花器官的决定[11-12]。所以,SOC1从营养生长阶段到生殖发育阶段,都发挥着重要的调控作用。在拟南芥中,SOC1的表达会对B功能基因造成压制[13],但在花分生组织出现后,SOC1的表达会受到短时的抑制,以利于其他花器官决定基因的表达,然而这种短时的抑制会很快消失,所以在拟南芥的雌雄蕊中,也可以检测到该基因的表达[14-15]。在调控方式上,SOC1的表达受到自身及其他转录因子的调控。有研究证明,SOC1本身可以与AG和AP1转录因子形成蛋白复合体,反过来结合于自身顺式元件上,抑制自身的表达[16-17]。除了AG和AP1这2个同源异型转录因子,SVP和FLC作为MADS家族一员,也可以蛋白二聚体的形式调控SOC1的表达,表现为与SOC1的启动子区域结合来抑制它的活性[18]。拟南芥SOC1虽然是一个转录因子,但是其亚细胞定位在细胞质中,必须依靠MADS转录因子AGL24的协助才能顺利进入细胞核,发挥其调控功能。可见,AGL24在SOC1的调控网络中具有十分重要的作用[19]。
鉴于SOC1的重要调控功能及其在花器官发育过程中扮演的重要角色,近年来,有越来越多的同源基因被发掘研究,如牡丹(Paeonia suffruticosa)、矮牵牛、石斛兰(Dendrobium)、草莓(Fragaria ananassa)、棉花(Gossypium hirsutum)等,涉及草本、木本、单子叶、双子叶植物,已为深入研究SOC1转录因子的进化、调控、功能等工作做了积极探索[10-11, 20-22]。本课题组已经对红掌中其他MADS基因的研究取得了一定的进展[23]。在此基础上开展SOC1基因的研究,既能丰富红掌MADS盒转录因子家族信息,也是解析该家族调控红掌器官发生、成花转换的必要环节,对深入开展红掌分子育种工作意义重大。
1 材料与方法 1.1 实验材料红掌品种“阿拉巴马”为第一作者所在单位的组培品种,取样时间为初花期。LA taq酶、pMD-18T载体、5'/3'末端快速扩增(rapid amplification of cDNA ends,RACE)试剂盒购自TaKaRa(大连)公司,RNA提取试剂(Plant RNA Reagent)购自美国Invitrogen公司,反转录cDNA第1链合成使用天根公司(北京)反转录试剂盒(Fastquant RT Kit)。荧光定量分析采用TaKaRa(大连)公司荧光染料混合液(SYBR Fast qRT-PCR Mix),使用仪器为7500 Fast型荧光定量PCR仪(AB公司,美国)。其他耗材、生化试剂均为国产。
1.2 RNA提取及反转录用于基因克隆的总RNA提取自红掌幼嫩的花梗、苞片、花序复合体。总RNA分为2份,1份用于合成5' RACE模板,1份用于合成3' RACE模板,操作步骤严格按照试剂盒说明书进行。模板合成后,保存备用。表达分析用总RNA提取自红掌各幼嫩器官,包括根(根尖)、茎、叶(未展开)、花梗(托叶包裹中)、苞片(托叶包裹中)、花被片(显微可见)、雄蕊(显微可见)、雌蕊(显微可见)、果实(未成熟),然后经由DNase处理,按照试剂盒说明书反转录合成cDNA,备用。
1.3 基因克隆及表达分析在前期转录组测序基础上,已经获得了2个SOC1基因的表达序列标签(expressed sequence tag,EST),分别长294 bp和280 bp。以此序列为核心,设计特异引物(P1-5'和P1-3'扩增AaSOC1-1;P2-5'和P2-3'扩增AaSOC1-2),结合试剂盒通用引物UPM,采用LA taq酶并严格按照其扩增体系进行扩增,获得2个基因的两端序列,然后通过拼接、验证,得到2个基因的完整编码区。所用引物序列如下。P1-5':ACATGATCCCAAACTTTCCCCCAGCAG,P1-3 ':TCTACGAGTTCTCCAGCAACAGCAT;P2-5 ':CAGCCTCTCCTTTTCCTTCAGCAGCGT;P2-3':TC ACGCTGCTGAAGGAAAAGGAGAGGC。通用引物为UPM:CTAATACGACTCACTATAGGGCAAGCAG TGGTAT CAACGCAGAGT。
在获得2个基因的完整编码区后,以2个基因C-端不保守区段为模板,设计特异引物(GSP1-F:AGCAGATTACATCGCTGAAGG,GSP1-R:TTCCCG CTGTCATTGTCATAG,产物长127 bp;GSP2-F:GG AAAAGGAGAGGCTGCTGTT,GSP2-R:ATCAA CG GTGTCCAGAGCATC,产物长97 bp),用于荧光定量反转录-聚合酶链式反应(reverse transcription-polymerase chain reaction,RT-PCR)。内参基因采用红掌UBQ基因,引物序列为UBQ-F:GGATT CTAGCT GCCAGGTTTG,UBQ-R:CGGGCATAATC GCAACTTG。反应体系(20 μL)严格按照试剂盒说明书进行,反应程序为:95 ℃ 30 s;95 ℃ 5 s,58 ℃ 10 s,40个循环。每个样本设置3个生物学重复。表达水平采用2-△△CT法计算。
1.4 序列生物信息学分析对获得的基因序列进行生物信息学分析,其中编码区分析采用NCBI在线网站ORF Finder(http://www.ncbi.nlm.nih.gov/gorf/gorf.html),编码蛋白质的保守区段分析采用NCBI Conserved Domain Search软件(http://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi),编码区蛋白质特征分析采用在线软件ProtParam(http://web.expasy.org/protparam/), TMHMM Server v2.0 (http://www.cbs.dtu.dk/services/TMHMM/),SignalP 4.1 Server(http://www.cbs.dtu.dk/services/SignalP/),iPSORT(http://ipsort.hgc.jp/),PSIPRED(http://bioinf.cs.ucl.ac.uk/psipred/)。多序列比对采用软件DNAman;系统进化树构建采用Clustal X+MEGA 6.0:氨基酸全序列参与分析,选择邻位连接法(neighbor-joining method),用Bootstrap法检验1 000次。
2 结果与分析 2.1 红掌2个SOC1基因的克隆通过RACE技术,在已知2个SOC1片段的基础上,分别获得了每个基因的上、下游序列,通过拼接以及全长PCR验证,确认了2个基因的核苷酸组成,分别命名为AaSOC1-1和AaSOC1-2。AaSOC1-1编码区长651 bp,AaSOC1-2编码区长642 bp(图 1)。从软件分析结果得出,2个基因序列的一致性只有75.6%,基本排除是同一基因不同剪接体的可能。
 |
| 图1 AaSOC1-1和AaSOC1-2基因编码区核苷酸序列比对 Fig. 1 Necleotide sequence alignment of gene coding region of AaSOC1-1 and AaSOC1-2 |
在获得2个SOC1基因核苷酸序列的基础上,对其编码的氨基酸以及相关特征属性进行分析。结果显示,AaSOC1-1编码216个氨基酸残基,其中谷氨酸(Glu)共23个,占比10.6%,比例最高,其次为亮氨酸(Leu),共计20个,占比9.3%,最少的为色氨酸(Trp),共1个,占比0.5%;所有负电荷氨基酸(Asp+Glu)共计29个,正电荷氨基酸(Arg+Lys+His)共计38个。AaSOC1-1编码蛋白的相对分子质量为24 896.2,理论等电点pI=9.16,理论化学分子式为C1 064H1 736N322O340S13,预测的亲水性平均系数(GRAVY)为-0.929:说明该结构亲水性能较好,属于亲水性蛋白质。通过TMHMM 2.0在线软件分析发现,该编码蛋白不具有跨膜螺旋结构,可认为是非跨膜蛋白。SignalP 4.1 Server和iPSORT软件分析显示,该蛋白不含有核定位信号,而含有线粒体定位信号,预测该基因编码产物可能定位于线粒体,通过其他途径(例如与互作蛋白结合)转移到细胞核发挥作用。该蛋白二级结构含有常见的螺旋(粉红色)和折叠(黄色)结构以及卷曲(无阴影)结构(图 2A);预测的三级结构如图 3A所示。
 |
| 图2 AaSOC1-1(A)和AaSOC1-2(B)预测氨基酸的二级结构组成 Fig. 2 Secondary structure composition of deduced amino acid from AaSOC1-1 (A) and AaSOC1-2 (B) |
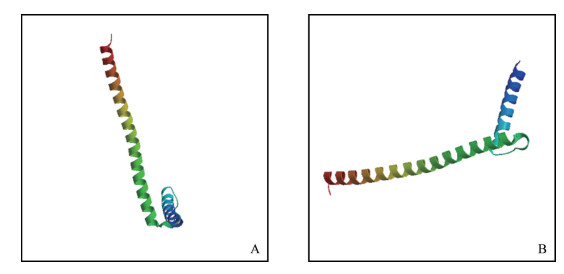 |
| 图3 AaSOC1-1(A)和AaSOC1-2(B)蛋白质三级结构预测 Fig. 3 Deduced protein tertiary structure of AaSOC1-1 (A) and AaSOC1-2 (B) |
AaSOC1-2编码213个氨基酸,其中亮氨酸(Leu)含量最高,共计29个,占比13.6%,其次为谷氨酸(Glu),共计27个,占比12.7%,含量最少的为色氨酸(Trp),共1个,占比0.5%;在该组成中,带负电荷氨基酸(Asp+Glu)共计35个,带正电荷氨基酸(Arg+Lys+His)共计41个。预测的相对分子质量为24 546.0,理论等电点pI=6.68,理论化学分子式为C1 064H1 754N314O334S8,预测的亲水性平均系数(GRAVY)为-0.642:说明AaSOC1-2编码蛋白也属于亲水性蛋白。通过TMHMM 2.0在线软件分析发现,该蛋白不具有跨膜螺旋结构,也可认为是非跨膜蛋白。SignalP 4.1 Server软件分析显示,该蛋白不含有信号肽。SignalP 4.1 Server和iPSORT软件分析显示,该蛋白不含有核定位信号,而含有线粒体定位信号,预测该基因编码产物定位于线粒体,也是通过其他途径转移到细胞核参与转录调控。从图 2B可看出,该蛋白的二级结构同样含有常见的螺旋、折叠和卷曲结构。预测的蛋白质三级结构如图 3B所示。
运用DNAman软件对这2个转录因子及不同物种的同源蛋白进行比对,结果如图 4所示。从中可见:红掌和其他物种的SOC1转录因子普遍具有氨基酸组成保守性,尤其在MADS区,无论是氨基酸数目还是组成,都保持了高度的保守性;在K区各序列之间保守区段较少,主要存在于零星的氨基酸残基中;在相对不保守的C端,各序列均含有一个较为保守的DVETELFIGLP区段,该区段被认为是SOC1家族共有的识别特征,本研究报道的2个SOC1序列均含有此保守区段,可以认定这2个转录因子确为红掌的SOC1同源成员。
 |
| 圆点线表示MADS结构域;实线表示AaSOC1-1的K结构域;虚线表示AaSOC1-2的K结构域;方框表示SOC1转录因子特征识别序列。 Dot line indicates the MADS domain; solid line indicates AaSOC1-1 K domain; dashed line indicates AaSOC1-2 K domain; rectangular frame indicates transcription factor recognition sequence. 图4 红掌SOC1与其他物种同源蛋白多序列比对 Fig. 4 Multiple sequence alignment of homologous SOC1 proteins from Anthurium and other species |
为了更好地说明红掌中2个SOC1转录因子与其他物种同源基因的进化关系,采用MEGA 6.0软件构建了不同物种SOC1转录因子的系统进化树,分析了总共25个不同来源的SOC1同源转录因子。结果(图 5)显示:单、双子叶植物区分明显,来源于棕榈、石斛、玉米、小麦、红掌的序列都归于单子叶植物组,其余都归于双子叶植物组,序列的聚类信息与植物所属类别完全一致;来源于红掌的2个SOC1,虽然聚于同一分支,然而置信分值只有86,间接说明二者在氨基酸组成、功能等方面存在较显著的差异。
 |
| 括号内的编号为在NCBI中的登录号。Ⅰ:双子叶植物;Ⅱ:单子叶植物。 Accession number in the NCBI gene database is included in the bracket.Ⅰ : Dicotyledon; Ⅱ :Monocotyledon. 图5 不同来源SOC1转录因子的系统进化树分析 Fig. 5 Phylogenetic tree analysis of SOC1 transcription factors from different sources |
为明确红掌中2个SOC1基因的表达情况,对根、茎、叶等9个有代表性的部位进行了荧光定量RT-PCR分析。结果(图 6)表明:AaSOC1-1的转录本在所检测的各部位都有表达,其中在根部表达水平极低,在苞片中表达水平最高,在刚刚形成的果实中也存在低水平表达;AaSOC1-2的最低表达同样在根部,其次是幼嫩的果实,而最高表达出现在花梗处;2个基因的表达在花器官中由外向内逐渐递减,表达主要集中于营养器官向生殖器官过渡的部位。
 |
| 图6 AaSOC1-1和AaSOC1-2在红掌不同部位的表达分析 Fig. 6 Expression analysis of AaSOC1-1 and AaSOC1-2 in different tissues of Anthurium |
在目前报道的有关SOC1的研究中,多数植株中只发现了1种转录本,也有在1种植物中发现多种转录本的情况[24]。在本研究中,已经发现的2个SOC1基因序列的相似度并不高,预测的氨基酸序列差异也比较大,但通过多序列比对、系统进化分析和氨基酸组成分析,都可以判定它们属于SOC1转录因子家族成员;因此,可以认定红掌中至少存在2个或2个以上的SOC1同源基因。
生物信息学分析显示,红掌中的这2个SOC1转录因子都不含有核定位信号,这就意味着它们发挥调控功能需要借助外力进入细胞核,进而调节下游基因的表达。在拟南芥以及草莓等物种上,SOC1定位于细胞质并与其他蛋白质互作进而发挥转录因子的调控功能已经被多次报道[11, 19]。据此推断,本研究的2个转录因子也有可能是通过类似的途径参与红掌的生长发育调控。
从表达分析结果看,源于红掌的这2个SOC1基因在不同的组织部位皆有表达,包括营养器官和生殖器官。这与前人在其他物种上报道的同源基因的表达模式类似[25-26]。特别之处在于,红掌的生殖器官着生在一根长长的花梗上,包括苞片、肉穗花序、花器官。本研究结果显示,红掌的2个SOC1基因在花梗中都有较高的表达水平,尤其是AaSOC1-2,这说明2个SOC1基因在成花转换中发挥着重要作用。在营养器官和生殖器官中的普遍表达,暗示红掌中2个SOC1基因除了扮演开花整合因子的角色,也很有可能在花器官分生组织决定方面起到某种作用,这一推论前人在其他物种的相关研究中也多有报道[20, 27]。
本研究虽未涉及2个SOC1转录因子的功能验证,但是从已报道的不同物种SOC1转录因子的研究结果来看,促进植物开花几乎是绝大多数SOC1的共有功能[28-30]。此外,在拟南芥中的SOC1转录因子还可以通过调控一些气孔开放相关基因的表达,进而间接控制气孔的开放[29]。对早竹SOC1的研究发现,它除了具有促进开花的功能外,还可以应答赤霉素信号,随赤霉素含量的增加而提高表达水平,且过表达该基因还能影响叶片的形态发育[30]。这些发现都丰富了SOC1的功能研究。我们对红掌中2个SOC1基因的功能研究也正在进行,二者是否存在功能特异性将在后续报道中呈现。
4 结论本研究采用经典RACE克隆法,以2条已获得的独立EST序列为模板,从红掌生殖器官cDNA中获得了2个SOC1类基因,通过序列比较和生物信息学软件分析,认定这2个基因均属于SOC1基因家族。这2个基因在长度、核苷酸相似度和预测蛋白质相似度方面的差异都可佐证二者不是由同一个RNA剪接而来。
将红掌的2个SOC1与其他物种的同源蛋白比较表明,它们均含有保守的SOC1模体(motif),系统进化分析结果与红掌所处的植物学分类地位一致。本研究还分析了2个SOC1基因在红掌不同组织部位的表达水平,发现它们在营养和生殖器官中均有表达,且在花器官中的表达水平由外向内递减,但它们的转录本表达丰度不一致。至于二者在组成、表达上的异同是否会导致其功能差异,尚有待进一步研究。
| [1] | SIMPSON G G, DEAN C. Arabidopsis, the Rosetta stone of flowering time?. Science, 2002, 296(5566): 285-289. DOI:10.1126/science.296.5566.285 |
| [2] | SUNG S, AMASINO R M. Vernalization and epigenetics: How plants remember winter. Current Opinion in Plant Biology, 2004, 7(1): 4-10. DOI:10.1016/j.pbi.2003.11.010 |
| [3] | BÄURLE I, DEAN C. The timing of developmental transitions in plants. Cell, 2006, 125(4): 655-664. DOI:10.1016/j.cell.2006.05.005 |
| [4] | PARCY F. Flowering: A time for integration. International Journal of Development Biology, 2005, 49(5/6): 585-593. |
| [5] | LEE H, SUH S S, PARK E, et al. The AGAMOUS-LIKE 20 MADS domain protein integrates floral inductive pathways in Arabidopsis. Genes and Development, 2000, 14(18): 2366-2376. DOI:10.1101/gad.813600 |
| [6] | ONOUCHI H, IGEÑO M I, PÉRILLEUX C, et al. Mutagenesis of plants overexpressing CONSTANS demonstrates novel interactions among Arabidopsis flowering-time genes. The Plant Cell, 2000, 12(6): 885-900. DOI:10.1105/tpc.12.6.885 |
| [7] | SAMACH A, ONOUCHI H, GOLD S E, et al. Distinct roles of CONSTANS target genes in reproductive development of Arabidopsis. Science, 2000, 288(5471): 1613-1616. DOI:10.1126/science.288.5471.1613 |
| [8] | NAKAMURA T, SONG I J, FUKUDA T, et al. Characterization of TrcMADS1 gene of Trillium camtschatcense (Trilliaceae) reveals functional evolution of the SOC1/TM3-like gene family. Journal of Plant Research, 2005, 118(3): 229-234. DOI:10.1007/s10265-005-0215-5 |
| [9] | ZHONG X F, DAI X, XU J H, et al. Cloning and expression analysis of GmGAL1, SOC1 homolog gene in soybean. Molecular Biology Reports, 2012, 39(6): 6967-6974. DOI:10.1007/s11033-012-1524-0 |
| [10] | WANG S L, BERUTO M, XUE J Q, et al. Molecular cloning and potential function prediction of homologous SOC1 genes in tree peony. Plant Cell Reports, 2015, 34(8): 1459-1471. DOI:10.1007/s00299-015-1800-2 |
| [11] | LEI H J, YUAN H Z, LIU Y, et al. Identification and characterization of FaSOC1, a homolog of SUPPRESSOR OF OVEREXPRESSION OF CONSTANS1 from strawberry. Gene, 2013, 531(2): 158-167. DOI:10.1016/j.gene.2013.09.036 |
| [12] | SREEKANTAN L, THOMAS M R. VvFT and VvMADS8, the grapevine homologues of the floral integrators FT and SOC1, have unique expression patterns in grapevine and hasten flowering in Arabidopsis. Functional Plant Biology, 2006, 33(12): 1129-1139. DOI:10.1071/FP06144 |
| [13] | GREGIS V, SESSA A, DORCA-FORNELL C, et al. The Arabidopsis floral meristem identity genes AP1, AGL24 and SVP directly repress class B and C floral homeotic genes. The Plant Journal, 2009, 60(4): 626-637. DOI:10.1111/tpj.2009.60.issue-4 |
| [14] | BORNER R, KAMPMANN G, CHANDLER J, et al. A MADS domain gene involved in the transition to flowering in Arabidopsis. The Plant Journal, 2000, 24(5): 591-599. DOI:10.1046/j.1365-313x.2000.00906.x |
| [15] | LIU C, ZHOU J, BRACHA-DRORI K, et al. Specification of Arabidopsis floral meristem identity by repression of flowering time genes. Development, 2007, 134(10): 1901-1910. DOI:10.1242/dev.003103 |
| [16] | LIU C, CHEN H, ER H L, et al. Direct interaction of AGL24 and SOC1 integrates flowering signals in Arabidopsis. Development, 2008, 135(8): 1481-1491. DOI:10.1242/dev.020255 |
| [17] | IMMINK R G H, POSÉ D, FERRARIO S, et al. Characterization of SOC1's central role in flowering by the identification of its upstream and downstream regulators. Plant Physiology, 2012, 160(1): 433-449. DOI:10.1104/pp.112.202614 |
| [18] | LI D, LIU C, SHEN L S, et al. A repressor complex governs the integration of flowering signals in Arabidopsis. Developmental Cell, 2008, 15(1): 110-120. DOI:10.1016/j.devcel.2008.05.002 |
| [19] | LEE J, OH M, PARK H, et al. SOC1 translocated to the nucleus by interaction with AGL24 directly regulates LEAFY. The Plant Journal, 2008, 55(5): 832-843. DOI:10.1111/tpj.2008.55.issue-5 |
| [20] | MA G Y, NING G G, ZHANG W, et al. Overexpression of Petunia SOC1-like gene FBP21 in tobacco promotes flowering without decreasing flower or fruit quantity. Plant Molecular Biology Reporter, 2011, 29(3): 573-581. DOI:10.1007/s11105-010-0263-4 |
| [21] | DING L H, WANG Y W, YU H. Overexpression of DOSOC1, an ortholog of Arabidopsis SOC1, promotes flowering in the orchid Dendrobium Chao Parya Smile. Plant and Cell Physiology, 2013, 54(4): 595-608. DOI:10.1093/pcp/pct026 |
| [22] | ZHANG X H, WEI J H, FAN S L, et al. Functional characterization of GhSOC1 and GhMADS42 homologs from upland cotton (Gossypium hirsutum L.). Plant Science, 2016, 242: 178-186. DOI:10.1016/j.plantsci.2015.05.001 |
| [23] |
马广莹, 刘慧春, 邹清成, 等. 红掌花发育B类基因AaPI的克隆及表达分析. 分子植物育种, 2014, 12(5): 950-955. MA G Y, LIU H C, ZOU Q C, et al. Cloning and expression analysis of B class gene AaPI from Anthurium andraeanum. Molecular Plant Breeding, 2014, 12(5): 950-955. (in Chinese with English abstract) |
| [24] | VOOGD C, WANG T C, VARKONYI-GASIC E. Functional and expression analyses of kiwifruit SOC1-like genes suggest that they may not have a role in the transition to flowering but may affect the duration of dormancy. Journal of Experimental Botany, 2015, 66(15): 4699-4710. DOI:10.1093/jxb/erv234 |
| [25] | LIU C, ZHU C, ZENG H M. Key KdSOC1 gene expression profiles during plantlet morphogenesis under hormone, photoperiod, and drought treatments. Genetics and Molecular Research, 2016, 15(1): gmr.15017579. |
| [26] | NA X F, JIAN B, YAO W W, et al. Cloning and functional analysis of the flowering gene GmSOC1-like, a putative SUPPRESSOR OF OVEREXPRESSION CO1/AGAMOUS-LIKE 20 (SOC1/AGL20) ortholog in soybean. Plant Cell Reports, 2013, 32(8): 1219-1229. DOI:10.1007/s00299-013-1419-0 |
| [27] | RUOKOLAINEN S, YAN P N, ALBERT V A, et al. Over-expression of the Gerbera hybrida At-SOC1-like1 gene Gh-SOC1 leads to floral organ identity deterioration. Annals of Botany, 2011, 107(9): 1491-1499. DOI:10.1093/aob/mcr112 |
| [28] | ZHAO S Z, LUO Y Z, ZHANG Z L, et al. ZmSOC1, a MADS-box transcription factor from Zea mays, promotes flowering in Arabidopsis. International Journal of Molecular Sciences, 2014, 15(11): 19987-20003. DOI:10.3390/ijms151119987 |
| [29] | KIMURA Y, AOKI S, ANDO E, et al. A flowering integrator, SOC1, affects stomatal opening in Arabidopsis thaliana. Plant and Cell Physiology, 2015, 56(4): 640-649. DOI:10.1093/pcp/pcu214 |
| [30] | LIU S N, MA T F, MA L Y, et al. Ectopic expression of PvSOC1, a homolog of SOC1, from Phyllostachys violascens, promotes flowering in Arabidopsis and rice. Acta Physiologiae Plantarum, 2016, 38(7): 166. DOI:10.1007/s11738-016-2186-7 |
 2017, Vol. 43
2017, Vol. 43


