| 检测乙酰微小杆菌的双重实时荧光定量聚合酶链式反应方法的建立 |
有关食品腐败菌的鉴定与检测研究已有许多,但是由于贮藏环境不同,食品中的主要腐败菌也不尽相同,其中,在低温及其他极端环境(极酸、冷冻等)贮藏的食品中微小杆菌为其主要腐败菌。例如:蒋荣荣等[1]研究发现,在直接接触食品的酸菜外包膜上的微生物中,相对于真菌与其他细菌,其主要污染菌为微小杆菌,并成为酸菜中的主要腐败菌;同样,崔慧玲等[2]在鲜切生菜的优势腐败菌分离研究中指出,贮藏在4 ℃条件下的鲜切生菜中的主要腐败菌是微小杆菌;在冷藏的南美白对虾[3]和盐水鸭[4]等产品中均有检出微小杆菌。此外,从多种其他极端环境中也分离得到微小杆菌,它们已进化出与其生存环境相对应的嗜极性,包括耐/嗜冷性、耐/嗜热性、耐/嗜盐性、耐/嗜碱性等[5]。可见,微小杆菌在冷藏条件及多数极端贮藏条件下可以正常代谢。该菌还具有较强的蛋白质水解能力[6-7],导致产品被降解,加速食物腐败;此外,它还能产生物膜[8],不仅增加了微生物去污剂、防腐剂的抗性,而且通过捕获营养物质,加速其生长繁殖,并包裹和保护其他有害细菌和真菌。就鲜切叶菜而言,由于加工和贮藏条件不同,其上的主要腐败菌不同于普通蔬菜。鲜切叶菜的生产需要经过多项组织破坏性工艺,导致汁液外流,该营养环境适合多种细菌生长[9-10];但是鲜切叶菜需贮藏在4 ℃冷藏条件下,该条件不利于一般腐败菌的代谢,而嗜冷菌得到了滋生[11-12],这也在一定程度上对其他腐败菌的生长产生了拮抗作用[13-14]。
对于有害细菌的检测已有多种方法被研究并成功应用。其中,实时荧光定量聚合酶链式反应(real-time polymerase chain reaction, RT-PCR)不仅具有普通PCR的优点:灵敏度高、操作简便、检测快速、重复性佳、特异性好[15-17],而且它可实现高通量检测[18];此外,它还可以通过不同的引物设计在同一反应体系中同时对多个靶基因进行扩增;最重要的是,它可以实现定量检测。KOBAYSHI等[19]对RTPCR法、组织学检验法、快速尿素酶检验法、细菌培养和尿素呼气试验在临床样品胃组织检测中的灵敏度和特异性的研究结果表明,RT-PCR方法的灵敏度和特异性均为100%,为各种方法之最佳。PATEL等[20]用分子信标为探针的RT-PCR方法与传统的生理生化检测方法对感染沙门菌的鸡肉进行检测,发现传统方法检测需要3~8 d,而RT-PCR检测仅需18 h,并且灵敏度高。RIBERIO等[21]在幽门螺旋杆菌(Helicobacter pylori)的检测中指出,RTPCR一次可以检测96个样品,实现了一次性高通量检测。ZHAI等[22]建立了检测海德尔堡沙门菌血清型B的双重RT-PCR方法,该方法具有较强的抗背景干扰能力,在被感染3.6×105 CFU/mL鼠伤寒沙门菌或3.3×104 CFU/mL大肠埃希菌背景下,对海德尔堡沙门菌的最低检出限为2.2 CFU/mL,表现出很好的特异性。可见,RT-PCR快速检测技术不仅显示出了更好的时效性、特异性等传统方法的优点,也逐步被开发并在实际中得到了应用。
本研究以鲜切叶菜中的乙酰微小杆菌(Exiguobacterium acetylicum)为研究对象,以前期试验发掘的特异性基因为检测靶点,运用已筛选到的特异性引物和设计的TaqMan探针建立并优化反应体系,最终建立快速、准确、灵敏、可重复、高效的检测E. acetylicum的双重RT-PCR方法,为在低温或其他极端环境中贮藏的食品的主要腐败菌控制提供快速检测方法,进而为食品货架期预测提供理论依据。
1 材料与方法 1.1 材料 1.1.1 菌株以12株乙酰微小杆菌为阳性对照,以4株微小杆菌属内其他种的菌株和10株非微小杆菌的腐败菌/致病菌作为阴性对照(表 1),检验引物的特异性。
| 表1 菌株及RT-PCR检测结果 Table 1 Strains used and their RT-PCR specificity |
 |
| 点击放大 |
Luria-Bertani(LB)液体培养基:胰蛋白胨10 g,酵母提取物5 g,氯化钠10 g,蒸馏水定容至1 L,完全溶解后分装,121 ℃灭菌20 min,待用。LB固体培养基:在LB液体培养基中加入1.8%琼脂,121 ℃灭菌20 min,待用。引物、T-291和T-2B探针(由上海生工生物工程技术服务公司合成);实时荧光定量试剂盒[2×Premix Ex TaqTM(Probe qPCR),购买自TaKaRa公司,大连];基因组DNA提取试剂盒(Omega Bio-Tek, Norcross, GA, 美国)。
1.1.3 仪器Nanodrop 2000核酸浓度测定仪(上海基因有限公司);LDZX-50 KBS立式压力蒸汽灭菌器(上海申安医疗器械厂);SW-CJ-1FD型单人单面净化工作台(江苏省苏州市净化设备有限公司);HYL-A全温摇瓶柜(江苏省太仓市强乐实验设备厂);5418高速离心机(Eppendorf公司,德国);DK-8 D型电热恒温水槽(上海森信实验仪器有限公司);WH-3微型旋涡混合仪(上海沪西分析仪器厂);实时荧光定量PCR仪(StepOnePlusTM system,ABI公司,美国);AY 120电子天平(SHIMADZU公司,日本);JS-380c全自动数码凝胶成像分析仪(上海培清科技有限公司)。
1.2 方法 1.2.1 细菌基因组DNA提取与测定参照细菌基因组提取试剂盒说明书提取细菌基因组,并通过核酸浓度测定仪测定基因组DNA提取液的浓度。
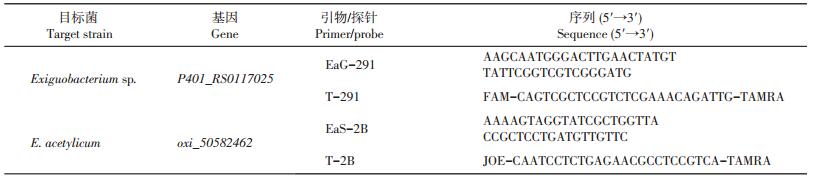
1.2.2 引物及探针的设计与合成在前期试验中参考ZHAI等[22]的方法,通过生物信息学比对与普通PCR检测,发掘出2组特异性引物(表 2):微小杆菌属特异性引物EaG-291(基因P401_RS0117025),以及乙酰微小杆菌特异性引物EaS-2B(基因oxi_50582462)。
| 表2 检测E. acetylicum的RT-PCR的引物及探针 Table 2 Primer and probe sets for RT-PCR detection of E. acetylicum |
 |
| 点击放大 |
本试验使用软件Primer Premier 5.0分别设计这2对引物对应的TaqMan探针,并对所设计的探针的特异性进行验证,最后,运用这2对引物与探针共同建立一个RT-PCR反应体系。
1.2.3 引物和探针浓度优化前期试验通过普通PCR方法对这2对引物的退火温度进行了优化。结果显示,退火温度为54 ℃时,条带亮度更为清晰且没有产生明显的二聚体;因此,本试验选择54 ℃为其最优退火温度。
根据实时荧光定量试剂盒[2×Premix Ex TaqTM (Probe qPCR)]使用说明书构建反应体系,其中,20 μL总反应体系中包含PCR混合物10 μL,ROX染料0.4 μL,模板DNA 2 μL,选择2个引物(EaG-291和EaS-2B)的浓度在0.2~0.6 μmol/L之间,2条探针(T-291和T-2B)的浓度在0.1~0.3 μmol/L之间,用ddH2O补至足量,分别组合进行RT-PCR,通过比较获得荧光信号所需阈值Ct的大小来判断引物和探针的最优浓度比例。每个反应设置3个平行,并以ddH2O作为阴性对照。反应参数:95 ℃预变性5 min;94 ℃变性15 s,54 ℃退火15 s,72 ℃延伸15 s,40个循环;同时,在延伸阶段采集荧光信号。
1.2.4 特异性评价用表 1中的细菌基因组DNA作为模板,按照1.2.3节建立的RT-PCR方法分别进行扩增,用以评价探针与所建体系的特异性。结果判定依据:以空白对照作为参照,Ct值大于35的反应判定为阴性,Ct值小于35的反应判定为阳性。
1.2.5 标准曲线制备为了确定该反应体系2个扩增曲线的有效性,将过夜培养的E. acetylicum按1.2.1节的方法提取基因组DNA与浓度测定,再用ddH2O进行10倍梯度稀释,分别作为模板进行RT-PCR扩增,建立标准曲线。
1.2.6 灵敏度评价DNA模板灵敏度评价:将过夜培养的E. acetylicum菌液提取基因组DNA,并测定样品基因DNA浓度,再用ddH2O进行10倍梯度稀释至n×10-9 ng/μL,分别进行RT-PCR扩增。
菌落灵敏度评价:将过夜培养的E. acetylicum菌液用无菌水10倍梯度稀释,并选择1×10-6、1×10-7和1×10-8 3个稀释梯度涂布平板计数,计算初始菌体浓度。同时,将每个稀释梯度(1×10-1~1×10-9)取1 mL提取基因组DNA,以此作为模板进行RT-PCR扩增。
1.2.7 重复性评价通过核酸定量仪测定样品基因DNA浓度,再用ddH2O进行10倍梯度稀释,使得样品中的DNA浓度为1.629、1.629×10-2和1.629×10-4 ng/μL,并进行RT-PCR扩增,分析不同梯度浓度的模板对整个反应体系的影响,以及在低浓度模板条件下反应体系的稳定性。
1.2.8 定量关系拟合取1 mL不同菌体浓度的E. acetylicum菌液提取的基因组作为模板进行RT-PCR扩增,再运用软件Origin 9对“Ct-菌体浓度(Nt)”之间的数量关系进行线性拟合。
1.2.9 RT-PCR对鲜切叶菜中E. acetylicum的检测应用参照文献[23]的方法制备鲜切叶菜,并按150 g分装在聚乙烯保鲜袋中,4 ℃贮藏。按照GB 47892—2010的方法洗脱收集鲜切叶菜中的细菌,按照1.2.1节的方法提取基因组DNA,按照1.2.3节建立的RT-PCR方法进行检测。在贮藏过程中,每2 d检测1次。
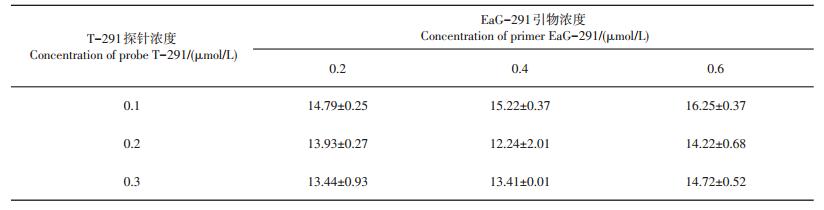
2 结果 2.1 引物和探针浓度优化 2.1.1 P401_RS0117025检测靶点的引物和探针浓度的优化由表 3可见,在RT-PCR反应体系中,引物浓度为0.4 μmol/L、探针浓度为0.2 μmol/L的组合对以P401_RS0117025基因为检测靶点的扩增曲线的Ct值达到最小,为12.24±2.01,明显优于其他试验组合。因此,引物EaG-291和探针T-291的最优浓度分别选择为0.4 μmol/L和0.2 μmol/L。
| 表3 引物和探针浓度对P401_RS0117025目标序列阈值的影响 Table 3 Effect of primer and probe concentrations on the threshold (Ct) of P401_RS0117025 target sequence in RT-PCR system |
 |
| 点击放大 |
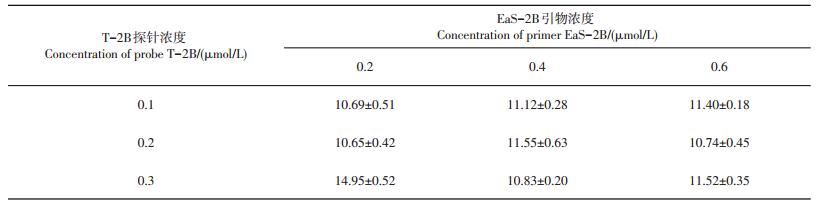
由表 4可见,在RT-PCR反应体系中,引物浓度为0.2 μmol/L、探针浓度为0.2 μmol/L的组合对以oxi_50582462基因为检测靶点的扩增曲线的Ct值达到最小,为10.65±0.42,优于其他试验组合。因此,引物EaS-2B和探针T-2B的最优浓度均选择为0.2 μmol/L。
| 表4 引物和探针浓度对oxi_50582462目标序列阈值的影响 Table 4 Effect of primer and probe concentrations on the threshold (Ct) of oxi_50582462 target sequence in RT-PCR system |
 |
| 点击放大 |
综上,检测乙酰微小杆菌的双重RT-PCR方法的最终反应体系为:2×PCR混合物10 μL,0.4 μmol/ L EaG-291前后引物各0.8 μL,0.2 μmol/L T-291探针0.4 μL,0.2 μmol/L EaS-2B前后引物各0.4 μL,0.2 μmol/L T-2B探针0.4 μL,ROX染料0.4 μL,模板DNA 2 μL,ddH2O 4.4 μL,总体积20 μL。
2.2 特异性评价以表 1中的细菌基因组DNA作为模板进行RT-PCR扩增。结果表明,所有的微小杆菌均能采集到P401_RS0117025基因扩增曲线的FAM荧光,但是只有12株E. acetylicum能同时采集到P401_RS0117025基因扩增曲线的FAM荧光和oxi_50582462基因扩增曲线的JOE荧光,而对10株非微小杆菌属细菌的DNA扩增中2种荧光信号均未检测到(表 1)。这表明本研究建立的RT-PCR检测体系具有良好的特异性。
2.3 标准曲线制备不同浓度梯度DNA扩增曲线见图 1,标准曲线见图 2。结果表明,用2个荧光扩增曲线绘制的标准曲线的R2值分别为0.994和0.999。说明建立的RT-PCR反应体系的有效性较高。
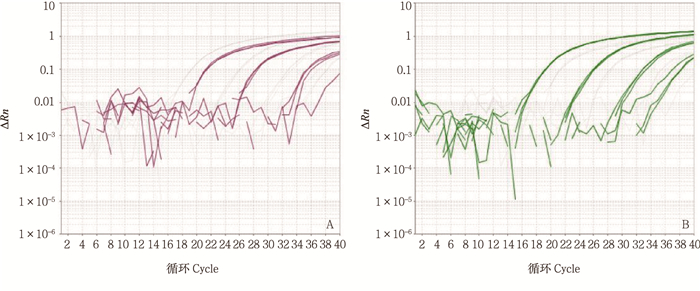 |
| ΔRn:荧光增量。 ΔRn: Fluorescence increment. 图1 检测靶点P401_RS0117025(A) 和oxi_50582462(B) 基因的RT-PCR扩增结果 Fig. 1 Result of RT-PCR for targets of P401_RS0117025 (A) and oxi_50582462 (B) genes |
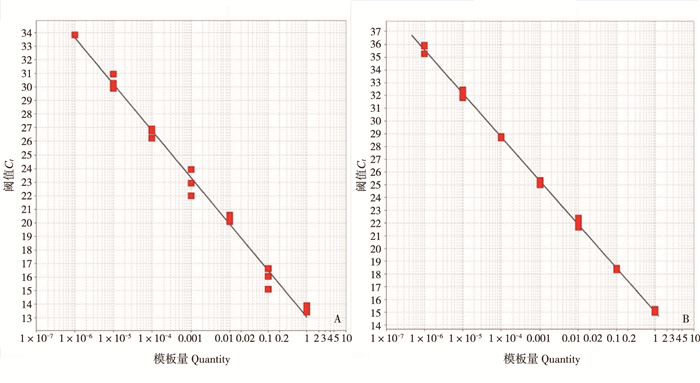 |
| 图2 检测靶点P401_RS0117025(A)和oxi_50582462(B)基因的RT-PCR标准曲线 Fig. 2 Standard curves of RT-PCR for targets of P401_RS0117025 (A) and oxi_50582462 (B) genes |
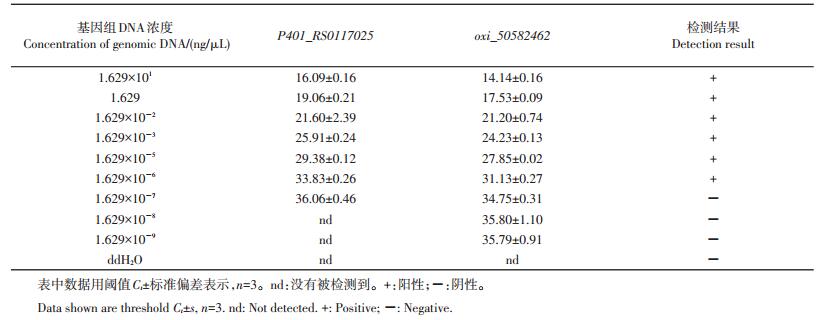
由表 5可见:当反应体系中基因组DNA浓度≥ 1.629×10-6 ng/μL时,2对引物均能获得阳性结果;当基因组DNA的浓度达到1.629×10-7 ng/μL时,P401_RS0117025基因扩增曲线的Ct值为36.06 ± 0.46,结果为阴性,oxi_50582462基因的检测结果为阳性。因此,该RT-PCR反应体系的DNA检测限为1.629×10-7 ng/μL。
| 表5 E. acetylicum DNA模板的RT-PCR灵敏度测定 Table 5 Sensitivity of RT-PCR assay for DNA template |
 |
| 点击放大 |
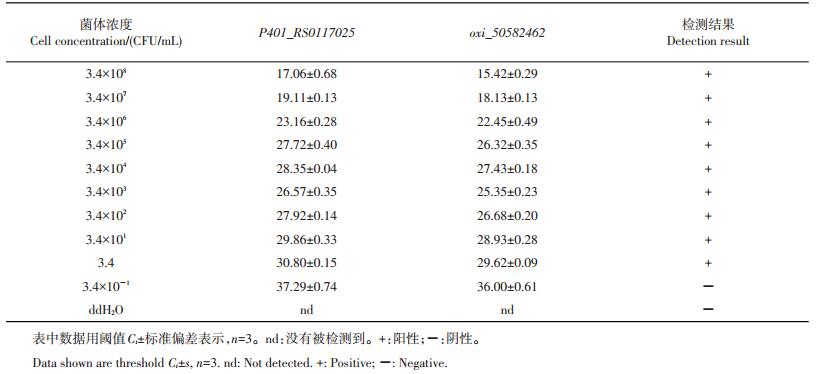
由表 6可见:当E. acetylicum的菌体浓度≥3.4 CFU/mL时,2对引物均能获得阳性结果;当其浓度为3.4×10-1 CFU/mL时,检测结果为阴性。因此,该RT-PCR反应体系的纯菌菌落检测限为3.4 CFU/mL。
| 表6 E. acetylicum纯菌菌落的RT-PCR灵敏度测定 Table 6 Sensitivity of RT-PCR assay in pure culture of E. acetylicum |
 |
| 点击放大 |
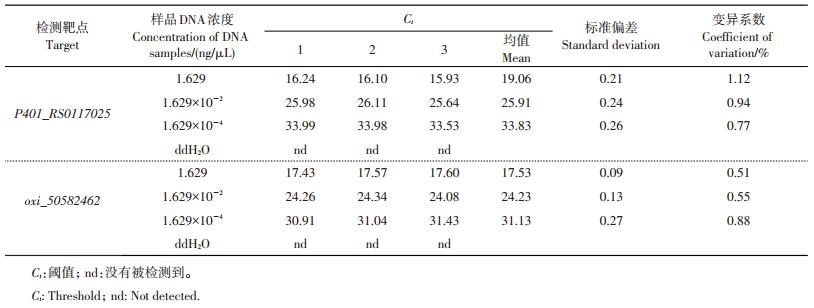
从表 7可以看出,当DNA浓度为1.629、1.629×10-2和1.629×10-4ng/μL时,随着样品中基因组DNA浓度的降低,RT-PCR检测体系中2条扩增曲线的Ct值升高;但是在不同的基因组DNA浓度条件下,该反应体系的2个Ct值的标准偏差没有明显变化,在0.30以内,变异系数在0.51~1.12之间变动。说明该RT-PCR检测体系的2对扩增引物与2条探针在DNA浓度较低(1.629×10-4 ng/μL)时仍能保持较好的重复性。
| 表7 RT-PCR检测体系的可重复性分析 Table 7 Repeatability analysis of RT-PCR assay |
 |
| 点击放大 |
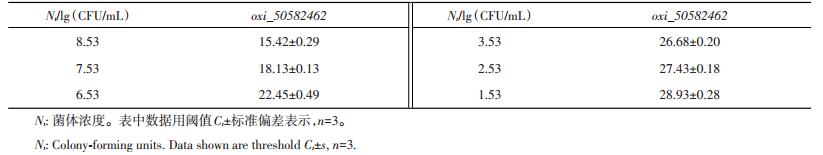
运用已建立的RT-PCR方法,对E. acetylicum不同菌体浓度菌液提取的基因组进行检测,结果见表 8。通过软件Origin 9对“Ct-Nt”之间的数量关系进行线性拟合,得到如下方程:Ct=-1.759×Nt+31.678,其R2=0.939,拟合效果较好。
| 表8 RT-PCR检测E. acetylicum不同菌体浓度的Ct值 Table 8 Ct value of RT-PCR detecting different cell concentrations of E. acetylicum |
 |
| 点击放大 |
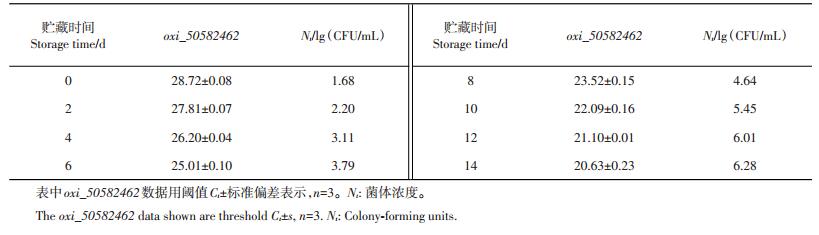
采用建立的RT-PCR方法对低温条件下不同贮藏时间的鲜切叶菜上的E. acetylicum进行检测。结果(表 9)表明,在无前增菌条件下,本文建立的RTPCR方法能够很好地对鲜切叶菜中的E. acetylicum进行检测;结合2.6节中的定量方程可对鲜切叶菜上的特定腐败菌E. acetylicum进行实时定量检测。
| 表9 低温条件下RT-PCR对鲜切叶菜中E. acetylicum的检测结果 Table 9 Detection result of E. acetylicum on fresh-cut leafy vegetables (FCLVs) at low temperature by RT-PCR |
 |
| 点击放大 |
本文以鲜切叶菜中的主要腐败菌E. acetylicum为研究对象,发掘了特异的TaqMan探针,并建立了检测该菌的双重RT-PCR。该方法特异性好、灵敏度高、检测快速,并且能对样品中的E. acetylicum进行定量检测。
本研究使用前期试验中发掘到的特异性靶点(P401_RS0117025和oxi_50582462)与引物(EaG-291和EaS-2B),对检测靶点设计了特异性TaqMan探针(T-291和T-2B),使用该探针建立了针对食品中E. acetylicum的RT-PCR检测方法。虽然在DEBORA等[24]的研究中建立了分离嗜常温与嗜热特性的微小杆菌的RT-PCR方法,但其主要是针对不同环境偏好特性的微小杆菌的区分。本研究建立的方法主要解决对食品中主要腐败菌E. acetylicum的检测,且对该腐败菌的研究在国内外也是首次提出,为低温或极端环境贮藏的食品的质量监督提供了新的、特定的检测指标与方法。
本研究建立的方法在供试的16株微小杆菌与10株非微小杆菌的致病菌/腐败菌中表现出良好的特异性,说明该方法对分离菌株与标准菌株都表现出较高的特异性。而且该检测体系在线性相关分析中2条扩增曲线的R2值均能达到0.99以上,与WALL等[25]建立的RT-PCR检测体系的相关性一致,即优化后的反应体系具有良好的有效性。此外,在重复性评价试验中该方法的变异系数在0.51~1.12之间变动,说明该方法具有良好的稳定性和准确性。
该RT-PCR方法对基因组DNA的最低检出限为1.629×10-7ng/μL;且在检测前无前增菌处理的情况下,对纯菌菌落的最低检出限即可达到3.4 CFU/ mL。而GALLELLI等[26]建立的双重RT-PCR检测方法对假单胞菌的最低检出限为5×104~5×105 CFU/ mL。说明本研究建立的检测方法灵敏度高。
传统的细菌分离鉴定方法需要前增菌与分离单菌落,并需生化实验结合分子生物学共同进行鉴定,检测耗时长,工作量大。本研究建立的检测方法操作简单、用时少,在很大程度上优于传统方法。虽然RT-PCR方法在一定程度上缩短了检测时间,但是王建昌等[27]建立的大肠埃希菌O157:H7的RT-PCR检测方法仍需要6~10 h的前增菌试验,这对于保质期极短的冷藏食品并不适宜。本研究建立的检测方法灵敏度高,在无前增菌条件下,对纯菌菌落的最低检出限可达3.4 CFU/mL,比普通的RT-PCR检测更加快速,这对于货架期短、需低温贮藏的食品中的E. acetylicum检测具有重要意义。
为防止假阳性结果出现,本研究采用微小杆菌属特异性基因P401_RS0117025和E. acetylicum特异性基因oxi_50582462作为检测靶点,同时进行检测。这种试验设计能够很好地排除假阳性结果,提高了方法的特异性和准确性[15-16]。同时,本研究还探索了Ct值与Nt(菌体浓度)之间的线性关系,并建立起“Ct-Nt”关系方程。该方程能对样品中的E. acetylicum进行绝对定量,从而为低温或极端环境贮藏的食品的货架期预测提供理论依据。并且,在对样品检测中,本研究建立的RT-PCR方法能够快速、灵敏地对鲜切叶菜中的E. acetylicum进行特异性检测,体现了很好的应用前景。
4 结论 4.1本研究设计了特异性探针。针对前期试验发掘到的Exiguobacterium sp.特异性基因P401_RS0117025设计TaqMan探针T-291,针对E. acetylicum特异性基因oxi_50582462设计TaqMan探针T-2B;经12株E. acetylicum、4株Exiguobacterium sp.和10株非微小杆菌验证显示,2条探针的特异性良好。
4.2建立了E. acetylicum的双重TaqMan探针的RT-PCR检测方法。对体系中引物EaG-291、EaS-2B与探针T-291、T-2B优化后,得到最优的反应体系为EaG-291 0.8 μL,EaS-2B 0.4 μL,T-291 0.4μL,T-2B 0.4 μL,PCR混合物10 μL,ROX染料0.4 μL,模板DNA 2 μL,以无菌水补足至20 μL。
4.3对建立的RT-PCR检测方法进行了评价。由该反应体系绘制的2条标准曲线中R2分别为0.994和0.999,且经12株E.acetylicum和4株Exiguobacterium sp.验证,呈现出良好的特异性。该方法的DNA灵敏度为1.629×10-7 ng/μL,纯菌菌落灵敏度为3.4 CFU/mL,且在不同浓度的DNA模板条件下均呈现出较好的可重复性。
4.4根据标准曲线建立了Ct值与Nt之间的数量关系方程:Ct=-1.759×Nt+31.678。同时,本研究建立的双重RT-PCR方法可对样品中的E. acetylicum进行绝对定量,从而可为低温或极端环境贮藏的食品的货架期预测提供理论依据。
| [1] |
蒋荣荣, 赵炜, 王洋, 等.酸菜包外包膜污染菌分析.
中国酿造,2013,32 (1):133–136.
JIANG R R, ZHAO W, WANG Y, et al. Analysis of polluted microorganisms on the outer packages of Chinese sauerkraut. China Brewing, 2013,32 (1):133–136. (in Chinese with English abstract) |
| [2] |
崔慧玲, 陈安钧, 罗婵, 等.鲜切生菜贮藏过程中优势腐败细菌的多样性分析.
食品与发酵工业,2014,40 (7):223–229.
CUI H L, CHEN A J, LUO C, et al. The diversity analysis of dominant spoilage bacteria in fresh-cut lettuce during storage. Food and Fermentation Industries, 2014,40 (7):223–229. (in Chinese with English abstract) |
| [3] |
赵海鹏, 谢晶, 严文蓉.南美白对虾冷藏过程中的细菌分离、初步鉴定及菌相分析.
江苏农业学报,2011,27 (1):164–168.
ZHAO H P, XIE J, YAN W R. Isolation and identification of bacteria in Penaeus vannammei during cold storage and analysis of composition of microorganism. Jiangsu Journal of Agricultural Sciences, 2011,27 (1):164–168. (in Chinese with English abstract) |
| [4] |
LIU F, WANG D Y, DU L H, et al. Diversity of the predominant spoilage bacteria in water-boiled salted duck during storage.
Journal of Food Science, 2010,75 (5):317–321. DOI: 10.1111/j.1750-3841.2010.01644.x. |
| [5] |
VISHNIVETSKAYA T A, KATHARIOU S, TIEDJE J M. The Exiguobacterium genus: Biodiversity and biogeography.
Extremophiles, 2009,13 (3):541–555. DOI: 10.1007/s00792-009-0243-5. |
| [6] |
HARADA J, TAKAKU S, WATANABE K. An on-demand metalloprotease from psychro-tolerant Exiguobacterium undae Su-1, the activity and stability of which are controlled by the Ca2+ concentration.
Bioscience, Biotechnology and Biochemistry, 2012,76 (5):986–992. DOI: 10.1271/bbb.110997. |
| [7] |
SOROKULOVA I, KRUMNOW A, GLOBA L, et al. Efficient decomposition of shrimp shell waste using Bacillus cereus and Exiguobacterium acetylicum.
Journal of Industrial Microbiology and Biotechnology, 2009,36 :1123–1126. DOI: 10.1007/s10295-009-0587-y. |
| [8] |
KUMAR C G, ANAND S K. Significance of microbial biofilms in food industry: A review.
International Journal of Food Microbiology, 1998,42 (1/2):9–27. |
| [9] |
FALAH A M F, NADINE M D, SURYANDONO A. Effects of storage conditions on quality and shelf-life of fresh-cut melon (Cucumis Melo L.) and papaya (Carica Papaya L.).
Procedia Food Science, 2015,3 :313–322. DOI: 10.1016/j.profoo.2015.01.034. |
| [10] |
MUKHOPADHYAY S, RAMASWAMY R. Application of emerging technologies to control Salmonella in foods: A review.
Food Research International, 2012,45 (2):666–677. DOI: 10.1016/j.foodres.2011.05.016. |
| [11] |
RANIERI M L, IVY R A, MITCHELL W R, et al. Real-time PCR detection of Paenibacillus spp. in raw milk to predict shelf life performance of pasteurized fluid milk products.
Applied and Environmental Microbiology, 2012,78 (16):5855–5863. DOI: 10.1128/AEM.01361-12. |
| [12] |
LEE D H, KIM J B, KIM M, et al. Microbiota on spoiled vegetables and their characterization.
Journal of Food Protection, 2013,76 (8):1350–1358. DOI: 10.4315/0362-028X.JFP-12-439. |
| [13] |
PINTO L, IPPOLITO A, BARUZZI F. Control of spoiler Pseudomonas spp. on fresh cut vegetables by neutral electrolyzed water.
Food Microbiology, 2015,50 :102–108. DOI: 10.1016/j.fm.2015.04.003. |
| [14] |
BOWER C K, DAESCHEL M A. Resistance responses of microorganisms in food environments.
International Journal of Food Microbiology, 1999,50 (1/2):33–44. |
| [15] |
HEID C A, STEVENS J, LIVAK K J, et al. Real time quantitative PCR.
Genome Research, 1996,6 (10):986–994. DOI: 10.1101/gr.6.10.986. |
| [16] |
NARANG N, FRATAMICO P M, TILLMAN G, et al. Performance comparison of a fliCh7 real-time PCR assay with an H7 latex agglutination test for confirmation of the H type of Escherichia coli O157:H7.
Journal of Food Protection, 2009,72 :2195–2197. DOI: 10.4315/0362-028X-72.10.2195. |
| [17] |
李彬, 栗寒, 李艳华, 等.豇豆花叶病毒和黑眼豇豆花叶病毒RTReal time PCR及IC-RT-Real time PCR检测方法研究.
南京农业大学学报,2010,33 (2):105–109.
LI B, SU H, LI Y H, et al. Research on RT-Real time PCR and ICRT-Real time PCR detection of Cow pea mosaic virus and Blackeye cow pea mosaic virus. Journal of Nanjing Agricultural University, 2010,33 (2):105–109. (in Chinese with English abstract) |
| [18] |
张小敏, 周斌, 何丹妮, 等.实时荧光定量PCR检测猪源Mx1方法的建立及应用.
南京农业大学学报,2013,36 (1):92–96.
ZHANG X M, ZHOU B, HE D N, et al. Establishment and application of the SYBR green Ⅰ real-time PCR assay for detection of porcine antiviral protein Mx1 gene. Journal of Nanjing Agricultural University, 2013,36 (1):92–96. (in Chinese with English abstract) DOI: 10.7685/j.issn.1000-2030.2013.01.016. |
| [19] |
KOBAYASHI D, EISHI Y, OHKUSA T, et al. Gastric mucosal density of Helicobacter pylori estimated by real-time PCR compared with results of urea breath test and histological grading.
Journal of Medical Microbiology, 2002,51 (4):305–311. DOI: 10.1099/0022-1317-51-4-305. |
| [20] |
PATEL J R, BHAGWAT A A, SANGLAY G C, et al. Rapid detection of Salmonella from hydrodynamic pressure-treated poultry using molecular beacon real-time PCR.
Food Microbiology, 2006,23 :39–46. DOI: 10.1016/j.fm.2005.01.011. |
| [21] |
RIBERIO M L, ECCLISSATO C C, MATTOS R G, et al. Quantitative real-time PCR for the clinical detection of Helicobacter pylori.
Genetics and Molecular Biology, 2007,30 (2):431–434. DOI: 10.1590/S1415-47572007000300022. |
| [22] |
ZHAI L G, YU Q, BIE X M, et al. Development of a PCR test system for specific detection of Salmonella Paratyphi B in foods.
FEMS Microbiology Letters, 2014,355 :83–89. DOI: 10.1111/fml.2014.355.issue-1. |
| [23] |
谢晶, 张利平, 高志立, 等.鸡毛菜的品质动力学分析及货架期预测模型.
食品科学,2014,35 (10):268–272.
XIE J, ZHANG L P, GAO Z L, et al. Kinetic quality changes and shelf life prediction of Chinese small cabbage (Brassica rapa L. Chinensis group). Food Science, 2014,35 (10):268–272. (in Chinese with English abstract) DOI: 10.7506/spkx1002-6630-201410050. |
| [24] |
DEBORA F R, JAMES M T. Multi-locus real-time PCR for quantitation of bacteria in the environment reveals Exiguobacterium to be prevalent in permafrost.
FEMS Microbiology Ecology, 2007,59 (2):489–499. DOI: 10.1111/fem.2007.59.issue-2. |
| [25] |
WALL S J, EDWARDS D R. Quantitative reverse transcriptionpolymerase chain reaction (RT-PCR): A comparison of primerdropping, competitive, and real-time PCRs.
Analytical Biochemistry, 2002,300 :269–273. DOI: 10.1006/abio.2001.5458. |
| [26] |
GALLELLI A, TALOCCI S, PILOTTI M, et al. Real-time and qualitative PCR for detecting Pseudomonas syringae pv. actinidiae isolates causing recent outbreaks of kiwifruit bacterial canker.
Plant Pathology, 2014,63 :264–276. DOI: 10.1111/ppa.2014.63.issue-2. |
| [27] |
王建昌, 王金凤, 段永生, 等.基于内参的大肠埃希菌O157:H7实时荧光定量PCR快速检测方法的建立.
食品科学,2015,36 (20):226–231.
WANG J C, WANG J F, DUAN Y S, et al. Development of realtime quantitative PCR assay for the detection of E. Coli O157:H7 based on internal amplification reference. Food Science, 2015,36 (20):226–231. (in Chinese with English abstract) DOI: 10.7506/spkx1002-6630-201520044. |
 2017, Vol. 43
2017, Vol. 43


