| 术前MRI影像组学预测模型在非增强成人弥漫性胶质瘤分级诊断中的应用 |
2. 中国人民解放军总医院第一医学中心放射诊断科, 北京 100853;
3. 中国人民解放军总医院第四医学中心放射诊断科, 北京 100048;
4. 中国人民解放军总医院第一医学中心神经外科, 北京 100853
2. Department of Radiology, First Medical Center of Chinese PLA General Hospital, Beijing 100853, China;
3. Department of Radiology, Forth Medical Center of Chinese PLA General Hospital, Beijing 100048, China;
4. Department of Neurosurgery, First Medical Center of Chinese PLA General Hospital, Beijing 100853, China
脑胶质瘤是中枢神经系统最常见的原发性肿瘤,约占原发恶性脑肿瘤的80%[1]。根据2021年WHO的中枢神经系统分类标准,其分级基于分子遗传特征和组织学表现[2]。高级别胶质瘤(WHO 3、4级)侵袭性强、预后差,需最大范围安全切除,因此术前精准分级至关重要[3-4]。MRI是胶质瘤术前评估的常规检查手段,但其更倾向于将非增强胶质瘤诊断为低级别胶质瘤。而8%~18%的非增强胶质瘤为高级别胶质瘤,传统影像评估存在误判风险,易导致治疗延误或术式选择不当,影响预后[5]。本研究基于术前非增强胶质瘤的MRI图像,结合影像组学构建模型,预测非增强胶质瘤的分级,为临床决策提供支持。
1 资料与方法 1.1 一般资料回顾性收集2016年1月至2023年12月在中国人民解放军总医院第一医学中心初诊为成人弥漫性胶质瘤且未经过相关治疗的患者153例(160个病灶),其中男91例,女62例;年龄20~77岁,平均(41.98±11.47)岁。160个病灶中,低级别胶质瘤103个(低级别组),高级别胶质瘤57个(高级别组)。将病灶按8∶2比例随机划分为训练集128个和验证集32个。
纳入标准:①年龄≥18岁;②术前MRI资料完整,包括T2WI、T2 FLAIR、T1WI增强扫描和ADC图;③术前T1WI增强扫描图像显示为非增强胶质瘤,即肿瘤内增强部分体积≤ 1 cm3[6]。排除标准:①病理诊断未明确级别;②根据2021年WHO分类标准被判定为未分类(NOS)或未评估(NEC);③图像质量不满足诊断要求。
本研究经中国人民解放军总医院医学伦理委员会批准(批号:2021-006-01)。
1.2 仪器与方法采用GE Discovery MR750 3.0 T MRI扫描仪、32通道头部线圈。扫描序列及参数:轴位T2WI,TR 5 642 ms,TE 93 ms,视野24 cm×24 cm,矩阵512×512,层厚5.5 mm,层距1.5 mm;轴位T1WI增强扫描,TR 1 743 ms,TE 27 ms,视野24 cm×24 cm,矩阵320×324,层厚5.5 mm,层距1.5 mm;DWI,TR 3 000 ms,TE 67.6 ms,视野24 cm×24 cm,矩阵160×160,层厚6 mm,层距1.5 mm,b值取1 000 s/mm2;ADC图在GE AW4.6工作站自动生成。
1.3 图像分析由1位具有3年工作经验的影像医师观察MRI图像上病灶的长径、短径、坏死、囊变、占位效应、边缘清晰、T2错配征、部位和水肿程度。其中占位效应分级基于脑室及中线结构受压程度判定:0级,无占位效应;1级,局部脑沟/脑池受压,但脑室及中线结构形态正常;2级,脑室受压变形,但中线无移位;3级,脑室受压伴中线向对侧移位≤ 1 cm;4级,中线移位>1 cm[7]。脑水肿程度判定:轻度,水肿宽度≤ 2 cm;中度,水肿宽度>2 cm,但未超过同侧大脑半球1/2区域;重度,水肿范围超过同侧大脑半球1/2区域。
1.4 影像组学分析 1.4.1 MRI图像预处理和肿瘤分割图像由DICOM格式转换为NIFTI格式后,依次行N4偏置场校正和归一化处理。基于MATLAB软件的SPM插件(https://www.fil.ion.ucl.ac.uk/spm),将T2WI和ADC图像配准至对应的T1WI增强扫描图像上。双盲条件下,由1位具有3年工作经验的影像医师使用ITK-SNAP(版本4.0.1)软件,在轴位T2WI图像上手动勾画全肿瘤区域ROI,包括钙化、出血、囊变及坏死区域,但不包含瘤周水肿区域(图 1)。为准确区分瘤周水肿和肿瘤,勾画时参考T2 FLAIR和T1WI增强扫描图像。为评估影像组学特征的可重复性,随机抽取30%病灶,由同一位影像医师在1个月后再次勾画ROI。
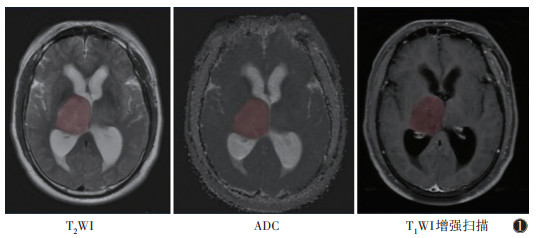 |
| 图 1 T2WI、ADC、T1WI增强扫描图像上ROI勾画示意图 |
1.4.2 特征提取及筛选
使用Python(版本3.9.0)中的Pyradiomics工具包提取影像组学特征,每个序列共提取1 132个影像组学特征,包括形状特征(14个)、一阶直方图特征(18个)、二阶纹理特征(68个)和小波特征(1 032个)。通过ICC行组间一致性分析,筛选出ICC ≥ 0.85的特征。采用弹性网络回归算法结合递归特征消除算法,计算最优特征数量。最终,根据最优特征子集的数量和特征权重排序,确定最具分级诊断价值的组学特征。
1.4.3 模型建立及验证采用逻辑回归(logistic regression,LR)、支持向量机(support vector machine,SVM)、K-最近邻(K-nearest neighbor,KNN)、决策树(decision tree,DT)和朴素贝叶斯(naive Bayes,NB)进行模型构建,其中SVM分类器包括径向基函数核(SVMrbf)和线性核(SVMlinear)2种。T2WI、T1WI增强扫描、ADC序列独立或组合生成7个影像特征组:单序列(T2WI、T1WI增强扫描、ADC)、双序列(T2WI+T1WI增强扫描、T2WI+ADC、T1WI增强扫描+ADC)、三序列(T2WI+T1WI增强扫描+ADC)。7个影像特征组在6种分类器中进行训练和验证,共建立42种模型,通过对比不同分类器和特征组的分类性能,筛选出诊断效能最佳的模型及影像特征组,以实现胶质瘤术前分级的精确预测。
1.5 统计学分析使用SPSS 26.0软件和R软件(版本4.1.0)分析数据。计量资料以x ± s表示,组间比较行独立样本t检验;计数资料以例(%)表示,组间比较行χ2检验。采用AUC、准确率、敏感度、特异度和F1分数评估模型的预测效能。以P < 0.05为差异有统计学意义。
2 结果 2.1 2组MRI特征比较2组病灶长径、短径、坏死、囊变、占位效应、边缘清晰、T2错配征、部位、水肿程度差异均无统计学意义(均P>0.05)(表 1,图 2)。
| 表 1 低级别与高级别成人弥漫性胶质瘤的MRI特征比较 |
 |
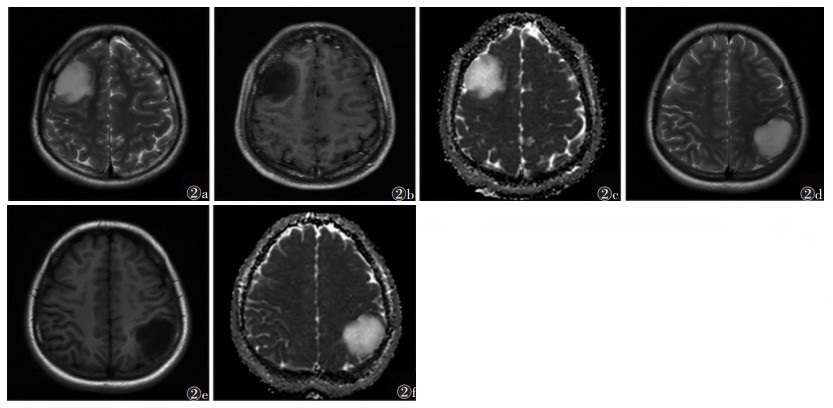 |
| 注:图2a~2c为高级别胶质瘤图像,患者,女,29岁;图 2a为T2WI图像,示右侧额叶高信号病灶伴轻度瘤周水肿;图2b为T1WI增强扫描图像,示病灶无明显强化;图2c为ADC图,病灶呈高信号。图2d~2f为低级别胶质瘤图像,患者,女,23岁;图2d为T2WI图像,示左侧顶叶高信号病灶伴轻度瘤周水肿;图2e为T1WI增强扫描图像,示病灶无明显强化;图2f为ADC图,病灶呈高信号 图 2 非增强成人弥漫性胶质瘤的MRI图像 |
2.2 影像组学特征筛选结果
经一致性分析,筛选ICC ≥ 0.85的特征,利用递归特征消除算法去除冗余特征,确定最佳特征数量,其中T1WI增强扫描序列2个,ADC序列6个,T2WI序列3个,T2WI+T1WI增强扫描序列3个,T2WI+ADC序列2个,T1WI增强扫描+ADC序列5个,T2WI+T1WI增强扫描+ADC序列7个。
2.3 模型的诊断效能评估单序列组中T1WI增强扫描影像组学特征KNN模型效能最高,训练集及验证集AUC分别为0.874、0.864;双序列组中基于T2WI+T1WI增强扫描特征的KNN模型效能最高,训练集及验证集AUC分别为0.887、0.839;三序列组中KNN模型效能最高,训练集及验证集AUC分别为0.892、0.805(表 2)。
| 表 2 7个影像特征组中效能最佳的影像组学模型在训练集和验证集中的诊断效能 |
 |
3 讨论
高级别成人弥漫性胶质瘤术前MRI常表现为花环样不均匀强化、周围水肿及坏死等特征,但有8%~18%病灶术前未见明显强化,呈低级别胶质瘤外观,为诊断带来挑战[5]。高、低级别胶质瘤在手术切除范围及预后管理上存在显著差异,因此,准确诊断对临床制订个性化方案至关重要。本研究应用机器学习方法开发了一种基于术前MRI的无创诊断非增强高级别成人弥漫性胶质瘤的影像组学模型,发现各模型均具有较好的诊断效能。
研究表明,对比增强图像中的强化常被视为肿瘤侵袭性的影像学特征,与预后较差相关[8]。对比增强在恶性肿瘤中常被解释为微血管增生和血-脑脊液屏障通透性增加,使得对比剂更易渗入肿瘤组织。既往研究显示,对比增强是预测异柠檬酸脱氢酶(isocitrate dehydrogenase,IDH)野生型胶质瘤的可靠影像学特征,但非增强并不是IDH突变型胶质瘤的有效预测指标[9-10]。另一项研究显示,所有非增强胶质瘤均为IDH突变型,其用于判断IDH1突变状态的准确率高达97.5%[11]。这一差异可能与不同版本的WHO中枢神经系统分类对胶质瘤分级标准的不同有关。自2016年WHO分类更新后,分子基因首次被纳入胶质瘤分级标准,胶质瘤的高级别判定不再仅依赖于组织学改变[2],这扩大了高级别胶质瘤的定义范围。
人工智能作为计算机科学的一个新兴分支,借助复杂的算法和训练模型,可从大量影像数据中集中挖掘隐藏信息,实现精准诊断及对生存与治疗反应的预测。目前,多项研究表明影像组学在术前预测胶质瘤分级方面具有重要潜力[12]。van der Voort等[13]开发了一种自动分割肿瘤区域并预测胶质瘤分级的模型,其在独立测试集中的AUC达0.81,并在IDH和1p19q缺失状态的预测上表现出良好的性能。Cheng等[14]基于多参数MRI及影像组学建立了术前预测胶质瘤分级的模型,该模型在使用肿瘤内特征、瘤周特征和融合特征时均表现出优异效能,AUC达0.975。然而,目前尚缺乏针对非增强胶质瘤术前分级的研究报道。
影像组学特征通过定量分析肿瘤强度、纹理等图像参数,可表征组织异质性和微环境特征[15-16]。本研究中多序列组筛选的7个关键特征中,3个一阶特征反映了肿瘤组织内像素强度的分布特征,可能与肿瘤的异质性相关;4个纹理特征则通过量化影像中灰度值的空间分布和纹理信息,反映肿瘤组织的异质性和微观结构[16-17]。其中ADC的90%百分位数及T1WI增强扫描的偏度特征反映了高级别胶质瘤的典型病理改变——高细胞密度及微血管增生[18-20],这从生物学机制层面解释了模型的分级鉴别能力。KNN是一种基于实例的监督学习算法,用于分类和回归任务,具有算法简单、高效、适合小数据集等优点。既往研究表明,KNN在肿瘤类型鉴别、基因分子改变预测、肿瘤级别与复发判断,以及预后评估方面的表现优于其他模型[21-24]。
本研究具有以下局限性:仅分析临床常用的T2WI、T1WI增强扫描和ADC序列,今后可考虑采用更先进的MRI技术,如DTI和MRS;使用6种机器学习方法(SVMrbf、SVMlinear、LR、KNN、DT和NB)构建最佳模型,未来可探索更多机器学习方法,如深度学习和集成学习方法,进一步优化MRI影像组学模型;样本来源于单中心,后续拟开展多中心研究,并扩大样本量,进一步提高预测模型的稳健性和普适性。
综上所述,基于术前常规MRI图像的影像组学结合KNN模型能非侵入性地鉴别诊断非增强高级别成人弥漫性胶质瘤,为患者的个性化诊疗和预后管理提供重要依据。
| [1] |
OSTROM Q T, PRICE M, NEFF C, et al. CBtrus statistical report: primary brain and other central nervous system tumors diagnosed in the united states in 2016—2020[J]. Neuro-Oncology, 2023, 25(Supplement_4): ⅳ1-ⅳ99. |
| [2] |
杨学军, 江涛, 陈忠平. 世界卫生组织中枢神经系统肿瘤分类的演变: 1979—2021年[J]. 中国现代神经疾病杂志, 2021, 21(9): 710-724. |
| [3] |
PARK Y W, VOLLMUTH P, FOLTYN-DUMITRU M, et al. The 2021 WHO classification for gliomas and implications on imaging diagnosis: part 1-key points of the fifth edition and summary of imaging findings on adult-type diffuse gliomas[J]. JMRI, 2023, 58(3): 677-689. |
| [4] |
NOORBAKHSH A, TANG J A, MARCUS L P, et al. Gross-total resection outcomes in an elderly population with glioblastoma: a SEER-based analysis[J]. J Neurosurg, 2014, 120(1): 31-39. |
| [5] |
EICHBERG D G, DI L, MORELL A A, et al. Incidence of high grade gliomas presenting as radiographically non-enhancing lesions: experience in 111 surgically treated non-enhancing gliomas with tissue diagnosis[J]. J Neurooncol, 2020, 147(3): 671-679. |
| [6] |
PARK Y W, HAN K, AHN S S, et al. Prediction of IDH1-mutation and 1p/19q-codeletion status using preoperative MR imaging phenotypes in lower grade gliomas[J]. AJNR Am J Neuroradiol, 2018, 39(1): 37-42. |
| [7] |
SAWAYA R, YAMAGUCHI S, ISHI Y, et al. Increased CD44 expression in primary meningioma: its clinical significance and association with peritumoral brain edema[J]. J Neurosurg, 2024, 141(1): 100-107. |
| [8] |
PIERALLINI A, BONAMINI M, BOZZAO A, et al. Supratentorial diffuse astrocytic tumours: proposal of an MRI classification[J]. Eur Radiol, 1997, 7(3): 395-399. |
| [9] |
MERENZON M A, GÓMEZ ESCALANTE J I, PROST D, et al. Preoperative imaging features: are they useful tools for predicting IDH1 mutation status in gliomas grades Ⅱ-Ⅳ?[J]. Surg Neurol Int, 2022, 13: 332. |
| [10] |
LASOCKI A, TSUI A, GAILLARD F, et al. Reliability of noncontrast-enhancing tumor as a biomarker of IDH1 mutation status in glioblastoma[J]. J Clin Neurosci, 2017, 39: 170-175. |
| [11] |
CARRILLO J A, LAI A, NGHIEMPHU P L, et al. Relationship between tumor enhancement, edema, IDH1 mutational status, MGMT promoter methylation, and survival in glioblastoma[J]. AJNR Am J Neuroradiol, 2012, 33(7): 1349-1355. |
| [12] |
FAN H, LUO Y, GU F, et al. Artificial intelligence-based MRI radiomics and radiogenomics in glioma[J]. Cancer Imaging, 2024, 24(1): 36. |
| [13] |
VAN DER VOORT S R, INCEKARA F, WIJNENGA M M J, et al. Combined molecular subtyping, grading, and segmentation of glioma using multi-task deep learning[J]. Neuro Oncol, 2023, 25(2): 279-289. |
| [14] |
CHENG J, LIU J, YUE H, et al. Prediction of glioma grade using intratumoral and peritumoral radiomic features from multiparametric MRI images[J]. IEEE/ACM Trans Comput Biol Bioinform, 2022, 19(2): 1084-1095. |
| [15] |
AERTS H J W L, VELAZQUEZ E R, LEIJENAAR R T H, et al. Decoding tumour phenotype by noninvasive imaging using a quantitative radiomics approach[J]. Nat Commun, 2014, 5(1). |
| [16] |
GILLIES R J, KINAHAN P E, HRICAK H. Radiomics: images are more than pictures, they are data[J]. Radiology, 2016, 278(2): 563-577. |
| [17] |
SONI N, PRIYA S, BATHLA G. Texture analysis in cerebral gliomas: a review of the literature[J]. AJNR Am J Neuroradiol, 2019, 40(6): 928-934. |
| [18] |
POUSSAINT T Y, VAJAPEYAM S, RICCI K I, et al. Apparent diffusion coefficient histogram metrics correlate with survival in diffuse intrinsic pontine glioma: a report from the pediatric brain tumor consortium[J]. Neuro Oncol, 2016, 18(5): 725-734. |
| [19] |
MEYER H J, HÖHN A K, HAMERLA G, et al. Histogram parameters derived from T2 weighted images are associated with histopathological findings in rectal cancer—a preliminary study[J]. Am J Transl Res, 2018, 10(11): 3790. |
| [20] |
CHANG P D, MALONE H R, BOWDEN S G, et al. A multiparametric model for mapping cellularity in glioblastoma using radiographically localized biopsies[J]. AJNR Am J Neuroradiol, 2017, 38(5): 890-898. |
| [21] |
YANG Z, CHEN M, KAZEMIMOGHADAM M, et al. Ensemble learning for glioma patients overall survival prediction using pre-operative MRIs[J]. Phys Med Biol, 2022, 67(24): 245002. |
| [22] |
REN J, ZHAI X, YIN H, et al. Multimodality MRI radiomics based on machine learning for identifying true tumor recurrence and treatment-related effects in patients with postoperative glioma[J]. Neurol Ther, 2023, 12(5): 1729-1743. |
| [23] |
ZHAN T, FENG P, HONG X, et al. An automatic glioma grading method based on multi-feature extraction and fusion[J]. Technol Health Care, 2017, 25(S1): 377-385. |
| [24] |
PATTANAIK B, ANITHA K, RATHORE S, et al. Brain tumor magnetic resonance images classification based machine learning paradigms[J]. Contemp Oncol (Pozn), 2022, 26(4): 268-274. |
 2025, Vol. 23
2025, Vol. 23


