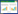文章信息
- 于明鑫, 杨爱明
- YU Mingxin, YANG Aiming
- 基于生物信息学的克罗恩病中炎症相关关键基因的筛选及验证
- Screening and validation of key inflammation-related genes in Crohn's disease based on bioinformatics
- 中国医科大学学报, 2024, 53(4): 324-331
- Journal of China Medical University, 2024, 53(4): 324-331
-
文章历史
- 收稿日期:2023-10-12
- 网络出版时间:2024-04-10 17:52:19
克罗恩病(Crohn’s disease,CD)是一种炎症性肠病(inflammatory bowel disease,IBD),主要表现为胃肠道不同部位的慢性炎症。近年来CD发病率呈上升趋势[1]。目前,CD的病因尚不明确。已有研究[2]表明,遗传、环境、炎症、感染与肠道菌群等因素增加了CD发生和进展的风险。CD不仅表现有肠道损伤,还可导致肠外全身性症状,严重影响患者的生活质量和预后[3]。研究[1]显示,特异性生物标志物可以帮助临床医生在CD的诊断和干预中获益,并在明确治疗反应和预测CD复发方面发挥作用。因此,明确CD发生的分子机制和关键生物标志物对CD的治疗具有重要意义。目前已发现100多个与CD相关的基因。MCGOVERN等[4]研究发现对肠道细菌的炎症反应是CD发病的关键因素,并指出NOD2、IL23R、ATG16L1是CD的强相关基因。而且NOD2/CARD15基因与回肠受累、肠道纤维化狭窄有关[5]。
本研究利用生物信息学方法对CD炎症相关关键基因及信号通路进行分析,筛选并验证CD炎症相关的关键基因,旨在为CD诊断治疗提供有利支持。
1 材料与方法 1.1 数据来源在基因表达综合(gene expression omnibus,GEO)数据库(https://www.ncbi.nlm.nih.gov/geo/)中以“Crohn’s disease”为关键词检索CD表达谱高通量测序/芯片数据。纳入标准:(1)数据集为肠组织样本测序数据;(2)同时含有健康人对照和CD患者信息;(3)数据集含有CD活动指数信息。排除标准:(1)儿童CD;(2)血液样本数据;(3)药物临床试验表达谱数据。获得GSE193677、GSE66407和GSE179285 3个数据集。GSE193677数据集(461例健康人和1 157例CD患者肠组织样本)为分析数据集,GSE66407数据集(99例健康人和103例CD患者肠组织样本)和GSE179285数据集(31例健康人和168例CD患者肠组织样本)为验证数据集。炎症反应相关基因(inflammator response related genes,IRGs)下载自分子特征数据库(molecular signatures database,Msigdb;https://www.gsea-msigdb.org),共200个基因用于后续分析。
1.2 炎症浸润分析利用Python3.80软件pydeseq2包对GSE193677数据集做标准化转换,然后使用GSEApy包的单样本基因集富集分析(single-sample gene set enrichment analysis,ssGSEA)获得CD患者和健康人16种炎症细胞和13种炎症分子通路的富集情况并比较,结果利用plotly包进行分析。
1.3 炎症相关差异表达基因(inflammation-related differentially expressed genes,IRDEGs)筛选利用Python3.80软件pydeseq2包对GSE193677数据集进行差异表达基因(differential expression genes,DEGs)分析,筛选标准:|log2FC|≥1且FDR校正P < 0.05。DEGs与IRGs取交集,获得IRDEGs。利用seaborn包分析IRDEGs在GSE66407数据集中的表达情况。
1.4 IRDEGs的富集分析利用Python3.80软件GSEApy包对IRDEGs进行基因本体(gene ontology,GO)和京都基因和基因组数据库(Kyoto encyclopedia of genes and genomes,KEGG)通路富集分析。
1.5 IRDEGs的蛋白质-蛋白质相互作用(protein-protein interaction,PPI)网络构建和关键IRDEGs筛选利用STRING数据库(http://string-db.org/)对IRDEGs进行PPI分析。PPI网络数据导入至Cytoscape 3.80软件构建网络图,利用Cytoscape软件的cytoHubba插件筛选鉴定关键基因,并对最大团中心性(maximum clique centrality,MCC)、邻域组件中心性(neighborhood component centrality,MNC)、最大邻域组件中心性(maximum neighborhood component centrality,DNMC)、紧密中心性、度中心性和边缘渗透组件(edge percolated component,EPC)6种拟合方法分别获得得分排名前10位的关键基因,取交集获得关键IRDEGs。
1.6 关键IRDEGs与CD活动指数的相关分析GSE193677数据集中包含健康人对照和CD患者的CD活动指数信息。分析GSE193677数据集中关键IRDEGs在5种活动指数(健康,非活动,轻度、中度、重度活动)CD患者中的表达情况。
1.7 关键IRDEGs的验证在GSE66407和GSE179285数据集中,比较关键IRDEGs在健康对照组和CD组、炎症组和非炎症组中的表达情况。
1.8 统计学分析2组比较由scipy包的Welch’s t检验完成,P值经statsmodels包FDR方法校正;多组比较方差齐性检验使用scipy包levene函数。其中,方差齐性的多组比较由statsmodels包的单因素方差分析完成,两两比较P值经statsmodels包TuKey方法校正;方差不齐多组比较由scipy包的Kruskal-Wallis检验完成,两两比较由scipy包的Mann-Whitney U检验完成并经bonferroni方法校正P值。P < 0.05为差异有统计学意义。
2 结果 2.1 CD患者炎症浸润情况结果显示,辅助性T细胞、中性粒细胞、调节性T细胞、树突状细胞(dentritic cells,DC)、巨噬细胞、NK细胞等在CD患者中表达均显著上调(图 1A);13种炎症分子通路均在CD患者中显著上调(图 1B)。
|
| A, inflammatory signaling pathway enrichment; B, inflammatory cell abundance. ** P < 0.01;*** P < 0.001, *P < 0.05. iDC, interdigitating dendritic cell; Tfh, follicular helper T cell; TIL, tumor infiltrating lymphocyte; Treg, regulatory T cells; aDC, actived dendritic cell; pDC, plasmacytoid dendritic cell; Th1, T helper cell 1;Th2, T helper cell 2;NK, natural killer; MHC, major histocompatibility complex; HLA, human leukocyte antigen; IFN, interferon; APC, antigen presenting cell; CCR, c-c motif chemokine receptor. 图 1 GSE193677数据集中CD患者炎症浸润情况 Fig.1 Inflammatory infiltration of CD patients in GSE193677 dataset |
2.2 GSE193677数据集中IRDEGs筛选及表达结果
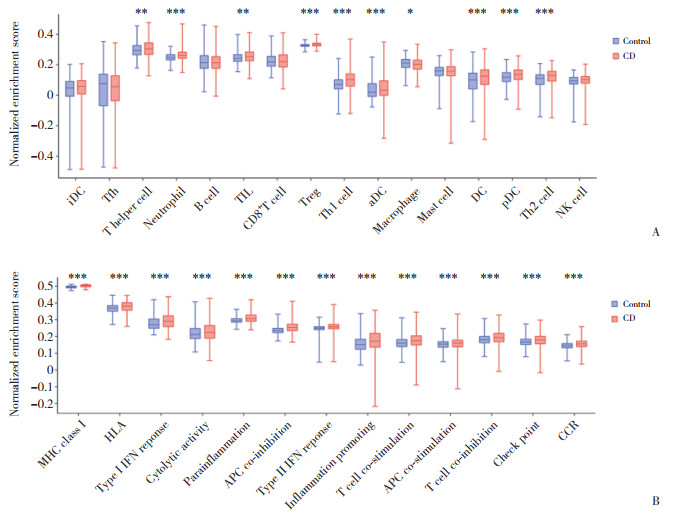
结果显示,共筛选出450个DEGs,其中378个上调基因,72个下调基因。同时从Msigdb数据库中获取200个IRGs。DEGs与IRGs取交集,获得24个IRDEGs。24个IRDEGs表达情况见图 2。
|
| 图 2 GSE193677数据集中24个IRDEGs表达情况 Fig.2 Expression of 24 IRDEGs in GSE193677 dataset |
2.3 IRDEGs富集分析
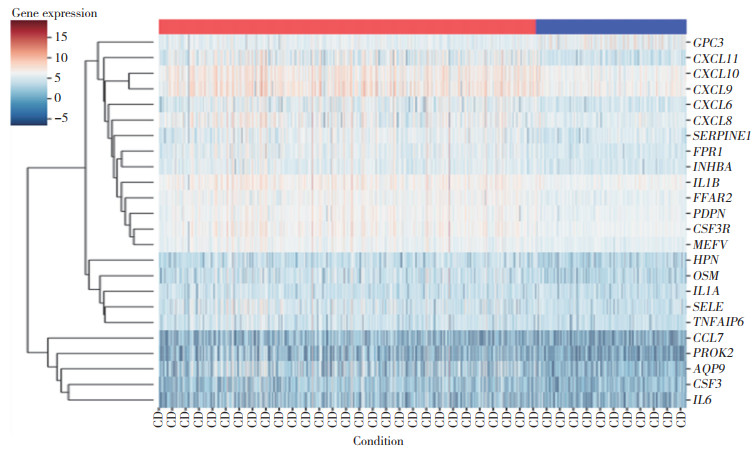
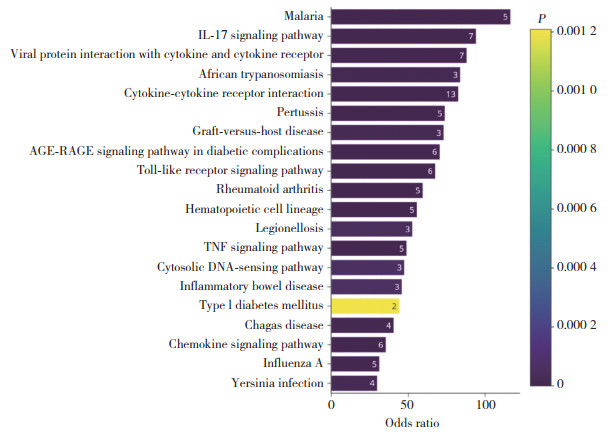
GO分析结果显示,IRDEGs在未成熟T细胞扩增调节、细胞黏附调节、CXCR趋化因子受体结合、丝氨酸蛋白抑制剂复合物等方面明显富集(图 3)。KEGG通路分析显示,IRDEGs在IL-17信号通路、TNF信号通路、细胞因子及其受体相互作用、IBD等信号通路显著富集(图 4)。
|
| A, biological process; B, molecular function; C, cellular component. The numbers in figure represened the number of genes involved. 图 3 IRDEGs的GO富集分析 Fig.3 GO enrichment analysis of IRDEGs |
|
| The numbers in figure represened the number of genes involved. 图 4 IRDEGs的KEGG富集分析结果 Fig.4 KEGG enrichment analysis of IRDEGs |
2.4 PPI网络构建和关键IRDEGs筛选
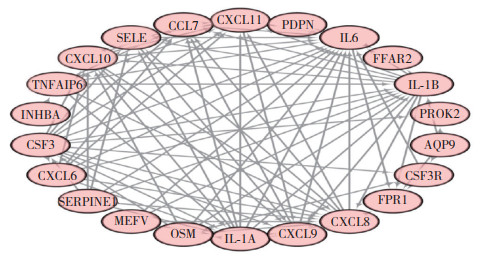
利用STRING数据库构建IRDEGs的PPI网络,见图 5。利用Cytoscape软件cytoHubba插件中的6种拟合方法(MCC、MNC、DNMC、紧密中心性、度中心性和EPC)筛选得分(由高到低排序)前10位基因,见表 1。取交集获得7个关键IRDEGs,包括IL1A、SELE、CXCL11、CSF3、CXCL9、CXCL10和CCL7。
|
| 图 5 PPI网络构建 Fig.5 PPI network construction |
| No. | MCC | MNC | EPC | DMNC | Degree | Closeness |
| 1 | IL1A | IL1A | IL1A | IL1A | IL1A | IL1A |
| 2 | IL1B | IL1B | IL1B | CXCL6 | IL1B | IL1B |
| 3 | CXCL8 | CXCL8 | CXCL8 | SELE | CXCL8 | CXCL8 |
| 4 | SELE | SELE | SELE | CXCL11 | SELE | SELE |
| 5 | CXCL11 | CXCL11 | CXCL11 | CSF3 | CXCL11 | CXCL11 |
| 6 | IL6 | IL6 | IL6 | SERPINE1 | IL6 | IL6 |
| 7 | CSF3 | CSF3 | CSF3 | CXCL10 | CSF3 | CSF3 |
| 8 | CXCL9 | CXCL9 | CXCL9 | CXCL9 | CXCL9 | CXCL9 |
| 9 | CXCL10 | CXCL10 | CXCL10 | CCL7 | CXCL10 | CXCL10 |
| 10 | CCL7 | CCL7 | CCL7 | TNFAIP6 | CCL7 | CCL7 |
2.5 关键IRDEGs与CD活动指数的相关分析
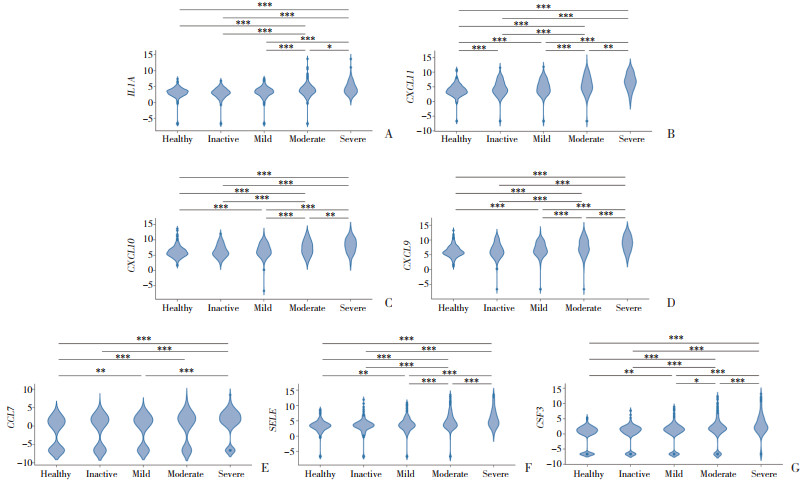
结果显示,7种关键IRDEGs在不同的临床病情活动状态下表达存在差异,并随着病情加重而呈现表达上升的趋势(图 6)。
|
| A, IL1A; B, CXCL11; C, CXCL10; D, CXCL9; E, CCL7; F, SELE; G, CSF3. *** P < 0.001;*P < 0.05;** P < 0.01. 图 6 关键IRDEGs在不同严重程度CD患者的表达水平比较 Fig.6 Comparison of expression levels of key IRDEGs in CD patients with different disease severity |
2.6 关键IRDEGs的验证
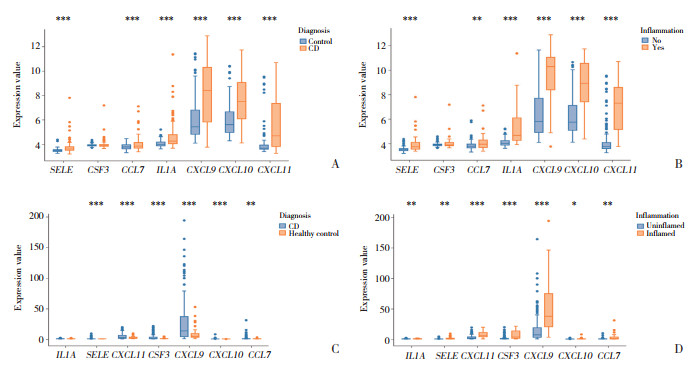
结果显示,GSE66407数据集中,与健康对照组比较,CD组中IL1A、SELE、CXCL11、CXCL9、CXCL10和CCL7表达明显升高(均P < 0.001);与非炎症组比较,炎症组中IL1A、SELE、CXCL11、CXCL9、CXCL10和CCL7表达也明显升高(均P < 0.01)。GSE179285数据集中,与健康对照组比较,CD组SELE、CSF3、CXCL11、CXCL9、CXCL10和CCL7表达均明显升高(均P < 0.01);与非炎症组比较,炎症组7种IRDEGs表达均明显升高(均P < 0.05)。见图 7。
|
| A, B, GSE66407 dataset; C, D, GSE179285 dataset. *** P < 0.001;** P < 0.01;*P < 0.05. 图 7 关键IRDEGs的验证结果 Fig.7 Validation results of key IRDEGs |
3 讨论
生物信息学研究手段有助于拓宽复杂多基因疾病的研究方向,有助于挖掘导致CD的新基因,为CD的发病机制提供新的见解。本研究通过一系列生物信息学分析方法,获得了CD中7个关键IRDEGs,包括IL1A、SELE、CXCL11、CSF3、CXCL9、CXCL10和CCL7;同时对7个关键IRDEGs进行了验证。GO功能富集分析中显示24个IRDEGs主要在未成熟T细胞扩增调节、细胞黏附调节、CXCR趋化因子受体结合、丝氨酸蛋白抑制剂复合物等方面明显富集;KEGG通路分析显示,IRDEGs在IL-17信号通路、TNF信号通路、细胞因子及其受体相互作用、IBD等信号通路显著富集。
多项研究[1]报道了IBD中持续的T细胞炎症激活。CD是促炎细胞因子(IL-12、IL-18、IL-23)导致过度的T辅助细胞(T-helper cell,TH)1和TH17细胞反应引起的。TH1和TH17细胞分泌促炎性细胞因子IL-17、IFN-γ和TNF,这些促炎性细胞因子通过刺激巨噬细胞、内皮细胞和单核细胞产生TNF、IL-1、IL-6、IL-8、IL-12和IL-18而使炎症持续存在[6]。此外,研究[7]显示CD患者上皮内淋巴细胞中IL-17A、IFN-γ和TNF增加。因此,IRDEGs的KEGG通路富集分析结果表明,这些IRDEGs可能通过IL-17信号通路、TNF信号通路、细胞因子及其受体相互作用等途径参与了CD的进展。
IL1A是一种多效细胞因子,参与各种炎症反应、炎症过程和造血。产生IL-23的单核巨噬细胞在IBD发病机制中起着核心作用,近期研究[8]发现,IL-1α/IL-1β是调节产生IL-23的单核细胞的关键细胞因子。本研究结果显示,IL1A表达水平随着CD严重程度加重而升高。因此,IL1A可能在CD发病机制中起着关键作用。SELE基因编码的蛋白存在于细胞因子刺激的内皮细胞中,被认为是导致白细胞在炎症部位聚集的原因[9-10]。目前,关于SELE对CD发生发展的影响尚不明确。
CXCL11、CXCL10、CXCL9均属于趋化因子超家族成员,该家族编码分泌蛋白,参与免疫调节和炎症反应。CXCL11、CXCL10、CXCL9均能与CXCR3结合[11]。其中,CXCL9与T细胞运输有关,CXCL9与CXCR3结合能趋化淋巴细胞。CXCL10蛋白与CXCR3结合可产生多效性作用,包括刺激单核细胞、自然杀伤细胞和T细胞迁移,以及调节黏附分子表达。近期研究[12]发现,CD未来风险比值最高的蛋白是CXCL9,CXCL9是唯一与其他CD风险生物标志物显著相关且作用方向一致的蛋白。此外,CXCR3配体(CXCL11和CXCL9)与回肠结肠切除术后CD复发密切相关[13]。以往也有研究[14]指出,IBD患者黏膜上皮细胞中TLR3能介导CXCL10释放,并在活动期IBD患者中观察到CXCL10基因在结肠黏膜中表达升高。因此,可以认为CXCL11、CXCL10、CXCL9与CD发生发展关系密切。
CSF3蛋白能控制粒细胞的产生、分化和功能。研究[15]发现与缓解期的结肠活检结果相比,活动期的结肠活检结果显示CSF3转录水平明显增加。另一项研究[16]显示,CSF3可以作为抗TNF治疗失败的CD患者潜在的治疗靶点。本研究结果显示CSF3是CD的关键IRDEGs,同时明确了CSF3表达水平随着CD疾病严重程度增高而升高。CCL7基因编码单核细胞趋化蛋白3,是一种在炎症和转移过程中吸引巨噬细胞的分泌趋化因子。目前,关于CCL7对CD疾病发生发展的影响尚不明确。
综上所述,本研究通过生物信息学方法,筛选并验证了与CD密切相关的关键IRDEGs(IL1A、SELE、CXCL11、CSF3、CXCL9、CXCL10和CCL7)。为CD的治疗和诊断研究提供了理论支持。本研究属于生物信息学研究,未对获得的关键基因进行实验验证,今后将收集CD患者和健康人的组织和血液样本,进一步对7个关键IRDEGs进行论证。
| [1] |
RODA G, CHIEN NG S, KOTZE PG, et al. Crohn's disease[J]. Nat Rev Dis Primers, 2020, 6(1): 22. DOI:10.1038/s41572-020-0156-2 |
| [2] |
TORRES J, MEHANDRU S, COLOMBEL JF, et al. Crohn's disease[J]. Lancet, 2017, 389(10080): 1741-1755. DOI:10.1016/S0140-6736(16)31711-1 |
| [3] |
ANANTHAKRISHNAN AN, BERNSTEIN CN, ILIOPOULOS D, et al. Environmental triggers in IBD: a review of progress and evidence[J]. Nat Rev Gastroenterol Hepatol, 2018, 15(1): 39-49. DOI:10.1038/nrgastro.2017.136 |
| [4] |
MCGOVERN DPB, KUGATHASAN S, CHO JH. Genetics of inflammatory bowel diseases[J]. Gastroenterology, 2015, 149(5): 1163-1176.e2. DOI:10.1053/j.gastro.2015.08.001 |
| [5] |
GAJENDRAN M, LOGANATHAN P, CATINELLA AP, et al. A comprehensive review and update on Crohn's disease[J]. Dis Mon, 2018, 64(2): 20-57. DOI:10.1016/j.disamonth.2017.07.001 |
| [6] |
UHLIG HH, POWRIE F. Translating immunology into therapeutic concepts for inflammatory bowel disease[J]. Annu Rev Immunol, 2018, 36: 755-781. DOI:10.1146/annurev-immunol-042617-053055 |
| [7] |
REGNER EH, OHRI N, STAHLY A, et al. Functional intraepithelial lymphocyte changes in inflammatory bowel disease and spondyloarthritis have disease specific correlations with intestinal microbiota[J]. Arthritis Res Ther, 2018, 20(1): 149. DOI:10.1186/s13075-018-1639-3 |
| [8] |
ASCHENBRENNER D, QUARANTA M, BANERJEE S, et al. Deconvolution of monocyte responses in inflammatory bowel disease reveals an IL-1 cytokine network that regulates IL-23 in genetic and acquired IL-10 resistance[J]. Gut, 2021, 70(6): 1023-1036. DOI:10.1136/gutjnl-2020-321731 |
| [9] |
ABBASSI O, KISHIMOTO TK, MCINTIRE LV, et al. E-selectin supports neutrophil rolling in vitro under conditions of flow[J]. J Clin Invest, 1993, 92(6): 2719-2730. DOI:10.1172/JCI116889 |
| [10] |
JANI PK, SCHWANER E, KAJDáCSI E, et al. Complement MASP-1 enhances adhesion between endothelial cells and neutrophils by up-regulating E-selectin expression[J]. Mol Immunol, 2016, 75: 38-47. DOI:10.1016/j.molimm.2016.05.007 |
| [11] |
KARIN N, WILDBAUM G. The role of chemokines in shaping the balance between CD4+ T cell subsets and its therapeutic implications in autoimmune and cancer diseases[J]. Front Immunol, 2015, 6: 609. DOI:10.3389/fimmu.2015.00609 |
| [12] |
LEIBOVITZH H, LEE SH, GARAY JAR, et al. Immune response and barrier dysfunction-related proteomic signatures in preclinical phase of Crohn's disease highlight earliest events of pathogenesis[J]. Gut, 2023, 72(8): 1462-1471. DOI:10.1136/gutjnl-2022-328421 |
| [13] |
WALSHE M, NAYERI S, JI JY, et al. A role for CXCR3 ligands as biomarkers of post-operative Crohn's disease recurrence[J]. J Crohns Colitis, 2022, 16(6): 900-910. DOI:10.1093/ecco-jcc/jjab186 |
| [14] |
OSTVIK AE, GRANLUND AVB, BUGGE M, et al. Enhanced expression of CXCL10 in inflammatory bowel disease: potential role of mucosal Toll-like receptor 3 stimulation[J]. Inflamm Bowel Dis, 2013, 19(2): 265-274. DOI:10.1002/ibd.23034 |
| [15] |
SÖDERMAN J, BERGLIND L, ALMER S. Gene expression-genotype analysis implicates GSDMA, GSDMB, and LRRC3C as contributors to inflammatory bowel disease susceptibility[J]. Biomed Res Int, 2015, 2015: 834805. DOI:10.1155/2015/834805 |
| [16] |
YAO Y, YANG L, ZHANG Z, et al. Identification of targets for subsequent treatment of Crohn's disease patients after failure of anti-TNF therapy[J]. J Inflamm Res, 2023, 16: 4617-4631. DOI:10.2147/JIR.S422881 |
 2024, Vol. 53
2024, Vol. 53