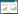文章信息
- 王馨羚, 肖琼, 付昱, 孙晨蕊, 李泓池, 张步晨, 宋永喜
- WANG Xinling, XIAO Qiong, FU Yu, SUN Chenrui, LI Hongchi, ZHANG Buchen, SONG Yongxi
- CCNA2在泛癌中的表达及其与预后和免疫浸润的关系
- Expression of CCNA2 in pan-cancer and its association with prognosis and immune infiltration
- 中国医科大学学报, 2023, 52(9): 791-797, 804
- Journal of China Medical University, 2023, 52(9): 791-797, 804
-
文章历史
- 收稿日期:2022-11-23
- 网络出版时间:2023-08-31 09:37:33
细胞周期蛋白A2(cyclin A2,CCNA2)是细胞周期蛋白家族中高度保守的成员之一,同时也是细胞周期的关键因子,与调节细胞周期和细胞衰老以及DNA复制和有丝分裂有关。作为一种新型致癌基因,CCNA2在调节癌细胞生长和凋亡中起作用,敲低CCNA2可以减缓细胞周期进程并诱导细胞凋亡,抑制相关癌细胞的生长,其在癌症转化和进展中也有潜在作用[1-2]。近年研究[3]表明,CCNA2过表达与某些癌症的预后不良有关,如肺腺癌、胰腺癌、乳腺癌、宫颈癌等。
越来越多的肿瘤研究[4]发现,CCNA2可作为肿瘤免疫微环境的潜在标志物。CCNA2在多种癌症中的表达与部分肿瘤杀伤免疫细胞的活化存在显著相关性。以结肠癌为例,CCNA2在肿瘤组织中表达较高,且在肿瘤微环境中正向调节肿瘤杀伤免疫细胞,有助于激活CD8+ T细胞,使CD8+ T细胞发挥对癌细胞的杀伤作用,从而延长结肠癌患者的生存时间[5]。此外,CCNA2能够调节巨噬细胞的功能,CCNA2高表达的肿瘤中M2型巨噬细胞表达水平更高[6]。因此,本研究综合分析了33种肿瘤中CCNA2的表达水平,旨在探究CCNA2与肿瘤预后及免疫浸润的相关性,从而为癌症治疗提供新的思路。
1 材料与方法 1.1 资料来源从UCSC Xena数据库(http://xena.ucsc.edu/)下载癌症基因组图谱计划(The Cancer Genome Atlas,TCGA)数据库中33种肿瘤的RNA测序数据(10 557例)、体细胞突变数据(10 380例)以及临床生存数据(13 694例)。通过R软件进行数据整理,根据预后随访信息的完整程度筛选临床生存数据,将其中缺失生存状态、生存时间为0的患者信息剔除。
1.2 基因表达分析从TCGA数据库中提取33种肿瘤中CCNA2表达的每百万测序碱基中基因外显子每千个碱基长度中所包含的测序片段数(fragments per kilobase of exon model per million mapped reads,FPKM),形成表达矩阵,通过患者ID匹配表达矩阵和临床信息。为了比较癌症样本和相邻正常样本的基因表达水平,对表达数据进行Wilcoxon检验。
1.3 生存分析使用单因素Cox回归模型,分析CCNA2的表达水平与总体生存率的相关性,计算95%CI的风险比。
1.4 免疫浸润分析肿瘤与免疫系统相互作用的数据库(tumor-immune system interactions database,TISIDB,http://cis.hku.hk/TISIDB/)[7]是一个肿瘤和免疫系统交互的门户网站。本研究利用TISIDB数据库分析不同类型肿瘤中CCNA2表达与肿瘤浸润淋巴细胞的相关性。
1.5 功能富集分析基因表达谱数据动态分析(Gene Expression Profiling Interactive Analysis,GEPIA)数据库(http://gepia.cancer-pku.cn/)[8]是一个基于TCGA和GTEx数据库中的样本进行基因表达分析的在线网站。本研究通过GEPIA2中的“Similar Genes Detection”筛选出相关性排名前100的基因,绘制前6个代表性基因的散点图。注释、可视化和集成发现的数据库(Database for Annotation,Visualization,and Integrated Discovery,DAVID,https://david.ncifcrf.gov/)[9]是一个常见的生物信息学数据库,用于基因列表功能注释和富集分析。本研究通过DAVID数据库对CCNA2共表达基因进行基因本体(Gene Ontology,GO)和京都基因与基因组数据库(Kyoto Encyclopedia of Genes and Genomes,KEGG)富集分析,GO通过生物过程、细胞组分和分子功能3个层面进行分析,KEGG对基因集进行信号通路分析。
1.6 统计学分析采用R软件(版本4.1.2)进行统计分析。应用Wilcoxon检验进行差异分析,应用Spearman相关进行相关性分析,应用log-rank检验进行生存分析。P < 0.05为差异有统计学意义。
2 结果 2.1 CCNA2在正常组织和肿瘤组织中的表达水平本研究纳入TCGA数据库中33种肿瘤组织中CCNA2的表达数据,其中24种肿瘤组织有相应的正常组织,作为对照。利用配对Wilcoxon检验分析肿瘤组织与正常组织中CCNA2的表达差异(图 1),结果表明,CCNA2在17种肿瘤组织中的表达水平显著高于正常组织(P < 0.001),如膀胱尿路上皮癌、乳腺浸润癌、胆管癌、结肠癌。
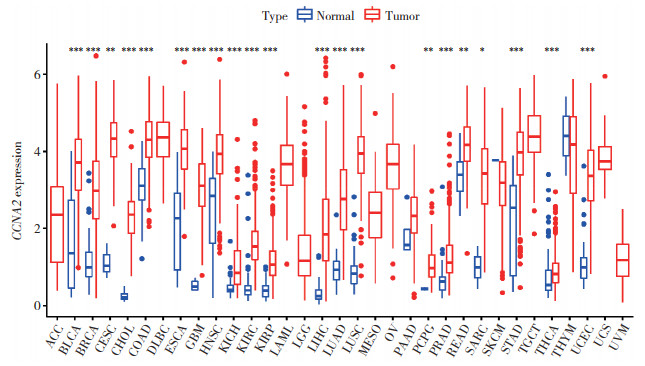
|
| *P < 0.05;**P < 0.01;***P < 0.001. ACC, adrenocortical carcinoma; BLCA, bladder urothelial carcinoma; BRCA, breast invasive carcinoma; CESC, cervical cancer; CHOL, cholangiocarcinoma; COAD, colon adenocarcinoma; DLBC, large B-cell lymphoma; ESCA, esophageal carcinoma; GBM, glioblastoma; HNSC, head and neck squamous cell carcinoma; KICH, kidney chromophobe; KIRC, kidney renal clear cell carcinoma; KIRP, kidney renal papillary cell carcinoma; LAML, acute myeloid leukemia; LGG, lower grade glioma; LIHC, liver cancer; LUAD, lung adenocarcinoma; LUSC, lung squamous cell carcinoma; MESO, mesothelioma; OV, ovarian cancer; PAAD, pancreatic cancer; PCPG, pheochromocytoma and paraganglioma; PRAD, prostate cancer; READ, rectal cancer; SARC, sarcoma; SKCM, melanoma; STAD, stomach cancer; TGCT, testicular cancer; THCA, thyroid cancer; THYM, thymoma; UCEC, endometrioid cancer; UCS, uterine carcinosarcoma; UVM, ocular melanoma. 图 1 CCNA2在33种肿瘤组织和正常组织中的表达水平 Fig.1 Expression levels of CCNA2 in 33 cancer and normal tissues |
2.2 CCNA2表达与临床分期的相关性
分析不同肿瘤中CCNA2表达水平与临床分期的相关性(图 2),结果表明,在食管癌、肾嫌色细胞癌、肾细胞癌、肾乳头状细胞癌、肝细胞性肝癌、肺腺癌、肺鳞状细胞癌、睾丸生殖细胞肿瘤、肾上腺皮质癌、乳腺浸润癌中,CCNA2表达随着TNM分期的增加而升高;而在皮肤黑色素瘤和结肠癌中,CCNA2表达随TNM分期的增加而降低。
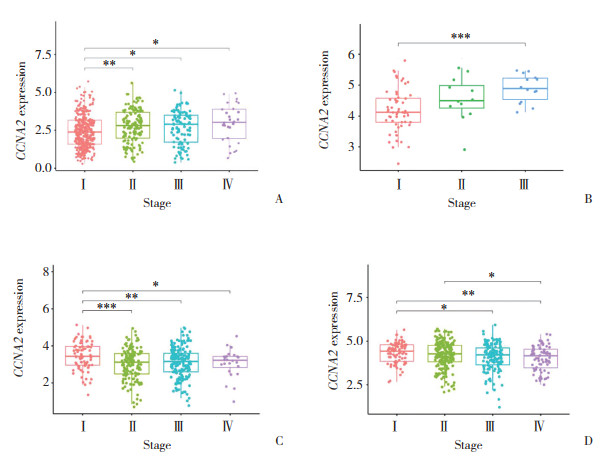
2.3 CCNA2在泛癌中的预后价值
通过Cox比例风险模型分析得出CCNA2在33种肿瘤中的预后森林图(图 3),结果表明,CCNA2高表达是多种肿瘤的危险因素,包括肾上腺皮质癌、肾嫌色细胞癌、肾细胞癌、肾乳头状细胞癌、脑低级别胶质瘤、肝细胞癌、肺腺癌、间皮瘤和胰腺癌(P < 0.001),而CCNA2高表达是胸腺瘤(P = 0.012)的保护因素。
2.4 CCNA2表达与免疫浸润的相关性
肿瘤浸润免疫细胞的数量和活性状态是肿瘤预后的重要预测指标[10]。应用TISIDB数据库分析CCNA2的表达量与肿瘤浸润淋巴细胞的相关性。热图(图 4A)显示,CCNA2与ACT‐CD4淋巴细胞有较强的相关性。其中,在肾细胞癌(r = 0.705,P < 0.001)、脑低级别胶质瘤(r = 0.704,P < 0.001)、肺腺癌(r = 0.664,P < 0.001)中CCNA2与ACT‐CD4淋巴细胞显著相关(图 4B~4D)。这表明CCNA2可能影响这些免疫检查点(immune checkpoint,ICP)基因的表达,并有望成为理想的免疫治疗靶点。
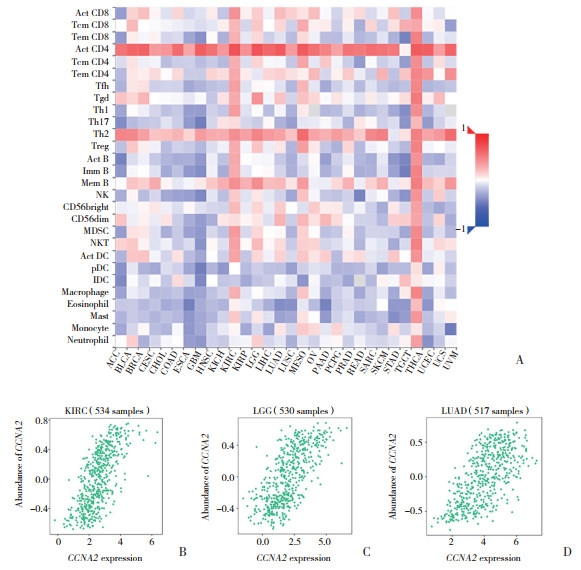
|
| A, heatmap of CCNA2 expression and immune infiltrating cells in 33 cancer types; B, correlation between CCNA2 expression and lymphocyte infiltration in KIRC; C, correlation between CCNA2 expression and lymphocyte infiltration in LGG; D, correlation between CCNA2 expression and lymphocyte infiltration in LUAD. Abbreviations as in Fig. 1. 图 4 TISIDB数据库中CCNA2表达与淋巴细胞的相关性 Fig.4 Correlation between CCNA2 expression and lymphocyte infiltration in TISIDB database |
2.5 CCNA2表达与ICP基因的相关性
ICP基因异常表达是众所周知的肿瘤免疫逃逸机制[11]。本研究分析了CCNA2表达与ICP基因的相关性(图 5),结果发现,47个ICP基因在多种肿瘤中与CCNA2表达强相关。特别是在甲状腺癌中,共46个ICP基因与CCNA2表达有关(P < 0.05)。提示CCNA2可协调这些ICP基因的活性,并作为理想的免疫治疗靶点。
2.6 CCNA2表达与肿瘤突变负荷和微卫星不稳定性的相关性
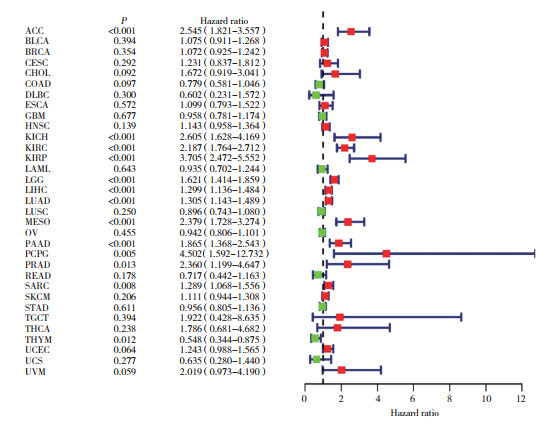
肿瘤突变负荷和微卫星不稳定性经常在肿瘤免疫治疗研究中作为预测性免疫标志物的指标[12]。本研究评估了肿瘤突变负荷和微卫星不稳定性与CCNA2表达的相关性。通过分析CCNA2在不同肿瘤中的突变数据,结果发现,在15种肿瘤中,CCNA2的表达水平与肿瘤突变负荷呈正相关,而在胸腺瘤中与肿瘤突变负荷呈负相关(图 6A);在9种肿瘤中,CCNA2的表达水平与微卫星不稳定性呈正相关,而在弥漫性大B细胞淋巴瘤、急性髓系白血病、皮肤黑色素瘤中与微卫星不稳定性呈负相关(图 6B)。
2.7 CCNA2表达与功能富集分析
使用GEPIA数据库分析100个CCNA2的相关基因,其中6个代表性靶基因(MAD2L1、PLK1、PLK4、KIF11、CCNB2、BUB1)的表达量与CCNA2的表达量在多种肿瘤中显著正相关(图 7A~7F)。应用R软件对CCNA2及其相关基因进行富集分析(图 7G~7H)。GO分析结果表明,CCNA2及其相关基因在肿瘤中主要与细胞分裂、细胞核、蛋白质结合等功能有关。KEGG分析结果表明,基因明显富集在细胞周期、卵母细胞减数分裂、孕酮介导的卵母细胞成熟等信号通路。

|
| A to F, six genes co-expressed with CCNA2 were identified from the Gene Expression Profiling Interactive Analysis database; G, Gene Ontology analysis of CCNA2 and its co-expressed genes; H, Kyoto Encyclopedia of Genes and Genomes analysis of CCNA2 and its co-expressed genes. 图 7 CCNA2在肿瘤中相关基因的功能富集分析 Fig.7 Functional enrichment analysis of CCNA2-related genes in tumors |
3 讨论
CCNA2是细胞周期蛋白家族中的成员之一。在哺乳动物细胞中,可通过与不同的细胞周期蛋白依赖性激酶的结合,参与控制G2/M期细胞周期转变和S期进展。研究[13]表明,CCNA2表达是肺腺癌、肺鳞状细胞癌和肾细胞癌预后模型的重要组成部分,与多种差异表达蛋白相互作用,被认为是多种肿瘤的核心基因。本研究通过对TCGA数据库的33种肿瘤数据进行统计分析,发现CCNA2在不同肿瘤中的表达存在差异,且多种CCNA2高表达的肿瘤预后较差,这说明CCNA2可作为一个肿瘤临床预后指标。
近年的研究表明,CCNA2在肿瘤免疫微环境中发挥重要作用。肿瘤微环境是肿瘤生长、进展和转移的关键调节器,主要包括免疫细胞、成纤维细胞、淋巴细胞等细胞和相关分子物质。肿瘤免疫微环境是免疫治疗的重要生物标志物,与肿瘤免疫逃逸机制息息相关[10]。研究[3]表明,CCNA2表达与CD4+ T细胞浸润显著相关,可能参与肿瘤免疫微环境的重塑,为肿瘤转移和进展奠定基础。此外,XU等[6]的研究证实了CCNA2可以激活巨噬细胞,从而增强肿瘤免疫浸润水平,提示CCNA2在免疫治疗中具有重要作用。本研究通过对CCNA2表达与免疫浸润进行相关性研究,发现CCNA2表达与肿瘤免疫微环境具有相关性,可作为肿瘤免疫治疗的一个潜在靶点。
肿瘤突变负荷和微卫星不稳定性是2种被广泛研究的生物标志物,可被用于肿瘤免疫治疗研究中。研究[14]表明,低肿瘤突变负荷的颈部鳞状细胞癌患者比高肿瘤突变负荷患者的预后更好,肿瘤突变负荷可影响B细胞和CD4+ T细胞浸润,说明肿瘤突变负荷可能对肿瘤患者的预后和免疫浸润有一定影响。高微卫星不稳定性可发生于多种肿瘤中,如子宫内膜癌、结直肠癌、胃癌等。随着微卫星不稳定性检测技术和免疫抑制剂在肿瘤治疗中的发展,高微卫星不稳定性肿瘤患者对免疫治疗反应良好[15]。本研究通过对基因突变数据进行分析,发现CCNA2表达在多种肿瘤中与肿瘤突变负荷和微卫星不稳定性具有相关性。本研究为CCNA2与肿瘤免疫浸润的相关性提供了有利的线索。
本研究分析了多种肿瘤中CCNA2与预后、免疫浸润、肿瘤突变负荷和微卫星不稳定等因素的相关性。虽然本研究对于CCNA2的泛癌研究结合了多个数据库中的信息,但仍具有局限性:(1)本研究仍停留在理论层面上,且得到的分析结果可能存在系统误差,仍需通过实验进一步验证;(2)本研究发现CCNA2的表达水平可能与肿瘤的免疫微环境和生存率相关,但仍不确定CCNA2是否通过免疫途径影响生存率。
综上所述,本研究阐明了CCNA2的表达水平与多种肿瘤的预后有关,且与肿瘤中不同程度的免疫浸润有关,进一步揭示了CCNA2作为肿瘤免疫治疗靶点生物标志物的潜力。因此,进行CCNA2与肿瘤免疫微环境的前瞻性研究具有重要意义,能够为未来的免疫治疗提供新的依据。
| [1] |
GAN Y, LI Y, LI T, et al. CCNA2 acts as a novel biomarker in regulating the growth and apoptosis of colorectal cancer[J]. Cancer Manag Res, 2018, 10: 5113-5124. DOI:10.2147/CMAR.S176833 |
| [2] |
HUO X, SUN H, CAO D, et al. Identification of prognosis markers for endometrial cancer by integrated analysis of DNA methylation and RNA-Seq data[J]. Sci Rep, 2019, 9(1): 9924. DOI:10.1038/s41598-019-46195-8 |
| [3] |
LU Y, SU F, YANG H, et al. E2F1 transcriptionally regulates CCNA2 expression to promote triple negative breast cancer tumorigenicity[J]. Cancer Biomark, 2022, 33(1): 57-70. DOI:10.3233/CBM-210149 |
| [4] |
WANG Y, ZHONG Q, LI Z, et al. Integrated profiling identifies CCNA2 as a potential biomarker of immunotherapy in breast cancer[J]. Onco Targets Ther, 2021, 14: 2433-2448. DOI:10.2147/OTT.S296373 |
| [5] |
LI J, ZHOU L, LIU Y, et al. Comprehensive analysis of cyclin family gene expression in colon cancer[J]. Front Oncol, 2021, 11: 674394. DOI:10.3389/fonc.2021.674394 |
| [6] |
XU Y, WU G, LI J, et al. Screening and identification of key biomar-kers for bladder cancer: a study based on TCGA and GEO data[J]. Biomed Res Int, 2020, 2020: 8283401. DOI:10.1155/2020/8283401 |
| [7] |
RU B, WONG CN, TONG Y, et al. TISIDB: an integrated repository portal for tumor-immune system interactions[J]. Bioinformatics, 2019, 35(20): 4200-4202. DOI:10.1093/bioinformatics/btz210 |
| [8] |
TANG Z, KANG B, LI C, et al. GEPIA2:an enhanced web server for large-scale expression profiling and interactive analysis[J]. Nucleic Acids Res, 2019, 47(W1): W556-W560. DOI:10.1093/nar/gkz430 |
| [9] |
SHERMAN BT, HAO M, QIU J, et al. DAVID: a web server for functional enrichment analysis and functional annotation of gene lists (2021 update)[J]. Nucleic Acids Res, 2022, 50(W1): W216-W221. DOI:10.1093/nar/gkac194 |
| [10] |
PANSY K, UHL B, KRSTIC J, et al. Immune regulatory processes of the tumor microenvironment under malignant conditions[J]. Int J Mol Sci, 2021, 22(24): 13311. DOI:10.3390/ijms222413311 |
| [11] |
WU SY, LIN KC, LAWAL B, et al. MXD3 as an onco-immunolo-gical biomarker encompassing the tumor microenvironment, disease staging, prognoses, and therapeutic responses in multiple cancer types[J]. Comput Struct Biotechnol J, 2021, 19: 4970-4983. DOI:10.1016/j.csbj.2021.08.047 |
| [12] |
MERINO DM, MCSHANE LM, FABRIZIO D, et al. Establishing guidelines to harmonize tumor mutational burden (TMB): in silico assessment of variation in TMB quantification across diagnostic platforms: phase Ⅰ of the Friends of Cancer Research TMB Harmonization Project[J]. J Immunother Cancer, 2020, 8(1): e000147. DOI:10.1136/jitc-2019-000147 |
| [13] |
CHEN S, ZHAO Z, WANG X, et al. The predictive competing endogenous RNA regulatory networks and potential prognostic and immunological roles of cyclin A2 in pan-cancer analysis[J]. Front Mol Biosci, 2022, 9: 809509. DOI:10.3389/fmolb.2022.809509 |
| [14] |
ZHANG L, LI B, PENG Y, et al. The prognostic value of TMB and the relationship between TMB and immune infiltration in head and neck squamous cell carcinoma: a gene expression-based study[J]. Oral Oncol, 2020, 110: 104943. DOI:10.1016/j.oraloncology.2020.104943 |
| [15] |
LI K, LUO H, HUANG L, et al. Microsatellite instability: a review of what the oncologist should know[J]. Cancer Cell Int, 2020, 20: 16. DOI:10.1186/s12935-019-1091-8 |
 2023, Vol. 52
2023, Vol. 52