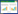文章信息
- 刘烨妹, 李梦玲, 吴胜男, 李思宇, 赵闯
- LIU Yemei, LI Mengling, WU Shengnan, LI Siyu, ZHAO Chuang
- 肺腺癌免疫微环境特征基因对患者生存时间及ICB疗效的预测价值
- Predictive value of characteristic genes of tumor immune microenvironment on survival time and immune checkpoint inhibitor efficacy in patients with lung adenocarcinoma
- 中国医科大学学报, 2023, 52(5): 438-446
- Journal of China Medical University, 2023, 52(5): 438-446
-
文章历史
- 收稿日期:2022-11-12
- 网络出版时间:2023-05-19 15:13:54
2. 中国医科大学附属第一医院临床流行病学与循证医学教研室,沈阳 110001;
3. 中国医科大学附属第一医院肿瘤内科二病房,沈阳 110001;
4. 中国医科大学附属第一医院全科医学教研室,沈阳 110001;
5. 中国医科大学附属盛京医院普通外科,沈阳 110004
2. Department of Clinical Epidemiology and Evidence-based Medicine, The First Hospital of China Medical University, Shenyang 110001, China;
3. Department of Oncology, The First Hospital of China Medical University, Shenyang 110001, China;
4. Department of General Medicine, The First Hospital of China Medical University, Shenyang 110001, China;
5. Department of General Surgery, Shengjing Hospital of China Medical University, Shenyang 110004, China
肺腺癌是最常见的肺癌类型,约占非小细胞肺癌的50%。早期肺腺癌往往没有明显症状,因此很难发现。许多患者确诊时已是晚期,严重影响患者的生存质量。目前,对于驱动基因突变的治疗仅适用于少数肺腺癌患者,由于肿瘤异质性和易产生耐药性等因素,其治疗效果不佳或者难以维持。免疫治疗已经在肺腺癌治疗中取得了很大的进展,但是并非所有患者都能够从免疫治疗中受益[1]。肿瘤免疫微环境(tumor immune microenvironment,TME)是指肿瘤细胞周围的组织环境,包括免疫细胞、炎症细胞、血管、基质细胞、生长因子等多种成分以及它们之间的相互作用。TME中的免疫细胞类型、数量以及它们与肿瘤细胞之间的相互作用,都会对肺腺癌的转移和复发产生影响[2]。因此,探究肺腺癌TME具有重要意义。
免疫检查点抑制剂(immune checkpoint inhibitors,ICB)治疗是一种新型的肺腺癌治疗方法,它是通过抑制免疫检查点分子(PD-1、PD-L1等)来增强免疫系统对肺腺癌的攻击能力。肺腺癌细胞会利用免疫检查点分子来逃避免疫系统的攻击,使得免疫细胞无法识别和攻击癌细胞,从而导致肺腺癌进展。ICB通过阻止免疫检查点分子的作用使免疫细胞能够再次识别和攻击癌细胞,从而抑制肺腺癌进展[3]。因此,明确肺腺癌的TME可以为肺腺癌患者的治疗和预后评估提供准确的依据。本研究利用生物信息学探讨肺腺癌TME特征基因以及相关基因对患者生存时间及ICB疗效的预测价值,构建免疫治疗以及预测免疫治疗疗效的相关基因调控网络。
1 材料与方法 1.1 数据采集和预处理使用2个具有ICB治疗信息的非小细胞肺癌公开测序数据集:(1)基因表达综合(Gene Expression Omnibus,GEO)数据库中GSE136961微阵列数据集(https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE136961);(2)癌症基因组图谱(The Cancer Genome Atlas,TCGA)数据库中肺腺癌患者的数据样本(TCGA-LUAD测序数据集,https://portal.gdc.cancer.gov/projects/TCGA-LUAD)。使用Oncomine Immune Response Research Assay筛选出与免疫应答相关的基因并下载以便进一步分析。
使用筛选出的免疫应答相关基因对TCGA-LUAD测序数据集进行基因表达的非负矩阵分解(non-negative matrix factorization,NMF)聚类分析,通过Kaplan-Meier生存分析绘制聚类分析结果的生存曲线,然后使用基因富集分析(Gene Set Enrichment Analysis,GSEA)对C1、C2 2种免疫亚型进行活性评分。
1.2 免疫浸润分析应用R语言中ESTIMATE [4]包来估计C1、C2 2种免疫亚型的肿瘤纯度(样本中肿瘤细胞的比例);TIDE [5]包进行免疫应答评分(TIDE评分)来预测潜在的ICB疗效,评分较低提示免疫应答效果较好;maftools [6]包进行肿瘤突变负荷(tumor mutational burden,TMB)分析以及基因组DNA拷贝数变异(copy number variations,CNV)分析。
1.3 转录调控网络分析加权基因共表达网络分析(weighted gene co-expression network analysis,WGCNA)[7]可以将基因表达数据转化为共表达网络来分析基因间的关系。WGCNA将相似表达模式的基因分组,并对它们进行功能注释。利用WGCNA对GSE136961微阵列数据集中筛选出的免疫应答相关基因进行分析,找到与PD-1免疫应答最相关的基因模块,同时对TCGA-LUAD测序数据集中筛选出的免疫应答相关基因进行批量生存分析,取两者交集,使用R语言中ClusterProfile[8]包进行基因本体(Gene Onotology,GO)富集分析和京都基因与基因组数据库(Kyoto Encyclopedia of Genes and Genomes,KEGG)通路分析,利用Cytoscape的regulon[9]模块进行转录调控网络分析。
1.4 统计学分析采用SPSS 25.0统计软件绘制Kaplan-Meier曲线,log-rank检验方法计算P值并进行生存分析,P < 0.05为差异有统计学意义。
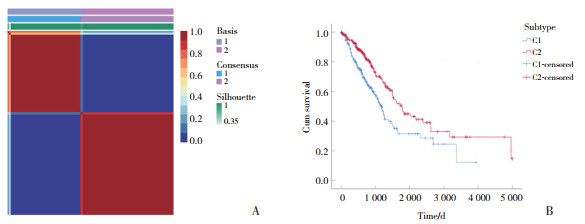
2 结果 2.1 免疫亚型聚类分析共获得395个免疫应答相关基因。将395个免疫应答相关基因作为输入数据,对来自TCGA-LUAD测序数据集中的510例原发性肺腺癌患者进行NMF聚类,获得C1和C2 2种免疫亚型(图 1A)。通过Kaplan-Meier生存分析绘制C1、C2 2种免疫亚型患者的生存曲线。结果显示,与C1免疫亚型患者比较,C2亚型患者具有更好的生存预后(图 1B)。GSEA分析结果显示,C2免疫亚型相对于C1免疫亚型表现出更高的得分(图 2),可见C2亚型患者的免疫系统更加活跃,并且对于肿瘤免疫更敏感,这也可能是C2亚型患者具有更好生存预后的原因之一。
|
| 图 1 TCGA-LUAD测序数据集中510例原发性肺腺癌患者NMF聚类分析结果(A)和C1和C2 2种免疫亚型的Kaplan-Meier生存分析(B) Fig.1 NMF clustering results of 510 primary lung adenocarcinoma patients from the TCGA-LUAD sequencing dataset (A) and Kaplan-Meier survival analysis for two immune subtypes, C1 and C2 (B) |
|
| C1 in blue, C2 in red. 图 2 C1、C2 2种免疫亚型的基因集富集分析GSEA分析 Fig.2 Gene set enrichment analysis of the two immune subtypes C1 and C2 GSEA analysis |
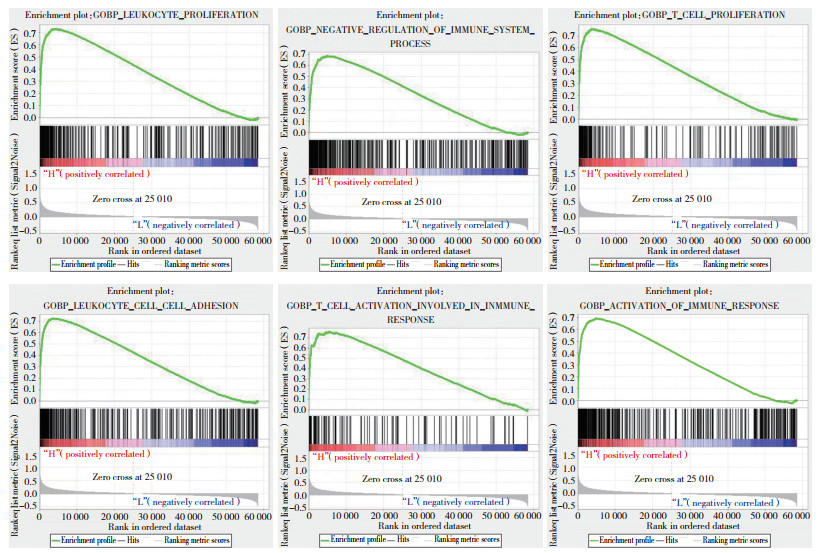
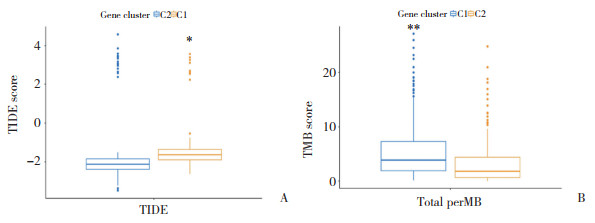
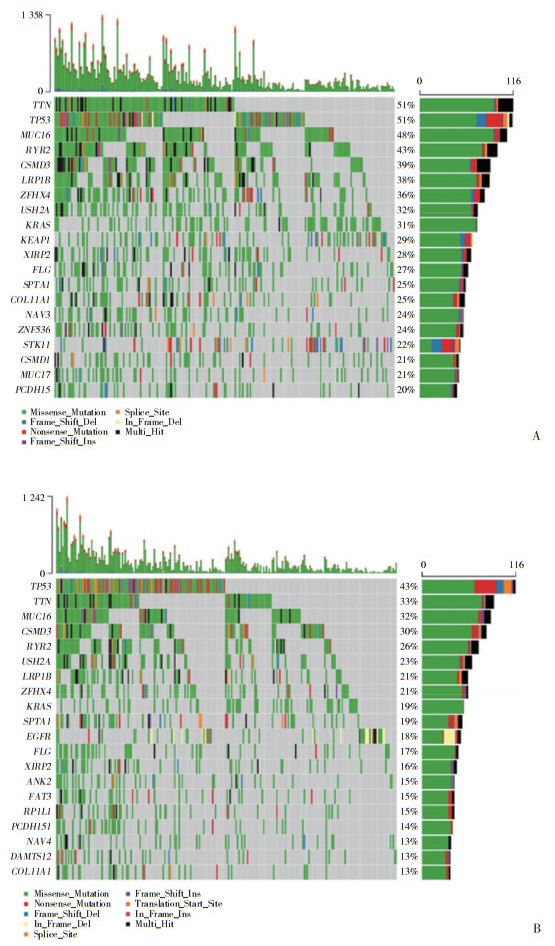
通过SITMATE和CIBERSORT对TCGA-LUAD测序数据量化的结果显示,2种免疫亚型中22种免疫细胞类型相对丰度不同。免疫细胞中巨噬细胞M2比例最高,多数免疫细胞在2种免疫亚型间存在差异分布(图 3A)。为了进一步明确2种免疫亚型在免疫治疗过程中的反应,本研究使用R语言中ESTIMATE包来评估2种免疫亚型的免疫和基质评分。结果显示,C2亚型免疫评分显著高于C1亚型(P < 0.05,图 3B)。利用TIDE算法对TCGA-LUAD测序数据集中的ICB反应进行评估,结果显示,C2亚型的TIDE评分显著低于C1亚型(P < 0.05,图 4A),TIDE评分越低表示免疫系统在肿瘤内部更加活跃,更容易对免疫治疗产生响应,C2亚型的肿瘤具有更好的免疫治疗响应能力。TMB是肿瘤细胞中突变的数量,是衡量肿瘤免疫治疗响应能力的指标,高TMB通常与更好的免疫治疗响应相关。与C2亚型比较,C1亚型具有更高的TMB(Wilcoxon秩和检验,P < 0.001,图 4B),说明C1亚型可能对ICB治疗亦具有良好的响应能力。但是与C2亚型相比,C1亚型TIDE评分相对较高,这意味着C1亚型的肿瘤内部免疫系统相对不活跃,可能需要更多的免疫刺激来激发对免疫治疗的响应。另外,CNV分析结果显示,2种免疫亚型中鉴定出20个显着突变基因,其中TTN、TP53、MUC16在C1亚型中突变频率更高。见图 5。
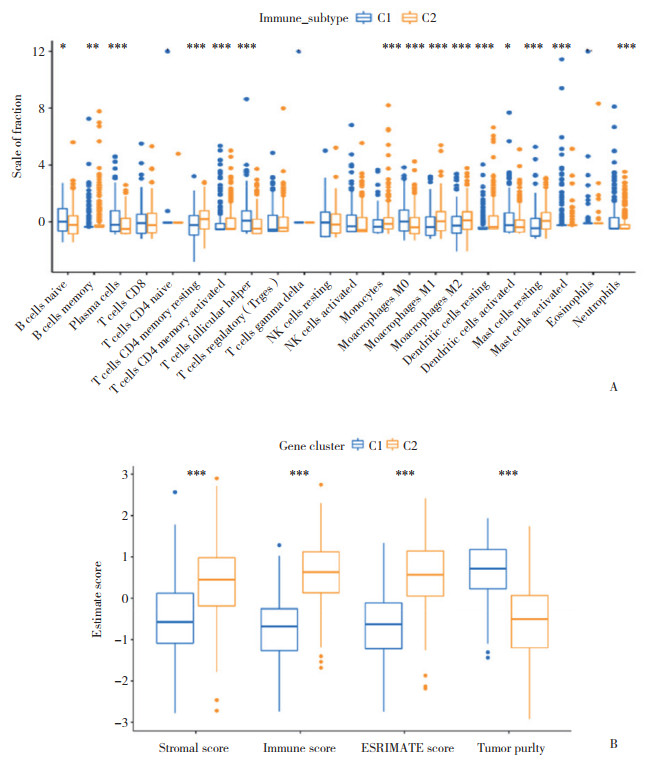
|
| A, proportions of 22 immune cells; B, immunity and matrix scores. * P < 0.05;** P < 0.01;*** P < 0.001. 图 3 2种免疫亚型的22种免疫细胞比例和免疫、基质评分 Fig.3 Proportions of 22 immune cells and scores of immunity and matrix for the two immune subtypes |
|
| A, IDE scores of immune responses; B, TMB analysis. * P < 0.05;** P < 0.001. 图 4 C1、C2 2种免疫亚型的免疫应答TIDE评分和肿瘤突变负荷TMB分析 Fig.4 TIDE scores of immune responses and TMB analysis for C1 and C2 immune subtypes |
|
| A, C1 subtype mutation; B, C2 subtype mutation. 图 5 CNV分析结果 Fig.5 R esults of CNV analysis |
2.2 转录调控分析
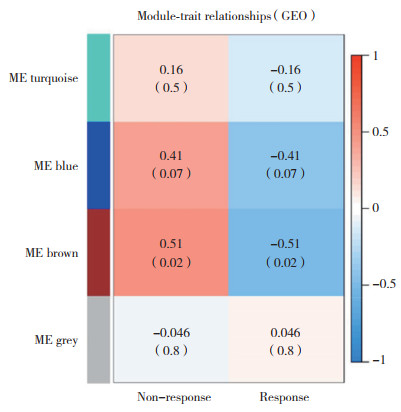
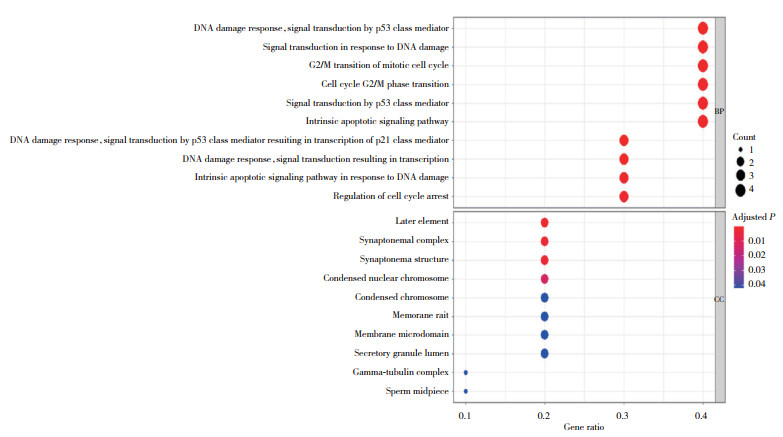
利用GSE136961微阵列数据集中395个免疫应答相关基因表达矩阵及其样本类别(免疫应答和非免疫应答)进行WGCNA分析,构建无标度共表达网络,生成了5个基因模块,以2的幂作为最佳软阈值。在这些模块中,棕色模块与免疫耐受样本的相关性最高(|r| = 0.51,P = 0.02),被认为是“免疫耐受模块”(图 6)。另一方面,对TCGA-LUAD数据集中的395个免疫应答相关基因进行批量生存分析,筛选出49个与生存有关的基因,将“免疫耐受特定模块”基因与这49个基因取交集,最后得到10个与免疫耐受以及生存相关的基因,分别为BRCA2、FOXM1、MELK、BRCA1、TUBB、CD52、SDHA、MIF、IRS1、NFATC1。这10个交集基因的KEGG和GO富集分析结果显示,主要与DNA损伤以及细胞周期的通路相关,见图 7。
|
| 图 6 GSE136961微阵列数据集的免疫应答相关模块WGCNA分析 Fig.6 WGCNA analysis of the immune response-related modules in the GSE136961 microarray dataset |
|
| BP, biological process; CC, cellular component. 图 7 10个交集基因的KEGG和GO富集分析 Fig.7 KEGG and GO enrichment analysis of the 10 intersection genes |
2.3 生存分析
利用Cytoscape的Regulon调控分析工具对BRCA2、FOXM1、MELK、BRCA1、TUBB、CD52、SDHA、MIF、IRS1、NFATC1 10个基因进行转录调控分析,结果显示,E2F4是调控BRCA1、BRCA2、FOXM1、MELK、TUBB 5个靶基因的转录因子;这5个与免疫耐受相关的靶基因在C1亚型显著高表达(图 8)。
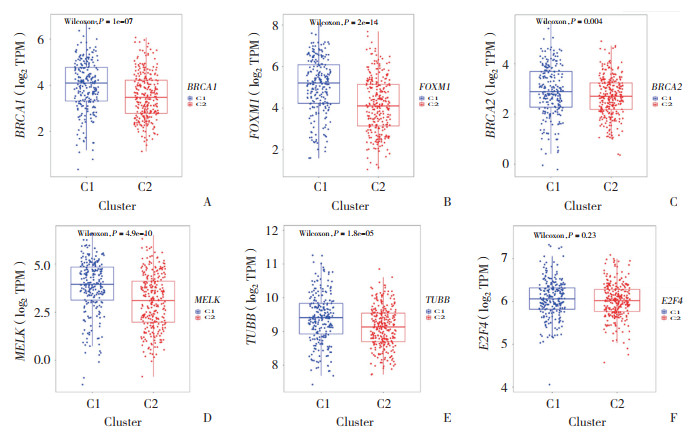
|
| A, BRCA1; B, BRCA2; C, FOXM1; D, MELK; E, TUBB; F, E2F4. 图 8 转录调控因子E2F4及5个靶基因在C1、C2 2种免疫亚型的表达 Fig.8 Expression of transcription factor E2F4 and its five target genes in C1 and C2 subtypes |
对TCGA-LUAD数据集的E2F4转录因子以及筛选出的5个与免疫耐受有关的靶基因(BRCA1、BRCA2、FOXM1、MELK、TUBB)进行生存分析。结果显示,转录调控因子E2F4的高表达与肺腺癌预后不良有关,5个与免疫耐受相关基因(BRCA1、BRCA2、FOXM1、MELK、TUBB)高表达患者预后差(均P < 0.05,图 9)。
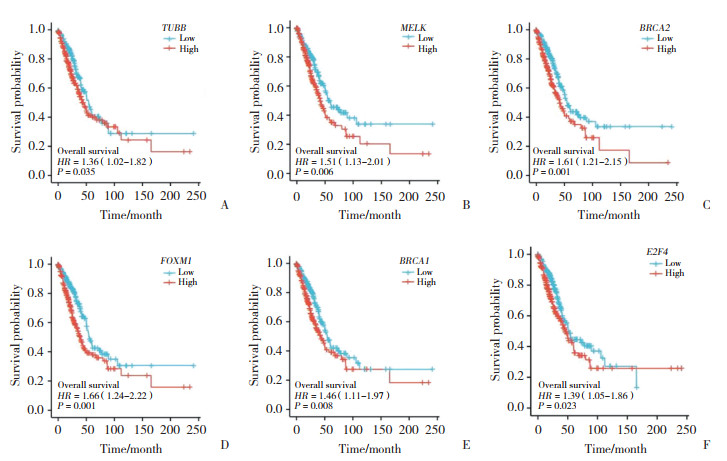
|
| A, TUBB; B, MELK; C, BRCA2; D, FOXM1; E, BRCA1; F, E2F4. 图 9 转录调控因子E2F4及5个靶基因的生存分析 Fig.9 Survival analysis of the transcriptional regulator E2F4 and its five target genes |
3 讨论
正常细胞变成癌细胞既需要内在因素(驱动基因的激活、无限制的增殖和对细胞凋亡的抵抗等),也需要外部因素(逃避免疫杀伤等)。当癌细胞形成并发展时会继续逃避免疫系统的攻击,从而形成恶性肿瘤[10]。另一方面,免疫细胞在TME内发挥免疫监视作用,防止癌症生长。因此,可以通过增强免疫监视来抑制肿瘤的发生和进展。目前,靶向PD-L1/PD-1 ICB治疗可以在非小细胞肺癌治疗领域使用[11]。然而,肺腺癌患者并非都对ICB治疗有良好的反应[12]。
本研究利用NMF算法,在TCGA-LUAD测序数据集中获得C1和C2 2种免疫亚型,并且这2种亚型呈现出不同的免疫活性[13]。本研究使用不同的算法来评估样本中异质免疫细胞的免疫评分、基质评分、绝对或相对浸润丰度,发现C2亚型的免疫评分更高,这意味着C2亚型患者具有更强的免疫反应;使用TIDE算法预测C2亚型中ICB治疗反应更敏感。这表明2种免疫亚型对ICB治疗的潜在反应不同。然而本研究结果显示2种免疫亚型中,C1亚型的TMB明显高于C2亚型,表明在肺腺癌数据集中TMB升高也许并不意味ICB治疗效果较好,TMB可能不是肺腺癌ICB治疗的生物学标志物[14]。
WGCNA分析结果显示,共获得10个与免疫耐受以及生存相关的基因,分别为BRCA2、FOXM1、MELK、BRCA1、TUBB、CD52、SDHA、MIF、IRS1、NFATC1。对这10个与免疫耐受以及生存相关的基因KEGG和GO富集分析结果显示,主要与DNA损伤以及细胞周期的通路相关。对这10个基因进行转录调控分析发现E2F4是调控5个靶基因(BRCA1、BRCA2、FOXM1、MELK、TUBB)的转录因子,对2种免疫亚型中E2F4和调控的5个靶基因(BRCA1、BRCA2、FOXM1、MELK、TUBB)进行表达及生存分析结果显示,E2F4和调控的5个靶基因高表达的肺腺癌患者生存时间较短,ICB治疗疗效较差。
研究发现E2F4上调与TP53失活显著相关,而TP53突变可能是ICB治疗耐药性差的机制之一[15]。另有研究[16]表明E2F4与肝癌的免疫细胞浸润显著相关,但E2F4表达与免疫纯度呈负相关。因此,E2F4可能通过转录调控的方式影响肺腺癌免疫微环境,从而影响肺腺癌免疫应答敏感性[16]。E2F4是调节BRCA1、BRCA2、FOXM1、MELK、TUBB 5个与免疫耐受相关基因的转录调控因子;这5个与免疫耐受相关基因高表达与预后不良、免疫活性低和免疫应答率低的C1亚型相关。其中,BRCA1和BRCA2是与遗传性乳腺癌和卵巢癌有关的重要基因,同时BRCA1调节RAD51的活性,而RAD51在同源DNA序列重组中具有催化活性[17]。TUBB的过表达与肺癌化疗耐药性有关,β-微管蛋白在细胞周期中发挥着重要的作用,同时TUBB的过表达与实体瘤的预后不良以及微管蛋白结合药物的耐药性有关[18]。FOXM1激活会促进免疫细胞功能受到抑制,FOXM1编码的蛋白在细胞的代谢、增殖、凋亡、迁移和侵袭等过程中均起重要的作用[19]。
综上所述,转录调控因子E2F4及与免疫耐受相关的5个靶基因可以作为临床免疫治疗用药以及预后不良的标靶基因。本研究基于生物信息学分析对肺腺癌的免疫应答和免疫浸润进行了全面论证,但还需要进行组织病理学检查和临床试验等进一步验证,以明确其作为生物标志物的临床效用[20]。另外,免疫逃避机制也是免疫系统失调的特征,在肺腺癌的进展中起重要作用。因此,今后研究应综合考虑免疫细胞浸润和癌症免疫逃避对肺腺癌的影响。
| [1] |
KARTOLO A, FEILOTTER H, HOPMAN W, et al. A single institution study evaluating outcomes of PD-L1 high KRAS-mutant advanced non-small cell lung cancer (NSCLC) patients treated with first line immune checkpoint inhibitors[J]. Cancer Treat Res Commun, 2021, 27: 100330. DOI:10.1016/j.ctarc.2021.100330 |
| [2] |
LEI X, LEI Y, LI JK, et al. Immune cells within the tumor microenvironment: biological functions and roles in cancer immunotherapy[J]. Cancer Lett, 2020, 470: 126-133. DOI:10.1016/j.canlet.2019.11.009 |
| [3] |
CHEN LP, HAN X. Anti-PD-1/PD-L1 therapy of human cancer: past, present, and future[J]. J Clin Investig, 2015, 125(9): 3384-3391. DOI:10.1172/jci80011 |
| [4] |
JIMÉNEZ-SÁNCHEZ A, CAST O, MILLER ML. Comprehensive benchmarking and integration of tumor microenvironment cell estimation methods[J]. Cancer Res, 2019, 79(24): 6238-6246. DOI:10.1158/0008-5472.CAN-18-3560 |
| [5] |
JIANG P, GU SQ, PAN D, et al. Signatures of T cell dysfunction and exclusion predict cancer immunotherapy response[J]. Nat Med, 2018, 24(10): 1550-1558. DOI:10.1038/s41591-018-0136-1 |
| [6] |
MAYAKONDA A, LIN DC, ASSENOV Y, et al. Maftools: efficient and comprehensive analysis of somatic variants in cancer[J]. Genome Res, 2018, 28(11): 1747-1756. DOI:10.1101/gr.239244.118 |
| [7] |
LANGFELDER P, HORVATH S. WGCNA: an R package for weighted correlation network analysis[J]. BMC Bioinformatics, 2008, 9: 559. DOI:10.1186/1471-2105-9-559 |
| [8] |
YU G, WANG LG, HAN Y, et al. clusterProfiler: an R package for comparing biological themes among gene clusters[J]. OMICS, 2012, 16(5): 284-287. DOI:10.1089/omi.2011.0118 |
| [9] |
JUNG H, HAN S, KIM S. The construction of regulatory network for insulin-mediated genes by integrating methods based on transcription factor binding motifs and gene expression variations[J]. Genomics Inform, 2015, 13(3): 76-80. DOI:10.5808/GI.2015.13.3.76 |
| [10] |
NADAL E, MASSUTI B, DÓMINE M, et al. Immunotherapy with checkpoint inhibitors in non-small cell lung cancer: insights from long-term survivors[J]. Cancer Immunol Immunother, 2019, 68(3): 341-352. DOI:10.1007/s00262-019-02310-2 |
| [11] |
CAO BP, GUO XC, HUANG LF, et al. Methylation silencing CDH23 is a poor prognostic marker in diffuse large B-cell lymphoma[J]. Aging, 2021, 13(13): 17768-17788. DOI:10.18632/aging.203268 |
| [12] |
YARCHOAN M, HOPKINS A, JAFFEE EM. Tumor mutational burden and response rate to PD-1 inhibition[J]. N Engl J Med, 2017, 377(25): 2500-2501. DOI:10.1056/nejmc1713444 |
| [13] |
LIU J, LIU Q, ZHANG X, et al. Immune subtyping for pancreatic cancer with implication in clinical outcomes and improving immunotherapy[J]. Cancer Cell Int, 2021, 21(1): 137. DOI:10.1186/s12935-021-01824-z |
| [14] |
SHUKUYA T, CARBONE DP. Predictive markers for the efficacy of anti-PD-1/PD-L1 antibodies in lung cancer[J]. J Thorac Oncol, 2016, 11(7): 976-988. DOI:10.1016/j.jtho.2016.02.015 |
| [15] |
SUN D, GUAN X, MORAN AE, et al. Identifying phenotype-associated subpopulations by integrating bulk and single-cell sequencing data[J]. Nat Biotechnol, 2022, 40(4): 527-538. DOI:10.1038/s41587-021-01091-3 |
| [16] |
ZHENG QX, FU Q, XU J, et al. Transcription factor E2F4 is an indicator of poor prognosis and is related to immune infiltration in hepatocellular carcinoma[J]. J Cancer, 2021, 12(6): 1792-1803. DOI:10.7150/jca.51616 |
| [17] |
LI Y, DONG W, ZHANG PQ, et al. Comprehensive analysis of regulatory factors and immune-associated patterns to decipher common and BRCA1/2 mutation-type-specific critical regulation in breast cancer[J]. Front Cell Dev Biol, 2021, 9: 750897. DOI:10.3389/fcell.2021.750897 |
| [18] |
CIERESZKO A, DIETRICH MA, SŁOWIŃSKA M, et al. Application of two-dimensional difference gel electrophoresis to identify protein changes between center, margin, and adjacent non-tumor tissues obtained from non-small-cell lung cancer with adenocarcinoma or squamous cell carcinoma subtype[J]. PLoS One, 2022, 17(5): e0268073. DOI:10.1371/journal.pone.0268073 |
| [19] |
KOO CY, MUIR KW, LAM EWF. FOXM1:From cancer initiation to progression and treatment[J]. Biochim Biophys Acta BBA Gene Regul Mech, 2012, 1819(1): 28-37. DOI:10.1016/j.bbagrm.2011.09.004 |
| [20] |
LIU XY, WU SC, YANG YH. The prognostic landscape of tumorinfiltrating immune cell and immunomodulators in lung cancer[J]. Biomed Pharmacother, 2017, 95: 55-61. DOI:10.1016/j.biopha.2017.08.003 |
 2023, Vol. 52
2023, Vol. 52