文章信息
- 仝洁, 陆进, 荀丽雪, 丁渡山, 杨帆, 张浩轩
- TONG Jie, LU Jin, XUN Lixue, DING Dushan, YANG Fan, ZHANG Haoxuan
- 乳腺癌中COL17A1表达的意义及其免疫相关性
- The significance of COL17A1 expression in breast cancer and its immune correlation
- 中国医科大学学报, 2023, 52(4): 318-326
- Journal of China Medical University, 2023, 52(4): 318-326
-
文章历史
- 收稿日期:2022-09-30
- 网络出版时间:2023-04-12 15:24:04
2. 中国科学院合肥肿瘤医院医学病理中心, 合肥 230088
2. Medical Pathology Center, Hefei Cancer Hospital, Chinese Academy of Sciences, Hefei 230088, China
乳腺癌是全球女性最常见的癌症,由于生活方式、生育年龄和遗传史等因素的变化,发病率呈逐年上升趋势[1]。乳腺癌分子分型主要有Luminal A、Luminal B、HER-2+和三阴性/Basal-like型4种,不同类型具体的临床表现和治疗方式也各有差异[2]。化疗和激素制剂的广泛使用降低了乳腺癌患者的死亡率,但仍具有一定的死亡风险。目前,生物标志物的应用已成为一种辅助乳腺癌诊断、治疗、预测预后的有效策略[3],因此,探索新的生物靶标将有助于提高乳腺癌的筛查效果,进而做到早发现、早诊断和早治疗。
ⅩⅤⅡ α1型胶原蛋白(collagen ⅩⅤⅡ α1,COL17A1)由COL17A1基因编码,是一种Ⅱ型细胞外基质跨膜蛋白。COL17A1是位于10q25.1区域的成熟Ⅰ型半桥粒的重要组成部分,它既可通过黏附在细胞基质,又可通过作为细胞表面受体来发挥作用[4]。由于COL17A1是参与转化细胞多层上皮结构形成的关键调控因子,因此被认为是癌前病变早期诊断和预防性治疗的潜在靶点[5]。众多学者[6-8]证明在胰腺癌、宫颈癌、胶质母细胞瘤中存在COL17A1高表达。但有研究[7]首次发现COL17A1基因在乳腺癌中存在高甲基化水平,并且与肿瘤细胞的侵袭和迁移有关。YODSURANG等[9]发现P53突变型乳腺癌更易发生侵袭现象,其主要机制是P53突变或缺失抑制了COL17A1的表达,从而降低了基底膜与肌上皮细胞之间的黏附能力。可见乳腺癌中COL17A1可能发挥着抑癌作用。关于COL17A1在乳腺癌组织中表达情况、临床预后意义及免疫相关性分析的深入研究尚未见报道,且缺乏相关的系统评价。本研究基于生物信息学分析乳腺癌中COL17A1表达的意义及其免疫相关性,旨在为乳腺癌的基础研究和诊疗策略提供理论依据和新的思路。
1 材料与方法 1.1 数据库选取通过UALCAN数据挖掘网站(http://ualcan.path.uab.edu/index.html)、在线肿瘤数据库基因表达谱交互分析(Gene Expression Profiling Interactive Analysis,GEPIA;http://gepia.cancer-pku.cn/)、cBioPortal数据库(http://www.cbioportal.org/)、MetaScape数据库(http://metascape.org/gp/index.html)、临床生信之家(https://www.aclbi.com/static/index.html#/)、癌细胞系百科全书(Cancer Cell Line Encyclopedia,CCLE;https://portals.broadinstitute.org/)、肿瘤免疫系统交互数据集数据库(Tumor Immune System Interactions Datasets Basis,TISIDB;http://cis.hku.hk/TISIDB/)和Kaplan-Meier Plotter数据库(http://kmplot.com /analysis/)8个数据库分析COL17A1的表达水平,在是否绝经、不同病理分期、年龄、淋巴结转移状态和不同分子分型乳腺癌患者中的表达差异,共表达基因及基因本体论(Gene Ontology,GO)功能注释和京都基因与基因组数据库(Kyoto Encyclopedia of Genes and Genomes,KEGG)通路富集分析,在不同乳腺癌细胞系中的表达,与淋巴细胞浸润和趋化因子相关性以及患者的生存预后分析。
1.2 细胞与试剂来源人正常乳腺上皮细胞(MCF-10A)及专用培养基、乳腺癌细胞MDA-MB-231、MCF-7和BT549均购自武汉普诺赛生命科技有限公司,RPMI-1640培养基和胎牛血清购自美国Gibco公司,胰蛋白酶、双抗(青霉素、链霉素)、BCA试剂盒(BL521A)和羊抗兔IgG-HRP(BL003A)均购自中国Biosharp公司,重组抗COL17A1抗体(ab184996)购自美国Abcam公司,GAPDH抗体(WH208790)购自美国ABclonal公司,RNA提取所用TRIzol(DP424)购自德国TIANGEN公司,逆转录试剂盒(R323)以及实时荧光定量预混PCR试剂盒(Q511-02)购自中国Vazyme公司。
1.3 方法 1.3.1 UALCAN数据库检索步骤及条件,(1)选择TGCA/CPTAC数据集;(2)enter gene symbol(s):COL17A1;TCGA dataset:breast invasive carcinoma;(3)Links for analysis:expression/total protein/pan-cancer view。
1.3.2 GEPIA数据库检索步骤及条件,(1)Gene:COL17A1;(2)Similar gene:200;(3)Datasets:breast cancer。
1.3.3 cBioPortal数据库检索步骤及条件,(1)Cancer type:breast cancer(TGCA firehose legacy,1 108例);(2)Enter genes:COL17A1;(3)Co-expression。
1.3.4 MetaScape数据库通过Metascape数据库对COL17A1在乳腺癌中的差异表达基因(differentially expressed gene,DEG)进行共表达GO功能注释和KEGG通路富集分析。检索步骤及设置条件:(1)输入COL17A1及其DEG集;(2)Input as species,H. sapiens (25);(3)选择“custom analysis”;(4)选择“enrichment”;(5)Min enrichment:1.5。
1.3.5 临床生信之家数据库此数据库整合了CCLE和Prognosis等公共数据库。其中,CCLE检索步骤及条件:(1)样本,breast cancer;(2)基因,COL17A1。Prognosis数据库用于研究某一个基因对某个样本的预后影响及预后效能,其检索步骤及条件:(1)样本,breast cancer;(2)基因,COL17A1;(3)分组方式,median;(4)生存时间,1、3、5年;(5)预后,总体生存(overall survival,OS)。
1.3.6 TISIDB数据库TISIDB数据库是分析肿瘤细胞和免疫系统相互作用关系的工具。检索步骤及条件:(1)Gene symbol,COL17A1;(2)Lymphocyte;(3)Chemokine。
1.3.7 Kaplan-Meier Plotter数据库采用此数据库分析COL17A1基因对乳腺癌患者生存预后的影响。检索步骤及条件:(1)Gene symbol,COL17A1;(2)Split patients by,median;(3)Survival,OS;(4)ER status/PGR status/HER2 status,positive/negative。
1.3.8 体外细胞培养提前将水浴锅预热至37 ℃,复苏液氮罐里冻存的MCF-10A、BT549、MCF-7和MDA-MB-231细胞,接种于T25培养瓶中,置于细胞培养箱中培养,2~3 d换液1次,待细胞密度达80%~90%时,胰蛋白酶消化传代后用于后续实验。
1.3.9 实时定量PCR(real-time quantitative PCR,qRT-PCR)检测用TRIzol试剂提取细胞总mRNA,酶标仪测定RNA的浓度和纯度,反转录为cDNA后采用实时荧光定量预混PCR试剂盒对cDNA进行扩增,采用2-ΔΔCt法计算各组细胞COL17A1的相对表达量(内参照为GAPDH)。引物序列为:COL17A1,正向5’-TTACCCGCCATGCGTATGAAG-3’,反向5’-CAGTCGAACTCGAATTTCACTCT-3’;GAPDH,正向5’-GGAGCGAGATCCCTCCAAAAT-3’,反向5’-GGCTGTTGTCATACTTCTCATGG-3’。
1.3.10 Western blotting检测收集细胞并用蛋白裂解液提取总蛋白,通过BCA法测定提取蛋白质的浓度。取20 μg蛋白进行SDS-PAGE(7.5%)电泳分离,湿转法将蛋白转移到聚偏二氟乙烯膜上,5%脱脂牛奶室温摇床封闭2 h,将膜置于相应的COL17A1兔单克隆抗体(1∶1 000)、GAPDH兔单克隆抗体(1∶10 000)中4 ℃摇床过夜,TBS-T洗去未结合的一抗,洗涤3次,每次5 min。再用辣根过氧化物酶标记的山羊抗兔IgG室温摇床孵育2 h。TBS-T洗去未结合的二抗,洗涤3次,10 min/次。利用凝胶成像系统观察各组细胞中蛋白表达情况,以目的蛋白与内参GAPDH的灰度值比值表示目的蛋白相对表达量,实验重复3次取平均值。
1.4 统计学分析生物信息学数据分析按各个数据库系统内部默认的运算方法得出分析结果。qRT-PCR和Western blotting检测结果采用GraphPad Prism软件进行统计学分析,数据均采用x±s表示,多组比较采用单因素方差分析,多组间两两比较采用LSD-t检验,P < 0.05为差异有统计学意义。
2 结果 2.1 COL17A1在乳腺癌中的表达UALCAN数据库分析结果显示,COL17A1在宫颈癌、膀胱癌和胰腺癌等肿瘤中的表达显著高于正常组织;而在子宫内膜癌、皮肤黑色素瘤和前列腺癌等肿瘤中,其表达显著低于正常组织(图 1A)。进一步对1 097例乳腺癌组织和114例正常乳腺组织分析的结果表明,COL17A1在乳腺癌组织的表达水平显著低于正常乳腺组织(图 1B,P < 0.05)。与正常乳腺组织比较,各年龄段,绝经前、中、后,不同病理分级,不同分子分型,肿瘤类型和淋巴结转移状态的乳腺癌组织中COL17A1 mRNA表达水平均显著降低(均P < 0.05,图 1C~1G)。

|
| A, pancarcinoma analysis; B, expression of COL17A1 in breast cancer tissues; C, expression of COL17A1 in breast cancer tissues of different age groups; D, expression of COL17A1 in breast cancer tissues at different postmenopausal states; E, expression of COL17A1 in breast cancer tissues with different pathological grades; F, expression of COL17A1 in different molecular subtypes of breast cancer tissues; G, expression of COL17A1 in breast cancer tissues with different lymph node metastases. SCLC, adrenocortical carcinoma; COAD, colon adenocarcinoma; DLBC, diffuse large B-celllymphoma; BLCA, bladder urothelial carcinoma; SARC, sarcoma; MM, multiple myeloma; LAML, acute myelocytic lymphoma; SKCM, skin cutaneous melanoma; BRCA, breast invasive carcinoma; LIHC, liver hepatocellular carcinoma; MESO, mesothelioma; OV, ovarian serous cystadenocarcinoma; ESCA, esophageal carcinoma; UCEC, uterine corpus endometrial carcinoma; GBM, glioblastoma multiforme; PAAD, pancreatic adenocarcinoma; NB, neuroblastoma; LUAD, lung adenocarcinoma; STAD, stomach adenocarcinoma; KIRC, kidney renal clear cell carcinoma; NSC, neurogenic stress cardiomyopathy; ALL, acute lymphoblastic leukemia; LUSC, lung squamous cell carcinoma; LGG, brain lower grade gliom; HNSC, head and neck squamous cell carcinoma; THCA, thyroid carcinoma; LCML, chronic myelomonocytic leukemia; MB, medulloblastoma; PRAD, prostate adenocarcinoma; CESC, cervical squamous cell carcinoma and endocervical adenocarcinoma; CLL, chronic lymphocytic leukemia. N0, no regional lymph node metastasis; N1, metastases in 1 to 3 axillary lymph nodes; N2, metastases in 4 to 9 axillary lymph nodes; N3, metastases in 10 or more axillary lymph nodes. 图 1 COL17A1的泛癌分析及其在乳腺癌组织中的表达水平 Fig.1 Pan-cancer analysis of COL17A1 and its expression in breast cancer tissues |
2.2 COL17A1的表达对乳腺癌患者预后分析
Kaplan-Meier Plotter数据库分析COL17A1的表达与乳腺癌患者预后关系的结果显示,COL17A1低表达显著影响患者的OS时间(P = 0.004 3,图 2A)。此外,在乳腺癌各亚型中,COL17A1低表达显著影响Luminal A型(P = 0.022,图 2B)和Basal-like型(P = 0.042,图 2C)患者的预后,导致OS时间缩短,而对Luminal B型和HER2+型患者的预后影响没有统计学意义(图 2D、2E)。
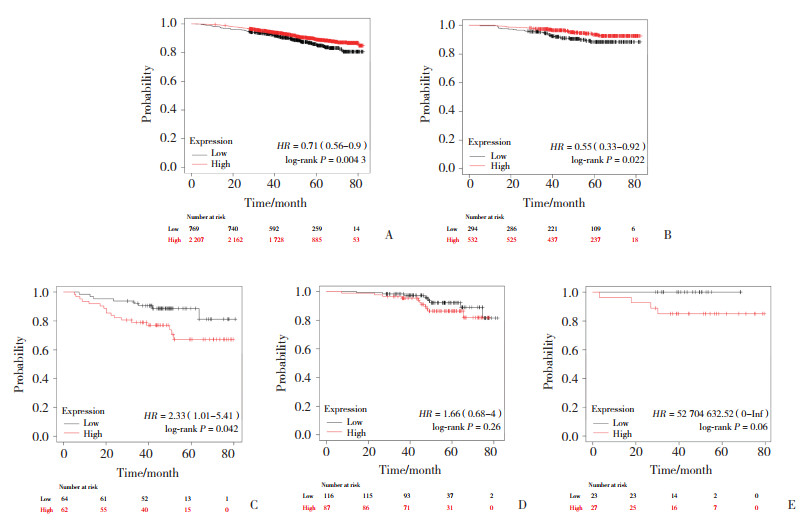
|
| A, breast cancer; B, Luminal A; C, Luminal B; D, HER2+;E, Basal-like. 图 2 COL17A1表达与乳腺癌患者生存预后的关系 Fig.2 Relationship between COL17A1 expression in breast cancer and patient survival and prognosis |
2.3 COL17A1在不同乳腺癌细胞系中的表达分析与验证
CCLE数据库分析结果显示,COL17A1在40种常见乳腺癌细胞系中均呈现低表达。其中,在CAL-85-1、HDQ-P1、HMC-1-8、MDA-MB-415、MDA-MB-231等乳腺癌细胞中呈较低水平表达;在UACC-812、BT549、HCC38、MCF-7、MDA-MB-453等乳腺癌细胞中呈极低表达甚至阴性表达(表 1)。
| Cell lines | COL17A1 expression | Cell lines | COL17A1 expression | |
| HMEL | 10.00 | MDA-MB-175-Ⅶ | 0.74 | |
| HCC1806 | 7.58 | HCC1395 | 0.73 | |
| CAL-85-1 | 5.39 | SK-BR-3 | 0.67 | |
| HDQ-P1 | 5.07 | Hs 606.T | 0.64 | |
| HMC-1-8 | 4.93 | HCC2218 | 0.62 | |
| MDA-MB-415 | 4.31 | ZR-75-30 | 0.58 | |
| MDA-MB-231 | 3.42 | BT-474 | 0.58 | |
| HCC202 | 3.07 | HCC1596 | 0.55 | |
| HCC1937 | 2.54 | HCC38 | 0.54 | |
| CAL-51 | 2.43 | BT-483 | 0.49 | |
| JIMT-1 | 2.39 | BT-20 | 0.41 | |
| HCC-1143 | 2.05 | HCC1428 | 0.38 | |
| MDA-MB-361 | 1.89 | CAMA-1 | 0.25 | |
| HCC1954 | 1.84 | AU565 | 0.23 | |
| MDA-MB-468 | 1.61 | UACC-893 | 0.21 | |
| Hs 578T | 1.53 | MDA-MB-157 | 0.19 | |
| HCC70 | 1.22 | KPL-1 | 0.08 | |
| UACC-812 | 0.84 | MCF-7 | 0.07 | |
| BT-549 | 0.84 | DU4475 | 0.07 | |
| ZR-75-1 | 0.77 | MDA-MB-453 | 0.01 |
Western blotting结果显示,与MCF-10A细胞相比,COL17A1蛋白在BT549、MCF-7、MDA-MB-231细胞中的表达均显著降低(均P < 0.001,图 3A)。同样,COL17A1 mRNA的表达趋势与此一致(均P < 0.001,图 3B)。
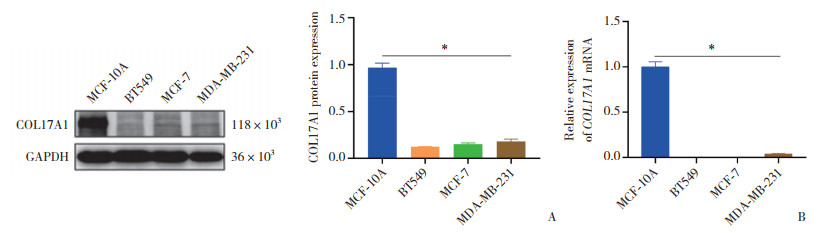
|
| A, Westerning blotting; B, qRT-PCR. *P < 0.001. 图 3 乳腺癌不同细胞系COL17A1的表达情况 Fig.3 Expression of COL17A1 in different breast cancer cell lines |
2.4 COL17A1的DEG分析(图 4)
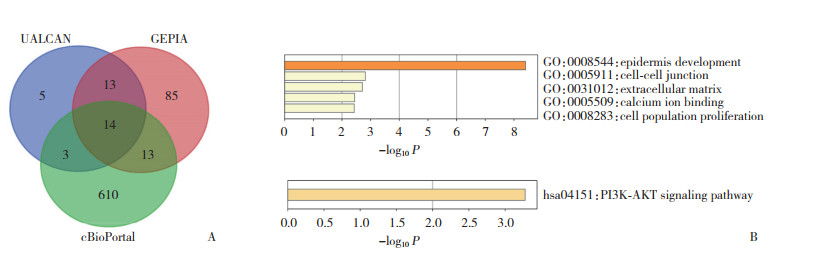
|
| A, Venn diagram; B, GO and KEGG. 图 4 乳腺癌中COL17A1的DEG分析 Fig.4 DEG analysis of COL17A1 in breast cancer |
采用UALCAN、GEPIA和cBioPortal数据库获取乳腺癌组织中与COL17A1基因共表达的、且相关系数≥0.4的基因,绘制维恩图,共得到14个DEG(图 4A)。这14个DEG的GO功能主要富集在表皮发育与形成、细胞外基质组织、细胞-细胞间连接和钙离子结合等;KEGG通路主要富集在PI3K-AKT信号通路(图 4B)。
2.5 COL17A1在乳腺癌中的免疫相关性分析TISIDB数据库分析结果显示,乳腺癌组织趋化因子CCL14与COL17A1相关性最大(r = 0.527,P < 0.001),其次是CCL21、CX3CL1和CCL19(图 5A)。乳腺癌组织趋化因子受体中,CCR6(r = 0.287,P < 0.001)和CX3CR1(r = 0.292,P < 0.001)与COL17A1的相关性大(图 5B)。
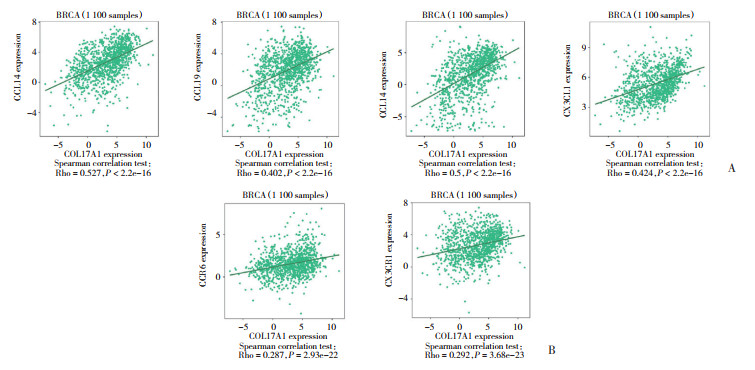
|
| A, correlation between COL17A1 and four chemokines including CCL14, CCL21, CX3CL1 and CCL19 in breast cancer; B, correlation between COL17A1 and two chemokine receptors, CCR6 and CX3CR1 in breast cancer. BRCA, breast cancer. 图 5 乳腺癌中COL17A1与趋化因子及趋化因子受体的相关性 Fig.5 Correlation of COL17A1 with chemokines and chemokine receptors in breast cancer |
乳腺癌组织中,COL17A1的表达与多种淋巴细胞浸润存在相关性,其中与17型辅助T(Th17)细胞(r = 0.301,P < 0.001)、活化B细胞(r = 0.328,P < 0.001)和肥大细胞(r = 0.312,P < 0.001)的相关性最明显(图 6)。
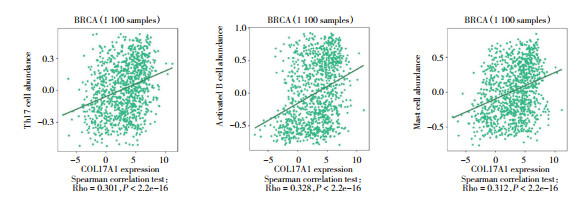
|
| 图 6 乳腺癌中COL17A1与3种免疫淋巴细胞浸润的相关性 Fig.6 Correlation of COL17A1 with three types of immunolymphocyte infiltration in breast cancer |
2.6 COL17A1基因对乳腺癌的综合预后分析
基于临床生信之家数据库的分析结果显示,COL17A1低表达与患者生存时间短及死亡状态相关(图 7A);Kaplan-Meier曲线也显示COL17A1作为乳腺癌的保护因素,其低表达与患者的预后较差相关(图 7B)。受试者操作特征(receiver operator characteristic curve,ROC)曲线分析显示,第1年、第3年、第5年的曲线下面积(area under the curve,AUC)为0.5~0.7(图 7C)。
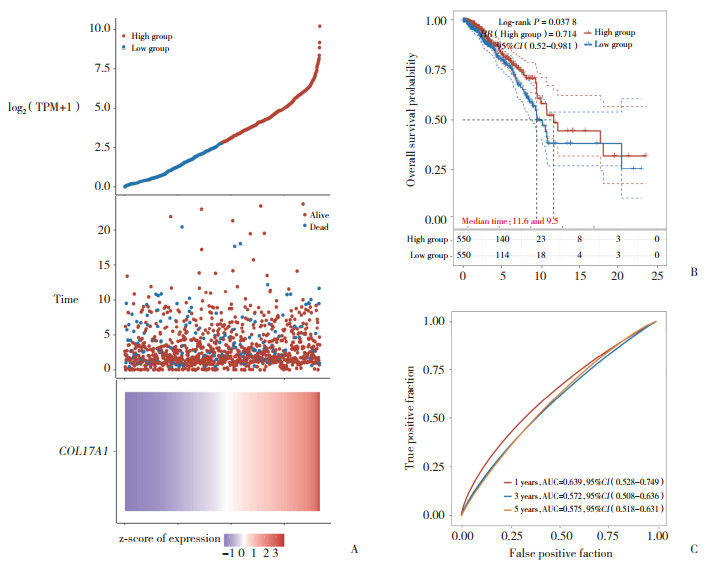
|
| A, relationship between COL17A1 and survival time and survival status in TCGA data; B, KM survival curve of COL17A1 gene in TCGA data; C, ROC curve and AUC value of COL17A gene at different time. If HR > 1, it means the gene is a risk factor; if HR < 1, it means the gene is a protective factor; In addition, the higher the AUC value, the stronger the predictive ability of the gene. 图 7 COL17A1表达对乳腺癌样本的预后影响及预测效能分析 Fig.7 Analysis of COL17A1 expression on the prognosis of patients with breast cancer positive samples and its predictive efficiency |
3 讨论
乳腺癌是女性最常见的恶性肿瘤之一,发病率和致死率分别占所有癌症的24.2%与15%,是女性癌症死亡的主要原因[10]。因此,在现有的研究基础上,探索新的分子靶标和更深层的分子机制,将有助于为乳腺癌的诊疗提供理论依据,从而改善患者治疗效果和预后。
COL17A1编码表皮与真皮连接处的跨膜糖蛋白,具有促进细胞稳定地附着在基底膜上的功能,表达于乳腺的皮肤、肌上皮和气管上皮细胞等[11],也可在多种癌症中表达上调,与本研究泛癌分析结果一致。在肿瘤微环境(tumor microenvironment,TME)中,上调的胶原蛋白可通过侵袭肿瘤周围组织并渗透到血流中来启动肿瘤细胞的转移[7]。但研究[12]发现,在乳腺癌中COL17A1可能作为一种保护因素,抑制了肿瘤细胞侵袭和迁移的过程。本研究通过肿瘤大数据库分析,对COL17A1在乳腺癌组织中的表达水平和预后情况等进行系统分析并进行验证和总结。
本研究表明,COL17A1在乳腺癌不同临床阶段均表达降低,且不利于患者的总体生存。其中,在乳腺癌各亚型中,COL17A1的低表达对Luminal A型和Basal-like型患者的生存预后有意义,而对其他亚型没有显著影响。以往研究[13-14]显示COL17A1在胰腺癌、膀胱癌等中均表达上调,与肿瘤侵袭发展相关,可作为此类癌症的独立预后因素。然而与原发性皮肤黑色素瘤相比,在转移性黑色素瘤中COL17A1表达较低[15]。肿瘤复发始终是一个难治性问题,有研究[16]发现,COL17A1在结直肠癌肿瘤干细胞的休眠过程中发挥作用,破坏COL17A1将有助于提高化疗效果。此外,综合预后分析结果显示,COL17A1可作为乳腺癌的预后生物标志物,这与SHI等[17]结果一致。ROC曲线分析结果显示,第1年、第3年、第5年的AUC为0.5~0.7,说明其独立预测效能较好,但仍需结合其他多种基因综合考量。结果还显示COL17A1在40种常见乳腺癌细胞系中均呈现低表达水平,与目前的研究[18]结论一致。本研究选取了MDA-MB-231细胞株(代表Basal-like型)、BT549细胞株(代表HER-2型)和MCF-7细胞株(代表Luminal A型),用MCF-10A细胞株做对照,从蛋白质和基因水平分别检测COL17A1的表达情况,表达趋势均与UALCAN和CCLE数据库结果一致,也证实了COL17A1在乳腺癌中呈现低表达。
本研究对COL17A1及其14个DEG(包括HAS3、KRT14、PAMR1、FAT2、S100A2、FLRT2、LAMB3、IL33、NGFR、LAMC2、KRT5、GPR87、MAMDC2、TP63)进行GO和KEGG分析,其中,GO功能主要富集在表皮发育与形成、细胞外基质组织、细胞-细胞间连接等,KEGG通路主要富集在PI3K-AKT信号通路。LOTHONG等[19]证实过表达COL17A1会通过抑制AKT/mTOR通路来抑制乳腺癌细胞的侵袭和增殖表型,但更多的分子机制仍需进一步探索。
趋化因子是细胞因子的亚家族,在发育、免疫监视、炎症、组织修复以及先天和适应性免疫中发挥着基本作用,在肿瘤生长、侵袭和转移等方面也发挥重要作用[20]。CCL14、CCL21、CX3CL1和CCL19等几类趋化因子在其他生物信息学分析中被证实与乳腺癌的临床预后显著相关,可作为辅助治疗乳腺癌患者的生物学标志物[18,21],与本研究结果一致。此外,CCR6和CX3CR1在乳腺癌组织中与COL17A1的相关性最大,并且CX3CL1及其同源性受体CX3CR1被证实在乳腺癌的转移性进展中发挥重要作用[22-23],但与COL17A1在肿瘤中的作用关系及机制目前尚未明确。TME中趋化因子通过与相应的受体结合,进而招募各种免疫淋巴细胞迁移和浸润,调节肿瘤免疫反应。本研究结果显示,乳腺癌组织中COL17A1的表达会导致Th17细胞、活化B细胞和肥大细胞的趋化作用明显增强,这几类淋巴细胞也被证实参与了乳腺癌的抗肿瘤免疫,并且与更好的预后水平相关[24-25]。因此,随着COL17A1在乳腺癌中的表达减少,抗肿瘤免疫作用也减弱,从而不利于患者的总体预后。
综上所述,COL17A1在乳腺癌组织中显著低表达,并与患者不良预后显著相关。COL17A1表达与趋化因子和淋巴细胞等的肿瘤免疫作用也有一定的相关性,可作为乳腺癌新的预后标志物,但需要更多的相关研究进一步验证。此外,COL17A1在乳腺癌中主要发挥的功能包括表皮发育与形成、细胞外基质组织、细胞-细胞间连接等,参与的信号通路包括PI3K-AKT信号通路。本研究也为明确乳腺癌分子机制和诊疗策略提供了新的思路。
| [1] |
AZAMJAH N, SOLTAN-ZADEH Y, ZAYERI F. Global trend of breast cancer mortality rate: a 25-year study[J]. Asian Pac J Cancer Prev, 2019, 20(7): 2015-2020. DOI:10.31557/APJCP.2019.20.7.2015 |
| [2] |
冯源, 兰晓莉, 张永学. 分子影像学用于早期诊断乳腺癌及其分子分型进展[J]. 中国医学影像技术, 2022, 38(7): 1086-1089. DOI:10.13929/j.issn.1003-3289.2022.07.028 |
| [3] |
BRADEN AM, STANKOWSKI RV, ENGEL JM, et al. Breast cancer biomarkers: risk assessment, diagnosis, prognosis, prediction of treatment efficacy and toxicity, and recurrence[J]. Curr Pharm Des, 2014, 20(30): 4879-4898. DOI:10.2174/1381612819666131125145517 |
| [4] |
SEPPÄNEN A. Collagen ⅩⅦ: a shared antigen in neurodermatological interactions?[J]. Clin Dev Immunol, 2013, 2013: 240570. DOI:10.1155/2013/240570 |
| [5] |
KOZAWA K, SEKAI M, OHBA K, et al. The CD44/COL17A1 pathway promotes the formation of multilayered, transformed epithelia[J]. Curr Biol, 2021, 31(14): 3086-3097. DOI:10.1016/j.cub.2021.04.078 |
| [6] |
HUANG WL, WU SF, HUANG X, et al. Integrated analysis of ECT2 and COL17A1 as potential biomarkers for pancreatic cancer[J]. Dis Markers, 2022, 2022: 9453549. DOI:10.1155/2022/9453549 |
| [7] |
THANGAVELU PU, KRENÁCS T, DRAY E, et al. In epithelial cancers, aberrant COL17A1 promoter methylation predicts its misexpression and increased invasion[J]. Clin Epigenet, 2016, 8(1): 120. DOI:10.1186/s13148-016-0290-6 |
| [8] |
YAN XY, ZHANG CB, LIANG TY, et al. A PTEN-COL17A1 fusion gene and its novel regulatory role in Collagen XVⅡ expression and GBM malignance[J]. Oncotarget, 2017, 8(49): 85794-85803. DOI:10.18632/oncotarget.20526 |
| [9] |
YODSURANG V, TANIKAWA C, MIYAMOTO T, et al. Identification of a novel p53 target, COL17A1, that inhibits breast cancer cell migration and invasion[J]. Oncotarget, 2017, 8(34): 55790-55803. DOI:10.18632/oncotarget.18433 |
| [10] |
BRAY F, FERLAY J, SOERJOMATARAM I, et al. Global cancer statistics 2018:globocan estimates of incidence and mortality worldwide for 36 cancers in 185 countries[J]. CA A Cancer J Clin, 2018, 68(6): 394-424. DOI:10.3322/caac.21492 |
| [11] |
HIEDA Y, NISHIZAWA Y, UEMATSU J, et al. Identification of a new hemidesmosomal protein, HD1:a major, high molecular mass component of isolated hemidesmosomes[J]. J Cell Biol, 1992, 116(6): 1497-1506. DOI:10.1083/jcb.116.6.1497 |
| [12] |
SROUR MK, GAO BW, DADMANESH F, et al. Gene expression comparison between primary triple-negative breast cancer and paired axillary and sentinel lymph node metastasis[J]. Breast J, 2020, 26(5): 904-910. DOI:10.1111/tbj.13684 |
| [13] |
WU MW, LI XB, ZHANG TP, et al. Identification of a nine-gene signature and establishment of a prognostic nomogram predicting overall survival of pancreatic cancer[J]. Front Oncol, 2019, 9: 996. DOI:10.3389/fonc.2019.00996 |
| [14] |
ELBADAWY M, USUI T, MORI T, et al. Establishment of a novel experimental model for muscle-invasive bladder cancer using a dog bladder cancer organoid culture[J]. Cancer Sci, 2019, 110(9): 2806-2821. DOI:10.1111/cas.14118 |
| [15] |
XIE RJ, LI BF, JIA LE, et al. Identification of core genes and pathways in melanoma metastasis via bioinformatics analysis[J]. Int J Mol Sci, 2022, 23(2): 794. DOI:10.3390/ijms23020794 |
| [16] |
OHTA Y, FUJⅡ M, TAKAHASHI S, et al. Cell-matrix interface regulates dormancy in human colon cancer stem cells[J]. Nature, 2022, 608(7924): 784-794. DOI:10.1038/s41586-022-05043-y |
| [17] |
SHI WJ, HU DJ, LIN S, et al. Five-mRNA signature for the prognosis of breast cancer based on the ceRNA network[J]. Biomed Res Int, 2020, 2020: 9081852. DOI:10.1155/2020/9081852 |
| [18] |
DU F, ZHENG FC, HAN Y, et al. Novel immune-related gene signature for risk stratification and prognosis of survival in ER (+) and/or PR (+) and HER2 (-) breast cancer[J]. Front Pharmacol, 2022, 13: 820437. DOI:10.3389/fphar.2022.820437 |
| [19] |
LOTHONG M, SAKARES W, ROJSITTHISAK P, et al. Collagen XVⅡ inhibits breast cancer cell proliferation and growth through deactivation of the AKT/mTOR signaling pathway[J]. PLoS One, 2021, 16(7): e0255179. DOI:10.1371/journal.pone.0255179 |
| [20] |
SAXENA S, SINGH RK. Chemokines orchestrate tumor cells and the microenvironment to achieve metastatic heterogeneity[J]. Cancer Metastasis Rev, 2021, 40(2): 447-476. DOI:10.1007/s10555-021-09970-6 |
| [21] |
HOZHABRI H, MOGHADDAM MM, MOGHADDAM MM, et al. A comprehensive bioinformatics analysis to identify potential prognostic biomarkers among CC and CXC chemokines in breast cancer[J]. Sci Rep, 2022, 12(1): 10374. DOI:10.1038/s41598-022-14610-2 |
| [22] |
LIANG Y, YI L, LIU P, et al. CX3CL1 involves in breast cancer metastasizing to the spine via the Src/FAK signaling pathway[J]. J Cancer, 2018, 9(19): 3603-3612. DOI:10.7150/jca.26497 |
| [23] |
DINATALE A, KAUR R, QIAN C, et al. Subsets of cancer cells expressing CX3CR1 are endowed with metastasis-initiating properties and resistance to chemotherapy[J]. Oncogene, 2022, 41(9): 1337-1351. DOI:10.1038/s41388-021-02174-w |
| [24] |
YANG LJ, QI YX, HU J, et al. Expression of Th17 cells in breast cancer tissue and its association with clinical parameters[J]. Cell Biochem Biophys, 2012, 62(1): 153-159. DOI:10.1007/s12013-011-9276-3 |
| [25] |
APONTE-LÓPEZ A, MUÑOZ-CRUZ S. Mast cells in the tumor microenvironment[J]. Adv Exp Med Biol, 2020, 1273: 159-173. DOI:10.1007/978-3-030-49270-0_9 |
 2023, Vol. 52
2023, Vol. 52




