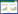文章信息
- 龙焕屏, 李光
- LONG Huanping, LI Guang
- 肺腺癌预后不良的重要基因CDC20、CCNB2和CDC45
- Important genes for poor prognosis of lung adenocarcinoma: CDC20, CCNB2, and CDC45
- 中国医科大学学报, 2021, 50(9): 846-850, 855
- Journal of China Medical University, 2021, 50(9): 846-850, 855
-
文章历史
- 收稿日期:2019-12-10
- 网络出版时间:2020-09-09 13:22
在癌症死亡患者中肺癌占27%,是癌症相关死亡的主要原因[1]。肺腺癌(lung adenocarcinoma,LUAD) 已经成为近数十年来最常见的肺癌类型之一,并且LUAD倾向于早期转移,能够导致很高的死亡率[2]。因此,寻找可靠的预后标志物和治疗靶点对提高LUAD的治疗效果非常重要。随着生物技术的发展,可以通过微阵列检测成千上万个基因的表达水平,进一步促进了癌症的研究。本研究通过分析基因表达微阵列数据(GSE33532、GSE32863和GSE43458) 探索了LUAD中差异表达基因的相互作用网络以及相关的信号传导途径,最终鉴定出3个核心基因CDC45、CCNB2和CDC20可以作为LUAD的候选生物标志,从而为LUAD的潜在生物学机制和治疗靶标提供有用的信息和方向。
1 材料与方法 1.1 微阵列数据信息从美国国立生物技术信息中心的基因表达汇编(Gene Expression Omnibus,GEO) 数据库中选取GSE33532、GSE32863和GSE43458的芯片数据,共178例LUAD和98例正常肺组织。
1.2 差异表达基因的筛选使用GEO2R筛选LUAD和正常肺组织之间的差异表达基因,筛选条件是| logFC |≥ 1和调整P < 0.001。然后,使用Venn diagram软件获得3个基因芯片的共同差异表达基因,log FC > 0定义为上调基因,log FC < 0定义为下调基因。
1.3 基因本体(Gene Ontology,GO) 分析和京都基因与基因组百科全书(Kyoto Encyclopedia of Genes and Genomes,KEGG) 分析使用注释、可视化和集成发现数据库(The Database for Annotation,Visualization,and Integrated Discovery,DAVID) 对基因进行分子功能(molecular function,MF)、细胞成分(cellular component,CC)、生物过程(biological process,BP) 和KEGG分析(P < 0.05)。
1.4 蛋白-蛋白相互作用网络(protein-protein interaction network,PPI) 的建立和重要模块的分析通过STRING获取PPI信息。然后,使用Cytoscape检查基因之间潜在的相关性,使用MCODE获得重要模块。
1.5 核心基因的生存分析通过Kaplan-Meier Plotter在线数据库评估基因对预后的影响(P < 0.05)。
2 结果 2.1 筛选LUAD中差异表达的基因使用GEO2R,分别从GSE33532、GSE32863和GSE43458中筛选出3 180、1 584、1 011个差异表达基因。使用Venn diagram获得331个共同差异表达基因,其中上调基因68个(图 1A),下调基因263个(图 1B)。
|
| A, 68 up-regulated genes were obtained in three data sets (log FC>0);B, 263 down-regulated genes were obtained in three data sets (log FC < 0). 图 1 通过使用Venn diagram获得331个共同差异表达基因 Fig.1 331 common differentially expressed genes obtained by Venn diagram |
2.2 差异基因的GO和KEGG分析
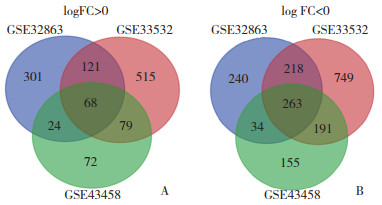
使用DAVID对331个基因进行GO分析。结果表明:(1) BP方面,上调的基因主要富集在胶原纤维组织、胞质分裂和凋亡过程的正向调控等,下调的基因主要富集在细胞黏附和细胞对激素刺激的反应等;(2) MF方面,上调的基因富集在细胞外基质结构组成和DNA结合、弯曲等,下调的基因则富集在肝素结合和碳水化合物结合等;(3) CC方面,上调的基因明显富集在蛋白质细胞外基质、中体和细胞外泌体等,下调的基因富集在膜筏和蛋白质细胞外基质等。KEGG分析结果显示,上调的基因主要富集在细胞外基质受体相互作用、蛋白质的消化吸收、黏着斑和细胞周期,下调的基因则富集在细胞黏附分子、药物代谢-细胞色素P450、酪氨酸代谢和PPAR信号通路(图 2)。
|
| A, GO and KEGG analysis of 68 up-regulated genes; B, GO and KEGG analysis of 263 down-regulated genes. 图 2 68个上调基因和263个下调基因的GO和KEGG分析 Fig.2 GO and KEGG analysis of 68 up-regulated genes and 263 down-regulated genes |
2.3 PPI的构建和重要模块分析
通过STRING获取331个共同差异表达基因的PPI信息。使用Cytoscape检查基因之间潜在的相关性,共有291个节点和934层边缘。此外,使用MCODE获得了1个重要模块。该模块包括16个基因,且均为上调基因(图 3)。

|
| 图 3 通过STRING和模块分析构建PPI Fig.3 Building PPI through STRING and module analysis |
2.4 使用Kaplan-Meier Plotter在线数据库进行核心基因的分析
使用Kaplan-Meier Plotter在线数据库对16个基因进行总生存分析(overall survival,OS),结果显示,13个基因(ASPM,CCNB2,CDC20,CDC45,CENPE,KIAA0101,KIF20A,MELK,PRC1,TOP2A,TPX2,TYMS,UBE2T)的高表达与LUAD的OS更差相关(均P < 0.05)。
2.5 13个核心基因的KEGG途径分析13个基因的KEGG分析结果表明,CDC45、CCNB2和CDC20显著富集于细胞周期途径中(P = 0.001 929 455,FDR=1.234 859 464)。
2.6 CDC45、CCNB2和CDC20的表达与不同临床病理特征肺癌患者预后的关系使用Kaplan-Meier Plotter在线数据库分析CDC45、CCNB2和CDC20的表达与不同临床病理特征肺癌患者临床预后的关系。结果显示,CDC20的高表达与男性、女性、TNM分期中的Ⅰ期、N2期、吸烟、腺癌和手术切缘阴性患者的OS和无进展生存期(free progression survival,FPS) 较差显著相关(表 1),CCNB2的高表达与男性、TNM分期中的Ⅰ期、吸烟、未吸烟、腺癌和手术切缘阴性患者的OS和FPS较差显著相关(表 2),CDC45的高表达与男性、TNM分期中的Ⅰ期、吸烟、腺癌、手术切缘阴性和未化疗患者的OS和FPS较差显著相关(表 3)。这些结果表明,CDC45、CCNB2和CDC20的表达水平可能影响肺癌患者的预后,可望成为LUAD的预后标志物。
| Clinicopathological characteristic | Overall survival (n = 1 928) | Progression-free survival (n= 982) | |||||
| n | Hazard ratio | P | n | Hazard ratio | P | ||
| Gender | |||||||
| Female | 715 | 2.09(1.64-2.65) | 8.1e-10 | 468 | 1.71(1.28-2.28) | 0.000 28 | |
| Male | 1 100 | 1.62(1.38-1.90) | 1.6e-9 | 514 | 1.74(1.34-2.26) | 2.6e-5 | |
| Stage | |||||||
| Ⅰ | 577 | 3.24(2.39-4.38) | 7.8e-16 | 325 | 2.97(1.83-4.81) | 3.7e-6 | |
| Ⅱ | 244 | 1.51(1.05-2.17) | 0.027 | 130 | 1.07(0.64-1.79) | 0.81 | |
| Stage T | |||||||
| 1 | 437 | 2.06(1.53-2.77) | 9.4e-7 | 177 | 1.14(0.69-1.88) | 0.61 | |
| 2 | 589 | 1.42(1.14-1.77) | 0.001 9 | 351 | 1.23(0.91-1.66) | 0.17 | |
| Stage N | |||||||
| 0 | 781 | 1.60(1.29-1.98) | 1.2e-5 | 374 | 1.37(0.99-1.89) | 0.059 | |
| 1 | 252 | 1.68(1.22-2.30) | 0.001 2 | 130 | 1.14(0.72-1.79) | 0.58 | |
| 2 | 111 | 1.33(0.89-2.00) | 0.017 | 51 | 2.13(1.03-4.42) | 0.037 | |
| Stage M | |||||||
| 0 | 681 | 1.76(1.43-2.17) | 9.4e-8 | 195 | 1.30(0.78-2.14) | 0.31 | |
| Smoking history | |||||||
| Smoking | 820 | 1.72(1.39-2.12) | 2.9e-7 | 603 | 1.52(1.19-1.94) | 0.000 79 | |
| Non-smoking | 205 | 1.54(0.88-2.70) | 0.13 | 193 | 2.42(1.46-4.00) | 0.000 39 | |
| Histology | |||||||
| Adenocarcinoma | 720 | 2.39(1.87-3.05) | 8.6e-13 | 461 | 2.58(1.85-3.59) | 5.5e-9 | |
| Squamous cell carcinoma | 524 | 1.12(0.89-1.42) | 0.34 | 141 | 0.97(0.58-1.61) | 0.89 | |
| Surgical margins negative | 726 | 2.26(1.78-2.87) | 4.6e-12 | 560 | 2.23(1.72-2.89) | 5e-10 | |
| Clinicopathological characteristic | Overall survival (n = 1 928) | Progression-free survival (n = 982) | |||||
| n | Hazard ratio | P | n | Hazard ratio | P | ||
| Gender | |||||||
| Female | 715 | 2.16(1.70-2.76) | 2e-10 | 468 | 1.80(1.34-2.40) | 6.4e-5 | |
| Male | 1 100 | 1.77(1.51-2.08) | 1.6e-12 | 514 | 1.61(1.24-2.09) | 0.000 27 | |
| Stage | |||||||
| Ⅰ | 577 | 3.26(2.40-4.44) | 1.9e-15 | 325 | 2.44(1.53-3.89) | 0.000 11 | |
| Stage T | |||||||
| 1 | 437 | 2.19(1.63-2.95) | 1.2e-7 | 177 | 1.83(1.09-3.07) | 0.02 | |
| 2 | 589 | 1.59(1.28-1.99) | 3.6e-5 | 351 | 1.15(0.85-1.55) | 0.36 | |
| Stage N | |||||||
| 0 | 781 | 1.74(1.41-2.16) | 2.7e-7 | 374 | 1.37(0.99-1.90) | 0.056 | |
| 1 | 252 | 1.70(1.24-2.33) | 0.000 87 | 130 | 0.98(0.62-1.54) | 0.93 | |
| Stage M | |||||||
| 0 | 681 | 1.93(1.56-2.39) | 5.6e-10 | 195 | 1.31(0.79-2.17) | 0.29 | |
| Smoking history | |||||||
| Smoking | 820 | 1.68(1.36-2.07) | 1e-6 | 603 | 1.66(1.30-2.13) | 4.1e-5 | |
| Non-smoking | 205 | 2.19(1.22-3.93) | 0.007 4 | 193 | 2.37(1.43-3.92) | 0.000 54 | |
| Histology | |||||||
| Adenocarcinoma | 720 | 2.36(1.84-3.02) | 2.9e-12 | 461 | 3.16(2.25-4.44) | 2.8e-12 | |
| Squamous cell carcinoma | 524 | 1.06(0.84-1.35) | 0.62 | 141 | 1.08(0.65-1.81) | 0.75 | |
| Surgical margins negative | 726 | 2.35(1.85-2.98) | 6.1e-13 | 560 | 2.07(1.60-2.68) | 1.4e-8 | |
| Clinicopathological characteristic | Overall survival (n = 1 928) | Progression-free survival (n = 982) | |||||
| n | Hazard ratio | P | n | Hazard ratio | P | ||
| Gender | |||||||
| Female | 715 | 1.81(1.43-2.30) | 8.1e-7 | 468 | 1.32(0.99-1.76) | 0.057 | |
| Male | 1 100 | 1.57(1.34-1.85) | 1.9e-8 | 514 | 1.53(1.18-1.98) | 0.001 3 | |
| Stage | |||||||
| Ⅰ | 577 | 3.26(2.39-4.44) | 2.9e-15 | 325 | 1.90(1.21-2.98) | 0.004 6 | |
| Stage T | |||||||
| 1 | 437 | 1.96(1.46-2.62) | 5.2e-6 | 177 | 1.33(0.80-2.19) | 0.27 | |
| 2 | 589 | 1.28(1.03-1.60) | 0.028 | 351 | 1.20(0.89-1.63) | 0.22 | |
| Stage N | |||||||
| 0 | 781 | 1.64(1.32-2.02) | 5.1e-6 | 374 | 1.21(0.87-1.67) | 0.25 | |
| 1 | 252 | 1.36(0.99-1.86) | 0.057 | 130 | 1.18(0.75-1.85) | 0.48 | |
| 2 | 111 | 1.24(0.83-1.85) | 0.3 | 51 | 2.79(1.33-5.85) | 0.004 9 | |
| Stage M | |||||||
| 0 | 681 | 1.64(1.33-2.02) | 3.2e-6 | 195 | 1.11(0.67-1.83) | 0.69 | |
| Smoking history | |||||||
| Smoking | 820 | 1.57(1.28-1.93) | 1.8e-5 | 603 | 1.65(1.29-2.11) | 6e-5 | |
| Non-smoking | 205 | 1.63(0.92-2.86) | 0.089 | 193 | 2.04(1.25-3.35) | 0.003 8 | |
| Histology | |||||||
| Adenocarcinoma | 720 | 2.24(1.74-2.87) | 8e-11 | 461 | 2.28(1.64-3.15) | 3.8e-7 | |
| Squamous cell carcinoma | 524 | 0.90(0.71-1.14) | 0.39 | 141 | 0.78(0.47-1.31) | 0.35 | |
| Surgical margins negative | 726 | 1.83(1.45-2.31) | 2.4e-7 | 560 | 1.90(1.47-2.45) | 6e-7 | |
| Chemotherapy | |||||||
| Yes | 196 | 1.03(0.69-1.55) | 0.88 | 125 | 0.95(0.62-1.44) | 0.8 | |
| No | 310 | 1.62(1.16-2.28) | 0.004 6 | 258 | 1.64(1.10-2.43) | 0.014 | |
3 讨论
为了确定在LUAD中有用的预后生物标志物,本研究使用生物信息学方法从GEO数据库中选取了GSE33532、GSE32863和GSE43458芯片,最终得到CDC45、CCNB2和CDC20在细胞周期途径中显著富集,并且这3个基因的表达与不同临床病理特征肺癌患者的预后有关,表明这3个基因可能作为LUAD的预后生物标志物和潜在治疗靶点。
CDC20突变可导致有丝分裂异常停止,从而导致后期染色体分离异常[3]。CDC20在多种肿瘤中高表达,如卵巢癌[4]、肝细胞癌[5]、胃癌[6]、乳腺癌[7],并与患者的预后有关。下调CDC20可增加癌细胞对放疗和化疗的敏感性[8]。CCNB2通过激活CDK1激酶来触发细胞周期G2/M的转变[9]。CCNB2在多种癌症中高表达,如前列腺癌[10]、乳腺癌[11]、。CCNB2 mRNA在癌症患者血清中高表达,且与癌症的分期和转移状态有关[12]。CDC45在启动DNA复制中起着至关重要的作用[13],且在多种肿瘤中高表达,如宫颈癌[14],并且CDC45可能是各种恶性肿瘤增殖的标志物[15]。
大量研究证明,CDC20、CCNB2和CDC45与多种肿瘤进展密切相关。本研究通过生物信息学分析发现,CDC20、CCNB2和CDC45在LUAD的预后过程中可能起关键作用,可能是潜在的治疗靶标。但是,这些预测还需要通过一系列实验加以验证。本研究的结果为LUAD的潜在生物学机制和治疗靶标提供了一些有用的信息和方向。
| [1] |
SIEGEL RL, MILLER KD, JEMAL A. Cancer statistics, 2016[J]. CA Cancer J Clin, 2016, 66(1): 7-30. DOI:10.3322/caac.21332 |
| [2] |
HOU S, ZHOU S, QIN Z, et al. Evidence, mechanism, and clinical relevance of the transdifferentiation from lung adenocarcinoma to squamous cell carcinoma[J]. Am J Pathol, 2017, 187(5): 954-962. DOI:10.1016/j.ajpath.2017.01.009 |
| [3] |
HARTWELL LH, CULOTTI J, REID B. Genetic control of the cell-division cycle in yeast, Ⅰ. detection of mutants[J]. Proc Natl Acad Sci U S A, 1970, 66(2): 352-359. DOI:10.1073/pnas.66.2.352 |
| [4] |
FEI H, CHEN S, XU C. Bioinformatics analysis of gene expression profile of serous ovarian carcinomas to screen key genes and pathways[J]. J Ovarian Res, 2020, 13: 82. DOI:10.1186/s13048-020-00680-1 |
| [5] |
MI N, CAO J, ZHANG J, et al. Identification of hub genes involved in the occurrence and development of hepatocellular carcinoma via bioinformatics analysis[J]. Oncol Lett, 2020, 20(2): 1695-1708. DOI:10.3892/ol.2020.11752 |
| [6] |
DING ZY, WU HR, ZHANG JM, et al. Expression characteristics of CDC20 in gastric cancer and its correlation with poor prognosis[J]. Int J Clin Exp Pathol, 2014, 7(2): 722-727. |
| [7] |
CHENG L, HUANG YZ, CHEN WX, et al. Cell division cycle proteinising prognostic biomarker of breast cancer[J]. Biosci Rep, 2020, 40(5): BSR20191227. DOI:10.1042/BSR20191227 |
| [8] |
WAN L, TAN M, YANG J, et al. APC (Cdc20) suppresses apoptosis through targeting Bim for ubiquitination and destruction[J]. Dev Cell, 2014, 29(4): 377-391. DOI:10.1016/j.devcel.2014.04.022 |
| [9] |
WU T, ZHANG X, HUANG X, et al. Regulation of cyclin B2 expression and cell cycle G2/M transition by menin[J]. J Biol Chem, 2010, 285(24): 18291-18300. DOI:10.1074/jbc.M110.106575 |
| [10] |
CAI F, LI J, ZHANG J, et al. Knockdown of Circ_CCNB2 sensitizes prostate cancer to radiation through repressing autophagy by the miR-30b-5p/KIF18A axis[J]. Cancer Biother Radiopharm, 2020. DOI:10.1089/cbr.2019.3538 |
| [11] |
RAMOS J, YOO C, FELTY Q, et al. Sensitivity to differential NRF1 gene signatures contributes to breast cancer disparities[J]. J Cancer Res Clin Oncol, 2020. DOI:10.1007/s00432-020-03320-9 |
| [12] |
MO ML, CHEN Z, LI J, et al. Use of serum circulating CCNB2 in cancer surveillance[J]. Int J Biol Markers, 2010, 25(4): 236-242. DOI:10.5301/JBM.2010.6088 |
| [13] |
APARICIO T, IBARRA A, MÉNDEZ J. Cdc45-MCM-GINS, a new power player for DNA replication[J]. Cell Div, 2006, 1(1): 1-5. DOI:10.1186/1747-1028-1-18 |
| [14] |
WEN X, LIU S, CUI M. Effect of BRCA1 on the concurrent chemoradiotherapy resistance of cervical squamous cell carcinoma based on transcriptome sequencing analysis[J]. Biomed Res Int, 2020, 2020: 3598417. DOI:10.1155/2020/3598417 |
| [15] |
POLLOK S, BAUERSCHMIDT C, SÄNGER J, et al. Human Cdc45 is a proliferation-associated antigen[J]. FEBS J, 2007, 274(14): 3669-3684. DOI:10.1111/j.1742-4658.2007.05900.x |
 2021, Vol. 50
2021, Vol. 50