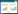文章信息
- 于淼, 吴思, 王爽, 崔畅婉, 路一平, 孙峥嵘
- YU Miao, WU Si, WANG Shuang, CUI Changwan, LU Yiping, SUN Zhengrong
- 中国东北地区人乳头瘤病毒31基因多态性分析
- Analysis of human papilloma virus 31 gene polymorphism in northeast China
- 中国医科大学学报, 2021, 50(12): 1086-1092
- Journal of China Medical University, 2021, 50(12): 1086-1092
-
文章历史
- 收稿日期:2021-01-27
- 网络出版时间:2021-12-07 13:47
宫颈癌是全球女性中常见的恶性肿瘤,人乳头瘤病毒(human papilloma virus,HPV)持续性感染在宫颈癌的发生、发展中发挥关键作用[1]。目前可鉴定出的HPV类型高达200多种,根据其致癌潜力将HPV分为高危型HPV和低危型HPV。HPV16、HPV18、HPV31、HPV33、HPV45、HPV52和HPV58是全世界范围内最常见的高危型HPV。2018年2月至2019年3月间,在中国医科大学附属盛京医院接受HPV检测的患者中,HPV31阳性率约为1.146%。
E6和E7基因编码的早期蛋白是目前高危型HPV研究中最多的癌蛋白。它们是参与人类上皮细胞永生化和转化的关键癌蛋白,并通过与多种宿主蛋白相互作用而发挥作用[2]。因此,存在于E6和E7基因上的突变很可能导致氨基酸序列突变,进而改变蛋白质的结构和功能,从而影响宿主的免疫应答情况[3-4]。CHAGAS等[5]对巴西东北部HPV31 E6和E7基因多态性进行研究,发现非同义突变C285T、A297G、A475G、C520T、C626T、G695A、A743G、C737G可以导致极性、亲水性电位和氨基酸侧链发生变化,进而可能改变癌蛋白折叠,从而影响病毒的致病性。ZHANG等[6]对四川地区HPV31 E6和E7基因多态性进行研究,也验证了这些非同义突变的存在。但是,有关中国东北地区HPV31 E6和E7基因多态性的研究较少。因此,本研究对中国东北地区HPV31 E6和E7基因多态性进行了相关研究。
1 材料与方法 1.1 标本采集收集2018年2月至2019年3月在中国医科大学附属盛京医院进行HPV检测、结果呈HPV31阳性的脱落宫颈细胞DNA标本。共采集到合格DNA标本204例,来源患者年龄为18~69岁,中位年龄41岁。标本采集前已取得患者知情同意,本研究承诺保护患者隐私。
1.2 PCR扩增 1.2.1 引物设计应用Primer 5.0生物信息学软件,以J04353.1为参考序列设计巢氏PCR引物。引物1,正义5’-TAAACTGCCAAGGTTGTG-3’,反义5’-TGTTCCTCCGCTTCCTGT-3’;引物2,正义5’-AAAAGTAGGGAGTGACCG-3’,反义5’-TTCGTCCTCTGAAATGTTG-3’。
1.2.2 扩增体系巢氏PCR第一步扩增体系25 μL(DNA样品3.0 μL,GoTaq® Green Master Mix,2×,12.5 μL,引物F1和R1各1.0 μL,无核酸酶水7.5 μL);第二步扩增体系50 μL(第一步25 μL体系所得扩增产物2.0 μL,GoTaq® Green Master Mix,2×,25 μL,引物F2和R2各2.0 μL,无核酸酶水19.0 μL)。
1.2.3 扩增条件95 ℃预变性2 min;95 ℃变性30 s,55 ℃退火30 s,72 ℃延伸1 min 30 s,重复30个循环,最后72 ℃延伸10 min。
1.3 扩增产物测序将扩增产物用1%琼脂糖凝胶电泳鉴定后,将合格产物送至美国Invitrogen公司北京分公司测序。
1.4 序列比对和系统发育树的构建应用MegaX软件将测序得到的结果与参考序列J04353.1进行序列比对分析,找出突变位点。将比对结果使用MegaX软件中的Neighbor-Joining法构建系统发育树。为确保实验数据准确性,PCR扩增和序列分析至少重复2次。
1.5 选择压力分析应用生物信息学软件包PAML4.9中的codeml程序估测HPV31 E6和E7序列中的阳性选择位点。该程序使用NEI等[7]的方法执行似然比测试,以推断编码区的非同义和同义核苷酸差异。HPV31 E6和E7蛋白序列应用MegaX进行比对。
1.6 蛋白质二级结构预测应用PSIPred server(http://bioinf.cs.ucl.ac.uk/psipred/)预测参考序列的二级结构,随后对突变位点所在的二级结构进行判断。
1.7 表位预测应用ProPred-Ⅰserver(http://www.imtech.res.in/raghava/propred1/)[8],在默认设置下预测人类白细胞抗原(human leukocyte antigen,HLA)Ⅰ类结合表位。应用ProPred server(http://www.imtech.res.in/raghava/propred/)[9],在默认设置下预测HLAⅡ类结合表位。应用ABCpred server(http://www.imtech.res.in/raghava/abcpred/ABC_submission.html),在默认参数下预测HPV31 E6和E7基因的B细胞表位[10]。
2 结果 2.1 HPV31 E6和E7基因突变204例标本中,成功扩增并测序151例,剩余53例因拷贝数低或DNA提取液纯度低等原因导致测序失败。与HPV31 E6参考序列比对,16株(10.60%)病毒株与参考序列完全相同,135株(89.40%)存在基因多态性。共发现25个(5.56%)单核苷酸突变位点,其中14个为同义突变,11个为非同义突变。全部病毒株根据突变情况可分为20个种类,命名为HPV31 E601~HPV31 E620,其中HPV31 E601与参考序列完全相同。HPV31 E6基因中的所有突变见表 1、表 2。在151个HPV31 E7病毒株中共发现8个(2.69%)单核苷酸突变位点,其中3个是同义突变,5个是非同义突变。所有病毒株与参考序列相比均存在核苷酸突变,未发现与参考序列完全相同的病毒株。全部病毒株根据突变情况可分为8个种类,命名为HPV31 E701~HPV31 E708。HPV31 E7基因中的所有突变见表 3。在HPV31 E6和E7中未观察到移码突变或是导致终止密码子提前的突变,并且发现E7基因较E6基因更为保守。
| Category | Nucleotide position | n | Lineage | ||||||||||||
| 11 | 21 | 27 | 69 | 81 | 90 | 141 | 154 | 178 | 190 | 202 | 207 | 213 | |||
| Reference J04353.1 | A | A | T | C | A | T | T | A | C | A | A | T | A | A | |
| HPV31 E601 | - | - | - | - | - | - | - | - | - | - | - | - | - | 16 | A |
| HPV31 E602 | - | - | - | - | - | - | - | - | T | - | - | - | T | 21 | C |
| HPV31 E603 | - | - | - | - | - | - | - | - | T | - | - | - | T | 2 | C |
| HPV31 E604 | - | - | - | - | - | - | - | - | T | - | - | - | T | 29 | C |
| HPV31 E605 | - | - | - | - | - | - | C | - | - | G | - | - | T | 6 | B |
| HPV31 E606 | - | - | - | T | - | - | - | - | - | - | - | - | - | 49 | A |
| HPV31 E607 | - | - | - | - | - | - | - | - | T | - | - | - | T | 1 | C |
| HPV31 E608 | - | - | - | - | - | - | - | C | - | G | - | - | T | 9 | B |
| HPV31 E609 | - | - | - | T | - | - | - | - | - | - | - | - | - | 1 | A |
| HPV31 E610 | - | - | - | - | G | - | - | - | T | - | - | - | T | 1 | C |
| HPV31 E611 | - | - | - | - | - | - | - | - | T | - | - | - | T | 1 | C |
| HPV31 E612 | - | - | - | - | - | - | - | - | T | - | - | - | T | 1 | C |
| HPV31 E613 | - | G | - | - | - | - | - | - | T | - | - | - | T | 1 | C |
| HPV31 E614 | - | - | - | - | - | C | - | - | T | - | - | - | T | 1 | C |
| HPV31 E615 | - | - | A | - | - | - | - | - | - | - | - | - | - | 6 | A |
| HPV31 E616 | - | - | - | - | - | - | - | - | T | - | - | C | T | 1 | C |
| HPV31 E617 | - | - | - | T | - | - | - | - | - | - | - | - | - | 1 | A |
| HPV31 E618 | - | - | - | - | - | - | - | - | T | - | - | - | T | 1 | C |
| HPV31 E619 | - | - | - | - | - | - | - | - | T | - | C | - | T | 2 | C |
| HPV31 E620 | C | - | - | - | - | - | - | - | - | - | - | - | - | 1 | A |
| aa mutation | |||||||||||||||
| N | E | P | Y | R | C | F | I | H | T | R | F | S | |||
| 4 | 7 | 9 | 23 | 27 | 30 | 47 | 52 | 60 | 64 | 68 | 69 | 71 | |||
| T | - | - | - | - | - | - | L | Y | A | - | - | - | |||
| Secondary structure | C | - | - | - | - | - | - | E | E | H | - | - | - | ||
| Nucleotide and amino acid mutations are presented by the corresponding letters. -,represent that nucleotides are identical to the reference sequence;aa,amino acid;n,the number of each species among the samples examined;C,random coil;E,extended strand;H,alpha helix. | |||||||||||||||
| Category | Nucleotide position | n | Lineage | |||||||||||
| 219 | 297 | 321 | 331 | 368 | 375 | 383 | 391 | 409 | 413 | 430 | 439 | |||
| Reference J04353.1 | A | G | A | T | A | C | T | A | A | C | C | A | A | |
| HPV31 E601 | - | - | - | - | - | - | - | - | - | - | - | - | 16 | A |
| HPV31 E602 | G | A | G | - | - | - | - | - | - | T | - | - | 21 | C |
| HPV31 E603 | - | A | - | - | - | - | - | - | - | T | - | - | 2 | C |
| HPV31 E604 | - | A | G | - | - | - | - | - | - | T | - | - | 29 | C |
| HPV31 E605 | - | - | - | - | G | - | - | - | - | T | - | - | 6 | B |
| HPV31 E606 | - | - | - | - | - | - | - | - | - | - | - | - | 49 | A |
| HPV31 E607 | - | A | G | - | - | - | - | - | G | T | - | - | 1 | C |
| HPV31 E608 | - | - | - | - | G | - | - | - | - | T | - | - | 9 | B |
| HPV31 E609 | - | - | - | - | - | T | - | - | - | - | - | - | 1 | A |
| HPV31 E610 | - | A | G | - | - | - | - | - | - | T | - | G | 1 | C |
| HPV31 E611 | G | A | G | - | - | T | - | - | - | T | - | - | 1 | C |
| HPV31 E612 | - | A | G | C | - | - | - | - | - | T | - | - | 1 | C |
| HPV31 E613 | - | A | G | - | - | - | C | - | - | T | - | - | 1 | C |
| HPV31 E614 | G | A | G | - | - | - | - | - | - | T | - | - | 1 | C |
| HPV31 E615 | - | - | - | - | - | - | - | - | - | - | - | - | 6 | A |
| HPV31 E616 | G | A | G | - | - | - | - | - | - | T | - | - | 1 | C |
| HPV31 E617 | - | - | - | - | - | - | - | - | - | - | T | - | 1 | A |
| HPV31 E618 | G | A | G | - | - | - | - | C | - | T | - | - | 1 | C |
| HPV31 E619 | G | A | G | - | - | - | - | - | - | T | - | - | 2 | C |
| HPV31 E620 | - | - | - | - | - | - | - | - | - | - | - | - | 1 | A |
| aa mutation | ||||||||||||||
| V | L | Q | C | K | F | I | R | I | A | R | T | |||
| 73 | 99 | 107 | 111 | 123 | 125 | 128 | 131 | 137 | 138 | 144 | 147 | |||
| - | - | - | R | R | - | T | - | V | V | C | A | |||
| Secondary structure | - | - | - | C | C | - | E | - | C | C | C | C | ||
| Abbreviations as in table 1 | ||||||||||||||
| Category | Nucleotide position | n | Lineage | |||||||
| 21 | 29 | 67 | 111 | 136 | 149 | 184 | 228 | |||
| Reference J04353.1 | G | A | C | C | G | C | A | T | A | |
| HPV31 E701 | - | - | - | - | - | - | G | - | 3 | A |
| HPV31 E702 | A | - | - | T | A | - | G | - | 62 | C |
| HPV31 E703 | - | - | T | - | - | - | G | - | 69 | A |
| HPV31 E704 | - | - | T | T | A | - | G | - | 6 | B |
| HPV31 E705 | - | - | - | T | A | - | G | - | 8 | C |
| HPV31 E706 | - | G | T | - | - | - | G | - | 1 | A |
| HPV31 E707 | - | - | T | - | - | - | G | C | 1 | A |
| HPV31 E708 | - | - | - | T | A | A | G | - | 1 | C |
| aa mutation | ||||||||||
| T | D | H | V | E | S | K | I | |||
| 7 | 10 | 23 | 37 | 46 | 50 | 62 | 76 | |||
| - | G | Y | - | K | Y | E | - | |||
| Secondary structure | - | H | C | - | C | C | C | - | ||
| Abbreviations as in table 1 | ||||||||||
2.2 系统发育分析
在全部的151例HPV31 E6序列中,A系出现频率最高(74例,49.01%),其次是C系(62例,41.06%),B系最低(15例,9.93%),见图 1A。在151个HPV31 E7序列中,同样是A系出现频率最高(74例,49.01%),其次是C系(71例,47.02%),B系最低(6例,3.97%),见图 1B。
|
| A, HPV31 E6;B, HPV31 E7. *, sequences obtained in this study. Others were the variants previously reported in the NCBI. Numbers above the branches are the bootstrap values. 图 1 HPV31 E6和HPV31 E7系统发育树 Fig.1 Neighbor-joining tree for HPV31 E6 and E7 |
2.3 选择压力分析
使用PAML4.9软件中的codeml程序推测所得突变是否为阳性选择位点,但在本研究所得突变中并未发现阳性选择位点。
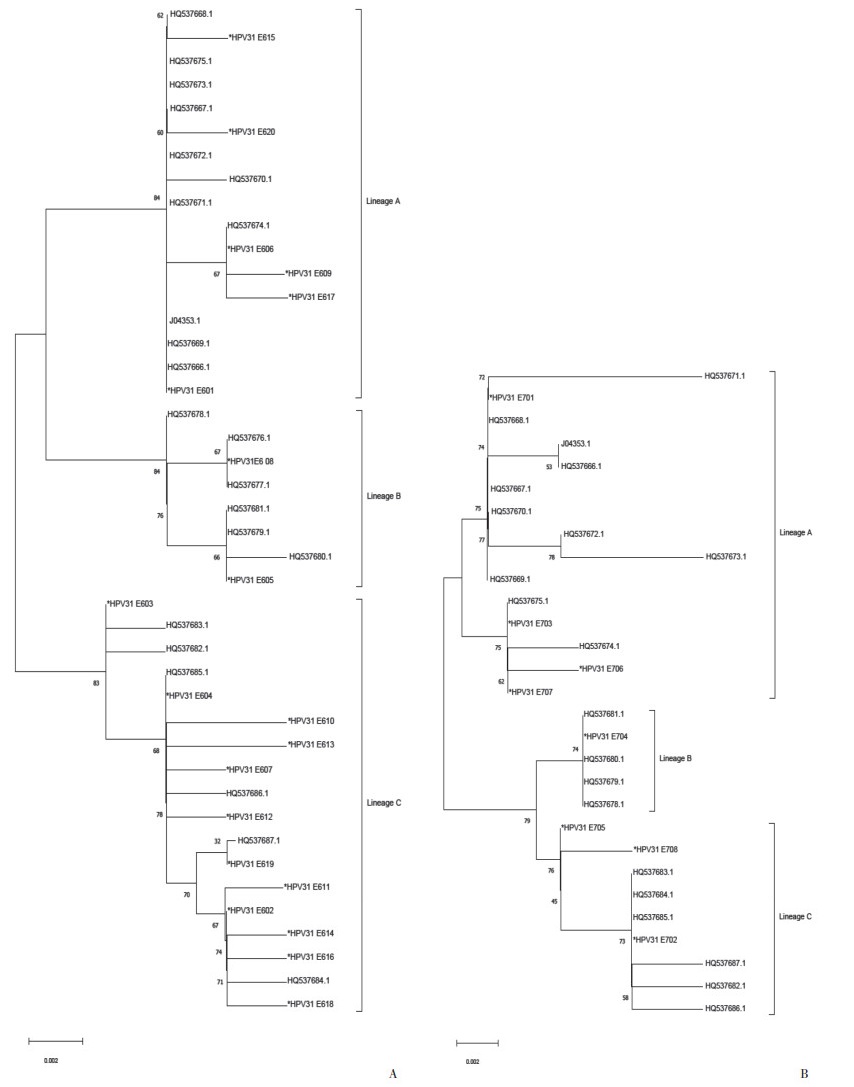
2.4 蛋白质二级结构预测HPV31 E6中共11个非同义突变,其中7个发生在无规则卷曲结构,3个发生在线性结构,1个发生在α螺旋结构。HPV31 E6和E7蛋白质二级结构见图 2。序列中非同义突变位点所在二级结构见表 1~3。
|
| A, HPV31 E6;B, HPV31 E7 图 2 HPV31 E6和HPV31 E7蛋白质二级结构 Fig.2 Protein secondary structure of HPV31 E6 and E7 |
2.5 表位预测
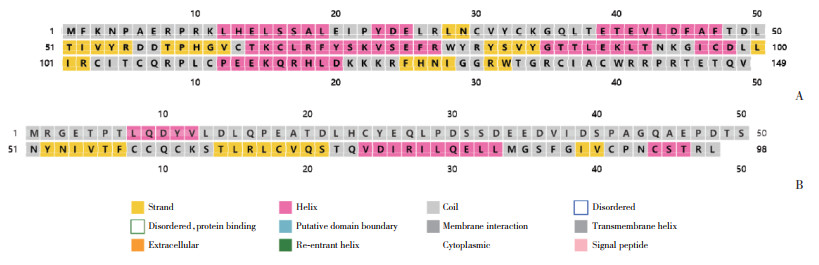
HPV31 E6中预测到MHCⅠ类表位9个,MHCⅡ类表位7个,B细胞表位5个。大部分HPV31 E6中的非同义突变存在于预测表位中(图 3A)。HPV31 E7中预测到MHCⅠ类表位4个,MHCⅡ类表位5个,B细胞表位2个。大部分HPV31 E7中的非同义突变存在于预测表位中(图 3B)。
|
| A, HPV31 E6; B, HPV31 E7. In blue, a consensus of the predicted MHCⅠ epitopes binding no fewer than 10 HLAⅠ alleles. In green, a consensus of the predicted MHCⅡ epitopes binding no fewer than 10 HLAⅡ alleles. In red, a consensus of the predicted B-cell epitopes (score ≥0.85). Black arrows indicate the non-synonymous mutation 图 3 HPV31 E6和HPV31 E7表位预测 Fig.3 Predicted MHC and B-cell epitopes of HPV31 E6 and E7 proteins |
3 讨论
持续感染高危型HPV是宫颈癌发生、发展的关键。E6和E7基因编码的早期蛋白是目前高危型HPV中研究最多的关键致癌蛋白。已有大量研究证实,HPV16和HPV18 E6和E7基因多态性在病毒的生物学功能和临床疾病进展中具有重要意义。HPV31作为较高流行类型,有研究[11]表明其可使宫颈上皮高级别病变的风险显著增加。本研究对中国东北地区HPV31 E6和E7基因多态性分布进行了研究。
本研究中,E7的突变频率较E6低,即与E6基因相比E7基因更为保守。在HPV31 E6基因中,A213T是最常见的同义突变,C413T(A138V)是最常见的非同义突变。在HPV31 E7基因中,C111T是最常见的同义突变,A184G(K62E)是最常见的非同义突变。有研究[12-13]表明,HPV的型内突变可使疫苗的保护作用降低,改变病毒的致病能力,使病毒逃逸风险和致癌风险增加。当选择E6、E7作为引物设计或诊断检测以及疫苗设计的靶标时,有必要将这些突变位点考虑在内。本研究中,发生在不同蛋白质二级结构中的非同义突变很可能使蛋白质极性、亲水性电位和氨基酸侧链发生变化,从而可能改变癌蛋白折叠[13],进而使蛋白质功能发生改变。因此,非同义突变可能会通过改变E6和E7癌蛋白的结构来影响病毒的致癌潜力、与宿主细胞的相互作用以及疫苗效果。
研究[14]报道HPV31 A谱系为美国女性中最广泛流行的谱系(41.7%),其次是C谱系(37.2%),B谱系最低(21.1%)。而在意大利,C谱系(65.8%)最为流行,其次是B谱系(29.3%),A谱系(4.9%)流行程度最低[15]。本研究证实中国东北地区的分布情况与美国大致相同,与意大利存在差异,表明HPV31 E6和E7基因多态性的分布情况与地域和种族有一定相关性。
MHCⅠ/Ⅱ类表位和B细胞表位在疫苗设计和诊断方法的研究中具有重要意义。本研究在E6基因中预测到MHCⅠ类表位9个,MHCⅡ类表位7个,B细胞表位5个。非同义突变A11C(N4T)、T331C(C111R)和A154C(I52L)存在于MHCⅠ类表位,C178T(H60Y)、C430T(R144C)、A439G(T147A)同时存在于MHCⅡ类表位和B细胞表位,A190G(T64A)、T383C(I128T)、A409G(I137V)、C413T(A138V)存在于B细胞表位。在E7基因中预测到MHCⅠ类表位4个,MHCⅡ类表位5个,B细胞表位2个。非同义突变A29G(D10G)、C149A(S50Y)存在于MHCⅠ类表位,G136A(E46K)同时存在于MHCⅠ类表位和B细胞表位。这些突变均可能使表位发生改变,从而使表位的结合能力发生变化,影响其与宿主细胞的免疫作用。
综上所述,HPV31 E6和E7基因在中国东北地区呈基因多态性分布,本研究结果为更深入了解基因多态性的地理分布情况以及基因探针、疫苗和治疗方案的设计提供了理论依据。
| [1] |
BOSCH FX, MANOS MM, MUÑOZ N, et al. Prevalence of human papillomavirus in cervical cancer: a worldwide perspective[J]. J Natl Cancer Inst, 1995, 87(11): 796-802. DOI:10.1093/jnci/87.11.796 |
| [2] |
RAMPIAS T, SASAKI C, PSYRRI A. Molecular mechanisms of HPV induced carcinogenesis in head and neck[J]. Oral Oncol, 2014, 50(5): 356-363. DOI:10.1016/j.oraloncology.2013.07.011 |
| [3] |
BERNARD HU, BURK RD, CHEN ZG, et al. Classification of papi- llomaviruses (PVs) based on 189 PV types and proposal of taxonomic amendments[J]. Virology, 2010, 401(1): 70-79. DOI:10.1016/j.virol.2010.02.002 |
| [4] |
MUÑOZ N, BOSCH FX, DE SANJOSÉ S, et al. Epidemiologic cla- ssification of human papillomavirus types associated with cervical cancer[J]. N Engl J Med, 2003, 348(6): 518-527. DOI:10.1056/NEJMoa021641 |
| [5] |
CHAGAS BS, BATISTA MV, GUIMARÃES V, et al. New variants of E6 and E7 oncogenes of human papillomavirus type 31 identified in northeastern Brazil[J]. Gynecol Oncol, 2011, 123(2): 284-288. DOI:10.1016/j.ygyno.2011.07.008 |
| [6] |
ZHANG JH, ZHANG SH, WANG MT, et al. Genetic variability in E5, E6, E7 and L1 genes of human papillomavirus type 31[J]. Mol Med Rep, 2018, 17(4): 5498-5507. DOI:10.3892/mmr.2018.8500 |
| [7] |
NEI M, GOJOBORI T. Simple methods for estimating the numbers of synonymous and nonsynonymous nucleotide substitutions[J]. Mol Biol Evol, 1986, 3(5): 418-426. DOI:10.1093/oxfordjournals.molbev.a040410 |
| [8] |
SINGH H, RAGHAVA GP. ProPred1:prediction of promiscuous MHC class-Ⅰbinding sites[J]. Bioinformatics, 2003, 19(8): 1009-1014. DOI:10.1093/bioinformatics/btg108 |
| [9] |
SINGH H, RAGHAVA GPS. ProPred: prediction of HLA-DR binding sites[J]. Bioinformatics, 2001, 17(12): 1236-1237. DOI:10.1093/bioinformatics/17.12.1236 |
| [10] |
SAHA S, RAGHAVA GP. Prediction of continuous B-cell epitopes in an antigen using recurrent neural network[J]. Proteins, 2006, 65(1): 40-48. DOI:10.1002/prot.21078 |
| [11] |
XI LF, SCHIFFMAN M, KOUTSKY LA, et al. Lineages of oncogenic human papillomavirus types other than type 16 and 18 and risk for cervical intraepithelial neoplasia[J]. J Natl Cancer Inst, 2014, 106(10): dju270. DOI:10.1093/jnci/dju270 |
| [12] |
CHEN Z, JING Y, WEN Q, et al. L1 and L2 gene polymorphisms in HPV-58 and HPV-33: implications for vaccine design and diagnosis[J]. Virol J, 2016, 13(1): 167.DOI: 10.1186s12985-016-0629-9.
|
| [13] |
CHEN Z, JING Y, WEN Q, et al. E6 and E7 gene polymorphisms in human papillomavirus types-58 and 33 identified in southwest China[J]. PLoS One, 2017, 12(1): e0171140. DOI:10.1371/journal.pone.0171140 |
| [14] |
LIU X, SCHIFFMAN M, HULBERT A, et al. Association of human papillomavirus 31 DNA load with risk of cervical intraepithelial neoplasia grades 2 and 3[J]. J Clin Microbiol, 2015, 53(11): 3451-3457. DOI:10.1128/jcm.01279-15 |
| [15] |
FERENCZI A, GYÖNGYÖSI E, SZALMÁS A, et al. Sequence variation of human papillomavirus type 31 long control region: phylogenetic and functional implications[J]. J Med Virol, 2013, 85(5): 852-859. DOI:10.1002/jmv.23542 |
 2021, Vol. 50
2021, Vol. 50