文章信息
- 刘双, 林征, 俞凯莉, 陈辉林, 饶雯清, 胡志坚
- LIU Shuang, LIN Zheng, YU Kaili, CHEN Huilin, RAO Wenqing, HU Zhijian
- ZNF568基因表达联合DNA甲基化分析与食管癌预后的关联
- Integrative analysis of ZNF568 gene expression and DNA methylation predicts prognosis of esophageal cancer
- 中国医科大学学报, 2021, 50(10): 890-893,898
- Journal of China Medical University, 2021, 50(10): 890-893,898
-
文章历史
- 收稿日期:2020-12-04
- 网络出版时间:2021-09-30 11:04
2. 泉州市安溪县医院放疗科, 福建 泉州 352400
2. Department of Radiation Therapy, Anxi County Hospital, Quanzhou 352400, China
我国是食管癌高发地区,2017年《国家癌症中心最新研究报告》报道,食管癌发病率为14.09/10万,居我国肿瘤发病第五位[1]。尽管治疗技术在不断改进[2],但患者总体预后仍然较差[3]。锌指蛋白(zinc finger protein,ZNF)是最大的DNA结合蛋白家族,ZNF DNA结合域通常与核酸酶或其他效应蛋白融合介导表观遗传反应[4],调节基因的表达,进而影响肿瘤的发生、发展。研究[5]发现,ZNF568基因低表达与胃癌不良预后相关,可作为判断肿瘤预后的标志物。然而,目前尚无ZNF568基因表达联合DNA甲基化与食管癌预后的关联研究。因此,本研究通过联合分析癌症基因组图谱(The Cancer Genome Atlas,TCGA)数据库ZNF568基因表达和DNA甲基化与食管癌预后的关联,为食管癌预后的预测提供理论依据。
1 材料与方法 1.1 TCGA数据库数据下载与预处理通过TCGA数据库官网(https://portal.gdc.cancer.gov),下载食管癌DNA甲基化数据、基因表达数据与临床数据,使用Perl脚本进行注释,并对注释后的数据进行预处理。
1.2 癌细胞系百科全书(Cancer Cell Line Encyclopedia,CCLE)数据库数据下载通过CCLE数据库官网(https://portals.broadinstitute.org/ccle/),下载食管癌ZNF568基因表达和DNA甲基化数据,验证ZNF568基因表达和DNA甲基化之间的关联。
1.3 统计学分析采用R软件中的limma包对DNA甲基化数据进行芯片归一化处理,并对食管癌癌组织与癌旁组织进行DNA甲基化差异分析;使用edgeR包分析食管癌癌组织与癌旁组织ZNF568基因的表达情况;筛选条件为错误发现率(false discovery rate,FDR)=0.05,fold change=1。ZNF568基因表达水平与DNA甲基化的关联分析使用Spearman秩相关。采用Kaplan-Meier法绘制生存曲线,通过log-rank检验进行生存曲线的比较;使用Cox回归模型对ZNF568基因表达和DNA甲基化与食管癌患者预后的关联进行单因素和多因素分析,计算风险比(hazard ratio,HR)和95%可信区间(confidence interval,CI)。建立预测Cox回归分析模型列线图,内部验证采用Bootstrap法,采用一致性指数(C指数)评价模型准确度。本研究使用R软件(3.5.3)进行统计分析,显著性检验为双侧检验,检验水准α=0.05。
2 结果 2.1 TCGA数据库食管癌癌组织和癌旁组织ZNF568基因表达和DNA甲基化差异分析采用R软件中的limma包对186例食管癌癌组织和16例癌旁组织进行ZNF568 DNA甲基化差异分析,发现ZNF568基因在癌组织中的甲基化水平高于癌旁组织(FDR < 0.001);采用edger包对159例食管癌癌组织与10例癌旁组织进行ZNF568基因表达差异分析,发现ZNF568基因在癌旁组织中的表达水平高于癌组织(FDR < 0.001)。见图 1。
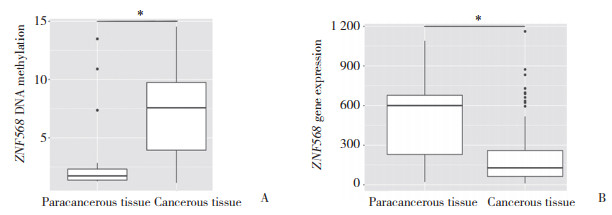
|
| A, ZNF568 DNA methylation; B, ZNF568 gene expression. *P < 0.05. 图 1 TCGA数据库中食管癌癌组织和癌旁组织ZNF568基因表达和DNA甲基化 Fig.1 ZNF568 gene expression and DNA methylation in esophageal cancerous and paracancerous tissues in the Cancer Genome Atlas database |
2.2 DNA甲基化与基因表达水平的关联分析
对159例癌组织中ZNF568基因表达水平与DNA甲基化进行关联分析,结果发现,ZNF568基因表达水平与DNA甲基化存在负相关(P < 0.001),见图 2。
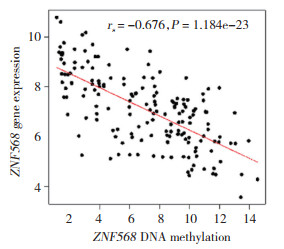
|
| 图 2 TCGA数据库中ZNF568基因表达水平与DNA甲基化的关联分析 Fig.2 Correlation analysis of ZNF568 gene expression level and DNA methylation in TCGA database |
2.3 CCLE数据库验证ZNF568基因表达水平与DNA甲基化的关联性
下载CCLE数据库中食管癌细胞系(17种食管癌细胞株)ZNF568基因表达、DNA甲基化数据,Spearman秩相关分析发现,ZNF568基因表达水平与DNA甲基化存在负相关(P = 0.002),见图 3。
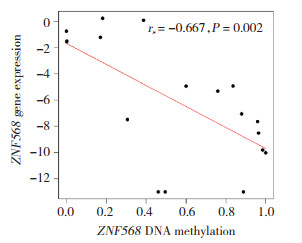
|
| 图 3 CCLE数据库中ZNF568基因表达水平与DNA甲基化的关联分析 Fig.3 Correlation analysis of ZNF568 gene expression level and DNA methylation in the Cancer Cell Line Encyclopedia database |
2.4 联合分析ZNF568基因表达和DNA甲基化与食管癌预后的关联
Kaplan-Meier法绘制的生存曲线和log-rank检验分析发现,ZNF568基因表达和DNA甲基化与食管癌生存无关联(P = 0.059,图 4)。调整性别、年龄、分化程度和TNM分期后,多因素Cox回归分析发现,ZNF568基因表达水平和DNA甲基化联合分析与食管癌预后存在关联,ZNF568基因高表达、DNA低甲基化能改善食管癌预后(HR = 0.70,95% CI:0.51~0.96,P = 0.043),见表 1。
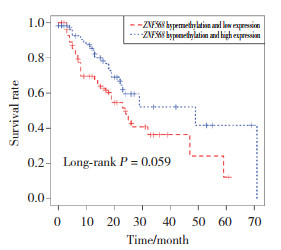
|
| 图 4 ZNF568基因表达水平和DNA甲基化联合分析与食管预后的生存曲线 Fig.4 Kaplan-Meier overall survival curves according to combined ZNF568 gene expression and DNA methylation |
| Variable | Univariate Cox regression | Multivariate Cox regression * | |||
| HR | 95% CI | HR | 95% CI | ||
| Sex | |||||
| Female | 1.00 | - | 1.00 | - | |
| Male | 2.02 | 0.80-5.14 | 1.46 | 0.56-3.80 | |
| Age(year) | |||||
| ≤ 60 | 1.00 | - | 1.00 | - | |
| > 60 | 0.97 | 0.55-1.76 | 0.82 | 0.45-1.50 | |
| Differentiation | |||||
| Well/moderate | 1.00 | - | 1.00 | - | |
| Poor | 1.59 | 0.86-2.95 | 1.58 | 0.83-3.05 | |
| TNM stage | |||||
| Ⅰ/Ⅱ | 1.00 | - | 1.00 | - | |
| Ⅲ/ Ⅳ | 2.56 | 1.41-4.65 | 2.29 | 1.22-4.31 | |
| ZNF568 expression | |||||
| Hypermethylation and low expression | 1.00 | - | 1.00 | - | |
| Hypomethylation and high expression | 0.75 | 0.55-1.01 | 0.70 | 0.51-0.96 | |
| * Adjusted by sex,age,differentiation,TNM stage,and ZNF568 expression. | |||||
2.5 构建ZNF568基因表达和DNA甲基化联合分析的列线图
使用多因素Cox回归分析的变量构建列线图(图 5),ZNF568基因表达和DNA甲基化联合分析能很好地预测食管癌预后,C指数为0.697(95% CI:0.543~0.851)。

|
| 图 5 ZNF568基因表达和DNA甲基化联合分析预测食管癌总生存的列线图 Fig.5 Nomogram for overall survival according to combined ZNF568 gene expression and DNA methylation |
3 讨论
TCGA是一个国际项目,收集了高质量的肿瘤和正常组织样本,并在多个高通量平台上进行了分子分析,收录了各种人类肿瘤的基因组变异、mRNA表达、DNA甲基化、临床信息等数据[6]。在大数据时代的背景下,生物信息学分析利用如TCGA数据库等高通量数据库,在肿瘤的全面研究中发挥了重要作用。本研究通过TCGA数据库联合分析食管癌DNA甲基化和基因表达的数据发现,ZNF568基因高表达、DNA低甲基化能改善食管癌预后,使用ZNF568基因表达和DNA甲基化联合分析构建的列线图能很好地预测食管癌预后,C指数为0.697,说明ZNF568基因表达和DNA甲基化联合分析可作为预测食管癌预后的标志物。
ZNF是最大的DNA结合蛋白家族,可作为真核生物中的转录因子,并通过特征性锌指结构域选择性结合靶基因启动子中的特定DNA序列[7-8]。ZNF DNA结合域通常与核酸酶或其他效应蛋白融合介导表观遗传反应[4]。不同肿瘤的研究[9]已经证明宿主DNA高甲基化导致肿瘤抑制基因沉默,而低甲基化区域与癌基因的表达增加。目前,尚无ZNF568基因表达联合DNA甲基化与食管癌预后的研究,但ZNF家族基因DNA甲基化和基因表达与肿瘤预后已有研究报道。使用TCGA数据库,ZHANG等[10]发现,ZNF726基因低表达能改善结肠癌的预后(HR = 0.55,95% CI:0.34~0.89),使用CCLE数据库进一步评估结肠癌细胞中ZNF726基因表达,发现甲基化的调节对ZNF726基因起重要作用。此外,在肺鳞状细胞癌的研究[11]中发现,ZNF454基因是肺鳞状细胞癌预后相关的甲基化驱动基因,通过联合分析ZNF454基因表达和DNA甲基化的数据,发现ZNF454基因低表达、DNA高甲基化能改善肺鳞状细胞癌的预后(P < 0.05);对肺鳞状细胞癌ZNF454甲基化驱动基因进行通路分析,发现ZNF454基因主要作用的途径为“通用转录”“ RNA聚合酶Ⅱ转录”和“基因表达”,提示ZNF454基因表达和DNA高甲基化存在关联。以上的研究说明ZNF家族基因甲基化与肿瘤预后存在关联,可作为判断肿瘤预后的标志物。
目前,仅有一篇研究报道了ZNF568基因表达与胃癌的关联,WANG等[5]使用TCGA数据库发现,ZNF568基因等5个基因是胃癌的突变基因,ZNF568基因低表达与胃癌预后存在关联(P < 0.05),ZNF568基因表达可作为预测胃癌预后的标志物。然而,目前尚无ZNF568基因表达和DNA甲基化与肿瘤预后的相关研究。本研究通过使用TCGA数据库,联合分析ZNF568基因表达和DNA甲基化发现,ZNF568基因高表达、DNA低甲基化与食管癌预后相关;构建ZNF568基因表达和DNA甲基化联合分析的列线图,结果显示,使用该基因能很好地预测食管癌的预后(C指数为0.697),说明ZNF568基因表达和DNA甲基化联合分析可用于预测食管癌预后。与经典遗传学改变不同,甲基化修饰的过程动态可逆[12],因此,抑制ZNF568 DNA甲基化作用可使上调ZNF568基因重新表达,这一特性也为食管癌的临床治疗提供新的思路。
综上所述,ZNF568基因高表达、DNA低甲基化与食管癌预后相关,检测食管癌ZNF568基因表达水平和DNA甲基化程度有助于判断食管癌的预后。
| [1] |
CHEN W, ZHENG R, ZHANG S, et al. Cancer incidence and mortality in China in 2013:an analysis based on urbanization level[J]. Chin J Cancer Res, 2017, 29(1): 1-10. DOI:10.21147/j.issn.1000-9604.2017.01.01 |
| [2] |
ZHENG Y, FU S, HE T, et al. Predicting prognosis in resected esophageal squamous cell carcinoma using a clinical nomogram and recursive partitioning analysis[J]. Eur J Surg Oncol, 2018, 44(8): 1199-1204. DOI:10.1016/j.ejso.2018.04.011 |
| [3] |
张思维, 郑荣寿, 左婷婷, 等. 中国食管癌死亡状况和生存分析[J]. 中华肿瘤杂志, 2016, 38(9): 709-715. DOI:10.3760/cma.j.issn.0253-3766.2016.09.014 |
| [4] |
GRIMMER MR, STOLZENBURG S, FORD E, et al. Analysis of an artificial zinc finger epigenetic modulator: widespread binding but limited regulation[J]. Nucleic Acids Res, 2014, 42(16): 10856-10868. DOI:10.1093/nar/gku708 |
| [5] |
WANG H, SHEN L, LI Y, et al. Integrated characterisation of cancer genes identifies key molecular biomarkers in stomach adenocarcinoma[J]. J Clin Pathol, 2020, 73(9): 579-586. DOI:10.1136/jclinpath-2019-206400 |
| [6] |
CANCER GENOME ATLAS RESEARCH NETWORK. Comprehensive molecular characterization of gastric adenocarcinoma[J]. Nature, 2014, 513(7517): 202-209. DOI:10.1038/nature13480 |
| [7] |
LAW DJ, LABUT EM, ADAMS RD, et al. An isoform of ZBP-89 predisposes the colon to colitis[J]. Nucleic Acids Res, 2006, 34(5): 1342-1350. DOI:10.1093/nar/gkl022 |
| [8] |
CHATTERJEE A, ECCLES MR. DNA methylation and epigenomics: new technologies and emerging concepts[J]. Genome Biol, 2015, 16: 103. DOI:10.1186/s13059-015-0674-5 |
| [9] |
HATTORI N, NIWA T, ISHIDA T, et al. Antibiotics suppress colon tumorigenesis through inhibition of aberrant DNA methylation in an azoxymethane and dextran sulfate sodium colitis model[J]. Cancer Sci, 2019, 110(1): 147-156. DOI:10.1111/cas.13880 |
| [10] |
ZHANG H, SUN X, LU Y, et al. DNA-methylated gene markers for colorectal cancer in TCGA database[J]. Exp Ther Med, 2020, 19(4): 3042-3050. DOI:10.3892/etm.2020.8565 |
| [11] |
ZHU Q, WANG J, ZHANG Q, et al. Methylation-driven genes PMPCAP1, SOWAHC and ZNF454 as potential prognostic biomarkers in lung squamous cell carcinoma[J]. Mol Med Rep, 2020, 21(3): 1285-1295. DOI:10.3892/mmr.2020.10933 |
| [12] |
HU LF. Epigenetic regulation of autophagy[J]. Adv Exp Med Biol, 2019, 1206: 221-236. DOI:10.1007/978-981-15-0602-4_11 |
 2021, Vol. 50
2021, Vol. 50




