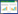文章信息
- 郑博文, 刘经纬, 孙丽萍, 邢承忠
- ZHENG Bowen, LIU Jingwei, SUN Liping, XING Chengzhong
- DDB2基因不同转录本在胃癌和正常胃组织中的表达差异及其对胃癌细胞增殖的影响
- Expression of different DDB2 gene transcripts in gastric cancer and normal gastric tissues and their effects on the proliferation of gastric cancer cells
- 中国医科大学学报, 2021, 50(1): 30-33, 39
- Journal of China Medical University, 2021, 50(1): 30-33, 39
-
文章历史
- 收稿日期:2020-05-18
- 网络出版时间:2020-12-22 8:55
2. 中国医科大学附属第一医院肿瘤病因与筛查研究室, 沈阳 110001;
3. 中国医科大学附属第一医院辽宁省教育厅消化道肿瘤病因与预防重点实验室, 沈阳 110001
2. Tumor Etiology and Screening Department of Cancer Institute and General Surgery, The First Hospital of China Medical University, Shenyang 110001, China;
3. Key Laboratory of GI Cancer Etiology and Prevention in Liaoning Province, The First Hospital of China Medical University, Shenyang 110001, China
胃癌是最常见的恶性肿瘤之一,其发病率和死亡率在所有癌症中分别位居第5位和第3位[1]。炎症和感染导致的DNA损伤积累会增加胃黏膜细胞的基因组不稳定性,进而诱发胃癌[2]。核苷酸切除修复系统(nucleotide excision repair,NER)是一种功能强大的DNA损伤修复系统,在保持胃黏膜细胞基因组的稳定性和完整性方面具有重要作用[3]。DNA损伤结合蛋白2 (DNA damage-binding protein 2,DDB2)是一种由DDB2基因编码的分子量为48×103的蛋白质,在NER修复通路中,它参与构成的DDB复合物能够识别DNA损伤部位,进而启动NER过程并完成损伤修复[4]。近期研究[5]表明,DDB2亦可通过NER通路之外的途径影响结直肠癌等多种癌症的发生和发展,其在调控肿瘤细胞生物学行为方面具有重要作用。
选择性剪切是将基因或mRNA前体转录产生的RNA外显子以多种方式剪切重连并产生多种不同长度mRNA的过程,这些不同长度的mRNA亦被称为剪接变异体[6]。选择性剪切是基因表达调控的关键步骤,通过选择在特定时间和特定细胞中产生剪接变异体,能够增加细胞的转录多样性;失调的选择性剪切经常出现在人类肿瘤中,并通过多种途径调控癌症的进程[7]。因此,阐明重要基因的不同剪接变异体在胃癌发生、进展中的功能和调控至关重要。检索NCBI数据库的子数据库Nucleotide可知,DDB2基因有2个已被证实的剪接变异体WT和D1。DDB2基因的野生型转录本WT含有10个外显子,全长1 870 bp,编码含427个氨基酸的蛋白;而变异型转录本D1长度较短,N端和C端与常见转录本相同,但缺少4个外显子(外显子4、5、6、7),全长717 bp,编码含238个氨基酸的蛋白。此外有研究[8]指出,DDB2基因rs830083多态突变型GG基因型显著提高了胃癌的发病风险,而rs830083正好位于D1转录本缺失的第4外显子附近,提示rs830083多态突变型GG基因型可能参与调控D1转录本的表达,进而影响胃癌的发生。目前的研究表明,DDB2基因D1转录本在胃癌中可能具有重要作用,且DDB2基因的2种转录本在胃癌中的表达水平与功能尚不明确。因此,本研究通过实时定量PCR (quantitative real-time PCR,RT-qPCR)、CCK-8、Western blotting等方法,深入探讨DDB2基因的2种剪接变异体在胃癌细胞中表达水平的差异及其对胃癌细胞生物学行为的影响。
1 材料与方法 1.1 材料 1.1.1 研究对象选择于中国医科大学附属第一医院肛肠外科接受胃癌手术的患者22例,其中男15例,女7例,平均年龄59.2岁。术后30 min内留取胃癌癌灶和远端正常胃组织,-80 ℃冻存待用。全部病例均经病理组织学检查确诊。本研究获得中国医科大学附属第一医院伦理委员会批准。
1.1.2 细胞株胃癌细胞株BGC823,来自中国医科大学附属第一医院肿瘤研究所第三研究室细胞库。
1.2 方法 1.2.1 组织RNA提取、反转录及RT-qPCR检测采用RNAiso Plus (日本TaKaRa公司)提取22对胃癌组织和远端正常胃组织的总RNA,然后用RT Master Mix (日本TaKaRa公司)将其反转为cDNA,最后将cDNA和TB Green Premix Ex Taq Ⅱ (日本TaKaRa公司)适量比例混合,以完成qPCR。根据PCR所得的△Ct值,计算标准化的2-△△Ct。DDB2基因WT转录本引物:上游5’-CCAACCAGTTTTACGCCTCCTC-3’,下游5’-TGTCTCCTGTGACCACCATTCG-3’;DDB2基因D1转录本引物:上游,5’-CACCTTCATCAAAGGGGCAG-3’,下游5’-GAACACGTCGATCGTCCTCA-3’;GAPDH基因引物:上游5’-TCTTCCAGGAGCGAGATCCCT-3’,下游5’-GCAAATGAGCCCCAGCCTTCT-3’。
1.2.2 质粒的构建和转染分别构建DDB2基因WT、D1转录本的过表达质粒和空载体(上海吉凯基因医学科技股份有限公司)。用含10%胎牛血清的1640培养基在37 ℃、5% CO2的恒温培养箱中,用6孔板培养胃癌BGC823细胞。待BGC823细胞生长至60%~80%时,按照转染说明书分别将1 μg质粒与2 μL转染试剂在200 μL缓冲液中混合,混匀后静置10 min,将混合液其加入到细胞中,4 h后换液。根据所加质粒不同,将BGC823细胞分为空载体组、WT组和D1组,以进行后续实验。
1.2.3 Western blotting收集各组BGC823细胞,在100 μL RIPA (上海索莱宝生物科技有限公司)中裂解并提取细胞总蛋白后,用BCA蛋白定量试剂盒(上海索莱宝生物科技有限公司)测定蛋白浓度并配平。加入5×loading buffer煮沸5 min。之后使用10%聚丙烯酰胺凝胶进行电泳,每孔加入蛋白30 μg,浓缩胶70 V 30 min,分离胶110 V 90 min。切下待用的凝胶后用三明治法湿转120 min,将蛋白转印于PVDF膜上。用10%脱脂牛奶室温封闭1 h。PBST清洗后孵育一抗(Flag抗体,日本GNI公司;GAPDH抗体,德国Bioworld公司;PARP抗体,美国Cell Signaling Technology公司;E-Cadherin抗体,美国Abcam公司),4 ℃过夜;PBST清洗后室温孵育二抗(兔二抗,美国Abcam公司) 2 h,PBST清洗后用发光液(上海碧云天生物技术有限公司)发光,显影后保存图片,观察Flag蛋白、PARP蛋白和E-cadherin蛋白的表达水平,以检测各组细胞的转染、凋亡和上皮-间充质转化(epithelial-mesenchymal transition,EMT)情况。
1.2.4 CCK-8增殖实验在各组BGC823细胞状态稳定后,用胰酶将细胞消化下来并计数。将细胞悬液浓度调整至3×104/mL,在96孔板中每孔加入100 μL细胞悬液。提取剩余细胞的总蛋白,通过Western blotting检测质粒上的3*Flag来检测各组的转染情况。分别在24、48和72 h后,向每孔加入10 μL CCK-8试剂(美国MCE公司),2 h后检测其在450 nm处的光密度(optical density,OD)值并进行比较。实验重复3次。
1.3 统计学分析应用SPSS 20.0软件进行统计学分析。计量资料以x±s表示,2组比较采用t检验。采用GraphPad Prism 7制作柱状图。P < 0.05为差异有统计学意义。
2 结果 2.1 DDB2基因WT、D1转录本在胃癌和远端正常胃组织中的RNA表达水平通过RT-qPCR检测22对胃癌组织及其对应的远端正常胃组织中DDB2基因WT、D1转录本的RNA表达水平。RT-qPCR结果显示:胃癌中DDB2基因D1转录本的RNA表达水平显著高于远端正常胃组织(P < 0.01),而WT转录本在胃癌和远端正常胃组织中的RNA表达水平无统计学差异。见表 1。
| Splice variant | n | △Ct | 2-△△Ct | P |
| WT | 0.792 | |||
| Normal gastric tissue | 22 | 6.32±1.37 | 1.00 | |
| Gastric cancer tissue | 22 | 6.31±1.55 | 1.01 | |
| D1 | 0.004 | |||
| Normal gastric tissue | 22 | 13.14±2.00 | 1.00 | |
| Gastric cancer tissue | 22 | 12.00±1.69 | 2.20 |
2.2 DDB2基因WT、D1转录本对胃癌细胞增殖能力的影响
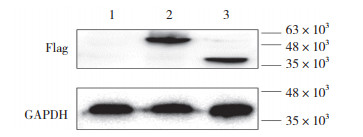
Western blotting结果显示:空载体组、WT组和D1组质粒均成功转染入BGC823细胞中。CCK-8实验发现,培养48和72 h后,WT组的OD值大于空载组(P < 0.05),而D1组的OD值小于空载组(P < 0.01)。表明DDB2基因WT转录本促进胃癌细胞增殖,而D1转录本抑制胃癌细胞增殖。见图 1、图 2。
|
| 1, empty vector group; 2, WT group; 3, D1 group. 图 1 BGC823细胞质粒转染情况 Fig.1 Plasmid transfection of BGC823 cells |
|
| * P < 0.05, ** P < 0.01, *** P < 0.001. 图 2 WT、D1转录本对BGC823细胞增殖能力的影响 Fig.2 Effects of WT and D1 transcripts on the proliferation of BGC823 cells |
2.3 DDB2基因WT、D1转录本对胃癌细胞的凋亡行为和EMT现象的影响
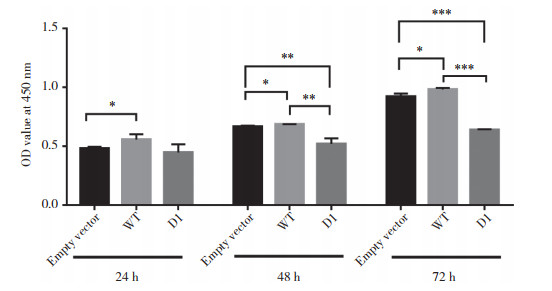
Western blotting结果显示:各组细胞中E-cadherin蛋白和PARP水解产物的表达量无明显差异,提示DDB2基因WT、D1转录本对胃癌细胞的凋亡行为和EMT现象无明显影响。见图 3。
|
| 1, empty vector group; 2, WT group; 3, D1 group. 图 3 BGC823细胞中E-cadherin与PARP水解产物的表达量 Fig.3 Expression of E-cadherin and PARP hydrolysate in BGC823 cells |
3 讨论
NCBI在线数据库显示,DDB2基因有WT、D1两种转录本。QIAO等[9]发现,在胃癌中DDB2基因WT转录本编码的DDB2野生型蛋白可以抑制PAQR3的泛素化进程,但并未提及其在胃癌和远端正常胃组织中表达量的差异,其表达量情况与本研究相符。DDB2基因的D1转录本与WT转录本相比缺少4个外显子,其编码蛋白发挥的功能可能有较大差异。RT-qPCR结果显示,D1转录本的RNA表达水平在胃癌和远端正常胃组织中有统计学差异(P < 0.01),这使得D1转录本有可能成为胃癌的筛查标志物,其具体应用仍需后续实验加以探索。
本研究中,CCK-8结果显示,与转染了空载质粒的BGC823细胞相比,转染WT转录本质粒的BGC823细胞增殖效应增强(P < 0.05),与QIAO等[9]的结果相符;而转染D1质粒的BGC823细胞增殖效应减弱(P < 0.01)。这种同一基因的不同转录本发挥不同功能的现象并非个例。检索PubMed数据库可知,其他基因如TERF1等[10]的剪接变异体可以影响癌细胞的生物学功能,甚至其多种转录本发挥的作用完全相反。结合RT-qPCR结果大胆推测:DDB2基因D1转录本可能在胃癌中发挥抑癌作用,在检测到细胞癌变倾向后D1转录本大量转录以抑制胃细胞癌变,并在胃细胞癌变不可逆之后仍然保持高转录效率,起到抑制胃癌细胞生长的作用。
凋亡是细胞重要的生物学行为之一,本研究通过Western blotting检测胃癌细胞中PARP水解产物的蛋白表达量,判断DDB2基因的2种转录本对细胞凋亡的影响。有研究[11]表明,DDB2基因WT转录本编码蛋白可以识别DNA损伤,并通过NER通路修复损伤;若DNA损伤积累过多,则WT转录本蛋白可下调P21蛋白并促进细胞凋亡。但是本研究在胃癌细胞中并未发现相似结论。结合本研究结果进行推断,DDB2基因WT编码的野生型蛋白主要负责DNA损伤的识别,仅能根据DNA损伤的程度决定是修复损伤还是促使细胞凋亡[12];其本身的表达量对细胞凋亡的进程可能影响不大。ROY等[13]发现,DDB2可以抑制VEGF、Snail和Zeb1基因的表达,进而抑制结肠癌细胞的EMT现象,但是本研究并未发现DDB2基因WT转录本对胃癌细胞的EMT现象产生影响。虽然胃癌与结肠癌均为消化道肿瘤,但是胃癌细胞与结肠癌细胞在形态、功能、蛋白组成方面均有较大差异。正是因为这些差异,DDB2在两者中发挥的作用可能并不相同,其具体原因仍需后续实验加以论证。
综上所述,本研究检测了DDB2基因的2种转录本在胃癌组织和远端正常胃组织中的RNA表达水平,并发现D1转录本在胃癌中高表达,而WT转录本在2种组织中的RNA表达水平并无明显差异。而后在体外实验中发现,DDB2基因WT转录本促进胃癌细胞增殖,而D1转录本抑制胃癌细胞增殖。目前DDB2基因的2种转录本在胃癌中的作用机制尚不明确,仍需后续实验加以探索。通过对胃癌中DDB2基因WT、D1转录本的深入研究,可以进一步解释胃癌的发生、发展机制,有利于发现更好的胃癌治疗手段。
| [1] |
LEVY J, GUPTA V, AMIRAZODI E, et al. Textbook outcome and survival in patients with gastric cancer:an analysis of the population registry of esophageal and stomach tumours in Ontario (PRESTO)[J]. Ann Surg, 2020, 10. DOI:10.1097/SLA.0000000000003849 |
| [2] |
THRIFT AP, EL-SERAG HB. Burden of gastric cancer[J]. Clin Gastroenterol Hepatol, 2020, 18(3): 534-542. DOI:10.1016/j.cgh.2019.07.045 |
| [3] |
MATEO J, CARREIRA S, SANDHU S, et al. DNA-repair defects and olaparib in metastatic prostate cancer[J]. N Engl J Med, 2015, 373(18): 1697-1708. DOI:10.1056/NEJMoa1506859 |
| [4] |
FELTES BC, PEDEBOS C, BONATTO D, et al. Dynamics of DDB2-DDB1 complex under different naturally-occurring mutants in xeroderma pigmentosum disease[J]. Biochim Biophys Acta Gen Subj, 2018, 1862(12): 2579-2589. DOI:10.1016/j.bbagen.2018.08.007 |
| [5] |
HUANG S, FANTINI D, MERRILL BJ, et al. DDB2 is a novel regulator of Wnt signaling in colon cancer[J]. Cancer Res, 2017, 77(23): 6562-6575. DOI:10.1158/0008-5472.CAN-17-1570 |
| [6] |
COOMER AO, BLACK F, GREYSTOKE A, et al. Alternative splicing in lung cancer[J]. Biochim Biophys Acta Gene Regul Mech, 2019, 1862(11/12): 194388. DOI:10.1016/j.bbagrm.2019.05.006 |
| [7] |
AGUADO C, GIMÉNEZ-CAPITÁN A, KARACHALIOU N, et al. Fusion gene and splice variant analyses in liquid biopsies of lung cancer patients[J]. Transl Lung Cancer Res, 2016, 5(5): 525-531. DOI:10.21037/tlcr.2016.09.02 |
| [8] |
LIU J, HE C, XING C, YUAN Y. Nucleotide excision repair related gene polymorphisms and genetic susceptibility, chemotherapeutic sensitivity and prognosis of gastric cancer[J]. Mutat Res, 2014, 765: 11-21. DOI:10.1016/j.mrfmmm.2014.04.008 |
| [9] |
QIAO S, GUO W, LIAO L, et al. DDB2 is involved in ubiquitination and degradation of PAQR3 and regulates tumorigenesis of gastric cancer cells[J]. Biochem J, 2015, 469(3): 469-480. DOI:10.1042/BJ20150253 |
| [10] |
MORCOS YAT, NAJJAR G, MEESSEN S, et al. A novel tissue and stem cell specific TERF1 splice variant is downregulated in tumour cells[J]. Int J Mol Sci, 2019, 21(1): 85. DOI:10.3390/ijms21010085 |
| [11] |
STOYANOVA T, ROY N, BHATTACHARJEE S, et al. p21 cooperates with DDB2 protein in suppression of ultraviolet ray-induced skin malignancies[J]. J Biol Chem, 2012, 287(5): 3019-3028. DOI:10.1074/jbc.M111.295816 |
| [12] |
ROY N, ELANGOVAN I, KOPANJA D, et al. Tumor regression by phenethyl isothiocyanate involves DDB2[J]. Cancer Biol Ther, 2013, 14(2): 108-116. DOI:10.4161/cbt.22631 |
| [13] |
ROY N, BOMMI PV, BHAT UG, et al. DDB2 suppresses epithelial-to-mesenchymal transition in colon cancer[J]. Cancer Res, 2013, 73(12): 3771-3782. DOI:10.1158/0008-5472.CAN-12-4069 |
 2021, Vol. 50
2021, Vol. 50