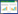文章信息
- 礼想, 杨博文, 宫丽宝, 臧丹, 张朝旭, 张闯, 李智, 曲秀娟
- LI Xiang, YANG Bowen, GONG Libao, ZANG Dan, ZHANG Chaoxu, ZHANG Chuang, LI Zhi, QU Xiujuan
- 基于共表达网络分析的Ⅰ期胃癌预后基因筛选
- Screening of prognostic genes for stageⅠgastric cancer based on co-expression network analysis
- 中国医科大学学报, 2021, 50(1): 1-8
- Journal of China Medical University, 2021, 50(1): 1-8
-
文章历史
- 收稿日期:2020-04-26
- 网络出版时间:2020-12-22 9:46
胃癌是最常见的消化道恶性肿瘤之一,也是全球癌症死亡的第三大原因[1]。胃癌患者的生存时间主要与确诊时分期密切相关,确诊时分期越早,生存时间越长,甚至可以治愈。既往研究[2]已确定了包括人类表皮生长因子受体2 (human epidermal growth factor receptor-2,HER2)、糖类抗原19-9 (carbohydrate antigen 19-9,CA19-9)和血管内皮生长因子(vascular endothelial growth factor,VEGF)等多种胃癌生物标志物,其中某些可用于临床判断预后、预测疗效或作为诊断的生物标志物。找到能够准确预测Ⅰ期胃癌预后的新型分子生物标志物,将有助于进一步研究Ⅰ期胃癌的发病机制及进行个性化治疗。
加权基因共表达网络分析(weighted correlation network analysis,WGCNA)是一种联合分析临床信息和基因表达数据的方法。通过分析与临床信息相关性高的基因模块,获得一系列在模块中具有高连接度的关键基因,这些基因对肿瘤的发生发展具有潜在的重要作用[3-4]。癌症基因组图谱(The Cancer Genome Atlas,TCGA)是由美国National Cancer Institute (NCI)和National Human Genome Research Institute (NHGRI)共同开发的数据库网站,包含有30余种癌症的数据信息。研究人员已利用WGCNA及TCGA发现了多种癌症的分子机制和特征。本研究拟利用胃癌的TCGA数据,构建基因共表达网络,识别网络中与胃癌预后相关的模块和基因,并对预后基因进行体外实验和通路富集,对该分子可能影响胃癌患者预后的机制进行分析和探讨。
1 材料与方法 1.1 数据获取与筛选从TCGA数据库(https://www.cancer.gov/about-nci/organization/ccg/research/structural-genomics/tcga)中下载胃癌患者的基因表达谱数据和临床数据。纳入研究的临床病例应包括病理性TNM分期、组织学等级、生存信息。排除病理分期或组织学分级信息不完整的病例。
1.2 构建基因共表达网络利用WGCNA对Ⅰ期胃癌数据进行分析。通过中位数绝对偏差(median absolute deviation,MAD)测量基因变异性,并按MAD进行排序,获取前3 600个基因。使用R包进行WGCNA分析,加权邻接矩阵定义如下:Amn = |Smn |β。其中,Smn为基因m和基因n之间的Pearson相关性;Amn为基因m和基因n之间的邻接。
在WGCNA中,需要选择合适的软阈值β才能构建网络。最佳软阈值需达到无尺度拟合指数0.9以上,且需要网络的平均连通性较高。以此软阈值构建拓扑重叠矩阵(topological overlap matrix,TOM)。WGCNA将具有高拓扑重叠指数的一组基因定义为同一模块,进行层次聚类,建立有嵌套的分层聚类树。基于WGCNA的R软件包中的MEs (module eigengenes)函数,计算其差异性。通过Cytoscape软件(v3.4.0)可将模块的共表达网络可视化。
1.3 筛选枢纽基因为了确定与Ⅰ期胃癌临床信息有关的模块,通过Pearson相关性检验计算出MEs与临床特征之间的相关性,P < 0.05被认为有统计学意义。测量每个模块的基因显著性(gene significance,GS),即基因与性状之间的相关性。模块的GS绝对值越高,表示该模块的生物学意义越强。通常,模块的GS倾向于与模块特征基因和临床特征之间高度相关。倾向于具有高模块成员(module membership,MM)定量值的高度连接的模块内基因被定义为枢纽基因,它在功能上具有重要作用。为了鉴定枢纽基因,使用Cytoscape软件(v3.4.0)中内置的cytoHubba进行网络分析。在Cytoscape软件中显示了前25个枢纽基因。为了验证枢纽基因的临床意义,依据基因表达量将胃癌患者分为低表达和高表达2组。通过生存分析确认影响胃癌预后的基因。此外,通过Kaplan-Meier在线数据分析(http://www.kmplot.com)进一步评估该基因对胃癌患者的生存的影响。
1.4 体外细胞学实验 1.4.1 实时定量PCR在4种胃癌细胞系(MKN-45,BGC-823,SGC-7901,MGC-803)中检测脑酸可溶性蛋白1 (brain acid soluble protein 1,BASP1)基因的mRNA表达。用Trizol试剂盒(美国Invitrogen公司)从培养的胃癌细胞中提取RNA,并测量其RNA浓度。用PrimeScript RT试剂盒(日本TaKaRa公司)进行逆转录。用SYBR premixed explosion-proof TaqTMⅡ (Tli RNaseH Plus,日本TaKaRa公司)进行实时PCR,并在Applied Biosystems 7500 Real-Time PCR Systems (美国Thermo Fisher公司)中检测mRNA表达量。18S用作内部对照。PCR条件为95 ℃10 min,95 ℃15 s、58 ℃34 s,45个循环。采用2-ΔΔCt计算mrna的相对表达量。PCR引物序列如下,BASP1 F,5’-GCAAC TCGTTTGCAGCG-3’,R,5’-TGCCTCCCATCTTGG AGTTC-3’;18S F,5’-CCCGGGGAGGTAGTGACGA AAAAT-3’,R,5’-CGCCCGCCCGCTCCCAAG AT-3’。
1.4.2 MTT法检测肿瘤细胞增殖将转染BASP1 siRNA和NC siRNA的MGC803细胞悬浮液以4 000/孔的密接度种于96孔板内,孵育72 h。加入MTT溶液(5 mg/mL,20 μL/孔)混匀,37 ℃静置4 h后,弃上清液,加入二甲基亚砜(200 μL/孔)溶解结晶,于波长570 nm处酶标仪(Model 550,美国Bio-Rad公司)测量光密度(optical density,OD)值。每组设置4个复孔,实验重复3次。
1.4.3 细胞迁移能力检测在24孔小室(8 μm,美国Corning公司)中进行迁移测定。用200 μL无血清RPMI1640培养基将预处理的对照组或转染siRNA的细胞(5×104/孔)制备成细胞悬液。将混匀的细胞悬浮液接种于上室中,下室则包含500 μL RPMI1640培养基和2.5%胎牛血清。37 ℃孵育24 h后,取出小室,用湿棉签将未迁移的细胞轻轻擦去,用Giemsa-Wright法染色培养皿下表面的细胞。在20倍显微镜下(BX53,日本Olympus公司)随机选择5个视野,计数迁移细胞的数量。
1.5 基因集富集分析(gene set enrichment analysis,GSEA)GSEA用于鉴定在预后因子高表达组中显著富集的途径。采用GSEA 3.0版本进行分析。用R中的Cluster Profiler程序包对该探索模块中基因的潜在生物学机制和途径进行探索。选择GSEA网站的分子特征数据库(Molecular Signatures Database,MSigDB)中的c2.cp.kegg.v5.1.symbols.gmt数据集作为参考基因集,以评估不同表达对每个参考集的影响。GSEA采用默认的加权富集统计方法进行,随机组合的数量设计为1 000次。
1.6 统计学分析采用R软件(version 3.6.1,www.r-project.org)进行统计分析,构建生存曲线,并通过Kaplan-Meier Plotter在线验证。正态分布变量使用独立的t检验,其他变量进行Mann-Whitney U检验。所有统计检验均为双侧检验,P < 0.05为差异有统计学意义。
2 结果 2.1 数据处理TCGA数据集中共纳入临床信息完整的胃癌病例330例,其中Ⅰ期胃癌51例,均为病理确诊,平均总生存时间为18.49个月。包括男性患者16例(31%)和女性患者35例(69%)。见表 1。
| Characteristic | n | Proportion (%) |
| Grade | ||
| G1-G2 | 31 | 61 |
| G3 | 20 | 39 |
| T stage | ||
| T1 | 18 | 35 |
| T2 | 33 | 65 |
| N stage | ||
| N0 | 49 | 96 |
| N1 | 2 | 4 |
| Gender | ||
| Male | 16 | 31 |
| Female | 35 | 69 |
| Primary diagnosis | ||
| C16.0 | 17 | 33 |
| C16.1 | 9 | 18 |
| C16.2 | 13 | 25 |
| C16.3 | 10 | 20 |
| C16.9 | 2 | 4 |
2.2 构建基因共表达网络
应用51例具有完整信息的Ⅰ期胃癌数据构建基因共表达网络。当软阈值功率β值=5时,基因之间的连通性满足无标度网络分布,见图 1A。通过层次聚类和动态分支切割识别出42个模块,为每个模块分配了唯一的颜色作为标识符,见图 1B。
|
| A, determination of soft-thresholding power in WGCNA; B, modules associated with the clinical traits of SIGC. 图 1 基因共表达网络构建 Fig.1 Construction of gene co-expression networks |
2.3 确定枢纽基因
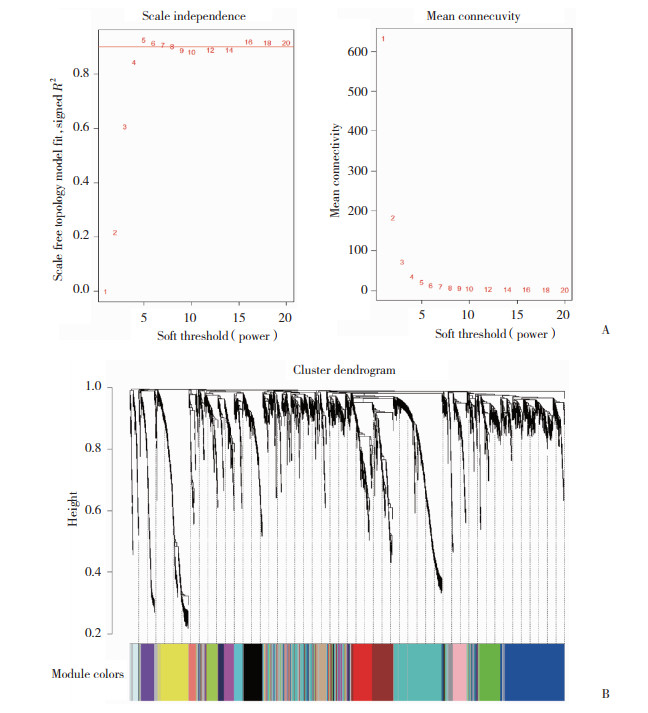
将模块和临床特征进行关联分析,寻找和临床特征显著相关的模块,见图 2。考虑Ⅰ期胃癌患者T、N分期较早,选择可能与胃癌预后相关性较大的组织学分化(Grade)临床特征进行进一步研究,该临床特征与Black模块相关性最高,相关性系数cor = 0.51,P < 0.05。
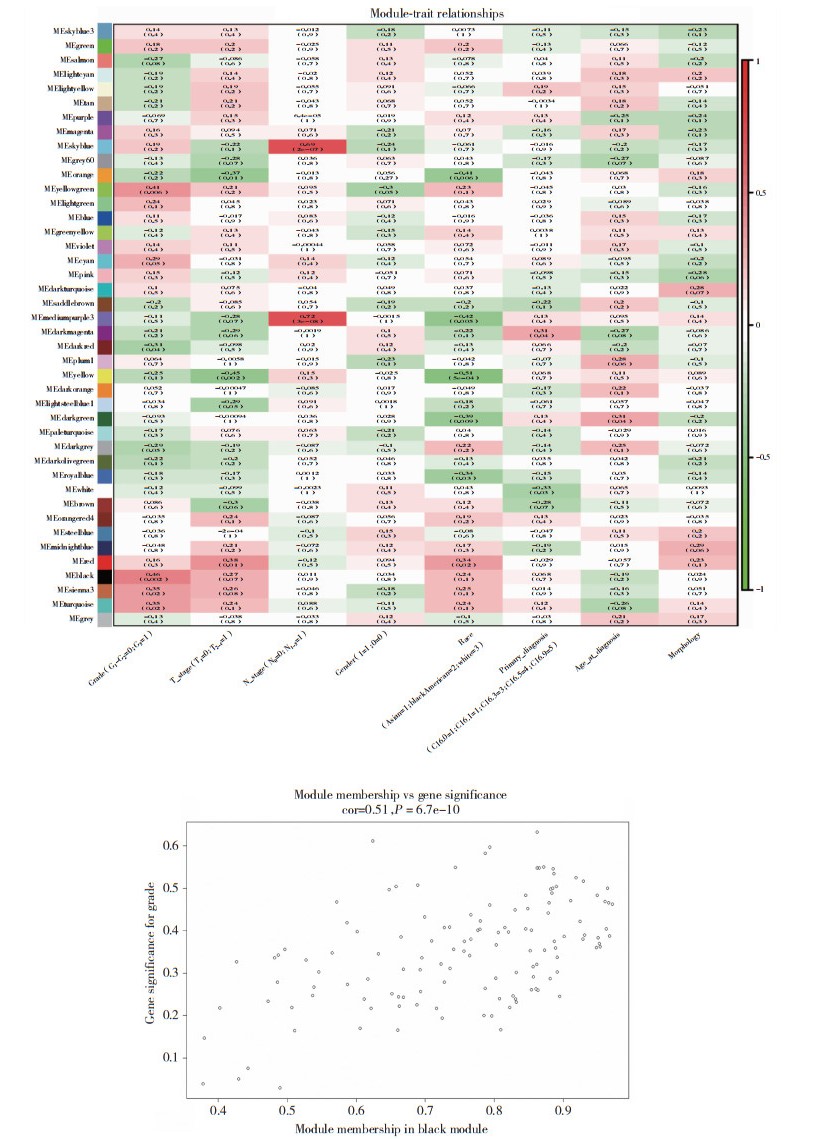
|
| 图 2 评估模块与临床特征的关联性 Fig.2 Evaluate the correlations between MEs and clinical traits |
模块中高度相关的枢纽基因具有至关重要的作用。在黑色模块中筛选了前25个基因为枢纽基因,并进一步分析其内部相关性,其相互关系网络见图 3A。通过对前25个枢纽基因逐个进行生存分析,获得了影响胃癌预后的基因BASP1。TCGA生存分析结果表明,BASP1基因高表达患者的总生存期(overall survival,OS)明显低于低表达患者(P = 0.016 25),见图 3B。利用Kaplan-Meier曲线对BASP1基因进行生存分析,Kaplan-Meier在线分析也获得了相似的结果,即高表达的患者的OS明显短于低表达患者(27.4个月vs 47.7个月,P = 0.000 51),见图 3C。表明BASP1可能是胃癌预后不良的预测指标。
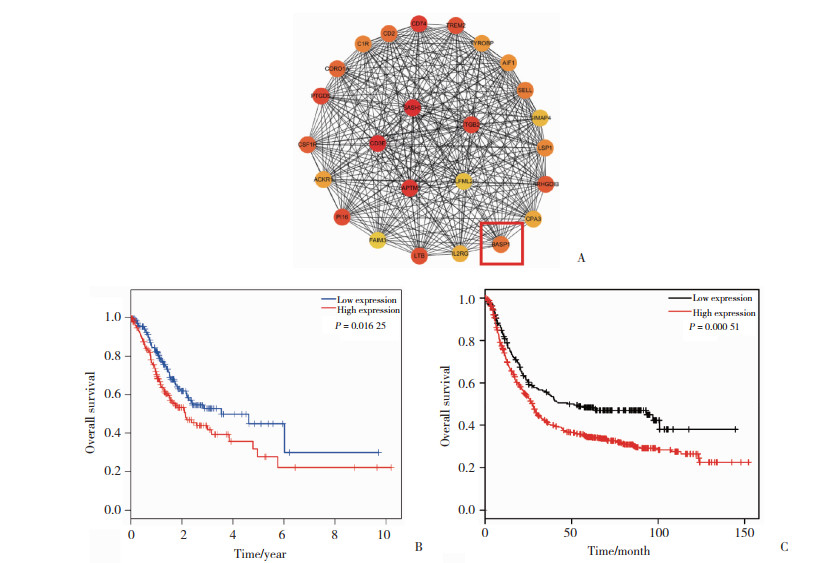
|
| A, 25 high-degree genes in black module screen from cytohubba; B, survival analysis by TCGA showed that overexpression of BASP1 confers a poor prognosis; C, Kaplan-Meier plotter online analysis showed the effect of BASP1 expression on the prognosis of gastric cancer. 图 3 BASP1高表达的胃癌患者预后差 Fig.3 Overexpression of BASP1 confers a poor prognosis in gastric cancer |
2.4 BASP1基因的功能
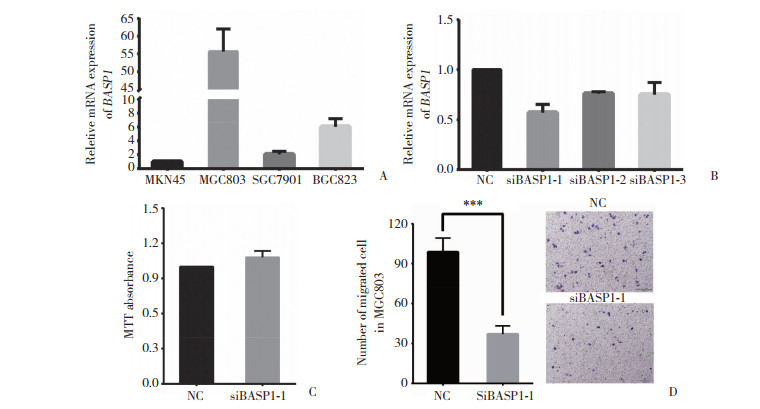
为了探索BASP1在胃癌细胞中的功能,用实时定量PCR检测了BASP1在不同胃癌细胞系中的mRNA表达。如图 4A所示,在胃癌细胞系MGC-803中,BASP1表达最高,因此选择该细胞株用于后续的细胞学实验。为了探讨BASP1的作用,利用siRNA敲低MGC-803细胞株中BASP1的表达。如图 4B所示,siRNA1敲减效果较好。TCGA数据库及Kaplan-Meier在线数据分析发现,BASP1是胃癌的预后不良因子,因此,对BASP1的作用进行了进一步体外细胞学实验研究。MTT分析结果如图 4C所示,抑制BASP1表达不会影响细胞增殖能力(P = 0.17)。但是,Transwell分析表明,与对照组相比,敲减BASP1表达后,胃癌细胞的迁移能力下降了62.2% (图 4D)。以上结果表明BASP1可能通过促进胃癌细胞迁移导致预后不良。
|
| A, mRNA expression of BASP1 in different gastric cancer cell lines; B, siRNA was used to knockdown BASP1 in MGC-803 and siRNA1 has a good result; C, MTT assay showed that suppression of BASP1 expression did not impact cell viability (P = 0.17);D, Transwell assays revealed that compared to control cells, knockdown of BASP1 significantly decreased cell migration ability of gastric cancer by 62.2% (×20). ***P = 0.001. NC, negative control. 图 4 BASP1基因的功能 Fig.4 The role of the hub gene BASP1 |
2.5 GSEA通路富集
为了探讨BASP1基因抑制细胞迁移的机制,进行了GSEA富集分析。结果显示,代表性肿瘤相关途径IL6-JAK-STAT3信号传导途径的富集结果较好(NOM p-val < 0.001;FDR q-val < 0.001),见表 2。
| GS follow link to MSigDB | Size | NOM p-val | FDR q-val |
| HALLMARK INFLAMMATORY RESPONSE | 196 | < 0.001 | < 0.001 |
| HALLMARK ALLOGRAFT REJECTION | 187 | < 0.001 | 0.001 |
| HALLMARK IL6 JAK STAT3 SIGNALING | 87 | < 0.001 | < 0.001 |
| HALLMARK TNFA SIGNALING VIA NFKB | 194 | < 0.001 | 0.001 |
| HALLMARK COAGULATION | 133 | < 0.001 | 0.001 |
3 讨论
胃癌生物学行为和分子机制的深入研究对准确预测胃癌的预后和疗效、筛选新的胃癌生物标志物具有重要意义。本研究通过WGCNA分析了TCGA数据库中Ⅰ期胃癌的数据,并从中筛选得到了预后影响因子BASP1。后续的生存分析结果显示,虽然BASP1筛选自Ⅰ期胃癌患者,但其高表达的胃癌患者,无论分期如何,预后都较差。这表明尽管TNM分期的不同可对胃癌的预后产生影响,但是其内在的分子机制和生物学行为才是影响胃癌预后的根本因素。
BASP1 (也称CAP-23,NAP-22)是一种从脑细胞质中提取的氨基末端十四烷酰化蛋白,主要表达于细胞质和细胞核中。既往文献[5-6]报道,BASP1大量表达于神经末梢中,睾丸、淋巴器官和肾脏等中也有表达。BASP1最重要的生物学功能是通过调节细胞骨架相关蛋白的运动,促进轴突的生长和发育。已有BASP1与癌症有关的相关报道[7-8],但关于它在癌症中作用的研究很少。
BASP1过表达对不同癌症患者生存的影响有所不同。BASP1在乳腺癌[8]、黑色素瘤[9]、甲状腺癌[10]、肝细胞癌[11]和胰腺癌[12]中表达下调时,预后较差。相反,BASP1高表达与子宫颈癌患者的整体生存能力差有关[13]。迄今为止,尚未见BASP1与胃癌预后有关的报道。本研究结果表明,BASP1可能是胃癌预后不良因子。
关于BASP1的作用机制,研究发现,乳腺癌中BASP1与雌激素受体α有相互作用[8];宫颈癌中,BASP1可能通过调节细胞周期和增殖发挥作用[13];甲状腺癌中,BASP1可能影响细胞增殖、细胞周期、细胞凋亡和迁移[10]。本研究结果显示,敲减BASP1后,胃癌细胞增殖能力未见明显变化,而迁移能力明显减弱。说明BASP1下调可改变胃癌细胞的迁移能力,提示BASP1主要通过影响胃癌细胞的迁移来影响胃癌的预后,与以往的研究结果不同。BASP1的富集分析结果显示,IL6-JAK-STAT3通路与胃癌相关。
白细胞介素-6 (interleukin-6,IL-6)是由IL-6基因产生的一种细胞因子。IL-6与其受体结合,通过JAK/STAT途径激活STAT3分子,刺激下游基因表达以执行多种生理功能[14]。文献[15-26]报道,在头颈部肿瘤、食道癌、乳腺癌、宫颈癌、卵巢癌、结直肠癌、胰腺癌、肾癌、前列腺癌及非小细胞肺癌患者中,均可观察到循环水平的IL-6升高。STAT3是STATs家族中重要的成员之一,其活化形式主要是通过酪氨酸磷酸化[27]。研究[28-32]发现,在鼻咽癌、乳腺癌、结直肠癌等多种肿瘤中均可检测到STAT3的异常活化。另有研究[33]发现,外源IL-6能促进胃癌的增殖、侵袭和转移,其潜在机制可能与JAK-STAT3- VEGF-C信号通路有关。BASP1在细胞核中表达丰富,因此推测它可能通过影响细胞核中IL-6分泌并调节IL6-JAK-STAT3途径,促进胃癌细胞迁移,具体机制仍有待进一步验证。
综上所述,BASP1是胃癌预后不良因子。BASP1可能通过影响IL6-JAK-STAT3通路,促进胃癌转移,从而导致胃癌预后不良。
| [1] |
TORRE LA, BRAY F, SIEGEL RL, et al. Global cancer statistics, 2012[J]. CA Cancer J Clin, 2015, 65(2): 87-108. DOI:10.3322/caac.21262 |
| [2] |
KARIMI P, ISLAMI F, ANANDASABAPATHY S, et al. Gastric cancer:descriptive epidemiology, risk factors, screening, and prevention[J]. Cancer Epidemiol Biomarkers Prev, 2014, 23(5): 700-713. DOI:10.1158/1055-9965.EPI-13-1057 |
| [3] |
SUN Q, ZHAO HY, ZHANG C, et al. Gene co-expression network reveals shared modules predictive of stage and grade in serous ovarian cancers[J]. Oncotarget, 2017, 8(26): 42983-42996. DOI:10.18632/oncotarget.17785 |
| [4] |
TIAN F, ZHAO JL, FAN XL, et al. Weighted gene co-expression network analysis in identification of metastasis-related genes of lung squamous cell carcinoma based on the Cancer Genome Atlas database[J]. J Thorac Dis, 2017, 9(1): 42-53. DOI:10.21037/jtd.2017.01.04 |
| [5] |
MAEKAWA S, MAEKAWA M, HATTORI S, et al. Purification and molecular cloning of a novel acidic calmodulin binding protein from rat brain[J]. J Biol Chem, 1993, 268(18): 13703-13709. DOI:10.1016/S0021-9258(18)86914-9 |
| [6] |
BOMZE HM, BULSARA KR, ISKANDAR BJ, et al. Spinal axon regeneration evoked by replacing two growth cone proteins in adult neurons[J]. Nat Neurosci, 2001, 4(1): 38-43. DOI:10.1038/82881 |
| [7] |
HARTL M, NIST A, KHAN MI, et al. Inhibition of Myc-induced cell transformation by brain acid-soluble protein 1 (BASP1)[J]. Proc Natl Acad Sci USA, 2009, 106(14): 5604-5609. DOI:10.1073/pnas.0812101106 |
| [8] |
MARSH LA, CARRERA S, SHANDILYA J, et al. BASP1 interacts with oestrogen receptor α and modifies the tamoxifen response[J]. Cell Death Dis, 2017, 8(5): e2771. DOI:10.1038/cddis.2017.179 |
| [9] |
RANSOHOFF KJ, WU WT, CHO HG, et al. Two-stage genome-wide association study identifies a novel susceptibility locus associated with melanoma[J]. Oncotarget, 2017, 8(11): 17586-17592. DOI:10.18632/oncotarget.15230 |
| [10] |
GUO RS, YU Y, CHEN J, et al. Restoration of brain acid soluble protein 1 inhibits proliferation and migration of thyroid cancer cells[J]. Chin Med J (Engl), 2016, 129(12): 1439-1446. DOI:10.4103/0366-6999.183434 |
| [11] |
MORIBE T, ⅡZUKA N, MIURA T, et al. Identification of novel aberrant methylation of BASP1 and SRD5A2 for early diagnosis of hepatocellular carcinoma by genome-wide search[J]. Int J Oncol, 2008, 33(5): 949-958. |
| [12] |
ZHOU QM, ANDERSSON R, HU DY, et al. Quantitative proteomics identifies brain acid soluble protein 1 (BASP1) as a prognostic biomarker candidate in pancreatic cancer tissue[J]. EBioMedicine, 2019, 43: 282-294. DOI:10.1016/j.ebiom.2019.04.008 |
| [13] |
TANG HR, WANG Y, ZHANG B, et al. High brain acid soluble protein 1 (BASP1) is a poor prognostic factor for cervical cancer and promotes tumor growth[J]. Cancer Cell Int, 2017, 17: 97. DOI:10.1186/s12935-017-0452-4 |
| [14] |
SONG ZQ, REN D, XU XH, et al. Molecular cross-talk of IL-6 in tumors and new progress in combined therapy[J]. Thorac Cancer, 2018, 9(6): 669-675. DOI:10.1111/1759-7714.12633 |
| [15] |
JINNO T, KAWANO S, MARUSE Y, et al. Increased expression of interleukin-6 predicts poor response to chemoradiotherapy and unfavorable prognosis in oral squamous cell carcinoma[J]. Oncol Rep, 2015, 33(5): 2161-2168. DOI:10.3892/or.2015.3838 |
| [16] |
RIEDEL F, ZAISS I, HERZOG D, et al. Serum levels of interleukin-6 in patients with primary head and neck squamous cell carcinoma[J]. Anticancer Res, 2005, 25(4): 2761-2765. |
| [17] |
CHEN MF, CHEN PT, LU MS, et al. IL-6 expression predicts treatment response and outcome in squamous cell carcinoma of the esophagus[J]. Mol Cancer, 2013, 12: 26. DOI:10.1186/1476-4598-12-26 |
| [18] |
DETHLEFSEN C, HØJFELDT G, HOJMAN P. The role of intratumoral and systemic IL-6 in breast cancer[J]. Breast Cancer Res Treat, 2013, 138(3): 657-664. DOI:10.1007/s10549-013-2488-z |
| [19] |
KOTOWICZ B, FUKSIEWICZ M, JONSKA-GMYREK J, et al. The assessment of the prognostic value of tumor markers and cytokines as SCCAg, CYFRA 21.1, IL-6, VEGF and sTNF receptors in patients with squamous cell cervical cancer, particularly with early stage of the disease[J]. Tumour Biol, 2016, 37(1): 1271-1278. DOI:10.1007/s13277-015-3914-0 |
| [20] |
MACCIÒ A, MADEDDU C. The role of interleukin-6 in the evolution of ovarian cancer:clinical and prognostic implications:a review[J]. J Mol Med (Berl), 2013, 91(12): 1355-1368. DOI:10.1007/s00109-013-1080-7 |
| [21] |
SANGUINETE MMM, OLIVEIRA PH, MARTINS-FILHO A, et al. Serum IL-6 and IL-8 correlate with prognostic factors in ovarian cancer[J]. Immunol Invest, 2017, 46(7): 677-688. DOI:10.1080/08820139.2017.1360342 |
| [22] |
CHUNG YC, CHANG YF. Serum interleukin-6 levels reflect the disease status of colorectal cancer[J]. J Surg Oncol, 2003, 83(4): 222-226. DOI:10.1002/jso.10269 |
| [23] |
MIURA T, MITSUNAGA S, IKEDA M, et al. Characterization of patients with advanced pancreatic cancer and high serum interleukin-6 levels[J]. Pancreas, 2015, 44(5): 756-763. DOI:10.1097/MPA.0000000000000335 |
| [24] |
ALTUNDAG O, ALTUNDAG K, GUNDUZ E. Interleukin-6 and C-reactive protein in metastatic renal cell carcinoma[J]. J Clin Oncol, 2005, 23(5): 1044. DOI:10.1200/JCO.2005.05.155 |
| [25] |
CULIG Z, PUHR M. Interleukin-6:a multifunctional targetable cytokine in human prostate cancer[J]. Mol Cell Endocrinol, 2012, 360(1/2): 52-58. DOI:10.1016/j.mce.2011.05.033 |
| [26] |
CHANG CH, HSIAO CF, YEH YM, et al. Circulating interleukin-6 level is a prognostic marker for survival in advanced nonsmall cell lung cancer patients treated with chemotherapy[J]. Int J Cancer, 2013, 132(9): 1977-1985. DOI:10.1002/ijc.27892 |
| [27] |
ZHANG Y, TURKSON J, CARTER-SU C, et al. Activation of Stat3 in v-Src-transformed fibroblasts requires cooperation of Jak1 kinase activity[J]. J Biol Chem, 2000, 275(32): 24935-24944. DOI:10.1074/jbc.M002383200 |
| [28] |
LUI VW, WONG EY, HO Y, et al. STAT3 activation contributes directly to Epstein-Barr virus-mediated invasiveness of nasopharyngeal cancer cells in vitro[J]. Int J Cancer, 2009, 125(8): 1884-1893. DOI:10.1002/ijc.24567 |
| [29] |
BOCHET L, MEULLE A, IMBERT S, et al. Cancer-associated adipocytes promotes breast tumor radioresistance[J]. Biochem Biophys Res Commun, 2011, 411(1): 102-106. DOI:10.1016/j.bbrc.2011.06.101 |
| [30] |
KUNIGAL S, LAKKA SS, SODADASU PK, et al. Stat3-siRNA induces Fas-mediated apoptosis in vitro and in vivo in breast cancer[J]. Int J Oncol, 2009, 34(5): 1209-1220. |
| [31] |
XU HY, LAI W, ZHANG Y, et al. Tumor-associated macrophage-derived IL-6 and IL-8 enhance invasive activity of LoVo cells induced by PRL-3 in a KCNN4 channel-dependent manner[J]. BMC Cancer, 2014, 14: 330. DOI:10.1186/1471-2407-14-330 |
| [32] |
YU H, LEE H, HERRMANN A, et al. Revisiting STAT3 signalling in cancer:new and unexpected biological functions[J]. Nat Rev Cancer, 2014, 14(11): 736-746. DOI:10.1038/nrc3818 |
| [33] |
ZHAO GB, ZHU GW, HUANG YJ, et al. IL-6 mediates the signal pathway of JAK-STAT3-VEGF-C promoting growth, invasion and lymphangiogenesis in gastric cancer[J]. Oncol Rep, 2016, 35(3): 1787-1795. DOI:10.3892/or.2016.4544 |
 2021, Vol. 50
2021, Vol. 50