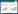文章信息
- 李露露, 蒲实, 范秋灵, 汪旭, 李卅立
- LI Lulu, PU Shi, FAN Qiuling, WANG Xu, LI Sali
- 糖尿病肾病患者循环miRNA表达谱的分析
- Analysis of Circulating miRNA Expression Profiles in Patients with Diabetic Nephropathy
- 中国医科大学学报, 2019, 48(8): 694-698, 708
- Journal of China Medical University, 2019, 48(8): 694-698, 708
-
文章历史
- 收稿日期:2018-08-09
- 网络出版时间:2019-07-15 10:13
糖尿病肾病(diabetic nephropathy, DN)是糖尿病严重的微血管并发症之一, 也是全世界范围内终末期肾衰竭的主要病因[1]。2015年, 我国糖尿病相关慢性肾脏病患者已超过肾小球肾炎相关慢性肾脏病患者, 成为我国住院慢性肾脏病患者的主要原因[2]。目前DN的治疗方法为药物及替代疗法, 但都不能做到有效治疗[3]。因此, 寻找用于早期诊断和有效治疗的新型生物标志物至关重要。
miRNA是一类非编码内源性RNA (20~30个核苷酸), 它通过与特定mRNA的3’非翻译区(3’UTR)结合来调控基因的表达, 诱导其降解或翻译抑制。已有研究[4]发现miRNA涉及许多基因表达的转录后调控, 在凋亡、DNA修复、氧化应激反应、细胞发育等过程中发挥重要作用。在DN中, 越来越多的研究[5]发现miRNA靶向炎症、纤维化和氧化应激等相关基因, 其表达变化不仅可以用于诊断DN, 还可以预测与疾病相关的分子信号机制。本研究利用miRNA芯片检测DN患者、糖尿病患者、健康对照者血清中miRNA的表达谱, 通过实时PCR验证结果, 并对其功能及可能作用的靶点进行分析和预测, 为进一步研究DN发生发展的分子机制及开发新的诊断和治疗靶点提供理论基础和依据。
1 材料与方法 1.1 一般资料及分组选取2012年2月至2013年3月中国医科大学附属第一医院肾内科行肾活检, 血肝酐 < 200 μmol/L, 病理确诊为结节性糖尿病肾小球硬化症患者为DN组(n = 5);同期本院住院治疗的2型糖尿病患者(尿微量白蛋白 < 30 mg/g)为DM组(n = 5);2组患者均符合WHO糖尿病诊断标准(空腹血糖≥7 mmol/L或口服75 g无水葡萄糖粉2 h后血糖≥11.1 mmol/L)。同期于本院健康体检者为对照组(n = 5), 对照组空腹血糖、餐后2 h血糖、糖化血红蛋白、血肝酐均在正常范围内, 尿微量白蛋白 < 30 mg/g。排除标准:恶性肿瘤、心血管疾病、肝损伤等可能影响miRNA表达的慢性疾病。DN组、DM组和对照组年龄、性别、吸烟史、血压、血管危险因素相匹配。
1.2 标本收集采用非抗凝管收集空腹外周血(5 mL), 室温下放置0.5~2 h凝固, 使用CS-15R离心机(德国BECKMAN公司), 4 ℃, 1 700 g离心10 min, 分组收集血清后再以2 000 g离心10 min完全去除细胞碎片。等分后-80 ℃储存。
1.3 血清总RNA提取和纯化按照TRIzol LS Reagent (美国Invitrogen公司)和mirvanaTM mirna isolation kit (美国Ambion公司)使用说明书进行RNA提取和纯化, nanodrop-2000检测纯化RNA浓度, 凝胶电泳检测RNA完整性。
1.4 miRNA芯片高通量筛选将提取血清总RNA进行靶标制备, 芯片杂交, 芯片清洗染色与扫描后, 应用miRNA芯片分析系统(Affymetrix® GeneChip® Command ConsoleTM 1.1, miRNA QC Tool软件)筛选3组血清中表达有差异的miRNAs。筛选标准:(1) B通道, Ratio≥2为上调 > 2倍的miRNA; (2) A通道, Ratio≤0.5为下调 > 2倍的miRNA。参照miRNA数据库the Sanger miRBase miRNA database V11确认miRNA的序列, 并应用Cluster 3.0软件进行聚类分析。
1.5 实时PCR检测按照miRcute miRNA cDNA第一链合成试剂盒和miRcute miRNA荧光定量检测试剂盒说明书进行反转录和扩增。
1.6 生物学信息分析应用互联网miRNA靶基因预测软件Pictar、Targetscan、MiRanda在线服务站点搜索差异表达miRNA的预测靶基因, 取至少2个软件预测到的基因作为主要靶基因。
1.7 统计学分析采用SPSS18.0软件进行统计学处理, 实验数据均以x±s表示。组间比较采用t检验, P < 0.05为差异有统计学意义。
2 结果 2.1 芯片实验检测结果 2.1.1 DM组与对照组miRNA表达比较结果显示, 与对照组比较, DM组患者血清中20个miRNA (miR-572、miR-603、miR-375等)表达上调, 其中miR-572上调最明显(4.22倍), 见图 1; 22个miRNA (miR-15b、miR-138、miR-875-5p等)表达下调, 其中miR-15b下调最明显(34.97倍), 见图 2。
|
| 图 1 与对照组比较DM组血清中上调的miRNAs Fig.1 Histogram representing miRNAs upregulated in the DM group compared with the control group |

|
| 图 2 与对照组比较DM组血清中下调的miRNAs Fig.2 Histogram representing miRNAs downregulated in the DM group compared with control group |
2.1.2 DN组与DM组miRNA表达比较
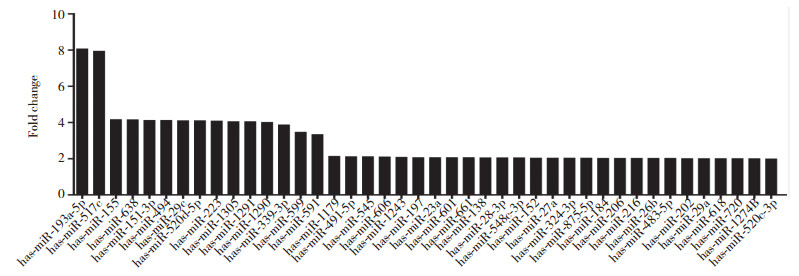
结果显示, 与DM组比较, DN患者血清中42个miRNA表达上调, 包括miR-193a-5p、miR-517c、miR-155、miR-638等, 其中miR-193a-5p与miR-517c上调最明显(分别为8.07倍、7.95倍), 见图 3; 19个miRNA表达下调, 包括miR-127-3p、miR-375、miR-572等, 其中miR-127-3p下调最明显(28.49倍), 见图 4。相反, DM组与对照组比较差异最显著的是miR-572和miR-15b (miR-572反而下调4.61倍, miR-15b上调1.88倍)。而DM组与对照组比较, 血清中miR-193a-5p下调2.09倍, miR-517c在对照组中未检测出, miR-127-3p上调3.97倍。
|
| 图 3 与DM组比较DN组血清中上调的miRNAs Fig.3 Histogram representing miRNAs that are upregulated in the DN group (compared to the DM group |

|
| 图 4 与DM组比较DN组血清中下调的miRNAs Fig.4 Histogram representing miRNAs that are downregulated in DN group compared to the DM group |
2.2 RT-PCR验证结果
为了验证miRNA芯片数据的可靠性, 选择部分差异性表达的miRNA进行荧光定量检测, 结果显示, 对照组、DM组和DN组中miR-1179呈递增式上调, miR-148b、miR-150呈递减式下调, 见表 1。
| Group | miR-1179 fold change | miR-148b fold change | miR-150 fold change |
| Control | 1.00 | 1.00 | 1.00 |
| DM | 2.031) | 2.041) | 2.031) |
| DN | 4.331), 2) | 4.111), 2) | 4.111), 2) |
| 1) compared with control group, P < 0.05;2) compared with DM group, P < 0.05. | |||
2.3 miR-1179、miR-148b和miR-150靶基因预测
miRNA靶基因预测软件结果显示, miR-148b调控763个预测靶基因(BCL2、USP33、ZFYVE26、TGFB2、HSPA4L、PODXL、FGF2等)的表达, 广泛参与信号转导、足细胞损伤、wnt-β通路调控的上皮间充质转分化等病理过程, 见表 2。miR-150调控287个预测靶基因(Notch信号通路、MAPK13、CBL、GLUT4等, 参与细胞凋亡、信号转导、葡萄糖转运等的表达)。miR-1179调控337个预测靶基因(TGFBR2、ZNF22、MTPN、FGF11、CDH6、SMAD等)的表达, 广泛参与细胞凋亡、细胞外基质增生等病理过程。
| Target genes | Function |
| BCL2 | apoptosis |
| MMP15 | extracellular matrix proliferation |
| LEPROTL1 | glycometabolism |
| ZFYVE26 | signal transduction |
| ZDHHC7 | signal transduction |
| USP33 | autophagy |
| TGFA | transforming growth factor |
| MAP3K9 | signal transduction |
| SMAD5 | signal transduction |
| SESN2 | CollagenⅡ |
| IGF1 | insulin-like growth factor |
| CABP7 | ca-binding protein |
| COL2A1 | collagen |
| TGFB2 | transforming growth factor |
| CADM1 | cell adhesion molecules |
| SERPINE1 | antiplasmin |
| CDK8 | signal transduction |
| RSBN1L | signal transduction |
| HSPA4L | heat shock protein |
| PODXL | podocyte injury |
| FGF2 | growth factor |
3 讨论
目前, 对于DN诊断和疾病进展检测依赖于尿微量白蛋白检测。然而并非所有微量白蛋白尿患者都会出现明显的蛋白尿和肾病。此外, 检测到微量蛋白尿时组织损伤和炎症可能已经发生, 微量蛋白尿对DN无特异性, 仅是肾小球损伤的一个标志。肾活检目前是诊断DN的主要方式, 但是这种侵入性且昂贵的手术仍有3%发生严重并发症的风险[6]。因此, 发现诊断和判断DN预后的新型生物标志物具有重要意义。
miRNA是一类能够调控mRNA表达的小分子非编码RNA。据报道, 在人类已鉴定出2 500余种成熟miRNA, 调节至少60%的蛋白质编码基因[7]。因此, miRNA可以调节许多基因表达, 以改变关键的细胞功能来影响各种疾病的进展。由于miRNA在生物流体(尿液和血浆)中稳定表达, 同时随着高通量测序、定量PCR、微阵列技术的建立, 作为DN敏感、非侵入性和阶段特异性精确诊断的生物标志物, miRNA研究已经变得非常有意义[5]。许多miRNA与DN的发生或微量白蛋白尿的出现相关, 并直接靶向肾脏纤维化途径, 包括转化生长因子-β和抑癌基因PTEN, 进而导致糖尿病肾脏病理损伤[8]。另一方面, 由于疾病进展的症状和分子机制可能因患者而异, 未来患者可能会根据其个体miRNA特征分离出个性化药物和治疗。课题组先前研究[9]发现, miR-503和miR-181d在糖尿病KKAy小鼠肾小球内的表达显著上调, 氯沙坦治疗可抑制其在糖尿病状态下的异常表达, 可能为DN提供新的有效治疗方法。
本研究发现3组血清中存在249个差异性表达的miRNA; 其中miR-1179呈递增式上调, miR-148b、miR-150呈递减式下调。miR-1179调控337个预测靶基因的表达, 包括TGFBR2、ZNF22、MTPN、FGF11、CDH6、SMAD等, 广泛参与细胞凋亡、细胞外基质增生等病理过程的发生。近期研究[10-12]发现miR-1179靶向细胞周期蛋白(cyclin E1, CCNE1)与非小细胞肺癌(non-small cell lung cancer, NSCLC)的细胞增殖相关, miR-1179靶向E2F转录因子5 (E2F5)控制细胞周期转变, miR-1179过表达可通过E2F5抑制胶质母细胞瘤细胞的增殖能力并诱导细胞周期停滞, 此外, 在胰腺癌细胞miR-1179的过表达抑制细胞迁移和侵袭, 而其在DN中的作用尚未见报道。
miR-148b调控763个预测靶基因的表达, 包括TGFB2、BCL2、USP33、ZFYVE26、TGFB2等, 广泛参与信号转导、足细胞损伤、wnt-β通路调控的上皮间充质转分化等病理过程的发生。研究[13]发现miR-148靶向TGF-β通路调节皮肤伤口愈合过程中的血管生成和内皮-间质转化。因此猜测miR-148的下调可能促进TGF-β信号增强, 与DN纤维化的发生密切相关。目前已有不少研究[14]报道miR-148b在不同疾病中的作用机制(miR-148/miR-152家族成员是自身免疫性疾病、慢性炎症性疾病和多种癌症的预后标志物和潜在治疗靶点)。SERINO等[15]在肾脏疾病中发现血清miR-148b和let-7b对于诊断IgA肾病具有特异性, 可能是一个新型、可靠、非侵入性的诊断方法; 此外, KUWAGATA等[16]发现在糖尿病状态下, 缺氧近端肾小管上皮细胞(proximal tubular cell, PTC)中mTORC1活化, 其通过减少miR-148b-3p表达使TNFR2过表达而导致细胞凋亡, 调节致病途径可能成为糖尿病PTC损伤的新疗法。miR-148可能在DN的发生发展中发挥重要作用, 其可能的作用机制有待进一步深入研究。
miR-150调控287个预测靶基因的表达, 包括Notch信号通路、MAPK13、CBL、GLUT4等, 参与细胞凋亡、信号转导、葡萄糖转运等。近年来, 许多临床研究发现miR-150具有潜在的预后价值。在慢性淋巴性白血病中, miR-150显著下调, 与疾病的发生相关[17]。在各种其他实体肿瘤组织(肺癌、乳腺癌、食道癌、结肠直肠癌和胰腺癌)中也发现了miR-150的异常表达。miR-150已被证实在食管鳞状细胞癌和原发性结直肠癌中显著下调, 预示着预后不良。在乳腺癌细胞系中, miR-150表达增加, 促进生长和克隆形成, 并减少细胞凋亡。另外的研究表明miR-150在其他肿瘤(前列腺癌和甲状腺癌)中表达增加。miR-150有可能成为监测癌症预后和进展的新的预测因子[18]。在狼疮性肾炎中, miR-150通过下调SOCS1促进肾脏纤维化, 是狼疮性肾炎肾损伤的生物标志物[19]。YANG等[20]发现miR-150可能在DN间充质干细胞的治疗中发挥重要作用。此外, XIE等[21]通过比较有和没有大量白蛋白尿的2型糖尿病患者尿外泌体中差异性表达的miRNA, 发现miR-150-5p上调, 可能是早期DN患者的新型生物标志物。
综上所述, 本研究结果丰富了DN miRNA差异表达谱, 这些差异性表达的miRNA可能涉及DN的发生和发展, 但需进一步对筛选出的差异miRNA进行多中心、大样本的临床验证和功能学研究, 进而将研究成果向临床应用转化, 找到DN的新型生物标志物和治疗新靶点。
| [1] |
XUE R, GUI D, ZHENG L, et al. Mechanistic insight and management of diabetic nephropathy:recent progress and future perspective[J]. J Diabetes Res, 2017, 2017: 1839809. DOI:10.1155/2017/1839809 |
| [2] |
ZHANG L, LONG J, JIANG W, et al. Trends in chronic kidney disease in China[J]. N Engl J Med, 2016, 375(9): 905-906. DOI:10.1056/nejmc1602469 |
| [3] |
BELLIZZI V, CONTE G, BORRELLI S, et al. Controversial issues in CKD clinical practice:position statement of the CKD-treatment working group of the Italian Society of Nephrology[J]. J Nephrol, 2017, 30(2): 159-170. DOI:10.1007/s40620-016-0338-x |
| [4] |
STEFANI G, SLACK FJ. Small non-coding RNAs in animal development[J]. Nat Rev Mol Cell Biol, 2008, 9(3): 219-230. DOI:10.1038/nrm2347 |
| [5] |
KATO M, NATARAJAN R. MicroRNAs in diabetic nephropathy:functions, biomarkers, and therapeutic targets[J]. Ann N Y Acad Sci, 2015, 1353: 72-88. DOI:10.1111/nyas.12758 |
| [6] |
SIMPSON K, WONNACOTT A, FRASER DJ, et al. MicroRNAs in diabetic nephropathy:from biomarkers to therapy[J]. Curr Diab Rep, 2016, 16(3): 1-7. DOI:10.1007/s11892-016-0724-8 |
| [7] |
KOZOMARA A, GRIFFITHSJONES S. miRBase:annotating high confidence microRNAs using deep sequencing data[J]. Nucleic Acids Res, 2014, 42(Database issue): D68-D73. DOI:10.1093/nar/gkt1181 |
| [8] |
GHAI V, WU X, BHEDA-MALGE A, et al. Genome-wide profiling of urinary extracellular vesicle micrornas associated with diabetic nephropathy in type 1 diabetes[J]. Kidney Inter Report, 2017, 3(3): 555-572. DOI:10.1016/j.ekir.2017.11.019 |
| [9] |
范秋灵, 张丛笑, 刘晓丹, 等. 自发性2型糖尿病KKAy小鼠肾小球microRNA表达谱和氯沙坦治疗效应[J]. 中华肾脏病杂志, 2012, 28(5): 397-402. DOI:10.7666/d.y2051850 |
| [10] |
LIN C, HU Z, YUAN G, et al. MicroRNA-1179 inhibits the proliferation, migration and invasion of human pancreatic cancer cells by targeting E2F5[J]. Chem Biol Interactions, 2018, 291: 65-71. DOI:10.1016/j.cbi.2018.05.017 |
| [11] |
Heller G, Altenberger C, Steiner I, et al. DNA methylation of microRNA-coding genes in non-small-cell lung cancer patients[J]. J Pathol, 2018, 245(4): 387-398. DOI:10.1002/path.5079 |
| [12] |
XU X, CAI N, ZHI T, et al. MicroRNA-1179 inhibits glioblastoma cell proliferation and cell cycle progression via directly targeting E2F transcription factor 5[J]. Am J Cancer Res, 2017, 7(8): 1680-1692. |
| [13] |
MISCIANINOV V, MARTELLO A, ROSE L, et al. MicroRNA-148b targets the TGF-β pathway to regulate angiogenesis and endothelial-to-mesenchymal transition during skin wound healing[J]. 2018, 26(8):1996-2007. DOI:10.1016/j.ymthe.2018.05.002.
|
| [14] |
FRIEDRICH M, PRACHT K, MASHREGHI M, et al. The role of the miR-148/-152 family in physiology and disease[J]. Eur J Immunol, 2017, 47(12): 2026-2038. DOI:10.1002/eji.201747132 |
| [15] |
SERINO G, PESCE F, SALLUSTIO F, et al. In a retrospective international study, circulating miR-148b and let-7b were found to be serum markers for detecting primary IgA nephropathy[J]. Kidney Inter, 2016, 89(3): 683-692. DOI:10.1038/ki.2015.333 |
| [16] |
KUWAGATA S, KUME S, CHIN-KANASAKI M, et al. MicroRNA148b-3p inhibits mTORC1-dependent apoptosis in diabetes by repressing TNFR2 in proximal tubular cells[J]. Kidney Inter, 2016, 90(6): 1211-1225. DOI:10.1016/j.kint.2016.06.036 |
| [17] |
MRAZ M, CHEN L, RASSENTI LZ, et al. miR-150 influences B-cell receptor signaling in chronic lymphocytic leukemia by regulating expression of GAB1 and FOXP1[J]. Blood, 2014, 124(1): 84. DOI:10.1182/blood-2013-09-527234 |
| [18] |
HUANG XL, ZHANG L, LI JP, et al. MicroRNA-150:a potential regulator in pathogens infection and autoimmune diseases[J]. Autoimmunity, 2015, 48(8): 503-510. DOI:10.3109/08916934.2015.1072518 |
| [19] |
ZHOU H, HASNI SA, PEREZ P, et al. miR-150 promotes renal fibrosis in lupus nephritis by downregulating SOCS1[J]. J Am Soc Nephrol, 2013, 24(7): 1073-1087. DOI:10.1681/ASN.2012080849 |
| [20] |
YANG H, ZHANG X, XIN G, et al. Investigation of mechanisms of mesenchymal stem cells for treatment of diabetic nephropathy via construction of a miRNA-TF-mRNA network[J]. Ren Fail, 2018, 40(1): 136-145. DOI:10.1080/0886022X.2017.1421556 |
| [21] |
XIE YJ, JIA YJ, XIE CH, et al. Urinary exosomal microRNA Profiling in incipient type 2 diabetic kidney disease[J]. J Diabates Res, 2017, 2017(5): 1-10. DOI:10.1155/2017/6978984 |
 2019, Vol. 48
2019, Vol. 48