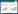文章信息
- 孙精文, 张蕊, 李艳
- SUN Jingwen, ZHANG Rui, LI Yan
- 急性髓系白血病SPRED1基因启动子甲基化状态的研究
- A Study of Methylation in the SPRED1 Gene Promoter in Acute Myeloid Leukemia
- 中国医科大学学报, 2019, 48(8): 688-693
- Journal of China Medical University, 2019, 48(8): 688-693
-
文章历史
- 收稿日期:2018-10-08
- 网络出版时间:2019-07-15 10:05
急性髓系白血病(acute myeloid leukemia, AML)是一组以造血干细胞增殖失控, 分化受阻, 同时抑制正常造血为特点的异质性造血系统恶性疾病。近年来, 随着基因组研究的不断深入, 发现表观遗传学的修饰作用, 如DNA甲基化状态的改变, 特别是抑癌基因启动子的甲基化, 引起抑癌基因沉默, 是影响白血病发生和发展的重要分子机制[1]。表观遗传修饰是指不涉及DNA序列改变的一种可逆的、动态的及可随细胞分裂而遗传的基因调节方法[2]。DNA甲基化是目前表观遗传学最主要及常见的基因转录前调控机制[3-4], 出现在不同的疾病中, 如胶质母细胞瘤[5]、淋巴细胞白血病[6]和AML[7]。已有文献[8]中, 大约50%的AML患者缺乏细胞遗传学异常, 处于中度风险组, 而很大比例的患者携带未知AML相关调控基因[9-10]。因此, 关于AML的发生和发展机制, 表观遗传学修饰机制比只用体内突变来解释更合理。
Ras/MAPK信号通路已被证实参与AML的发病机制, SPRED1 (human sprout-related EVH1 domain-containing 1)是参与调控AML Ras/MAPK信号通路的抑癌基因[11-12]。SPRED1于2001年由YOSHIMURA等[13]在鼠的破骨细胞cDNA文库中发现。SPRED1位于染色体15q13.2, 编码含有444个氨基酸的蛋白质, 具有3个功能域, 即N端的EVH1域、中间的c-kit结合域和C端的SPRY相关域, 与SPRED2、SPRED3同属于SPRED家族[14]。人类SPRED1在肺、脑、脊髓、肾脏和乳腺中高表达, 在肝、胰腺、前列腺、甲状腺、肌肉、骨骼、骨髓中表达相对较低[15-17]。SPRED1与神经纤维蛋白1 (protein-neurofibromin, NF1)相互作用, 下调Ras/MAPK信号通路[14]。在肝细胞癌、前列腺癌和淋巴瘤中, SPRED1可抑制Ras/MAPK信号通路和恶性细胞转移。另外, SPRED1已被证实在儿童急性白血病中下调, 是Ras/MAPK信号通路的抑癌基因[12]。然而SPRED1的下调机制目前仍未知。研究[18]发现, 伴有SPRED1突变者具有白血病倾向, 但后续研究[12, 19]表明, SPRED1突变和缺失在AML并不常见。本研究拟探讨AML患者SPRED1的表观遗传学状态, 分析其与SPRED1 mRNA水平和预后参数的相关性。
1 材料与方法 1.1 材料 1.1.1 研究对象选择2015年10月至2017年6月于中国医科大学附属第一医院住院的AML患者共50例(病例组), 其中, 男27例, 女23例, 年龄16~80岁(中位年龄45岁)。所有患者依据2016年世界卫生组织AML分型标准[20]进行诊断、分型。对照组为20例健康志愿者, 年龄18~75岁(中位年龄54岁)。本研究获得中国医科大学伦理委员会批准(#AF-SOP-07-1.0-01)以及研究对象和家属的知情同意。
1.1.2 主要试剂AxyPrep血基因组DNA小量试剂盒[康宁生命科学(吴江)有限公司]; M-MLV逆转录酶试剂盒(美国Promega公司); Trizol试剂盒、SYBR GREEN试剂盒、单链cDNA合成试剂盒(日本TaKaRa公司); EZ DNA Methylation-Gold Kit、热启动的DNA聚合酶(美国Zymo公司)。
1.1.3 主要仪器NanoDropTM 2000分光光度计(美国Thermo公司); PCR仪(7500, 美国ABI公司); K960热循环仪(杭州晶格科学仪器有限公司); 低温高速离心机(德国Eppendorf公司); MassARRAY平台(美国Sequenom公司)。
1.2 方法 1.2.1 RNA的提取及实时荧光定量PCR抽取骨髓样本, 用Trizol试剂盒从骨髓样本的单个核细胞中提取RNA, 紫外分光光度仪检测A260/A280均为1.8~2.0, 以确定其浓度与纯度。用单链cDNA合成试剂盒逆转录合成cDNA, 置于-20 ℃保存备用。行实时定量PCR, β-actin为内参照基因。PCR引物由Invitrogen公司合成, SPRED1正义引物序列为5’-GATGAGCGAGAGACGGAGAC-3’, 反义引物序列为5’-GTCTCTGAGTCTCTCCACGGA-3’; β-actin正义引物序列为5’-GTGGACATCCGCAAAGAC-3’, 反义引物序列为5’-AAAGGGTGTAACGCAACTAA-3’。反应条件为95 ℃ 30 s; 95 ℃ 5 s, 60 ℃ 34 s, 40个循环; 95 ℃ 15 s, 60 ℃ 1 min, 95 ℃ 15 s, 60 ℃ 15 s。随后进行熔解曲线的获取和分析, 以确定PCR反应的特异性。SPRED1的相对表达量用2-ΔΔCt法计算。
1.2.2 DNA的提取及甲基化定量PCR用AxyPrep血基因组DNA小量试剂盒从骨髓样本的单个核细胞中提取DNA, 紫外分光光度仪检测A260/A280均为1.8~2.0。所有DNA立即硫化修饰处理或-20 ℃保存备用。参照EZ DNA Methylation Kit说明书, 对抽提的DNA进行亚硫酸氢盐处理, 在MassArray平台进行DNA甲基化的定量检测。应用Sequenom EpiDesigner software (www.epidesigner.com)设计引物, 正义引物序列为5’-AGGATAATGTTGTTGTTGAGGTAGGaggaagagag-3’, 反义引物序列为5’-CTAAATCCCAAATACTCCCAAATTCcagtaatacgactcactatagggagaaggct-3’。反应条件为94 ℃ 4 min; 94 ℃ 20 s, 64 ℃ 30 s, 72 ℃ 1 min, 40个循环; 72 ℃ 5 min。
1.3 统计学分析采用SPSS 22.0软件和GraphPad Prism软件(version 5.0, 美国GraphPad Software公司)进行统计分析及数据绘图。采用Mann-Whitney检验评估病例组与对照组之间的SPRED1基因启动子甲基化差异, 采用Spearman相关系数r评估SPRED1基因启动子甲基化状态与mRNA水平和预后参数之间的相关性。所有检验均设定为双侧检验, P < 0.05为差异有统计学意义。
2 结果 2.1 AML患者SPRED1基因启动子甲基化水平显著升高SPRED1基因(#1:310-723 bp)包含12个CpG位点(图 1A), 用Sequenom MassARRAY平台分析50例AML患者和20例健康对照的甲基化水平。分析之前, 进行严格的质量控制, 删除潜在的不可靠的测量数据[21], 包括少于30%的样本检测到的CpG位点(unreliable CpG units)和丢失数据超过30%的样本(unreliable samples), 最后所获得的有效CpG位点为9个。
|
| A, red CpG units could not be detected due to sequential problems; blue CpG units were analyzed for DNA methylation; B, average methylation levels for SPRED1 of 20 controls and 50 patients with AML. *P < 0.05 (Mann-Whitney test). 图 1 AML患者与健康对照组SPRED1启动子甲基化水平的比较 Fig.1 Comparison of SPRED1 gene promoter methylation levels between control individuals and patients with AML |
#1_CpG_1在AML患者中的甲基化水平(86.94%± 8.61%)明显高于健康对照组(82.70%±2.85%, P < 0.01);#1_CpG_2在AML患者中的甲基化水平(86.18%± 29.40%)明显高于健康对照组(80.90%±28.98%, P = 0.031);#1_CpG_11在AML患者中的甲基化水平(88.68%±15.38%)高于健康对照组(86.00%±7.71%, P = 0.039, 图 1B), 其他位点在AML病例组和健康对照组中均无统计学差异(P > 0.05)。
2.2 AML患者SPRED1基因启动子甲基化水平与SPRED1 mRNA表达水平呈负相关AML患者的SPRED1 mRNA水平(0.92±2.73)明显低于健康对照组(1.77±4.79, P < 0.01)。在AML患者中, #1_CpG_11甲基化水平与SPRED1 mRNA表达水平呈负相关(r = -0.427, P = 0.004) (图 2A); #1_CpG_2、#1_CpG_1甲基化水平与SPRED1 mRNA表达水平无统计学相关性(r = -0.033, P = 0.822;r = -0.005, P = 0.972) (图 2B、2C)。
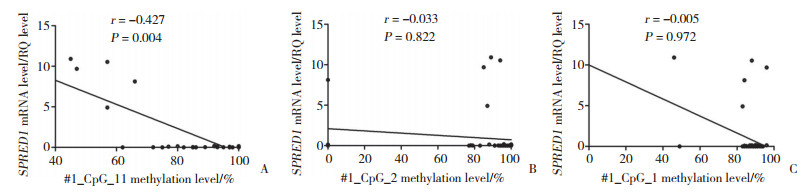
|
| A, association of methylation status and mRNA levels for #1_CpG_11 in 44 patients with AML. RQ values represent relative mRNA expression levels for SPRED1 gene. *P < 0.05 (Spearman); B, association of methylation status and mRNA levels for #1_CpG_2 in 50 patients with AML; C, association of methylation status and mRNA levels for #1_CpG_1 in 50 patients with AML. 图 2 AML患者SPRED1基因启动子甲基化水平与SPRED1 mRNA水平的相关性 Fig.2 Correlation between SPRED1 gene promoter methylation level and SPRED1 mRNA expression level in AML patients |
2.3 SPRED1基因启动子甲基化水平与AML患者的预后参数缺乏相关性
分析SPRED1#1_CpG_11甲基化水平与一系列AML患者预后相关的临床和实验室检查参数之间的关系, 包括年龄、性别、French-American-British (FAB)分型、白细胞(white blood cell, WBC)数、血红蛋白(hemoglobin, Hb)含量、血小板(platelet, PLT)数、骨髓原始细胞数、染色体核型和基因突变, 发现#1_CpG_11甲基化水平与上述预后参数之间均无相关性(P > 0.05)。依据2017年第3版AML美国国立综合癌症网络(national comprehensive cancer network, NCCN)指南, 基于细胞遗传学和分子学异常, AML风险分层为低危、中危和高危3组, 本研究结果表明低危组、中危组和高危组之间的SPRED1基因启动子甲基化水平无统计学差异(P > 0.05), 见表 1。
| Parameters | n (%) | #1_CpG_11 methylation level of SPRED1 (x±s, %) |
| Age (year) | ||
| ≥60 | 7(14) | 77.00±23.34 |
| < 60 | 43(86) | 89.54±14.26 |
| Gender | ||
| Male | 27(54) | 89.21±12.57 |
| Female | 23(46) | 88.05±18.18 |
| FAB classification | ||
| M2 | 14(28) | 88.38±13.08 |
| M5 | 28(56) | 88.84±16.22 |
| Others | 8(16) | 88.67±16.39 |
| Peripheral blood count | ||
| WBC > 10×109/L | 34(68) | 91.09±14.17 |
| WBC≤10×109/L | 16(32) | 82.25±16.60 |
| Hb≥80 g/L | 30(60) | 90.48±12.92 |
| Hb < 80 g/L | 20(40) | 86.32±17.85 |
| PLT≥50×109/L | 14(28) | 91.62±12.49 |
| PLT < 50×109/L | 36(72) | 87.45±16.29 |
| Bone marrow | ||
| Blast≥65.8 % | 25(50) | 89.70±16.31 |
| Blast < 65.8% | 25(50) | 87.57±14.21 |
| Number of karyotypic abnormalities | ||
| Normal | 18(37.50) | 84.85±14.80 |
| 1-2 | 22(43.75) | 90.29±15.53 |
| Complex | 10(18.75) | 90.30±14.93 |
| Number of gene mutation | ||
| None | 2(12.25) | 89.00±4.00 |
| Isolated | 4(62.50) | 98.00±3.46 |
| More than two | 10(12.50) | 96.38±5.55 |
| Gene mutations | ||
| FLT3/C-KIT | 15(30) | 93.08±10.76 |
| CEBPA | 8(16) | 89.50±17.82 |
| NPM1 | 8(16) | 98.50±7.80 |
| DNMT3A | 9(18) | 91.00±14.24 |
| TET2 | 9(18) | 95.50±9.20 |
| IDH1/2 | 24(48) | 85.60±15.46 |
| Risk stratification* | 17(34) | 87.50±18.01 |
| Low | ||
| Medium | 12(24) | 76.55±22.28 |
| High | 22(44) | 91.00±10.42 |
| Number of karyotypic abnormalities | 16(32) | 95.38±6.00 |
| * risk status based on cytogenetic and molecular abnormalities according to national comprehensive cancer network (NCCN) guideline version 3.2017 acute myeloid leukemia. WBC, white blood cell; Hb, hemoglobin; PLT, platelet. | ||
根据在白血病发生发展中的作用, 突变基因分为FLT3和c-KIT (促进增殖), CEBPA (减少分化), NPM1 (参与细胞周期调控)以及DNMT3A、TET2和IDH1/2 (调控表观遗传学) 4类。50例AML患者中, 46例发生了基因突变, 其中, FLT3-ITD突变15例, c-KIT突变8例, CEBPA突变8例, NPM1突变9例, DNMT3A突变9例, TET2突变24例, IDH1/2突变17例。本研究显示, #1_CpG_11甲基化水平与上述预后相关突变基因之间均无相关性(P > 0.05)。
3 讨论表观遗传学调控机制在疾病的发生发展中起重要作用, 主要包括染色体重塑、组蛋白修饰、DNA甲基化、非编码RNA调控等。DNA甲基化在真核细胞中是重要的表观遗传学调控机制, 已被广泛研究[22]。Sequenom MassARRAY平台具有高度的准确性、敏感性和高通量, 是近几年新兴的基因甲基化定量检测方法。MassARRAY系统相比亚硫酸盐测序PCR (bisulfite sequencing PCR, BSP)能更真实地反映低甲基化区域的甲基化水平, 最低可以检测到5%[23], 因此也更能准确地反映与基因表达水平之间的关系。本研究结果显示, AML患者SPRED1基因启动子甲基化水平增高, 且与SPRED1 mRNA水平呈负相关, 表明SPRED1基因启动子甲基化状态的改变可能影响其表达并促进白血病的发生与发展。本研究证实了抑癌基因SPRED1启动子甲基化水平在AML患者中明显增高, 同时SPRED1基因表观沉默。真核生物启动子区域包括2部分, 一是核心启动子区, 具备结合、控制转录起始前复合物的装配、定位转录起始点、控制转录方向和响应胞内激活子或抑制子等功能, 二是上游序列区, 决定基因转录的特异性、活性和效率, 确保精准转录。启动子区元件构成及其功能对于转录水平的调控至关重要, 因此笔者推测这可能是SPRED1基因不同位点甲基化水平与基因表达的相关性存在差异的原因, 有待进一步研究。有研究[24]发现, SPRED1基因的表达水平与肿瘤浸润和转移的发生率呈负相关。体内外研究[25-26]也发现, SPRED1基因高表达可通过抑制制细胞外调节蛋白激酶(extracellular regulated protein kinases, ERK)的激活有效抑制肿瘤细胞。在儿童AML中已证实SPRED1基因表达的下调与Ras/MAPK通路的激活有关[12]。因此, 推测SPRED1基因启动子高甲基化状态引起基因表达水平降低, 促进Ras/MAPK通路激活, 从而导致白血病的发生。
AML的发生发展与甲基化调控基因, 如DNMT3A、TET2及IDH1/2的突变密切相关。DNMT3A是AML细胞DNA甲基化的重要介质, 与不良预后密切相关[9, 27]。IDH1/2和TET2突变与导致基因组高甲基化相关[27-28]。有研究[12]发现, FLT3突变与SPRED1基因低表达密切相关。CEBPA基因编码一种造血干细胞分化相关的转录因子[29], 其突变与AML患者的良好预后密切相关[19]。在本研究中, 未发现SPRED1基因启动子甲基化状态与突变基因之间的关系具有统计学意义, 考虑可能是由于样本量不足所致。未来还需研究SPRED1甲基化状态与治疗反应和预后的关系, 全面了解SPRED1高甲基化状态在AML发生发展中的作用。
| [1] |
MOHAMMADI S, GHAFFARI SH, SHAIEGAN M, et al. Curcumin veto the effects of osteopontin (OPN) specific inhibitor on leukemic stem cell colony forming potential via promotion of OPN overexpression[J]. Int J Hematol Oncol Stem Cell Res, 2016, 10(3): 120-129. |
| [2] |
NOWACKA-ZAWISZA M, WI S'NIK E. DNA methylation and histone modifications as epigenetic regulation in prostate cancer[J]. Oncol Rep, 2017, 38(5): 2587-2596. DOI:10.3892/or.2017.5972 |
| [3] |
STEPHEN JK, CHEN KM, MERRITT J, et al. Methylation markers for early detection and differentiation of follicular thyroid cancer subtypes[J]. Cancer Clin Oncol, 2015, 4(2): 1-12. DOI:10.5539/cco.v4n2p1 |
| [4] |
REPPE S, DATTA H, GAUTVIK KM. The influence of DNA methylation on bone cells[J]. Curr Genomics, 2015, 16(6): 384-392. DOI:10.2174/1389202916666150817202913 |
| [5] |
MAZOR T, PANKOV A, JOHNSON B E, et al. DNA methylation and somatic mutations converge on the cell cycle and define similar evolutionary histories in brain tumors[J]. Cancer Cell, 2015, 28(3): 307-317. DOI:10.1016/j.ccell.2015.07.012 |
| [6] |
VAITKIENE P, SKIRIUTE D, SKAUMINAS K, et al. GATA4 and DcR1 methylation in glioblastomas[J]. Diagn Patho, 2013, 8(1): 7-7. DOI:10.1186/1746-1596-8-7 |
| [7] |
AL-ALI HK, JAEKEL N, NIEDERWIESER D. The role of hypomethylating agents in the treatment of elderly patients with AML[J]. J Geriatr Oncol, 2014, 5(1): 89-105. DOI:10.1016/j.jgo.2013.08.004 |
| [8] |
ARDIMAN JW, THIELE J, ARBER DA, et al. The 2008 revision of the World Health Organization (WHO) classification of myeloid neoplasms and acute leukemia:rationale and important changes[J]. Blood, 2009, 114(5): 937-951. DOI:10.1182/blood-2009-03-209262 |
| [9] |
PATEL JP, GÖNEN M, FIGUEROA ME, et al. Prognostic relevance of integrated genetic profiling in acute myeloid leukemia[J]. N Engl J Med, 2012, 366(12): 1079-1089. DOI:10.1056/NEJMoa1112304 |
| [10] |
SHEN Y, ZHU YM, FAN X, et al. Gene mutation patterns and their prognostic impact in a cohort of 1185 patients with acute myeloid leukemia[J]. Blood, 2011, 118(20): 5593-5603. DOI:10.1182/blood-2011-03-343988 |
| [11] |
BREMS H, CHMARA M, SAHBATOU M, et al. Germline loss-of-function mutations in SPRED1 cause a neurofibromatosis 1-like phenotype[J]. Nat Genet, 2007, 39(9): 1120-1126. DOI:10.1038/ng2113 |
| [12] |
PASMANT E, GILBERT-DUSSARDIER B, PETIT A, et al. SPRED1, a RAS MAPK pathway inhibitor that causes Legius syndrome, is a tumour suppressor downregulated in paediatric acute myeloblastic leukaemia[J]. Oncogene, 2015, 34(5): 631-638. DOI:10.1038/onc.2013.587 |
| [13] |
BREMS H, LEGIUS E. Legius syndrome, an update. molecular pathology of mutations in spred1[J]. Keio J Med, 2013, 62(4): 107-112. DOI:10.2302/kjm.2013-0002-RE |
| [14] |
HIRATA Y, BREMS H, SUZUKI M, et al. Interaction between a domain of the negative regulator of the Ras-ERK Pathway, SPRED1 protein, and the gtpase-activating protein-related domain of neurofibromin is implicated in legius syndrome and neurofibromatosis type 1[J]. J Biol Chem, 2016, 291(7): 3124-3134. DOI:10.1074/jbc.M115.703710 |
| [15] |
YANAI I, BENJAMIN H, SHMOISH M, et al. Genome-wide midrange transcription profiles reveal expression level relationships in human tissue specification[J]. Bioinformatics, 2005, 21(5): 650-659. DOI:10.1093/bioinformatics/bti042 |
| [16] |
SHMUELI O, HORNSABAN S, CHALIFACASPI V, et al. GeneNote:whole genome expression profiles in normal human tissues[J]. CR Biol, 2003, 326(10): 1067-1072. DOI:10.1016/j.crvi.2003.09.012 |
| [17] |
JIANG CF, SHI ZM, LI DM, et al. Estrogen-induced miR-196a elevation promotes tumor growth and metastasis via targeting SPRED1 in breast cancer[J]. Mol Cancer, 2018, 17(1): 83. DOI:10.1186/s12943-018-0830-0 |
| [18] |
PASMANT E, BALLERINI P, LAPILLONNE H, et al. SPRED1 disorder and predisposition to leukemia in children[J]. Blood, 2009, 114(5): 1131. DOI:10.1182/blood-2009-04-218503 |
| [19] |
ZHANG R, LEE JY, WANG X, et al. Identification of novel genomic aberrations in AML-M5 in a level of array CGH[J]. PLos One, 2014, 9(4): e87637. DOI:10.1371/journal.pone.0087637 |
| [20] |
ARBER DA, ORAZI A, HASSERJIAN R, et al. The 2016 revision to the World Health Organization (WHO) classification of myeloid neoplasms and acute leukemia[J]. Blood, 2016, 127(20): 2391-2405. DOI:10.1182/blood-2016-03-643544 |
| [21] |
OLLIKAINEN M, SMITH KR, JOO EJ, et al. DNA methylation analysis of multiple tissues from newborn twins reveals both genetic and intrauterine components to variation in the human neonatal epigenome[J]. Hum Mol Genet, 2010, 19(21): 4176-4188. DOI:10.1093/hmg/ddq336 |
| [22] |
LI S, GARRETTBAKELMAN FE, CHUNG SS, et al. Distinct evolution and dynamics of epigenetic and genetic heterogeneity in acute myeloid leukemia[J]. Nat Med, 2016, 22(7): 792-799. DOI:10.1038/nm.4125 |
| [23] |
OHTANI K, DIMMELER S. Epigenetic regulation of cardiovascular differentiation[J]. Cardiovas Res, 2011, 90(3): 404-412. DOI:10.1093/cvr/cvr019 |
| [24] |
YOSHIDA T, HISAMOTO T, AKIBA J, et al. Spreds, inhibitors of the Ras/ERK signal transduction, are dysregulated in human hepatocellular carcinoma and linked to the malignant phenotype of tumors[J]. Oncogene, 2006, 25(45): 6056-6066. DOI:10.1038/sj.onc.1209635 |
| [25] |
KATO R, NONAMI A, TAKETOMI T, et al. Molecular cloning of mammalian Spred-3 which suppresses tyrosine kinase-mediated Erk activation[J]. Biochem Biophy Res Commun, 2003, 302(4): 767-772. DOI:10.1016/s0006-291x(03)00259-6 |
| [26] |
NOBUHISA I, KATO R, INOUE H, et al. Spred-2 suppresses aorta-gonad-mesonephros hematopoiesis by Inhibiting MAP kinase activation[J]. J Exp Med, 2004, 199(5): 737-742. DOI:10.1084/jem.20030830 |
| [27] |
WOUTERS BJ, DELWEL R. Epigenetics and approaches to targeted epigenetic therapy in acute myeloid leukemia[J]. Blood, 2016, 127(1): 42-52. DOI:10.1182/blood-2015-07-604512 |
| [28] |
KROEZE LI, ASLANYAN MG, VAN ROOIJ A, et al. Characterization of acute myeloid leukemia based on levels of global hydroxymethylation[J]. Blood, 2014, 124(7): 1110-1118. DOI:10.1182/blood-2013-08-518514 |
| [29] |
MUELLER BU, PABST T. C/EBPalpha and the pathophysiology of acute myeloid leukemia[J]. Curr Opin Hematol, 2006, 13(1): 7-14. DOI:10.1097/01.moh.0000190110.08156.96 |
 2019, Vol. 48
2019, Vol. 48