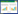文章信息
- 温行, 李章富, 王辉, 孙绍华, 郭星, 李福才
- WEN Xing, LI Zhangfu, WANG Hui, SUN Shaohua, GUO Xing, LI Fucai
- miR-200c调控肽基脯氨酰顺反异构酶对喉癌Hep-2细胞生物学行为的影响
- Effect of miR-200c Regulation of Peptidyl-prolyl Cis/trans Isomerase on the Biological Behavior of Hep-2 Cells
- 中国医科大学学报, 2019, 48(1): 17-22, 28
- Journal of China Medical University, 2019, 48(1): 17-22, 28
-
文章历史
- 收稿日期:2018-06-14
- 网络出版时间:2018-12-29 14:08
2. 中国医科大学附属第一医院耳鼻喉科, 沈阳 110001
2. Department of Otolaryngology, The First Hospital, China Medical University, Shenyang 110001, China
喉鳞状细胞癌(简称喉癌)是人类最常见的头颈部肿瘤之一,伴有高发病率和死亡率,而且预后不良[1-2]。因此,探究喉癌的发生发展机制,寻找其诊断及预后的生物分子标记物至关重要。miR-200c最早发现在多种癌细胞系中表达失调,目前,miR-200c是否参与喉癌的发生和发展尚不明确。肽基脯氨酰顺反异构酶(peptidyl-prolyl cis/trans isomerase,NIMA-interacting 1,PIN1)在中心体复制及分离等过程中发挥着重要作用,在绝大多数人类肿瘤中高表达[3]。前期,本课题组发现过表达PIN1加剧了Hep-2细胞中心体扩增、细胞分裂异常和细胞迁移。本研究将探讨miR-200c对喉癌细胞系Hep-2细胞生物学行为的影响,以及miR-200c是否通过调控PIN1发挥其生物学功能,为喉癌的诊断、治疗和预后分析提供科学依据和线索。
1 材料与方法 1.1 材料人喉癌细胞系Hep-2;人喉癌组织标本(取自中国医科大学附属第一医院耳鼻喉科喉癌患者,全部患者术前未行放化疗,标本保存于-80 ℃冰箱中,所有标本均经病理学检查确诊,并经医学伦理委员会批准及患者知情同意);RPMI1640培养基(美国HyClone公司);qRT-PCR试剂盒(日本TaKaRa公司);PIN1和β-actin抗体(美国Proteintech公司);BCA测定蛋白浓度试剂盒(中国碧云天生物技术公司);苏木素伊红(HE)染色试剂盒(中国碧云天生物技术公司);Transwell小室(美国Corning公司)。
1.2 方法 1.2.1细胞培养与转染:人喉癌细胞系Hep-2细胞培养于RPMI1640培养基(10%胎牛血清),5% CO2,37 ℃孵箱中孵育,24 h更换新鲜培养液。当细胞长满瓶底后,用0.25%的胰酶消化,进行传代。取对数生长期的Hep-2细胞接种于6孔板中,待细胞密度达到80%时,进行质粒和小RNAs(miR-200c模拟物、模拟物对照、miR-200c抑制物、抑制物对照)转染。
1.2.2质粒提取:将含有空载质粒和重组质粒pcDNA3.1-PIN1的菌株按照1:100的比例接种到200 mL LB培养基(氨苄青霉素,100 μg/mL)中,37 ℃,水平震荡培养箱中过夜培养。第2天,将200 mL菌液加入离心管中,按TIANGEN质粒提取试剂盒说明书提取质粒。
1.2.3实时荧光定量PCR:转染48 h后,加入TRIzol试剂提取RNA。使用全式金miRNA反转试剂盒和TaKaRa反转试剂盒合成cDNA。选择U6和GAPDH作为内参对照,反应条件:95 ℃ 30 s,1个循环;95 ℃ 5 s,60 ℃ 34 s,40个循环。
1.2.4Western blotting:转染48 h后,裂解细胞提取总蛋白。BCA试剂盒测定各样品蛋白浓度,上样量为20 μg。电泳,转膜,封闭,孵一抗PIN1(1:500),β-actin(1:2 000),4 ℃摇床过夜,二抗(1:5 000)37 ℃恒温孵育1 h,ECL发光。应用Image J软件进行灰度分析。
1.2.5双荧光素酶报告基因实验:将野生型wt-pGL3-PIN1-3’UTR和突变型mut-pGL3-PIN1-3’UTR质粒分别与miR-200c模拟物和模拟物对照共同转染至Hep-2细胞,pRL-TK作为内对照,24 h后收集细胞,应用Promega双荧光素酶报告基因检测试剂盒检测荧光素酶活性。
1.2.6CCK8检测:接种细胞于6孔板中并做相应的转染后收集细胞,胰酶消化细胞,调整细胞浓度。96孔板每孔加入100 μL细胞悬液,间隔24 h,检测前每孔加入100 μL新鲜培养基(含10 μL CCK8试剂),培养箱内孵育2 h。测定450 nm处的吸光度(optical density,OD)值。
1.2.7Transwell实验:接种细胞于6孔板中进行转染,转染后24 h消化细胞,双无培养基吹成细胞悬液,调整细胞浓度。小室上室加入100 μL细胞悬液,下室加入650 μL完全培养基,37 ℃培养24 h。取出小室,甲醛溶液固定,苏木素染色。PBS清洗后,用棉签擦干净小室,中性树脂封片。干燥后,显微镜10倍物镜下观察拍照计数。
1.2.8免疫荧光实验:无菌条件下,将TC处理细胞爬片置于12孔板内,并将细胞接种至孔内,24 h内进行细胞转染。48 h后取出玻片,甲醇溶液固定,丙酮溶液再固定。PBS清洗后,1%BSA封闭液室温封闭2 h。一抗孵育,4 ℃过夜。PBS清洗玻片,孵二抗,避光37 ℃ 50 min。DAPI复染,PBS避光清洗后进行封片,荧光显微镜观察拍照(400倍),统计中心体异常扩增比例。
1.2.9细胞凋亡实验:接种细胞于6孔板中并进行转染,48 h后胰酶消化细胞。1 000 r/min离心10 min,收集细胞。加入1 mL PBS溶液,轻震悬浮细胞,1 000 r/min离心10 min,弃上清。加入500 μL结合缓冲液重悬细胞,加入5 μL FITC和5 μL PI,轻轻混匀,室温避光反应20 min,1 h内进行流式细胞仪检测。
1.3 统计学分析采用SPSS 16.0统计软件进行统计分析,应用独立样本t检验进行统计学分析,每组实验数据均以x±s表示,P < 0.05为差异有统计学意义。
2 结果 2.1 miR-200c与喉癌发生和发展的关系以U6为对照,应用实时PCR检测了43例喉癌组织和癌旁正常组织中miR-200c的表达水平。其中,32例(74%)癌组织中miR-200c的表达量与癌旁组织相比升高(图 1)。统计分析结果显示,喉癌组织中miR-200c的平均表达量(1.86±0.33)高于相应的癌旁组织(0.92±0.46),差异有统计学意义(P < 0.05)。此外,在伴有淋巴结转移的喉癌组织中miR-200c的平均表达量(0.16±0.06)显著低于对照组(0.92±0.46),差异有统计学意义(P < 0.05)。
|
| 图 1 miR-200c在喉癌组织中的表达水平 Fig.1 Expression level of miR-200c in laryngeal carcinoma tissues |
2.2 miR-200c抑制喉癌Hep-2细胞迁移、减弱中心体异常扩增
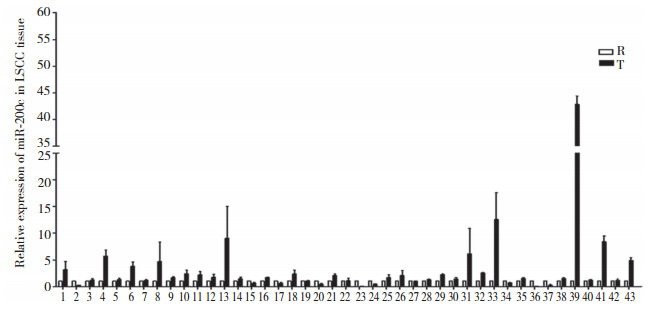
在Hep-2细胞中分别转染miR-200c模拟物、模拟物对照、miR-200c抑制物、抑制物对照,并设空白对照组。应用实时PCR检测转染效率,模拟物转染组中miR-200c的表达水平(0.61±0.02)与对照组(0.40±0.06)相比升高,抑制物转染组中miR-200c的表达水平(0.37±0.02)与对照组(0.40±0.01)相比降低,差异有统计学意义(P < 0.05),提示转染成功。CCK8检测结果显示,转染miR-200c相关小RNAs后,Hep-2细胞的生长速率无明显差异(图 2A,P > 0.05)。Transwell小室实验结果显示,miR-200c模拟物转染组穿过小室的Hep-2细胞数(56.33±16.65)与对照组(118±19.31)相比显著减少,而miR-200c抑制物转染组(321.67±32.72)与对照组(109±15.10)相比增多(图 2B,P < 0.05)。免疫荧光实验结果显示,与对照组(9.34%±0.72%)相比,miR-200c模拟物转染组中心体异常扩增的细胞比例(7.15%±0.35%)显著降低,而miR-200c抑制物转染组(11.56%±1.29%)与对照组(9.02%±0.86%)相比显著增加(图 2C,P < 0.05)。细胞凋亡实验结果表明,转染miR-200c相关小RNAs后,Hep-2细胞的早期凋亡率无显著变化(图 2D,P > 0.05)。
|
| A, proliferation of Hep-2 cells detected by CCK8 assay; B, migration of Hep-2 cells detected by Transwell assay (×100);C, abnormal centrosome amplification of Hep-2 cells detected by immunofluorescence assay (×400);D, early apoptosis of Hep-2 cells. 图 2 miR-200c对喉癌Hep-2细胞生物学行为的影响 Fig.2 Effect of miR-200c on the biological behavior of Hep-2 cells |
2.3 miR-200c靶基因的预测和鉴定
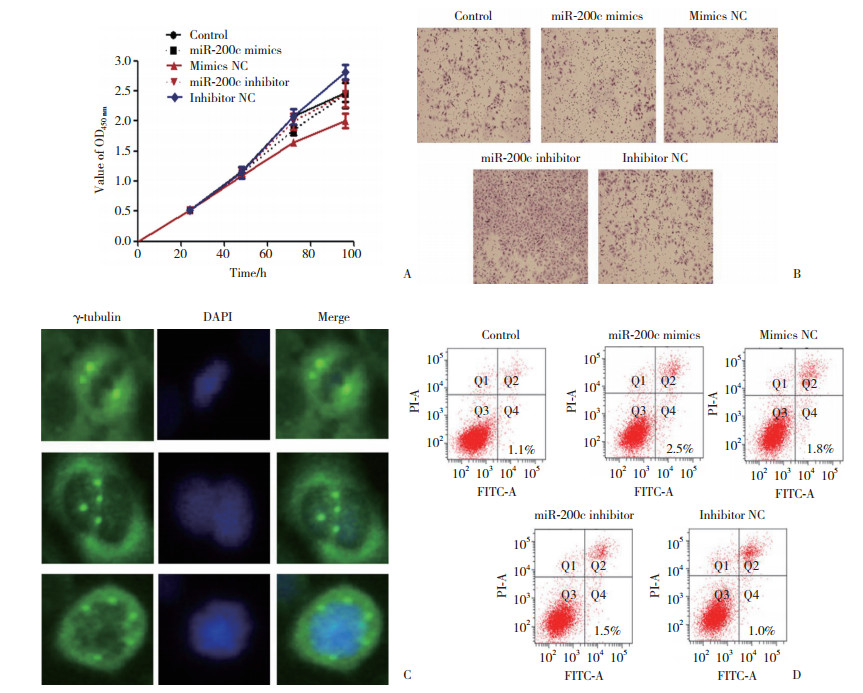
应用TargetScan等软件预测,发现PIN1 mRNA 3’非编码区(3’UTR)111-117位点为miR-200c的结合部位(图 3A)。构建野生型和突变型pGL3-PIN1-3’UTR报告基因载体,双荧光素酶报告基因实验结果显示,实验组荧光素酶活性(0.05±0.02)与对照组(0.10±0.01)相比显著降低,差异有统计学意义(P < 0.05)。同时,实时PCR和Western blotting结果显示,miR-200c模拟物组中PIN1蛋白表达水平(0.52±0.16)与对照组(0.83±0.06)相比降低,抑制物转染组中PIN1蛋白表达水平(1.62±0.16)与对照组(1.08±0.24)相比升高(图 3B,P < 0.05),而相应的mRNA表达水平无显著差异(P > 0.05)。

|
| A, complementary sequences of miR-200c and PIN1 3'UTR; B, protein expression of PIN1 in Hep-2 cells. 1, miR-200c mimics; 2, mimics NC; 3, miR-200c inhibitor; 4, inhibitor NC. 图 3 miR-200c靶基因的预测和鉴定 Fig.3 Prediction and identification of miR-200c target gene |
2.4 miR-200c通过调控PIN1抑制喉癌Hep-2细胞的迁移和中心体异常扩增
在Hep-2细胞中共转染PIN1和miR-200c模拟物,同时分别转染PIN1、miR-200c模拟物和模拟物对照,并设空白对照组。实时PCR结果显示,与对照组(0.41±0.04)相比,模拟物转染组中miR-200c的表达水平(0.63±0.06)升高(P < 0.05),PIN1转染组中PIN1 mRNA表达水平(0.62±0.08)与对照组(0.47±0.03)相比升高(P < 0.05),提示转染成功。Western blotting结果显示,共转染组中PIN1的蛋白表达水平(1.31±0.24)与PIN1组(2.11±0.42)相比降低(图 4A,P < 0.05)。CCK8检测结果显示,Hep-2细胞的生长速率在各组中无明显差异(图 4B,P > 0.05)。Transwell小室实验结果显示,共转染组中穿过小室的Hep-2细胞数(399±43.86)与模拟物组(72±19.31)相比显著增多,与PIN1组(523±61.80)相比显著减少(图 4C,P < 0.05)。
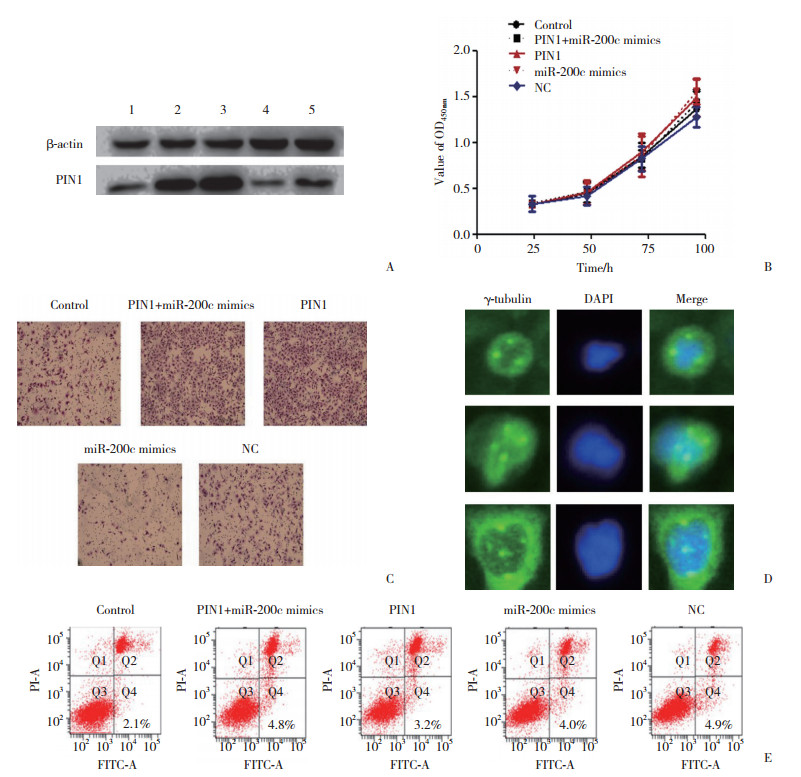
|
| A, PIN1 protein expression level detected by western blotting; B, proliferation of Hep-2 cells detected by CCK8 assay; C, migration of Hep-2 cells detected by Transwell assay (×100);D, abnormal centrosome amplification of Hep-2 cells detected by immunofluorescence assay (×400);E, early apoptosis of Hep-2 cells. 1, Control; 2, PIN1+miR-200c mimics; 3, PIN1;4, miR-200c mimics; 5, NC. 图 4 PIN1对Hep-2细胞增殖、迁移、中心体扩增和凋亡的影响 Fig.4 Effect of PIN1 on proliferation, migration, centrosome, and apoptosis in Hep-2 cells |
免疫荧光实验结果显示,共转染组与模拟物组(7.34%±0.27%)相比,中心体异常扩增的细胞比例(9.95%±0.74%)升高,与PIN1组(12.93%±1.37%)相比显著降低,差异有统计学意义(图 4D,P < 0.05)。细胞凋亡实验结果表明,Hep-2细胞的早期凋亡率在各组中无显著变化(图 4E,P > 0.05)。
3 讨论miRNAs作为细胞信号通路的一个重要组成部分,越来越多的证据表明,miRNAs是许多生命活动的主要调控因子,如细胞增殖、分化、凋亡、应激反应和血管生成,它们通过结合多个靶基因的3’UTR区发挥作用[4]。研究[5-6]发现,miR-200家族在不同肿瘤中的表达水平不同,例如,miR-200家族在结直肠癌中表达下调,在卵巢癌中高表达。
miR-200c最先发现在多种癌细胞系中表达失调[7]。本研究发现miR-200c在喉癌组织中高表达,提示其参与喉癌的发生。有研究[8, 10]显示,miR-200c还参与胰腺癌、鼻咽癌和前列腺癌的发生。此外,miR-200c在伴有淋巴结转移患者的喉癌组织中低表达,提示miR-200c在肿瘤转移过程中发挥作用。Transwell小室实验结果表明,miR-200c能够抑制喉癌Hep-2细胞迁移的能力。有研究[11]发现,结肠癌中,miR-200c过表达后能够减弱细胞的侵袭和转移能力。
研究[12-13]表明,miR-200c在肿瘤中是通过其下游靶基因介导发挥作用的,已被证实的miR-200c靶基因有TKS5、JUN等。本研究中,通过TargetScan等生物信息学软件预测发现PIN1 mRNA 3’UTR存在miR-200c结合位点,荧光素酶报告基因检测结果证实PIN1是miR-200c的靶基因之一。本研究还发现,Hep-2细胞中miR-200c能够在翻译水平抑制PIN1的表达。
在多数人类癌症中,PIN1呈现出表达上调的现象[14, 16]。本课题组前期研究发现PIN1在喉癌中表达上调,下调PIN1的表达抑制了Hep-2细胞的迁移能力,减弱了细胞中心体的异常扩增。有研究[17]发现,乳腺癌中PIN1能够通过Ras信号通路增强c-Jun的转录活性,增强细胞的迁移能力。研究[18]显示,中心体在微管组织、信号传导、极性和细胞分裂中发挥关键作用。目前,中心体失调的时机、机制和影响尚不清楚[19]。有研究[20]报道,PIN1在中心体复制过程中扮演重要角色,PIN1过表达能够诱导中心体扩增、染色体不稳定性和肿瘤发生。
本研究中,发现miR-200c能够通过调控PIN1抑制Hep-2细胞的迁移能力,并降低细胞的中心体异常扩增比例。有研究[21]显示,过表达miR-200c能够通过下调E2F3的表达抑制膀胱癌细胞的增殖能力。miR-200c能够通过下调EDNRA的表达调控胃癌细胞的凋亡过程[22]。本研究结果显示,miR-200c对喉癌Hep-2细胞的增殖和凋亡过程无显著影响,推测此差异可能存在组织特异性。
综上所述,本研究首次发现miR-200c参与喉癌的发生,证实PIN1是miR-200c的靶基因之一,发现miR-200c能够通过调控PIN1抑制喉癌Hep-2细胞的迁移能力并减弱中心体异常扩增。此研究成果为进一步探讨miR-200c作为喉癌诊治的新靶标奠定了重要的科学基础。
| [1] |
JEMAL A, SIEGEL R, XU J, et al. Cancer statistics, 2010[J]. CA Cancer J Clin, 2010, 60(5): 277-300. DOI:10.3322/caac.20073 |
| [2] |
YANG CW, WANG SF, YANG XL, et al. Identification of gene expression models for laryngeal squamous cell carcinoma using co-expression network analysis[J]. Medicine (Baltimore), 2018, 97(7): e9738. DOI:10.1097/MD.0000000000009738 |
| [3] |
YEH ES, MEANS AR. PIN1, the cell cycle and cancer[J]. Nat Rev Cancer, 2007, 7(5): 381-388. DOI:10.1038/nrc2107 |
| [4] |
JI W, SUN B, SU C. Targeting microRNAs in cancer gene therapy[J]. Genes (Basel), 2017, 8(1): E21. DOI:10.3390/genes8010021 |
| [5] |
PATERSON E L, KAZENWADEL J, BERT AG, et al. Down-regulation of the miRNA-200 family at the invasive front of colorectal cancers with degraded basement membrane indicates EMT is involved in cancer progression[J]. Neoplasia, 2013, 15(2): 180-191. DOI:10.1593/neo.121828 |
| [6] |
NAM EJ, YOON H, KIM SW, et al. MicroRNA expression profiles in serous ovarian carcinoma[J]. Clin Cancer Res, 2008, 14(9): 2690-2695. DOI:10.1158/1078-0432.CCR-07-1731 |
| [7] |
HURTEAU GJ, SPIVACK SD, Brock GJ. Potential mRNA degradation targets of hsa-miR-200c, identified using informatics and qRT-PCR[J]. Cell Cycle, 2006, 5(17): 1951-1956. DOI:10.4161/cc.5.17.3133 |
| [8] |
LI L, LI B, CHEN D, et al. miR-139 and miR-200c regulate pancreatic cancer endothelial cell migration and angiogenesis[J]. Oncol Rep, 2015, 34(1): 51-58. DOI:10.3892/or.2015.3945 |
| [9] |
CHEN P, GUO X, ZHANG L, et al. MiR-200c is a cMyc-activated miRNA that promotes nasopharyngeal carcinoma by downregulating PTEN[J]. Oncotarget, 2017, 8(3): 5206-5218. DOI:10.18632/oncotarget.14123 |
| [10] |
TAO T, LIU D, LIU C, et al. Autoregulatory feedback loop of EZH2/miR-200c/E2F3 as a driving force for prostate cancer development[J]. Biochim Biophys Acta, 2014, 1839(9): 858-865. DOI:10.1016/j.bbagrm.2014.07.001 |
| [11] |
PAN Q, MENG L, YE J, et al. Transcriptional repression of miR-200 family members by Nanog in colon cancer cells induces epithelial-mesenchymal transition (EMT)[J]. Cancer Lett, 2017, 392: 26-38. DOI:10.1016/j.canlet.2017.01.039 |
| [12] |
SUNDARARAJAN V, GENGENBACHER N, STEMMLER MP, et al. The ZEB1/miR-200c feedback loop regulates invasion via actin interacting proteins MYLK and TKS5[J]. Oncotarget, 2015, 6(29): 27083-27096. DOI:10.18632/oncotarget.4807 |
| [13] |
GUO J, FANG W, SUN L, et al. Reduced miR-200b and miR-200c expression contributes to abnormal hepatic lipid accumulation by stimulating JUN expression and activating the transcription of srebp1[J]. Oncotarget, 2016, 7(24): 36207-36219. DOI:10.18632/oncotarget.9183 |
| [14] |
SHINODA K, KUBOKI S, SHIMIZU H, et al. Pin1 facilitates NF-kappaB activation and promotes tumour progression in human hepatocellular carcinoma[J]. Br J Cancer, 2015, 113(9): 1323-1331. DOI:10.1038/bjc.2015.272 |
| [15] |
SHI M, CHEN L, JI J, et al. Pin1 is overexpressed and correlates with poor prognosis in gastric cancer[J]. Cell Biochem Biophys, 2015, 71(2): 857-864. DOI:10.1007/s12013-014-0274-0 |
| [16] |
XU C, KIM NG, GUMBINER BM. Regulation of protein stability by GSK3 mediated phosphorylation[J]. Cell Cycle, 2009, 8(24): 4032-4039. DOI:10.4161/cc.8.24.10111 |
| [17] |
WULF GM, RYO A, WULF GG, et al. Pin1 is overexpressed in breast cancer and cooperates with Ras signaling in increasing the transcriptional activity of c-Jun towards cyclin D1[J]. EMBO J, 2001, 20(13): 3459-3472. DOI:10.1093/emboj/20.13.3459 |
| [18] |
BORNENS M. The centrosome in cells and organisms[J]. Science, 2012, 335(6067): 422-426. DOI:10.1126/science.1209037 |
| [19] |
GODINHO SA, PELLMAN D. Causes and consequences of centrosome abnormalities in cancer[J]. Philos Trans R Soc Lond B Biol Sci, 2014, 369(1650): 20130467. DOI:10.1098/rstb.2013.0467 |
| [20] |
SUIZU F, RYO A, WULF G, et al. Pin1 regulates centrosome duplication, and its overexpression induces centrosome amplification, chromosome instability, and oncogenesis[J]. Mol Cell Biol, 2006, 26(4): 1463-1479. DOI:10.1128/MCB.26.4.1463-1479.2006 |
| [21] |
LIU L, QIU M, TAN G, et al. miR-200c inhibits invasion, migration and proliferation of bladder cancer cells through down-regulation of BMI-1 and E2F3[J]. J Transl Med, 2014, 12: 305. DOI:10.1186/s12967-014-0305-z |
| [22] |
WEI W, SHI L, CHEN W, et al. miR-200c regulates the proliferation, apoptosis and invasion of gastric carcinoma cells through the downregulation of EDNRA expression[J]. Int J Mol Med, 2018, 41(3): 1619-1626. DOI:10.3892/ijmm.2017.3317 |
 2019, Vol. 48
2019, Vol. 48