文章信息
- 孙溢华, 邢佳鑫, 宣金锋, 姚军, 夏皙, 王保捷
- SUN Yihua, XING Jiaxin, XUAN Jinfeng, YAO Jun, XIA Xi, WANG Baojie
- X-STR侧翼序列SNPs在特殊亲缘关系及混合样品鉴定中的意义
- Significance of Using SNPs Located in the X-STR Flanking Region for the Identification of Kinships and Mixed Samples
- 中国医科大学学报, 2018, 47(12): 1072-1076
- Journal of China Medical University, 2018, 47(12): 1072-1076
-
文章历史
- 收稿日期:2018-03-08
- 网络出版时间:2018-11-28 16:04
2. 苏州市公安局吴中分局, 江苏 苏州 215104
2. Wuzhong Public Security Sub-Bureau, Suzhou Public Security Bureau, Suzhou 215104, China
目前短串联重复序列(short tandem repeats,STR)是应用于法医DNA分型的主要遗传标记[1],其中常染色体STR遗传标记和Y染色体STR遗传标记应用较为成熟,广泛用于个体识别与亲缘关系鉴定中,已经有多种商品化试剂盒。而X染色体STR(X-STR)遗传标记在法医学领域的研究与应用相对滞后[2]。
X-STR遗传标记具有独特的遗传方式,在某些特殊的亲缘关系鉴定中可发挥独特的应用价值[3-5]。人类女性有2条X染色体,而男性只有1条,X染色体遗传标记的遗传特征既不同于常染色体,也不同于Y染色体,母亲的2条X染色体上的等位基因可随机传递给子女;父亲则只能将其X染色体的基因传递给女儿,其传递的X染色体必定来源于女儿的祖母[6]。
在某些特殊的亲缘关系(父女关系、母女或母子关系、祖母与孙女关系、同父的姐妹关系等)鉴定时,当检测结果≥50%的X-STR型别一致时,结论是不能否定也不能肯定具有上述亲缘关系。超过50%的X-STR型别一致时,可能是源于亲缘的遗传,也可能是随机的一致,如能证明是亲缘的遗传,则增加了亲缘关系指数;如能证明是随机的一致,则排除了亲缘关系[7]。X-STR侧翼序列单核苷酸多态性(single nucleotide polymorphism,SNP)的型别可能在证明是亲缘遗传或是随机一致的较好指标。
2人混合样品的个人识别鉴定一直是法医物证检验的难题[8]。对混合样品STR检测谱型的分析在不能排除嫌疑人时也很难确定是嫌疑人。最好的办法是在混合样品中能够分别检测不同嫌疑人的遗传标记型别[9]。嫌疑人如STR侧翼序列的SNP型别不同,通过序列特异性引物扩增的方法可能实现分别检测不同嫌疑人的STR型别,从而达到个人识别目的。
本研究拟以X染色体DXS7423基因座侧翼序列SNPs(rs762822、rs546652、rs762822和rs546652)为指标,探讨其用于某些特殊亲缘关系及2人混合样品的个人识别鉴定的可能性。
1 材料与方法 1.1 材料1组三代四口家系(祖母、父亲、母亲、孙女)和1组三代五口家系(祖母、父亲、母亲、两个异卵双生孙女)血液样本;2个DXS7423基因座及侧翼序列rs17279108分型不同的男性个体血液样本。所有样品提供者均已签署知情同意书。蛋白酶K(德国Merck公司)、Chelex 100(美国Bio-Rad公司)、琼脂糖(西班牙Biowest公司)、Investigator Argus X-12试剂盒(德国Qiagen公司)。NanoDrop 2000超微量分光光度计、ABI-9700型PCR扩增仪和ABI-3500遗传分析仪均购自美国Thermo公司。
1.2 方法 1.2.1 DNA提取及定量采用chelex-100法提取DNA[10],经NanoDrop 2000超微量分光光度计检测后稀释至适合浓度。
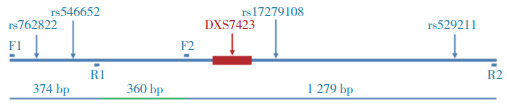
1.2.2 SNP位点的筛选及引物设计选取DXS7423基因座附近的rs762822、rs546652、rs17279108和rs529211 4个频率较好的SNPs位点。采用Primer Premier 5.0软件设计引物,并由宝生物工程(大连)有限公司合成。见表 1、图 1。
|
| 图 1 含DXS7423基因座上下游的扩增片段及SNPs位置示意图 Fig.1 Locations of the DXS7423 locus, primers, and SNPs |
| Primer | Sequence(5’-3’) | Temperature(℃) | GC(%) | Product length(bp) |
| DXS7423F1 | AGAAGCCACTAGCCTTTTTAGC | 58.4 | 45 | 374 |
| DXS7423R1 | GAACACTTCCCTCTACCATAAACC | 59.3 | 46 | |
| DXS7423F2 | TCTCCTGACACTGAGAGATCATAGC | 61.3 | 48 | 1 279 |
| DXS7423R2 | GACATGGGGTGTGGATGAGG | 63.6 | 60 |
1.2.3 PCR扩增
上游片段PCR扩增体系20 μL,含rTaqDNA聚合酶(5 U)2 μL;10×PCR Buffer(含Mg2+)2 μL;dNTP 2 μL;引物各1.5 μL;灭菌去离子水9 μL;DNA模板2 μL。94 ℃ 5 min;94 ℃ 30 s,58 ℃ 30 s,72 ℃ 30 s,30个循环;72 ℃ 10 min。下游片段PCR扩增体系20 μL,含LA TaqDNA聚合酶(5 U)2 μL;10×LA PCR Buffer(含Mg2+)2 μL;dNTP 2 μL;引物各1.5 μL;灭菌去离子水9 μL;DNA模板2 μL。94 ℃ 5 min;94 ℃ 1 min,68 ℃ 1 min,72 ℃ 20 s,30个循环;72 ℃ 10 min。Investigator Argus X-12试剂盒的反应体系及反应条件参照使用手册。
1.2.4 扩增产物检测DXS7423基因座上下游扩增产物委托宝生物工程(大连)有限公司进行测序。Investigator Argus X-12试剂盒的复合扩增产物使用ABI-3500遗传分析仪进行检测。
1.2.5 序列特异性引物(sequence specific primer,SSP)PCR根据rs17279108(A/T)SNP位点分别设计序列特异性引物,并设计1条公共引物。公共引物为5’-TCTCCTGACACTGAGAGATCATAGC-3’;序列特异性引物(A)为5’-CTCCAGAATGCGAGCCCTCT-3’,产物长度286 bp;序列特异性引物(T)为5’-CCAGAATGCGAGCCCTCA-3’,产物长度284 bp。选取2个男性样品,二者的rs17279108位点和DXS7423位点分型结果分别为A/15和T/14,及二者DNA不同比例的混合液(前者为高组分,后者为低组分)。
Taq HS Perfect Mix 2×10 μL;公共引物3 μL;序列特异性引物(A或T)3 μL;灭菌去离子水1.3 μL;DMSO1.7 μL;DNA模板1 μL。94 ℃ 3 min;94 ℃ 5 s,64 ℃ 1 s,68 ℃ 10 s,30个循环;68 ℃ 10 min。
1.2.6 不同比例混合样品的检测将2例男性样品DNA按照1:5、1:10、1:20、1:50和1:100进行混合。用Investigator Argus X-12试剂盒对混合样品进行扩增,不同比例混合的样品进行扩增时,所加入扩增体系中的DNA模板总量均为10 ng,并采用毛细管电泳对扩增产物进行检测分型。应用PCR-SSP方法对混合样品进行扩增,扩增产物用琼脂糖凝胶检测。
2 结果 2.1 家系样品检测结果通过对2个家系的检测,获得了9个样品的分型结果,见表 2。
| Item | Family 1 | Family 2 | ||||||||
| Paternal grandmother | Father | Mother | Granddaughter | Paternal grandmother | Father | Mother | Granddaughter 1 | Granddaughter 2 | ||
| rs762822 | A/G | A | A/A | A/A | A/A | A | A/G | A/A | A/G | |
| rs546652 | G/T | G | G/G | G/G | G/G | G | G/T | G/G | G/T | |
| rs17279108 | A/T | A | A/A | A/A | A/A | A | A/A | A/A | A/A | |
| rs529211 | C/G | C | C/C | C/C | C/C | C | C/G | C/C | C/G | |
| DXS7423 | 15/15 | 15 | 15/15 | 15/15 | 14/15 | 15 | 14/15 | 14/15 | 15/15 | |
2.2 PCR-SSP扩增产物电泳检测结果
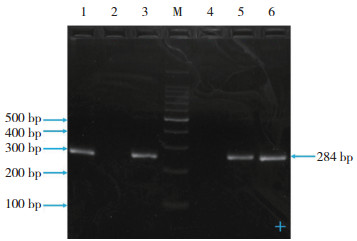
PCR-SSP扩增产物采用2%琼脂糖凝胶电泳进行检测,见图 2。
|
| 1,A product of male sample 1;2,T product of male sample 1;3,A product of male sample 2;4,T product of male sample 2;5,A product of mixed sample;6,T product of mixed sample;M,100 bp DNA ladder. 图 2 PCR-SSP扩增产物琼脂糖凝胶电泳图谱 Fig.2 Agarose gel electrophoresis of PCR-SSP products |
2.3 PCR-SSP扩增产物的DXS7423位点测序分型结果
测序结果如表 3所示。根据样品的测序结果,参考国际法医遗传学会(international society of forensic genetics,ISFG)命名规范[11],将2个样品分别命名为“15”和“14”,这与该样品经Investigator Argus X-12试剂盒检测分型结果相一致。
| Item | Sequencing data | Allele |
| A PCR product of male sample 1 | Pr1-Nn-(TCCA)3-tctgtcct-(TCCA)12-Nn-Pr | 15 |
| T PCR product of male sample 2 | Pr2-Nn-(TCCA)3-tctgtcct-(TCCA)11-Nn-Pr | 14 |
2.4 不同比例混合样品检测结果
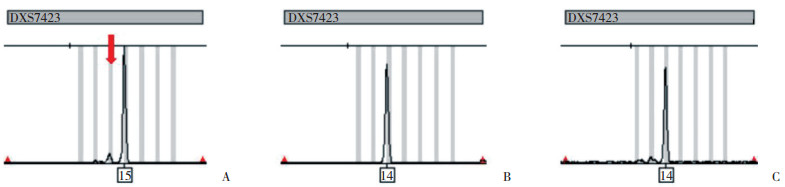
采用Investigator Argus X-12试剂盒对不同混合比例的样品进行检测后发现,在1:20时,低组分隐约可见;在1:50的时候低组分在图谱中已无法显示。在采用PCR-SSP方法对不同混合比例的样品进行检测后发现,在1:50,甚至1:100的比例时,低组分样品对应的扩增体系依然有扩增产物出现,见图 3。
|
| A,the genotype of 1:20 mixed sample directly;B,the genotype of 1:50 mixed sample after PCR-SSP;C,the genotype of 1:100 mixed sample after PCR-SSP. 图 3 不同混合比例样品毛细管电泳图谱 Fig.3 DXS7423 genotype of mixed samples in different proportions |
3 讨论
在遗传过程中,父亲全部X染色体都传递给女儿;母亲随机将其中的1条X染色体传递给女儿或儿子;祖母随机将1条X染色体传递给了儿子,儿子又将其全部传递给了孙女;同父的姐妹将具有来自父亲的相同的X染色体,X-STR侧翼序列SNPs不受染色体交叉的影响[12]。
当2个个体在某一基因座表现出相同的等位基因,那么这种一致性产生的原因可能是偶然或者遗传导致的。如果该基因座侧翼序列的SNPs表现出不一致的分型结果,则说明该相同等位基因的出现是随机的,在进行亲缘关系计算时应排除掉该位点。如果侧翼序列的SNPs分型结果一致,则支持了相同等位基因是由遗传获得的这种可能性。如果采用高通量测序等新一代的测序技术,可以同时检测多个X-STR侧翼序列内的大量SNPs位点[13-14],测序结果有助于特殊亲缘关系推断。DXS7423基因座侧翼序列的4个SNPs有超过50%的可能区分2个无关个体。如果将检测的侧翼序列的范围扩大,会有新SNPs位点被检出,可进一步提高二者区分概率。
本研究应用PCR-SSP方法,制备已知X-STR型别的2个个体混合样品。在检测样品DXS7423基因座侧翼序列SNPs的基础上,根据SNPs型别差异,设计序列特异性引物对含有DXS7423基因座的片段分别进行扩增,对扩增片段进行X-STR基因座分型检测,分别成功地获得了2个个体的DXS7423基因座型别。根据本实验结果,如果能够筛选足够多的侧翼序列的SNPs有差别的X-STR基因座位进行检测,可能解决2个混合斑的个人识别问题。
在筛选SNPs的过程中,应注意SNPs位置,选用的SNPs所在位置应在X-STR商品化试剂盒中的引物结合区外侧,以避免在进行X-STR分型时可能出现基因座丢失的问题;由于不同案件中的嫌疑人差别,不可能使用固定的复合扩增体系进行不同案件的检测,因此需要根据具体案情采用不同的条件构建复合扩增体系;应用新一代测序技术,可同时对较多STR侧翼序列的SNPs进行检测,可达到筛选足够的待测STR基因座目的。但要注意此方法中的错误率,必要时可以多次检测或者辅以其他方法进行检测,从而完成SNPs筛选。
| [1] |
WEISSENBACH J, GYAPAY G, DIB C, et al. A second-generation linkage map of the human genome[J]. Nature, 1992, 359(6398): 794-801. DOI:10.1038/359794a0 |
| [2] |
SZIBOR R. X-chromosomal markers:past, present and future[J]. Forensic Sci Int Genet, 2007, 1(2): 93-99. DOI:10.1016/j.fsigen.2007.03.003 |
| [3] |
FREITAS NS, RESQUE RL, RIBEIRO-RODRIGUES EM, et al. X-linked insertion/deletion polymorphisms:forensic applications of a 33-markers panel[J]. Int J Legal Med, 2010, 124(6): 589-593. DOI:10.1007/s00414-010-0441-9 |
| [4] |
CHEN DP, TSENG CP, TSAI SH, et al. Use of X-linked short tandem repeats loci to confirm mutations in parentage caseworks[J]. Clin Chim Acta, 2009, 408(1/2): 29-33. DOI:10.1016/j.cca.2009.06.042 |
| [5] |
MARISCAL RAMOS C, MARTINEZ-CORTES G, RAMOS-GONZALEZ B, et al. Forensic parameters of the X-STR decaplex system in Mexican populations[J]. Leg Med (Tokyo), 2017, 31(2): 38-41. DOI:10.1016/j.legalmed.2017.12.013 |
| [6] |
ZARRABEITIA MT, MIJARES V, RIANCHO JA. Forensic efficiency of microsatellites and single nucleotide polymorphisms on the X chromosome[J]. Int J Legal Med, 2007, 121(6): 433-437. DOI:10.1007/s00414-007-0169-3 |
| [7] |
NOTHNAGEL M, SZIBOR R, VOLLRATH O, et al. Collaborative genetic mapping of 12 forensic short tandem repeat (STR) loci on the human X chromosome[J]. Forensic Sci Int Genet, 2012, 6(6): 778-784. DOI:10.1016/j.fsigen.2012.02.015 |
| [8] |
BUDOWLE B, ONORATO AJ, CALLAGHAN TF, et al. Mixture interpretation:defining the relevant features for guidelines for the assessment of mixed DNA profiles in forensic casework[J]. J Forensic Sci, 2009, 54(4): 810-821. DOI:10.1111/j.1556-4029.2009.01046.x |
| [9] |
HU N, CONG B, LI S, et al. Current developments in forensic interpretation of mixed DNA samples (Review)[J]. Biomed Rep, 2014, 2(3): 309-316. DOI:10.3892/br.2014.232 |
| [10] |
WALSH PS, METZGER DA, HIGUCHI R. Chelex 100 as a medium for simple extraction of DNA for PCR-based typing from forensic material[J]. Biotechniques, 1991, 10(4): 506-513. |
| [11] |
BAR W, BRINKMANN B, BUDOWLE B, et al. DNA recommendations. Further report of the DNA commission of the ISFH regarding the use of short tandem repeat systems. International society for forensic haemogenetics[J]. Int J Legal Med, 1997, 110(4): 175-176. DOI:10.1007/s004140050061 |
| [12] |
TOMAS C, SANCHEZ JJ, CASTRO JA, et al. Forensic usefulness of a 25 X-chromosome single-nucleotide polymorphism marker set[J]. Transfusion, 2010, 50(10): 2258-2265. DOI:10.1111/j.1537-2995.2010.02696.x |
| [13] |
HENDRE PS, KAMALAKANNAN R, VARGHESE M. High-throughput and parallel SNP discovery in selected candidate genes in eucalyptus camaldulensis using illumina NGS platform[J]. Plant Biotechnol J, 2012, 10(6): 646-656. DOI:10.1111/j.1467-7652.2012.00699.x |
| [14] |
WU SH, RODRIGO AG. Estimation of evolutionary parameters using short, random and partial sequences from mixed samples of anonymous individuals[J]. BMC Bioinformatics, 2015, 16(2): 357. DOI:10.1186/s12859-015-0810-y |
 2018, Vol. 47
2018, Vol. 47