文章信息
- 罗枫雪, 李佛生, 姚民, 徐莺.
- LUO Feng-xue, LI Fo-sheng, YAO Min, XU Ying.
- 水稻OsHAK26启动子的克隆及瞬时表达分析
- The Cloning and Transient Expression Analysis of Promoter of OsHAK26 from Oryza sativa
- 中国生物工程杂志, 2017, 37(2): 33-39
- China Biotechnology, 2017, 37(2): 33-39
- http://dx.doi.org/DOI:10.13523/j.cb.20170206
-
文章历史
- 收稿日期: 2016-08-19
- 修回日期: 2016-09-10
钾离子是主要的营养元素和含量最丰富的阳离子,在植物的许多基础生命活动,例如酶活性的激活、跨膜运输、离子中和反应以及渗透压的调节等过程中起着重要的作用[1]。土壤中钾离子的浓度很低,只有植物细胞的千分之一到十分之一[2-3],因此,根细胞是逆浓度梯度从土壤中吸收钾离子。这个过程是由两种机制介导的,第一种机制是钾离子转运蛋白在微摩尔的钾离子浓度下进行的高亲和性有效吸收,第二种机制是钾离子通道在一毫摩尔及以上钾离子浓度时进行的低亲和性吸收。
植物体内钾离子转运蛋白由四个多基因家族KT/KUP/HAK, Trk/HKT, KEA和CHX转运蛋白组成,KT/KUP/HAK是其中最大的一个家族,水稻中KT/HAK/KUP家族由27个成员组成[4-5],根据系统进化关系可分为四大亚类[6-7]。大部分KT/KUP/HAK家族转运蛋白属于Ⅰ类和Ⅱ类。前者主要介导高亲和力钾离子摄入[8],后者则介导低亲和力的钾离子转运作用,是对钾离子通道作用的补充[9]。目前对于该家族Ⅲ类和Ⅳ类的功能研究较少[10]。通过进化树和PSORT蛋白质定位分析发现OsHAK26属于Ⅳ亚类,位于水稻基因组第8号染色体上,其编码的转运蛋白极有可能定位在植物细胞膜上[11]。
植物的生长发育和生命周期是基因在时间和空间上有序表达的结果,通过启动子调控元件和转录因子相互协调作用,能够调控基因在特定的时空或环境条件下的表达[12]。因此对启动子活性及启动子中顺式作用元件功能的鉴定是了解高等植物基因表达和调控机制的有效手段之一。本文主要在生物信息学分析的基础上,通过5′系列缺失法获得OsHAK26基因5′上游片段-1 473bp、-963bp、-441bp、-193bp四种不同的缺失突变,利用荧光素酶 (LUC) 基因作为报告基因,首次对OsHAK26启动子的结构和功能进行了初步的研究,以期为将来进一步解析OsHAK26基因结构和功能提供有价值的线索。
1 材料与方法 1.1 材料供试水稻 (Oryza sativa L.) 材料为普通籼稻桂朝二号,种植于四川大学江安校区实验基地,按常规方法进行田间管理。拟南芥 (Arabidopsis thaliana) 为哥伦比亚型 (Col-O),培养于实验室22℃光照培养箱, 16h光照,8h黑暗。大肠杆菌 (Escherichia coli) Top10和pBI221-35S-LUC质粒载体由本实验室保存。
1.2 试剂盒和药品pEASY-T1 Simple试剂盒购自北京全式金生物科技有限公司,植物总DNA提取试剂盒和DNA柱式胶回收试剂盒均购于天根生化科技 (北京) 有限公司,PCR试剂盒PrimeSTAR® Max DNA Polymerase、Taq DNA聚合酶、各种DNA限制性内切酶、氨苄青霉素均购自宝生物工程 (大连) 有限公司,质粒小量提取试剂盒Plasmid Mini Kit Ⅰ为美国Omega公司产品,质粒大量提取试剂盒购于威格拉斯生物技术 (北京) 有限公司,引物由成都擎科梓熙生物技术有限公司合成,拟南芥原生质体提取和培养过程中所用到各种溶液均参考Yoo等[13]的方法进行配制,其他化学药品均为国产或进口分析纯试剂。
1.3 水稻基因组DNA的提取以水稻叶片为材料,按照植物总DNA提取试剂盒中的说明书提取水稻基因组DNA,并以1.0%的琼脂糖凝胶电泳鉴定DNA。
1.4 水稻OsHAK26基因启动子及不同缺失片段的克隆登陆水稻基因组数据库 (http://rice.plantbiology.msu.edu/) 进行检索,获得OsHAK26基因翻译起始位点上游2 064bp的启动子序列,用引物设计软件Pmier 5设计5′端不同缺失的上游引物和3′端下游引物。以水稻叶片中提取的DNA为模板,通过上游特异引物OsHAK26PF、OsHAK26PF1、OsHAK26PF2、OsHAK26PF3、OsHAK26PF4和共同的下游引物OsHAK26PR进行PCR扩增,获得五种不同长度的启动子片段 (表 1)。50μl的PCR扩增体系为:DNA模板2 μl,2×Primer Star Mix 25μl,上游引物2μl (10μmol/L),下游引物2μl (10μmol/L),ddH2O 19 μl。扩增条件为:98℃预变性5 min;98℃变性10 s,61℃退火15 s,72℃延伸30s,共35个循环;72℃延伸5 min,4℃保存。对PCR产物用Taq酶进行加A尾。
| Primer | Sequence (5′~3′) | Fragment length (bp) |
| BamH Ⅰ-OsHAK26PR | cgGGATCCGGCTGCTGATCGGATCGACCAC | - |
| Pst Ⅰ-OsHAK26PF | aaCTGCAGCAATGGCAGATCACCCAATGCTGAAG | 2 064 |
| Pst Ⅰ-OsHAK26PF1 | aaCTGCAGTGGTTGAAGCAGGAGTTCTCGTCT | 1 568 |
| Pst Ⅰ-OsHAK26PF2 | aaCTGCAGGGAGGAGGCAATGATGAGATACTAC | 1 058 |
| Pst Ⅰ-OsHAK26PF3 | aaCTGCAGCGTGACCCTTGATTGTCAGTATCACC | 536 |
| Pst Ⅰ-OsHAK26PF4 | aaCTGCAGTGGGTCTAATAAGAAGATGACCAGTCAAG | 288 |
| Straight lines: restriction enzyme cutting site. Small letters: protective base | ||
将加过A尾的PCR产物经1.0%的琼脂糖凝胶电泳检测并切胶回收。将回收产物与pEASY-T1 Simple克隆载体连接,并用热激法转化DH5α感受态细胞,并涂布于Amp抗性的LB固体培养基 (100μg/ml),37℃过夜培养12h。挑取单菌落于含有Amp抗性的LB液体培养基中,37℃摇床震荡培养2h后菌液PCR验证。菌液PCR验证的阳性克隆菌株送成都擎科生物公司测序。选择测序正确的菌液扩大培养进行后续试验,同时用80%的甘油保菌。
1.5 启动子序列生物信息学分析通过在线软件PlantCAR (http://bioinformatic.psb.ugent.be/webtools/plantcare/html/)[14]、PLAC (http://www.dna.affrc.go.jp/PLACE/)[15]和SoftBerry (http://linux1.softberry.com/all.htm) 分析OsHAK26启动子所含有的顺式作用元件。
1.6 启动子不同缺失片段瞬时表达载体的构建从测序正确的阳性菌株中提取质粒,用PstⅠ和BamHⅠ双酶切回收目的片段。将目的片段分别与PstⅠ和BamHⅠ双酶切回收的PBI221载体大片段连接,转化大肠杆菌,菌液PCR及质粒双酶切鉴定筛选阳性克隆,获得重组载体OsHAK26P∷LUC、OsHAK26P1∷LUC、OsHAK26P2∷LUC、OsHAK26P3∷LUC和OsHAK26P4∷LUC。
1.7 报告基因检测和启动子不同缺失片段活性的确定从测序正确的阳性菌株中大量提取质粒,利用Yoo等[13]的方法制备与转化拟南芥叶肉细胞原生质体,避光过夜培养检测报告基因。LUC报告基因检测按照Promage的LUC检测试剂盒的protocol进行,然后用LmaxII384荧光读板机读取荧光强度。GUS报告基因通过与检测底物 (1mmol/L 4-MUG,10mmol/L pH 8.0 Tris-HCl,2mmol/L MgCl2) 反应,然后用SpectraMax M2酶标仪检测GUS酶的活性。以LUC报告基因活性和内参质粒中GUS报告基因活性的比值代表启动子不同缺失片段相对活性。不同缺失片段启动子的活性跟LUC报告基因活性成正相关。
2 结果与分析 2.1 OsHAK26基因启动子序列生物信息学分析本实验将水稻OsHAK26基因翻译起始位点上游2 000bp的序列作为研究对象。通过SoftBerry软件预测,位于翻译起始位点上游95bp处的腺嘌呤A可能为OsHAK26启动子的转录起始位点,因为其周围序列符合转录起始位点YR (C/T A/G) 规律。将该处的A标记为+1bp。通过PlantCAR和PLACE软件进一步分析上游片段可能含有的顺式作用元件 (表 2和图 1)。发现除了基本的核心启动子元件,包括TATA-Box和CAAT-Box元件,该段序列中可能的顺式元件可以分为四类包括组织特异性元件、非生物胁迫相关元件、激素响应元件和功能相关元件。
| 元件分类 | 元件名称 | 序列 | 功能 | 数量 |
| 组织特异性元件 | GTGANTG10 | GTGA | 花粉G10元件 | 14 |
| SITEIIATCYTC | TGGGCY (Y=C/T) | 花药和分生组织特异性表达元件 | 1 | |
| POLLEN1LELAT52 | AGAAA | 花粉特异表达元件 | 7 | |
| QELEMENTZMZM13 | AGGTCA | 单子叶植物花粉特异表达元件 | 1 | |
| Skn-1-motif | GTCAT | 胚乳特异表达元件 | 4 | |
| 激素响应元件 | P-box | CCTTTTG | 赤霉素响应元件 | 3 |
| TCA-element | GAGAAGAATA | 水杨酸响应元件 | 1 | |
| ERE | ATTTCAAA | 乙烯响应元件 | 1 | |
| TGACG-motif | TGACG | 甲基茉莉酸响应元件 | 2 | |
| ABRE | TACGTG | 脱落酸响应元件 | 1 | |
| 非生物胁迫相关元件 | MYC | CANNTG | 干旱和脱落酸应答元件 | 19 |
| MBS | CGGTCA | 干旱响应元件 | 1 | |
| CCAAT-box | CAACGG | 干旱响应元件 | 1 | |
| HSE | AAAAAATTTC | 高温响应元件 | 3 | |
| LTR | CCGAAA、CCGAC | 低温响应元件 | 2 | |
| GT1GMSCAM4 | GAAAAA | 盐胁迫响应元件 | 6 | |
| 功能相关元件 | TAAAGSTKST1 | TAAAG | 保卫细胞钾离子通道蛋白元件 | 3 |
| ABRERATCAL | GACGAGA | 钙离子响应元件 | 1 | |
| MYBGAHV | TAACAAA | 蔗糖合成抑制元件 | 1 | |
| BOXLCOREDCPAL | ACCATCC | 转录激活因子结合元件 | 1 | |
| Ⅰ-box | GGATAAGGTG | 光响应元件 | 2 |
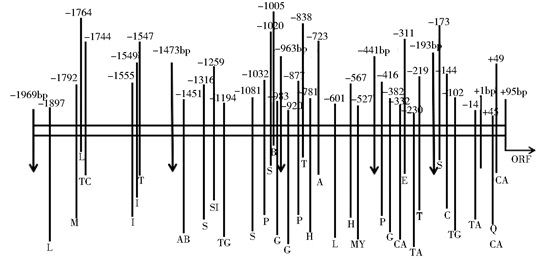
|
| 图 1 启动子中存在的不同顺式作用元件 Figure 1 Element-structure representation of the promoter Q: QELEMENTZMZM13;SI: SITEIIATCYTC; S: Skn-1-motif; P: P-box; TC: TCA-element; A: ABRE; E: ERE; TG: TGACG-motif; H: HSE; L: LTR; G: GT1GMSCAM4;M: MBS; C: CCAAT-box; T: TAAAGSTKST1;AB: ABRERATCAL; B: BOXLCOREDCPAL; MY: MYBGAHV; Ⅰ: Ⅰ-box; CA: CAATbox; TA: TATAbox; +1 represents the predicted transcription initiation site; MYC, GTGANTG10 and POLLEN1LELAT52 are abound in different deletion fragments and therefore not shown in the figure |
根据生物信息学分析的结果,设计了5对特异性引物,对该上游片段进行了不同程度的缺失,获得了全长启动子片段 (2 064bp) 和4种5′端缺失长度不同的片段 (1 568bp、1 058bp、536bp、288bp),由长至短分别命名为OsHAK26P、OsHAK26P1、OsHAK26P2、OsHAK26P3和OsHAK26P4。分别连接入pBI221-35S-LUC载体,替换该载体原有的35S启动子,成功地构建瞬时表达载体,并依次命名为OsHAK26P∷LUC、OsHAK26P1∷LUC、OsHAK26P2∷LUC、OsHAK26P3∷LUC、OsHAK26P4∷LUC (图 2)。
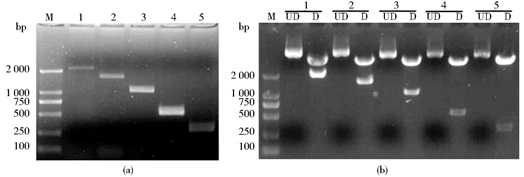
|
| 图 2 启动子不同缺失片段瞬时表达载体的构建 Figure 2 Construction of plant transient expression vectors of different deletion mutations products from promoter (a) PCR amplification of different deletion fragments of promoter (b)PstⅠ/ BamHⅠdouble digested products from transient expression vectors containing different deletion fragments; M:DNA Marker (DL2 000);UD:transient expression vectors; D:PstⅠ/ BamHⅠdouble digestion process; 1:OsHAK26P; 2:OsHAK26P1; 3:OsHAK26P2; 4:OsHAK26P3; 5:OsHAK26P4 |
将构建的瞬时表达载体采用PEG4000介导法转化拟南芥叶片原生质体,暗培养12h后,检测原生质体中LUC报告基因和GUS报告基因的活性,活性比值作为启动子不同缺失片段相对活性值。
由结果可知,OsHAK26全长启动子只有CaMV35S启动子活性的1/140,具有较弱的启动活性。启动子四个不同缺失片段和全长启动子相比的能力强弱关系和所占百分比为:OsHAK26P > OsHAK26P1(68.5%)≈ OsHAK26P3(67.3%) > OsHAK26P2(32.5%) > OsHAK26P4(21%)。表明四种缺失片段都具有一定的启动活性,随着长度减小,活性下降,但缺失-963bp~-441bp之间的片段却导致活性显著回升34.8%, 缺失-441bp~-193bp之间的片段导致活性大幅下降46.3%(图 3)。
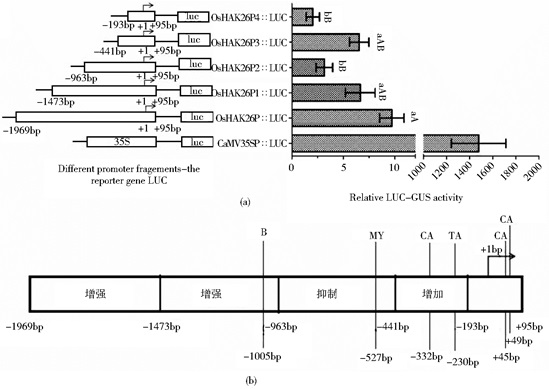
|
| 图 3 启动子及不同缺失片段的瞬时表达结果 Figure 3 Transient expression result of different deletion mutations products from promoter (a) Diagram of plant expression vectors containing the different promoter segments drived the report gene of LUC (left) and Relative LUC/GUS activity of different promoter segments (right) Bars represent mean±SE based on three replicates.Values with the same uppercase and lowercase letters within columns are not significantly different according to Duncan's multiple range tests at P < 0.05 and P < 0.01 respectively.Since CaMV35S promoter activity is much greater than the full length promoter of OsHAK26, significant difference analysis does not contain the data (b) Structure diagram of promoter of OsHAK26 CA: CAATbox; TA: TATAbox; MY: MYBGAHV; B: BOXLCOREDCPAL, The arrow represents the transcription initiation site and transcription direction |
启动子作为一种重要的调控元件,精确调控着植物体内基因的表达,影响基因功能的正确发挥。本文对水稻OsHAK26启动子序列的预测分析和瞬时表达分析为进一步研究OsHAK26基因的功能和调控机制提供了有价值的理论依据[16]。
本研究成功克隆出OsHAK26基因ATG上游2 064bp的启动子序列,发现除了基本的核心启动子元件,该段序列中可能的顺式元件可以分为以下四类。
组织特异性元件包括大量的花粉特异性元件GTGANTG10、POLLEN1LELAT52[17-18]和QELEMENTZMZM13,以及四个胚乳特异性元件Skn-1-motif。水稻基因组注释项目 (http://rice.plantbiology.msu.edu/) 和BAR (http://www.bar.utoronto.ca/) 软件分析结果均表明OsHAK26基因只在花粉中高表达 (GenBank登录号为LOC_Os08g39950.1),由此推测OsHAK26启动子的启动活性可能具有花粉偏好性,并且对水稻花粉正常发育起着重要作用。因此在拟南芥叶肉原生质体系统中,OsHAK26基因启动子启动活性可能受到抑制,而表现为弱启动子。有研究表明KT/HAK/KUP家族某些基因表达也具有花粉偏好性,OsHAK10和OsHAK15在水稻几乎所有组织中均有表达,但在开花前一天的雄蕊中表达量最高,推测其在水稻授粉和受精过程中起重要作用[19],OsHAK26可能具有类似的表达模式和功能。
非生物胁迫应答元件包括MYC[20]、MBS、HSE、LTR和GT1GMSCAM4等,其中MYC和GT1GMSCAM4数量较多,推测OsHAK26基因可能在水稻逆境胁迫响应中起作用,尤其响应盐胁迫和干旱胁迫。BAR (http://www.bar.utoronto.ca/) 软件分析结果也表明七天的水稻幼苗在冷胁迫和盐胁迫情况下OsHAK26基因表达量显著上调,而在干旱胁迫下表达量反而下调。
激素响应元件包括P-box、TCA-element、ERE、TGACG-motif和ABRE[21]等。研究表明27个OsHAK基因可以响应NAA, GA3和KT激素应答,但不同的基因表达量的变化有所不同[22],说明OsHAK26基因在激素响应过程中起作用。
功能相关元件包括TAAAGSTKST1、ABRERATCAL、MYBGAHV、BOXLCOREDCPAL和Ⅰ-box[23]。研究表明KT/HAK/KUP基因家族成员在2 000bp左右的上游启动子序列中含有1个钙离子响应元件、2个光响应元件和一个生物节律响应元件[22],OsHAK26基因启动子生物信息学分析结果与此相符,表明OsHAK26基因很可能在水稻体内钾离子转运过程中发挥作用。
研究表明许多核心启动子元件在水稻和拟南芥都具有功能保守性[24],水稻花粉特异启动子在拟南芥中呈现相似的表达模式[25]。因此本文选择拟南芥叶肉原生质体瞬时表达系统研究OsHAK26基因启动子不同缺失片段的启动活性。本研究表明启动子不同的缺失片段均存在多种胁迫元件,另外-441bp~-193bp区段发现了一个CAAT盒和一个TATA盒,瞬时表达结果表明该区段对于启动活性最为重要,此区段缺失后,启动活性骤降46.3%,因此-441bp~-193bp区段可能是OsHAK26基因启动子的核心启动区域。而-963~-441bp区段具有显著抑制效应,可能是含有蔗糖合成抑制元件MYBGAHV或者其他的抑制元件;-1 473~-963bp区段具有增强效应,可能是含有转录激活元件BOXLCOREDCPAL;由图 3可见OsHAK26P1与OsHAK26P3启动活性相同,可能是-963~-441bp区段的抑制作用和-1 473~-963bp区段的激活作用刚好抵消。未来可以进一步探究-963~-441bp和-1 473~-963bp区段所含有的抑制元件和增强元件的核心序列及定位。也可以利用OsHAK26基因启动子驱动β-葡萄糖醛酸酶报告基因表达,从而研究OsHAK26基因在不同组织器官中的差异性表达以及胁迫情况下的表达量变化[9]。
本文对OsHAK26基因启动子的结构进行研究,表明启动子四种不同的缺失片段均具有启动活性,且不同片段缺失对启动子活性影响不同。为进一步探究OsHAK26基因的生物学功能和通过基因工程提高或者改变外源基因的表达奠定了理论基础。
| [1] | Clarkson D T, Hanson J B. The mineral nutrition of higher plants. Annual Review of Plant Physiology, 1980, 311(1) : 239–298. |
| [2] | Robert H, Ralf R M. Physiological functions of mineral micronutrients (Cu, Zn, Mn, Fe, Ni, Mo, B, Cl). Current Opinion in Plant Biology, 2009, 12(3) : 259–266. DOI:10.1016/j.pbi.2009.05.006 |
| [3] | Schroeder J I, Ward J M, Gassmann W. Perspectives on the physiology and structure of inward-rectifying K+ channels in higher plants:biophysical implications for K+ uptake. Annu Rev Biophys Biomol Struct, 1994, 23 : 441–471. DOI:10.1146/annurev.bb.23.060194.002301 |
| [4] | Gupta M, Qiu X, Wang L, et al. KT/HAK/KUP potassium transporters gene family and their whole-life cycle expression profile in rice (Oryza sativa). Molecular Genetics and Genomics, 2008, 280 : 437–452. DOI:10.1007/s00438-008-0377-7 |
| [5] | Zhang Z, Zhang J, Chen Y, et al. Genome-wide analysis and identification of HAK potassium transporter gene family in maize (Zea mays L.). Molecular Biology Reports, 2012, 39 : 8465–8473. DOI:10.1007/s11033-012-1700-2 |
| [6] | Rubio F, Santa-María G E, Rodríguez-navarro A. Cloning of Arabidopsis and barley cDNAs encoding HAK potassium transporters in root and shoot cells. Physiologia Plantarum, 2000, 109 : 34–43. DOI:10.1034/j.1399-3054.2000.100106.x |
| [7] | Yang Z, Gao Q, Sun C, et al. Molecular evolution and functional divergence of HAK potassium transporter gene family in rice (Oryza sativa L.). Journal of Genetics and Genomics, 2009, 36 : 161–172. DOI:10.1016/S1673-8527(08)60103-4 |
| [8] | Yang T, Zhang S, Hu Y, et al. The role of a potassium transporter OsHAK5 in potassium acquisition and transport from roots to shoots in rice at low potassium supply levels. Plant Physiology, 2014, 166 : 945–959. DOI:10.1104/pp.114.246520 |
| [9] | Bañuelos M A, Garciadeblas B, Cubero B, et al. Inventory and functional characterization of the HAK potassium transporters of rice. Plant Physiology, 2002, 130 : 784–795. DOI:10.1104/pp.007781 |
| [10] | Grabov A. Plant KT/KUP/HAK potassium transporters:single family-multiple functions. Annals of Botany, 2007, 99 : 1035–1041. DOI:10.1093/aob/mcm066 |
| [11] | Amrutha R N, Sekhar P N, Varshney R K, et al. Genome-wide analysis and identification of genes related to potassium transporter families in rice (Oryza sativa L.). Plant Sci, 2007, 172 : 708–721. DOI:10.1016/j.plantsci.2006.11.019 |
| [12] | 聂丽娜, 夏兰琴, 徐兆师, 等. 植物基因启动子的克隆及其功能研究进展. 植物遗传资源学报, 2008, 9(3) : 385–391. Nie L N, Xia L Q, Xu Z S, et al. Progress on cloning and functional study of plat gene pomoters. Journal of Plant Genetic Resources, 2008, 9(3) : 385–391. |
| [13] | Yoo S, Cho Y, Sheen J. Arabidopsis mesophyll protoplasts:a versatile cell system for transient gene expression analysis. Nature Protocols, 2007, 2(7) : 1565–1572. DOI:10.1038/nprot.2007.199 |
| [14] | Lescot M, Déhais P, Thijs G, et al. PlantCARE, a database of plant cis-acting regulatory elements and a portal to tools for in silico analysis of promoter sequences. Nucleic Acids Res, 2002, 30 : 325–327. DOI:10.1093/nar/30.1.325 |
| [15] | Higo K, Ugawa Y, Iwamoto M, et al. Plant cis-acting regulatory DNA elements (PLACE) database. Nucleic Acids Res, 1999, 27 : 297–300. DOI:10.1093/nar/27.1.297 |
| [16] | Yi W, Wei H W. Potassium transport and signaling in higher plants. Annu Rev Plant Biol, 2013, 64 : 451–476. DOI:10.1146/annurev-arplant-050312-120153 |
| [17] | Rogers H J, Bate N, Combe J, et al. Functional analysis of cis-regulatory elements within the promoter of the tobacco late pollen gene g10. Plant Mol Biol, 2001, 45 : 577–585. DOI:10.1023/A:1010695226241 |
| [18] | Twell D, Wing R, Yamaguchi J, et al. Isolation and expression of an anther-specific gene from tomato. Mol Gen Genet, 1989, 217 : 240–245. DOI:10.1007/BF02464887 |
| [19] | Chen G, Hu Q D, Luo L, et al. Rice potassium transporter OsHAK1 is essential for maintaining potassium-mediated growth and functions in salt tolerance over low and high potassium concentration ranges. Plant, Cell and Environment, 2015, 38 : 2747–2765. DOI:10.1111/pce.12585 |
| [20] | Abe H, Urao T, Ito T, et al. Arabidopsis AtMYC2 (bHLH) and AtMYB2 (MYB) function as transcriptional activators in abscisic acid signaling. The Plant Cell, 2003, 15(1) : 63–78. DOI:10.1105/tpc.006130 |
| [21] | Simpson S D, Nakashima K, Narusaka Y, et al. Two divergent novel cis-acting elements of erd1, a clpA homologous Arabidopsis gene function in induction by dehydration stress and dark-induced senescence. Plant J, 2003, 33 : 259–270. DOI:10.1046/j.1365-313X.2003.01624.x |
| [22] | Gupta M, Qiu X H, Wang L, et al. KT/HAK/KUP potassium transporters gene family and their whole-life cycle expression profile in rice (Oryza sativa). Mol Genet Genomics, 2008, 280 : 437–452. DOI:10.1007/s00438-008-0377-7 |
| [23] | Aida M H, Luisa L O, Gerardo A A, et al. Functional properties and regulatory complexity of a minimal RBCS light-responsive unit activated by phytochrome, cryptochrome, and plastid signals. Plant Physiol, 2002, 128 : 1223–1233. DOI:10.1104/pp.010678 |
| [24] | Yamamoto Y Y, Ichida H, Matsui M, et al. Identification of plant promoter constituents by analysis of local distribution of short sequences. BMC Genomics, 2007, 8 : 67. DOI:10.1186/1471-2164-8-67 |
| [25] | Oo M, Bae H K, Nguyen T, et al. Evaluation of rice promoters conferring pollen-specific expression in a heterologous system, Arabidopsis. Plant Reproduction, 2014, 27 : 47–58. DOI:10.1007/s00497-014-0239-x |
 2017, Vol. 37
2017, Vol. 37




