文章信息
- 陈建武, 任红艳, 华文君, 刘西梅, 綦世金, 周黎, 欧阳艳, 毕延震, 杨烨, 郑新民.
- CHEN Jian-wu, REN Hong-yan, HUA Wen-jun, LIU Xi-mei, QI Shi-jin, ZHOU Li, OU Yang-yan, BI Yan-zhen, YANG Ye, ZHENG Xin-min.
- 一种用于提高基因打靶效率的双荧光筛选策略
- A Double Fluorescence Screening Strategy to Enhance the Efficiency of Gene Targeting
- 中国生物工程杂志, 2017, 37(1): 58-63
- China Biotechnology, 2017, 37(1): 58-63
- http://dx.doi.org/DOI:10.13523/j.cb.20170109
-
文章历史
- 收稿日期: 2016-07-12
- 修回日期: 2016-10-12
2. 湖北省农业科学院畜牧兽医研究所 动物胚胎工程及分子育种湖北省重点实验室 武汉 430064;
3. 湖北三峡职业技术学院农学院 宜昌 443000
2. Hubei Key Lab of Animal Embryo Engineering and Molecular Breeding, Institute of Animal Science and Veterinary, Hubei Academy of AgroSciences, Wuhan 430064, China;
3. College of Agriculture, Hubei Three Groges Vocational and Technical College, Yichang 443000, China
基因打靶技术兴起于20世纪80年代[1-3],它的产生和发展是建立于基因同源重组技术和胚胎干细胞(ES)技术上的一种分子生物学技术。通过利用细胞基因组DNA可与外源性DNA的同源序列发生同源重组的特性,来人为定向改造细胞染色体上的某一基因称为基因打靶[4]。2007年科学家Capecchi等因在基因打靶技术上的开创性成就而获得了诺贝尔生理学或医学奖。当前,运用同源重组介导的基因敲除依然是构建基因敲除动物模型准确性最高的方法。
基因敲除的技术路线并不复杂,但由于真核生物内外源DNA与靶细胞DNA序列自然发生同源重组的概率非常低,约为百万分之一[5],要把基因敲除成功的细胞筛选出来是一件非常困难的工作。因此,同源重组的筛选和检测就成了基因敲除技术所要解决的关键问题。所以,提高同源重组的筛选效率是提高基因打靶效率的一个重要途径,目前已有多种筛选方法,包括1988年犹他大学的Capecchi等[6]的研究组发展的正负双向选择系统(positive-negative-selection,PNS)、启动子陷阱法(promoter trap)[7]及外显子陷阱法(exon-trapping)[8]。但是这些技术因筛选效率不高或广谱性不强,极大的限制了基因打靶技术的发展。所以,建立一个广谱性强的高效筛选系统尤为重要。本研究将传统的正负筛选改良为“两正一负”筛选策略,并将筛选“可视化”。将增强绿色荧光蛋白(EGFP)基因和新霉素抗性(NeoR)基因作为正筛选,红色荧光蛋白(RFP)基因作为负筛选,从而建立起一个广谱性强的可视化筛选策略。通过运用CRISPR/Cas9基因编辑技术对湖北白猪肌肉生长抑制素基因(Myostatin-MSTN)进行定点敲除[9],期望得到高阳性率的细胞系,验证本策略的可行性,从而建立一个广谱性强的高筛选策略。
1 材料与方法 1.1 材 料限制性内切核酸酶、dNTP Mix、高保真酶rTaq、pMD-18T载体和T4 DNA连接酶,购自宝生物工程(大连)有限公司;质粒DNA小量提取试剂盒、琼脂糖凝胶DNA回收试剂盒购自天根生化科技(北京)有限公司;大肠杆菌DH5α为湖北省农业科学院畜牧兽医研究所生物技术室存留;HRX-2MCS载体为本实验室保存,PX330载体购自美国Addgene公司;引物合成和基因测序由华大(武汉)基因生物科技有限公司完成。细胞培养相关试剂DMEM、胎儿血清、G418、电转液均购自GIBCO公司。电转染仪ECM 2001 购自BTX公司。
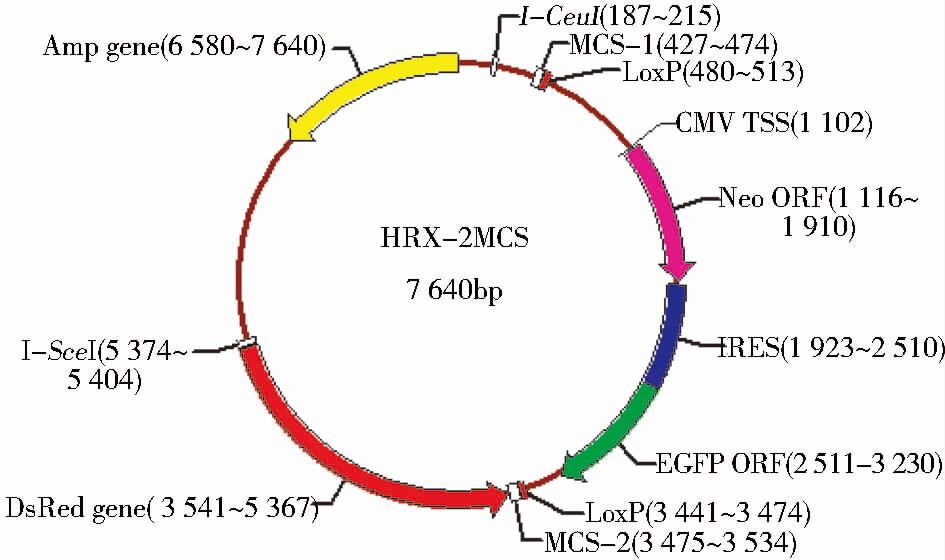
1.2 方 法 1.2.1 双荧光打靶系统的建立选用pUC19作为骨架载体。在pUC19载体Nde l和Hind III酶切位点分别克隆进I-Ceu I和I-Sce I两个稀有的酶切位点,形成hpUC19载体。再将RFP-wt质粒上的RFP表达框(CMV+RFP+polyA)通过hpUC19上预留的Hind III位点克隆进RFP表达框,形成hRpUC19。再则通过酶 切位点Sac I 和Sal I将LoxP-CMV-Neo-IRES-EGFP-LoxP表达框克隆进hRpUC19形成HRX载体。最后在Sac I酶切位点后引入酶切位点Sgs I-BC II-Pac I-Cla I-SfaA I形成多克隆位点1(MCS-1),在Sal I酶切位点后引入Mre I-Nsi I-BbvC I形成多克隆位点2(MCS-2),最终形成HRX-2MCS(图 1)。
|
| 图 1 HRX-2MCS载体 Figure 1 HRX-2MCS vector |
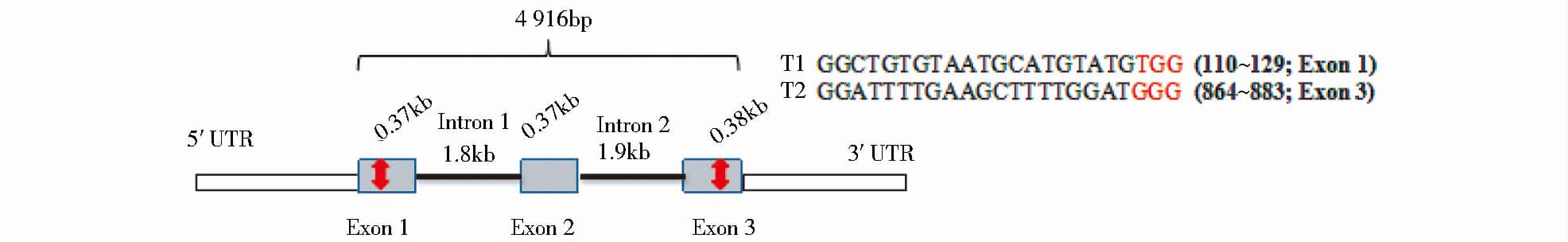
从NCBI获得湖北白猪MSTN基因组序列,利用ZiFit软件预测靶位点,选取其中外显子1和外显子3的2个位点(图 2)。
|
| 图 2 猪MSTN基因打靶示意图 Figure 2 Diagram of gene targeting in pig MSTN Target site |
利用HRX-2MCS载体作为骨架载体。采取湖北白猪耳组织,提取基因组并以此为模板,用LA Taq 酶扩增同源臂,引物序列见表 1。根据GenBank中公布的猪MSTN序列设计引物扩增同源臂。T1、T2位点的同源左臂分别用C1Lf/C1Lr和C2Lf/C2Lr,同源右臂分别用C1Rf/C1Rr和C2Rf/C2Rr进行扩增。再通过PCR方法在左臂的上游和下游分别引入酶切位点EcoR I和Cla I,在右臂的上游和下游分别引入酶切位点Sal I和Nsi I。将同源臂进行TA克隆后,送公司测序。同源臂测序完成后,用EcoR I和Cla I双酶切左臂TA克隆,Sal I和Nsi I双酶切右臂TA克隆,回收同源臂,再分别用相同的酶双酶切HRX-MCS2载体之后与同源臂连接。完成供体载体dT1和dT2的构建。
| Primer name | Primer sequence(5′-3′) | Size(bp) |
| C1Lf C1Lr C2Lf C2Lr C1Rf C1Rr C2Rf C2Rr T1-UP T2-UP T1-Down T2-Down MKR EGFP-QF1 | GAGGAAAAAGACATTTCAAC GGAAAAAGAGGGGCTGTG CCTAGTGAATGGAGGAAGG CTAACTGTGGATTTTGAAGC TGGAGACAAAACACTAAATC TCCAGGCAGGCACATGCTTAA ATGGGACTGGATTATTGC GCAATGGTTGGCATTTAACC CTGGAAATCTGAGGCAAACTGC CTACCACTCCCTTCATCACCTAC CAGGCACCTTAACAGAGAAGGC GCCTGCCTTTGCAACACTGCAG CGGGGTCATTAGTTCATAGCCC CAAGCAGAAGAACGGCATCAAGGT | 20 18 19 20 20 21 18 20 22 23 22 22 21 23 |
通过公司合成带有Bbs I酶切位点黏性末端T1、T2靶序列的Oligo。以PX330(hSpCas9+CRISPR Array+tracrRNA)作为骨架载体,将PX330用Bbs I单酶切后,与靶位点Oligo连接,制得Cas9载体T11和T21。
1.2.4 打靶载体电穿孔转染PK-15细胞及细胞筛选在12孔细胞板中培养PK-15细胞至70%~90%汇合时,用胰酶消化悬浮细胞。吸取悬浮液,离心后弃上清液,再用DPBS将细胞清洗两遍。用50μl电转液悬浮细胞,分别按照1∶1的比例加入5μg dT1/T11和dT2/T31的混合质粒,混匀,室温放置5min,转移至0.4cm的点转杯,在350V/cm,3ms条件下,电穿孔1次。电击完毕后加2ml培养液,然后置于39℃、5%CO2、100%湿度培养箱培养。电转24h后,加1 000ng/ml G418进行筛选,之后每天观察细胞死亡状况,并根据细胞存活状况依次降低G418浓度。每2~3天更换一次培养液,筛选20天左右后会发现克隆斑形成。用荧光显微镜观察,挑选出在红光显微镜下发红光且在绿光显微镜下不发绿光的克隆斑。在荧光显微镜下,将边缘清晰、活力旺盛的细胞用胰酶稍许消化后,用克隆环挑取出来并转移至48孔细胞培养板中。当细胞克隆斑长至90%密度时,用胰酶消化、分离出一半细胞用于PCR检测用,剩余细胞继续扩增培养。
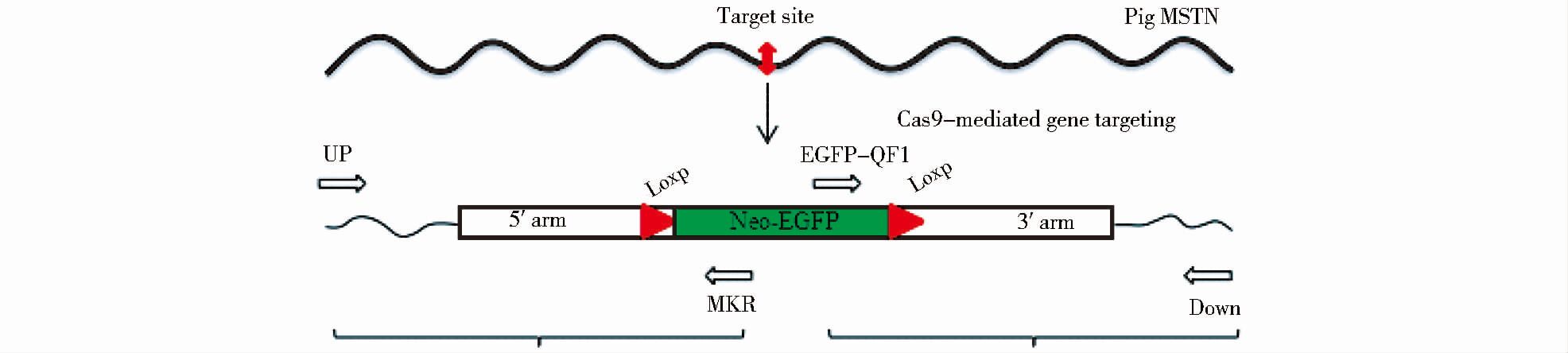
1.2.5 细胞单克隆的PCR鉴定及测序根据猪MSTN基因组序列和供体载体dT1/dT2设计5′端和3′端鉴定引物UP/MKR和EGFP-QF1/Down,如图 3所示。其中UP/Down序列位于打靶位点左右臂外侧方向猪MSTN基因上,MKR/EGFP-QF1位于供体载体dT1/dT2的Neo-EGFP表达框上。将UP/MKR和EGFP-QF1/Down扩增出来的PCR产物纯化回收后,做TA克隆,送公司测序,验证定点打靶是否成功(图 3)。
|
| 图 3 基因打靶检测示意图 Figure 3 Detection diagram of gene targeting |
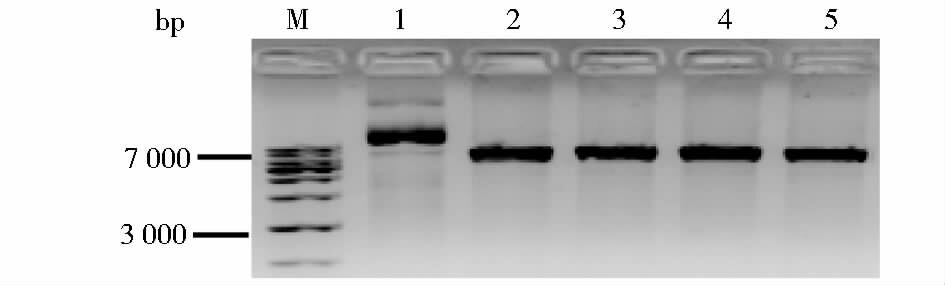
本实验主要以PUC19为骨架构建了双荧光打靶系统HRX-2MCS载体。该载体引入两个稀有酶切位点I-Ceu I和I-Sce I,方便质粒的线性化;在Hind III酶切位点引入RFP表达框,作为负筛选在发生同源重组后RFP表达框会脱落;在Sac I 和Sal I酶切点间引入EGFP-Neo表达框,在发生同源重组后表达框会置换打靶位点序列形成正筛选作用,表达绿色荧光蛋白和具有G418耐药性;最后引入两个多克隆位点方便插入同源臂。分别用MSC1上的EcoR I、Cla I,MSC2上的Sal I、Nsi I酶切HRX-2MCS载体,验证载体构建的正确性(图 4)。
|
| 图 4 HRX-2MCS载体酶切鉴定图 Figure 4 Identification of HRX-2MCS vector by enzyme digestion M: DNA marker 1kb; 1:HRX-2MCS vector; 2~5:HRX-2MCS respective digested with EcoR I/Cla I/Sal I/Nsi I |
将dT1和T11及dT2和T21分别共转染PK-15细胞,经过G418和绿色荧光蛋白正筛选及红色荧光蛋白负筛选,T1、T2位点分别得到了36个和30个具有药物抗性及表达绿色荧光蛋白的细胞克隆斑(图 5)。细胞经过扩增培养,消化部分细胞准备进行PCR扩增。
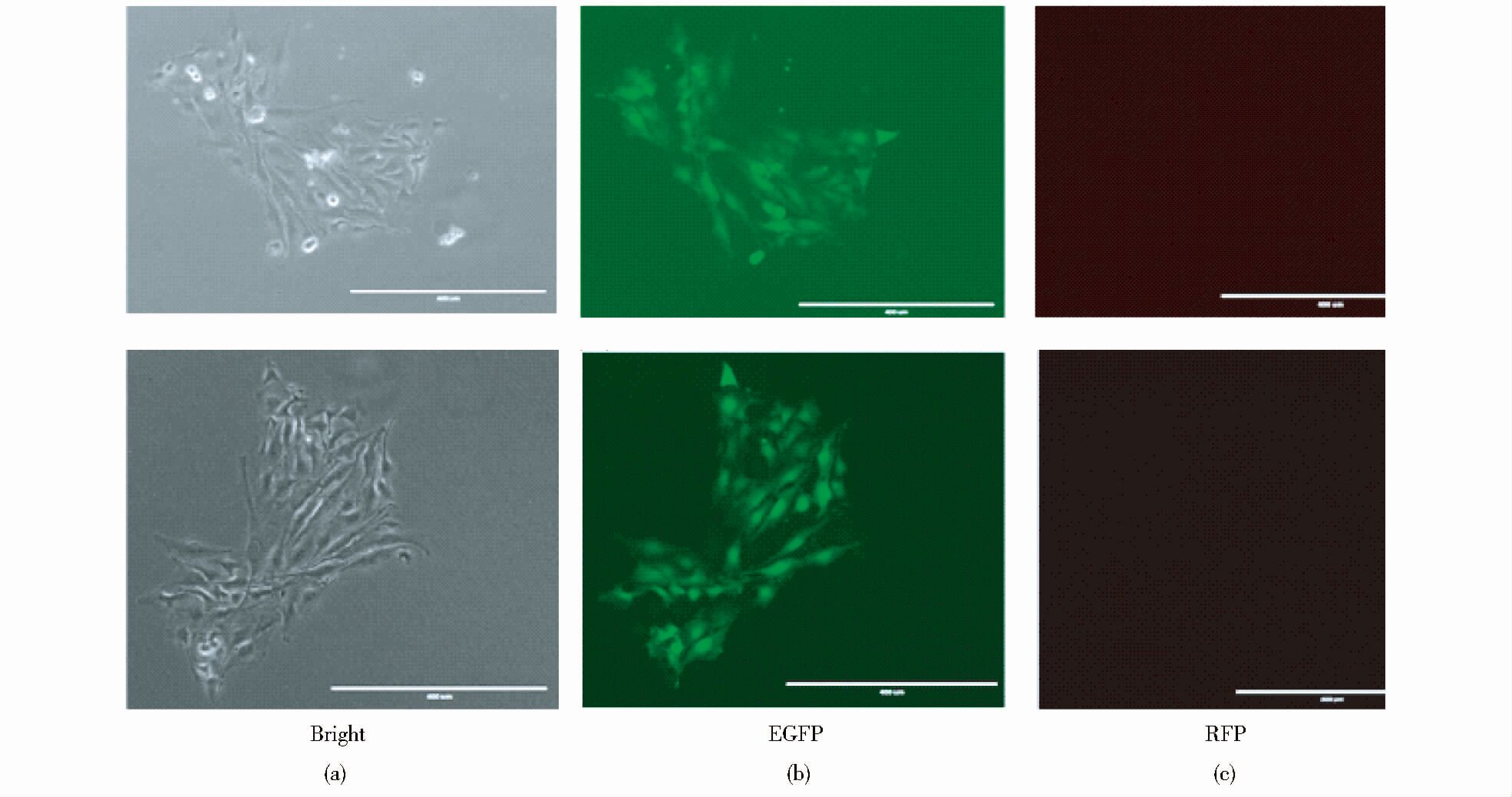
|
| 图 5 克隆斑在不同光照下的形态 Figure 5 The shape of cell colony under different lighting (a) Under the bright lighting (b) Under the green fluorescent lighting (c) Under the red fluorescent lighting. |
提取克隆斑的基因组DNA,分别用UP/MKR和EGFP-QF1/Down引物检测5′端和3′端。T1位点5′端能扩增出1 196bp片段,3′端能扩增出1 797bp片段;T2位点5′端能扩增出962bp片段,3′端能扩增出1 503bp片段。经过PCR检测,T1、T2位点分别有阳性克隆斑29个和26个(图 6)。将PCR检测阳性克隆斑的PCR产物回收后做TA克隆,送公司测序,结果均是阳性。

|
| 图 6 克隆斑的PCR鉴定结果和测序图谱 Figure 6 PCR and sequencing analysis of cell colony (a) T1 site 5′ sequence analysis (b) T1 site 3′ sequence analysis (c) T2 site 5′ sequence analysis (d) T2 site 3′ sequence analysis. The sequence included by red arrows is from targeting vector,the black arrows is from wild type gene |
基因打靶技术是现代分子生物学的一项重要的技术手段,它无论是在基础理论研究还是在实际应用中都有广阔的前景[10]。利用同源重组介导的基因打靶是目前构建动物模型常用的方法,但是自然状况下发生同源重组概率极低,要把发生打靶细胞筛选出来极为困难[5],这样大大阻碍了人类对基因功能的研究,所以建立一种高效的筛选策略极为重要。
本研究以pUC19为骨架载体,通过载体上的酶切位点克隆进RFP、EGFP、Neo等筛选基因,从而构建一个通用型高筛选效率的载体HRX-2MCS。RFP表达框位于同源区外侧,转入细胞后会随着载体的表达而表达红色荧光蛋白,当细胞发生同源重组后同源区外侧的RFP基因脱落,从而起到负筛选作用。LoxP-CMV-Neo-IRES-EGFP-LoxP表达框位于同源区内,发生同源重组后细胞会表达EGFP基因和具有G418药性,G418作为正筛选能杀死大多数未中靶的细胞,作为第二个筛选基因EGFP表达的绿色荧光蛋白能筛选出中靶细胞。
本研究的筛选策略具有“可视性”,当转染初期,质粒进入细胞,细胞会表达质粒上的RFP和EGFP基因,呈现红光和绿光,可以根据这一特点掌握转染效率,可以探索出转染效率较高的条件,从而提高基因打靶效率;伴随着CRISPR/Cas9系统的作用,细胞发生同源重组,此时已同源重组的细胞就会有G418抗性和绿色荧光蛋白的表达,细胞在荧光显微镜下呈现只发绿光不发红光的特性。通过加G418药筛和保留只表达绿色荧光蛋白的细胞,本实验得到猪MSTN基因T1位点克隆斑36个、T2位点克隆斑30个。通过PCR检测和产物测序证实T1位点打靶成功克隆斑有29个,阳性率为80.5%;T2位点打靶成功克隆斑有26个,阳性率为86.7%。两个位点的阳性率均为80%以上。在传统的正负双向选择系统中,由于细胞产生耐药性,使得筛选出来的细胞阳性率较低,在小鼠ES细胞中效率只有0.5%~5%,大大地增加了检测的成本和难度[6],然而在传统正负筛选上改良的“双荧光”筛选法在猪体细胞中的筛选效率达到80%以上,筛选效率得到极大的提升。启动子陷阱法的筛选效率较高,在小鼠ES细胞中约为50%,但是该方法要求打靶基因有较高的表达量,极大的限制了使用范围[11]。外显子陷阱打靶系统中,由于打靶位点仅限于外显子位点,对内含子和非编码区无法进行人为修饰,广谱性并不强[8]。本研究将传统的正负双向选择系统进行升华,运用“可视化”的两正一负筛选策略,极大的提高了筛选效率,而且在编码区和非编码区都能进行打靶,广谱性极强。本筛选策略通过与新一代内切核酸酶系统ZFN[12]、TALEN[13]、Cas9[14]、NgAgo[15]结合可以大大提高基因打靶的效率,为基因功能的探索、家畜品种的改良、疾病的治疗等提供了一种新策略。
| [1] | Thomas K R, Capecchi M R. Site-directed mutagenesis by gene targeting in mouse embryo-derived stem cells. Cell , 1987, 51 (3) : 503–512. DOI:10.1016/0092-8674(87)90646-5 |
| [2] | Doetschman T, Gregg R G, Maeda N, et al. Targeted correction of a mutant HPRT gene in mouse embryonic stem cells. Nature , 1987, 330 (6148) : 576–578. DOI:10.1038/330576a0 |
| [3] | Koller BH, Smithies O. Inactivating the beat 2-microglobin locus in mouse embryonic stem cells by homologous recombination. Proc Natl Acad Sci USA , 1989, 86 : 8932–8935. DOI:10.1073/pnas.86.22.8932 |
| [4] | Mansour S L, Thomas K R, Deng C, et al. Introduction of a lacZ reporter gene into the mouse int-2 locus by homologous recombination. Proc Natl Acad Sci USA , 1990, 87 (19) : 7688–7692. DOI:10.1073/pnas.87.19.7688 |
| [5] | Kan Y, Ruis B, Lin S. The mechanism of gene targeting in human somatic cells. PLoS Genet , 2014, 10 (4) : e1004251. DOI:10.1371/journal.pgen.1004251 |
| [6] | Mansour S L, Thomas K R, Capecchi M R. Disruption of the proto-oncogene int-2 in mouse embryo-derived stem cells:a general strategy for targeting mutations to non-selectable gene. Nature , 1988, 336 (6197) : 348–352. DOI:10.1038/336348a0 |
| [7] | Stanford W L, Cohn J B, Cordes S P. Gene-trap mutagenesis:past, present and beyond. Nat Rev Genet , 2001, 2 (10) : 756–768. DOI:10.1038/35093548 |
| [8] | Shinta S, Kiyoe U, Miho K, et al. Construction and applications of exon-trapping gene-targeting vectors with a novel strategy for negative selection. BMC Res Notes , 2015, 8 (1) : 278–287. DOI:10.1186/s13104-015-1241-6 |
| [9] | Mussolino C, Cathomen T. RNA guides genome engineering. Nat Biotechnol , 2013, 31 (3) : 208–209. DOI:10.1038/nbt.2527 |
| [10] | Thomson A J, Mcwhir J. Biomedical and agricultural applications of animal transgenesis. Mol Biotechnol , 2004, 27 (3) : 231–244. DOI:10.1385/MB:27:3 |
| [11] | Friedel R H, Plump A, Lu X, et al. Gene targeting using a promoterless gene trap vector ("targeted trapping") is an efficient method to mutate a large fraction of genes. Proc Natl Acad Sci USA , 102 (37) : 13188–13193. DOI:10.1073/pnas.0505474102 |
| [12] | Kim Y G, Cha J, Chandrasegaran S. Hybrid restriction enzymes:zinc finger fusions to Fok I cleavage domain. Proc Natl Acad Sci USA , 1996, 93 (3) : 1156–1160. DOI:10.1073/pnas.93.3.1156 |
| [13] | Li T, Huang S, Zhao X, et al. Modularly assembled designer TAL effector nucleases for targeted gene knockout and gene replacement in eukaryotes. Nucleic acids Res , 2011, 39 (14) : 6315–6325. DOI:10.1093/nar/gkr188 |
| [14] | Qi LS, Larson M H, Gilbert L A, et al. Repurposing CRISPR as an RNA-guided platform for sequence-specific control of gene expression. Cell , 2013, 152 (2) : 1173–1183. |
| [15] | Gao F, Shen X Z, Jiang F, et al. DNA-guided genome editing using the natronobacterium gregoryi argonaute. Nature Biotechnology , 2016, 34 (7) : 768–713. DOI:10.1038/nbt.3547 |
 2017, Vol. 37
2017, Vol. 37




