文章信息
- 李博, 梁楠, 刘夺, 刘宏, 王颖, 肖文海, 姚明东, 元英进.
- LI Bo, LIANG Nan, LIU Duo, LIU Hong, WANG Ying, XIAO Wen-hai, YAO Ming-dong, YUAN Ying-jin.
- 合成8二甲基异戊烯基柚皮素的人工酿酒酵母菌株构建
- Metabolic Engineering of Saccharomyces cerevisiae for Production of 8-Dimenthylally Naringenin
- 中国生物工程杂志, 2017, 37(9): 71-81
- China Biotechnology, 2017, 37(9): 71-81
- http://dx.doi.org/DOI:10.13523/j.cb.20170910
-
文章历史
- 收稿日期: 2017-03-15
- 修回日期: 2017-05-29
2. 天津化学化工协同创新中心合成生物学平台 天津 300072
2. SynBio Research Platform, Collaborative Innovation Center of Chemical Science and Engineering, Tianjin 300072, China
黄酮类化合物通常作为植物的防卫元素,在植物抵御外来因素侵扰过程中发挥重要作用。鉴于该类物质具有抗菌、抗炎、抗癌、解痉和利胆等功效,因此在人类疾病研究和药理研究中具有重要应用价值[1-4]。其中,以8二甲基异戊烯基柚皮素(8DN)作为代表的黄酮类产物,不仅具有药理研究价值,同时还是淫羊藿素等药物合成的重要前体,越来越被人们广泛关注和研究[5-7]。
8DN的合成方法目前主要分为化学合成法和生物合成法,其中化学合成法是以间苯三酚为原料与苄氧乙腈发生酰化反应,再经过羟醛缩合及多步的脱羟保护反应,最终得到产物[8-10]。该方法的局限性在于环境污染严重,生产效率低。目前8DN的生物合成法受限于合成路径及相关基因的复杂性,主要通过饲喂8DN直接前体的方式来实现[11]。目前日本学者Sasaki等[12]利用8DN合成酶N8DT通过柚皮素细胞饲喂的方式获得了8DN,产量达20.2μg/g。而浙江大学的傅明亮等[13-14]通过饲喂异黄腐酚,在脱甲基酶的作用下合成了8DN,产物转化率达到6.62%。该方法在一定程度上避免了化学合成中多步反应的低效率和环境污染的问题[15-18],但存在着饲喂原料成本昂贵,可利用率低等问题,限制了其工业应用价值和生物合成路径的可操作性。
针对上述情况,本研究利用合成生物学思想和技术[19-20],拟在安全模式微生物酿酒酵母中[21-24]优化组合8DN合成的关键外源基因,构建生产8DN的完整合成途径。鉴于8DN合成路径及相关基因的复杂性,我们首先通过引入不同来源的柚皮素代谢路径基因TAL、4CL、CHS和CHI,在酿酒酵母体内构建一条8DN前体物柚皮素的合成路径,利用多基因组合优化筛选出柚皮素高产菌株,并进一步借助Delta位点整合的方式将高产菌的基因线路组合转移到酵母基因组中[25],从而获得稳定的柚皮素高产底盘菌。在此基础上导入最后一个关键酶基因模块——异戊烯基转移酶基因N8DT[26],初步实现8DN在酿酒酵母中的生物全合成(图 1)。此外我们针对关键酶N8DT的低活性问题,通过蛋白质二级结构的分析,提出了蛋白质结构优化方案,对蛋白质的两组特定结构序列进行截断:① 定位信号肽区域(1~40aa);② disorder区域(1~84aa)。实验结果显示,截断后N8DT的活性都反映出正向效果,其中定位信号肽区域截断后的N8DT蛋白具有更好的催化效果,促使8DN的产量进一步提高了44.2%,摇瓶产量高达52.6μg/L。
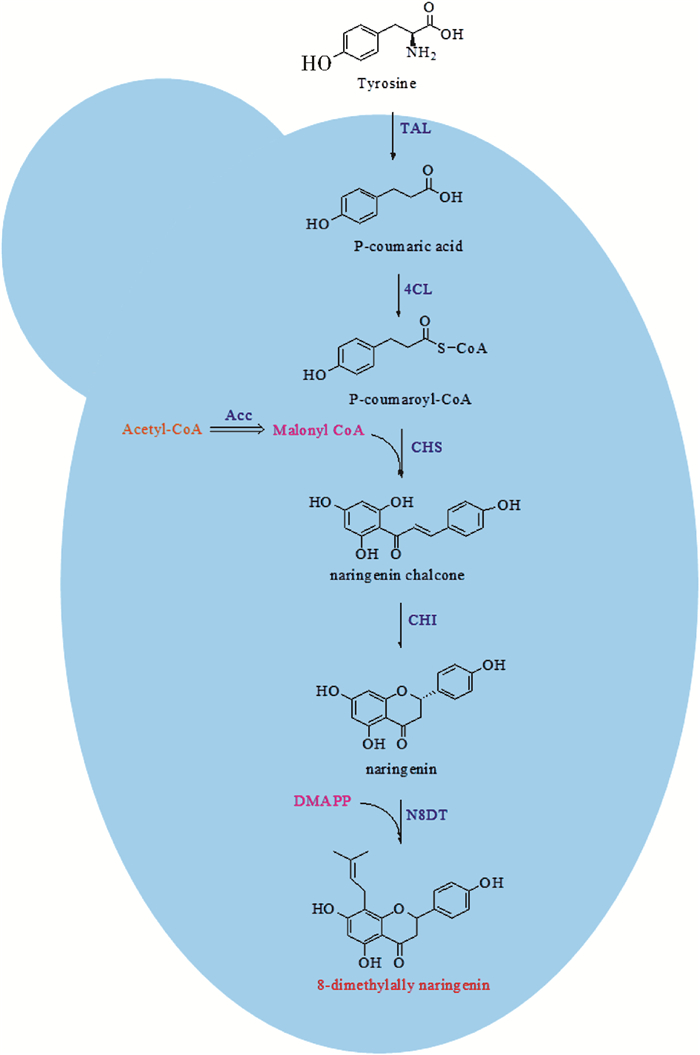
|
| 图 1 酿酒酵母体内8DN合成路径的构建 Figure 1 Construction of 8-dimenthylally naringenin pathway in S.cerevisiae The five exogenous genes TAL, 4CL, CHS, CHI and N8DT were introduced in the pathway (malonyl-CoA and DMAPP are the products of endogenous metabolism) |
大肠杆菌感受态细胞DH5α购自Biomed公司,TransStart FastPfu高保真DNA聚合酶购自TransGen公司;T4 DNA连接酶;限制性内切核酸酶Bsa I-HF、Not I-HF、Sac I-HF和Sal I-HF购自NEB公司;质粒小提试剂盒、普通DNA产物纯化试剂盒、琼脂糖凝胶DNA回收试剂盒购自天根公司;柚皮素和8DN标品购自上海源叶生物有限公司;甲醇、乙腈和二氯甲烷购自Fisher公司;丙酮购自天津市元立化工有限公司;二甲基亚砜(DMSO)、PEG3350、鲑鱼精DNA、各类氨基酸粉末购自鼎国昌盛公司;乙酸锂购自北方天医化学试剂厂;去氨基酸酵母氮源(YNB)购自Genview公司。
1.2 菌株、质粒和培养基宿主菌E. coli DH5α [endA1; hsdR17; gyrA96;thi-1; relA1; supE44; recA1; ΔlacU169(φ80lac ΔZM15)]和酿酒酵母菌株CENpK2-1d(MATa,his3Δ1,leu2Δ0,trp3Δ0,ura3Δ0) 购自Euroscarf公司,酵母表达载体质粒pRS425K为本实验室构建,详见表 1。
| Strains | Chassis | Description | Source |
| CENpK2-1d | - | MATa,his3 Δ 1,leu2 Δ 0,trp3 Δ 0,ura3 Δ 0 | Purchased |
| SyBE_Sc02050001-SyBE_Sc02050030 | CENpK2-1d | pRS415: TGPM1-PPG1K-TAL-TGPD1 pRS423: TGPD1-PPGI1-4CL-TFBA1 pRS414: TFBA1-CHS-PTPI1-TPGK1 pRS416: TPGK1-PHXT7-CHI-TCYC1 |
This study |
| SyBE_Sc02050031 | CENpK2-1d | Delta::ura3_PPGK1-TAL-TGPD1-PPGI3-4CL-TFBA1-PTPI1-CHS-TPGK1-PHXT7-CHI-TCYC1 | This study |
| SyBE_Sc02050032 | SyBE_Sc02050031 | pRS425K-PENO2-Sf_N8DT-TGPM1 | This study |
| SyBE_Sc02050033 | SyBE_Sc02050031 | pRS425K-PENO2-Sf_N8DTjie1-40-TGPM1 | This study |
| SyBE_Sc02050034 | SyBE_Sc02050031 | pRS425K-PENO2-Sf_N8DTjie1-84-TGPM1 |
LB培养基(10g/L氯化钠、10g/L胰蛋白胨、5g/L酵母提取物,固体培养基添加1.5%琼脂粉)用于大肠杆菌的培养,YPD培养基(40g/L葡萄糖、20g/L胰蛋白胨、10g/L酵母提取物,固体培养基添加2%琼脂粉)用于酿酒酵母的培养,SC四缺培养基(20g/L葡萄糖、6.7g/L YNB、2g/L drop-out的氨基酸混合物,固体培养基添加2%琼脂粉)、SC-leu培养基(20g/L葡萄糖、6.7g/L YNB、2g/L drop-out去除异亮氨酸的氨基酸混合物,固体培养基添加2%琼脂粉)和SC-ura培养基(20g/L葡萄糖、6.7g/L YNB、2g/L drop-out去除尿嘧啶的氨基酸混合物,固体培养基添加2%琼脂粉)用于酵母转化子筛选和培养。
1.3 酿酒酵母表达载体的构建和基因组模块整合与筛选基因表达载体模块的构建如图 2(a)、(b)所示。以BY4741基因组DNA为模板,通过特异性引物PCR扩增CYC1、GPM1、GPD、FBA1、PGK1和ENO2终止子,PGK1、PGI1、TPI1、HXT7和PDC启动子,L-arm(Pst I酶切位点上游250bp),R-arm(Bsa I终止密码子下游254bp)和ura3 ORF框;通过OE-PCR扩增得到delta1-URA3-TGPM片段并以Pst I和BamH I酶切连接到pRS425K,命名为Module1;通过OE-PCR扩增得到TGPM1-PPGK1-TGPD1片段并以Pst I和BamH I酶切连接到pRS425K,命名为Module2;将TAL基因以Bsa I酶切连接到Module2中,命名为Module3;将TGPM1-PPGK1-TAL-TGPD1片段Not I酶切连入pRS415中,命名为Module4;相同的,通过OE-PCR扩增得到TGPD1-PPGI1-TFBA1片段并以Pst I和BamH I酶切连接到pRS425K,命名为Module5;将4CL以Bsa I酶切连接到Module5中,命名为Module6;将TGPD1-PPGI1-4CL-TFBA1片段Not I酶切连入pRS423中,命名为Module7;通过OE-PCR扩增得到TFBA1-PTPI1-TPGK1片段并以Pst I和BamH I酶切连接到pRS425K,命名为Module8;将CHS基因以Bsa I酶切连接到Module8中,命名为Module9;将TFBA1-PTPI1-CHS-TPGK1片段Not I酶切连入pRS414中,命名为Module10;通过OE-PCR扩增得到TPGK1-PHXT7-TCYC1片段并以Pst I和BamH I酶切连接到pRS425K,命名为Module11;将CHI基因以Bsa I酶切连接到Module11中,命名为Module12;将TPGK1-PHXT7-CHI-TCYC1片段Not I酶切连入pRS416中,命名为Module13;通过OE-PCR扩增得到TPGK1-delta2片段并以Pst I和BamH I酶切连接到pRS425K,命名为Module14;通过OE-PCR扩增得到TENO2-PPDC1-TGPM1片段并以Pst I和BamH I酶切连接到pRS425K,命名为Module15;将N8DT基因以Bsa I酶切连接到Module15中,命名为Module16(表 2)。
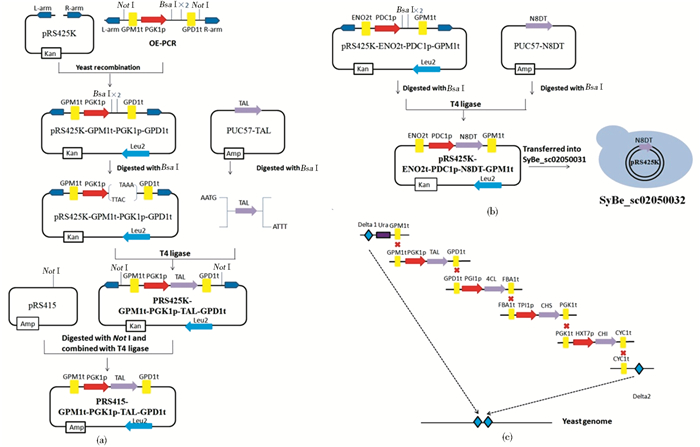
|
| 图 2 柚皮素及8DN生产菌的构建策略 Figure 2 The construction strategy of the strains produced naringenin and 8DN (a) Construction of the exgenous genes modules for synthesis of naringenin (for example TAL) (b) Construction and transformation of N8DT gene module for 8DN production (c) Schematic diagram of integrating naringenin genes into the chromosome by Delta integration |
| Plamids | Description | Source |
| pRS425K | Replace AmpKX with KanMX in pRS425 | This lab |
| pRS415 | Single copy plasmid with LEU2 and Amp marker | Purchased |
| pRS423 | Multi-copy plasmid with LEU2 and Amp marker | Purchased |
| pRS414 | Single copy plasmid with LEU2 and Amp marker | Purchased |
| pRS416 | Single copy plasmid with LEU2 and Amp marker | Purchased |
| Module1 | pRS425K: delta1-URA3-TGPM | This study |
| Module2 | pRS425K: TGPM1-PPG1K -TGPD1 | This study |
| Module3 | pRS425K: TGPM1-PPG1K-TAL-TGPD1 | This study |
| Module4 | pRS415: TGPM1-PPG1K-TAL-TGPD1 | This study |
| Module5 | pRS425K: TGPD1-PPGI1-TFBA1 | This study |
| Module6 | pRS425K: TGPD1-PPGI1-4CL-TFBA1 | This study |
| Module7 | pRS423: TGPD1-PPGI1-4CL-TFBA1 | This study |
| Module8 | pRS425K: TFBA1-PTPI1-TPGK1 | This study |
| Module9 | pRS425K: TFBA1-CHS-PTPI1-TPGK1 | This study |
| Module10 | pRS414: TFBA1-CHS-PTPI1-TPGK1 | This study |
| Module11 | pRS425K: TPGK1-PHXT7-TCYC1 | This study |
| Module12 | pRS425K: TPGK1-PHXT7-CHI-TCYC1 | This study |
| Module13 | pRS416: TPGK1-PHXT7-CHI-TCYC1 | This study |
| Module14 | pRS425K: TPGK1-delta2 | This study |
| Module15 | pRS425K: TENO2-PPDC1-TGPM1 | This study |
| Module16 | pRS425K: TENO2-PPDC1-Sf_N8DT-TGPM1 | This study |
| Module17 | pRS425K: TENO2-PPDC1-Sf_N8DTjie1-40-TGPM1 | This study |
| Module18 | pRS425K: TENO2-PPDC1-Sf_N8DTjie1-84-TGPM1 | This study |
利用包含4种外源基因(TAL、4CL、CHS和CHI)的模块Module4、Module7、Module10和Module13采用乙酸锂转化法转化酿酒酵母菌株CENpK2-1D,先构建出一系列柚皮素生产菌。并通过SC四缺固体培养基筛选单菌落,在SC四缺液体培养基中培养,30℃、200r/min培养至OD600约为4,存甘油菌。这些柚皮素生产菌的分别命名为SyBE_Sc02050001~SyBE_Sc02050030。随后选取柚皮素产量最高的SyBE_Sc02050003的4种基因模块组合用Not I酶切纯化后与Module1和Module14用乙酸锂转化法利用Delta位点整合至酿酒酵母CENpK2-1D基因组,提高柚皮素生产菌的稳定性,如图 2(c)所示。用SC-ura培养基筛选存菌,该菌株命名为SyBE_Sc02050031。然后以SyBE_Sc02050031作为底盘菌株,将Module16质粒用乙酸锂转化法转化至酵母体内,在SC-leu平板上30℃恒温倒置培养2~3d后,在SC-leu固体培养基上划线分纯,至长出单菌落,挑取单菌落到SC-leu液体培养基,30℃、200r/min培养至OD600约为4,存甘油菌。该菌株命名为SyBE_Sc02050032。菌株信息详见表 1。
本研究中,合成柚皮素生产菌的外源基因有TAL、4CL、CHS和CHI,其中TAL的来源为圆红冬孢酵母(R.toruloides);4CL的来源为欧芹(Petroselinum crispum);CHI有6种来源,包括大豆(Glycine max)、橙子(Citrus sinensis)、蜜柑(Citrus unshiu)、拟南芥(Arabidopsis thaliana)、玉米(Zea mays)、花生(Arachis hypogaea);CHS有5种来源,包括拟南芥、葡萄(Vitis vinifera)、虎杖(Polygonum cuspidatum)、海洋细菌(Rhodopirellula baltica)、大豆。用于合成8DN的异戊烯基转移酶N8DT来源于苦参(Sophora flavescens)。上述基因按照酿酒酵母密码子偏好性进行优化并规避常用酶切位点,并在两端添加Bsa I酶切位点后交由金斯瑞公司合成。合成后的基因质粒与相应模块用Bsa I酶切,获得基因片段,纯化回收后经T4连接酶连接,得到相应的基因表达载体。
1.4 蛋白质截断的设计和构建通过在线工具(http://bioinf.cs.ucl.ac.uk/psipred)对N8DT蛋白序列进行分析,考察蛋白质的定位区域和disorder区域,通过该区域的截断策略改善蛋白质的异源表达优化。以已有PUC57-Sf_N8DT为模板,通过两组特异性引物(表 3)PCR扩增得到N端截断后的N8DT基因,命名为Sf_N8DTjie1~40和Sf_N8DTjie1~84,并与Module15质粒经过酶切连接后得到模块Module17和Module18(表 2)。将两个模块采用乙酸锂转化法分别导入SyBE_Sc02050031菌株中,经筛选分纯后,存甘油菌,命名为SyBE_Sc02050033、SyBE_Sc02050034。
| Primers name | Sequence |
| N8DTjie1-40-F | GGTCTCCATGGTACAAAAAGAGAAAGATCCAA |
| N8DTjie1-40-R | GGTCTCCTTTATCTGAATAAAGG |
| N8DTjie1-84-F | GGTCTCCAGTTGTCAATGCTATTTCCGAA |
| N8DTjie1-84-R | GGTCTCCTTTATCTGAATAAAGG |
1.5 人工酵母菌株发酵
柚皮素筛选菌株的发酵:将SyBE_Sc02050001~SyBE_02050030菌株接种于3ml种子培养基中,在30℃、220r/min培养24h,以初始菌体浓度OD600=0.2转接于新鲜的3ml种子培养基中,于30℃、220r/min条件下培养12h,以初始菌体浓度OD600=0.1分别接种于50ml SC四缺培养基(20g/L葡萄糖、6.7g/L YNB、2g/L drop-out的氨基酸混合物,固体培养基添加2%琼脂粉)中。培养条件为220r/min,30℃培养96h,发酵结束时测定菌株浓度OD600。
柚皮素整合菌株的发酵:SyBE_Sc02050031菌株的培养条件与上述菌株相同,发酵培养基选择YPD培养基(40g/L葡萄糖,20g/L胰蛋白胨,10g/L酵母提取物,固体培养基添加2%琼脂粉)。
8DN生产菌株的发酵:SyBE_Sc02050032、SyBE_Sc02050033和SyBE_Sc02050034菌株的培养条件与上述菌株相同,发酵培养基选取50ml Sc-leu(20g/L葡萄糖、6.7g/L YNB、2g/L drop-out去除异亮氨酸的氨基酸混合物、0.5g/L酪氨酸)。
1.6 柚皮素与8DN的提取和检测柚皮素与8DN提取:取600μl发酵液,加入等体积的乙酸乙酯和石英砂,置于涡旋振荡器上5 000r/min振荡30min后,以10 000r/min的速率离心10min,用枪头吸出上清液放置在干净的EP管中,重复3次。将收集好的上清液进行氮吹蒸发,大约20min后确认无液体残留停止,获得浅黄色固体。加入600μl色谱甲醇溶解,用2μm有机滤膜过滤后,装入进样小瓶中。
柚皮素与8DN的检测采用HPLC(Waters2489 UV/Vis Detector, Waters Corp.,USA)测定,色谱柱为Ascentis Express C18(10cm×2.1mm, 2.7μm, Thermo),流动相为甲醇和0.1%甲酸,比例随时间梯度变化。流速为0.2ml/min,检测波长290/310nm,柱温30℃,进样量10μl。
2 结果与分析 2.1 生产8DN酿酒酵母的构建在菌株构建中合成的模块都通过Not I酶切验证了正确性,如图 3(a)所示,其中前体柚皮素合成模块pRS414-CHS,pRS425K-TAL,pRS423-4CL,pRS416-CHI以及8DN合成模块pRS425K-Sf_N8DT质粒的酶切凝胶电泳条带都分别被标注。

|
| 图 3 柚皮素和8DN生产菌构建和发酵验证 Figure 3 The construction and the detected results of the strains producing naringenin and 8DN (a) The agarose gel electrophoresis identification of the genes by the restriction enzyme of Not I in this study. pRS414-CHS were indicated with Line 1-5 and the sources of CHS were Arabidopsis thaliana, Vitis vinifera, Polygonum cuspidatum, Rhodopirellula baltica and Glycine max, respectively; pRS425K-TAL from R.toruloides was indicated with Line 6; pRS423-4CL from Petroselinum crispum was indicated with Line 7; pRS416-CHI were indicated with Line 8-13 and the sources of CHI were Glycine max, Citrus sinensis, Citrus unshiu, Arabidopsis thaliana, Zea mays and Arachis hypogaea, respectively; wild type pRS425K-N8DT from Sophora flavescens was indicated with Line 14, and the two truncated N8DT, pRS425K-N8DTjie1-40 and pRS425K-N8DTjie1-84, were indicated with Line 15 and 16 (b) HPLC chrotomagraphy of the standard products of naringenin and 8DN, in addition of the yeast extracting solution of SyBe_sc02050031 and SyBe_sc02050032 (c) Naringenin production in shake flask (d) 8DN production of SyBe_sc02050031 and SyBe_sc02050032 in shake flask |
对生产8DN的人工酿酒酵母构建分为两部分,一是体内合成柚皮素路径的构建,二是异戊烯基转移酶基因N8DT的表达。其中柚皮素作为重要的前体物质[27],其体内合成各个模块的适配性是我们关注的重点,通过对不同来源的功能模块进行组合,最终我们得到了合成柚皮素的最适基因组合,并将它们通过Delta位点整合到酵母基因组上得到了高产柚皮素的稳定菌株SyBE_Sc02050031,该底盘菌不仅为后面的合成提供了前体物质柚皮素,同时为反应提供了适合的胞内环境,避免了饲喂柚皮素过程中底物利用率低,以及胞外高浓度柚皮素对该步反应的抑制等问题。
另外,为了最大限度提高N8DT基因的表达强度,我们选择将该基因连入pRS425K这一多拷贝质粒中[28-29],以此增加其表达量,将该质粒导入柚皮素生产菌SyBE_Sc02050031中,实现8DN在酿酒酵母中的合成。
2.2 柚皮素和8DN生产菌的摇瓶发酵定量和筛选柚皮素与8DN的检测与定量是通过使用高效液相色谱(HPLC)在一定测定条件下,测量出生产菌中目标产物的保留时间和峰面积,并与各自标准品进行比对,如图 3(b)所示。通过检测得到柚皮素与8DN的保留时间分别为8.32min和12.00min,它们的标准曲线为y=1 388.6x-499.87和y=25 196x+979.46。
经过不同来源的基因模块相互组合得到的30株柚皮素生产菌经过96h摇瓶发酵后,测定柚皮素产量如图 3(c)所示,产量从0.36mg/L到22.33mg/L动态分布,差异近62倍,其中SyBE_Sc02050003的摇瓶发酵产量最高,达到22.33mg/L。将基因组合优化构建的稳定的柚皮素生产菌SyBE_Sc02050031作为底盘细胞,导入Module 16质粒后得到8DN生产菌,8DN产量达到36.7μg/L,如图 3(d)所示。
2.3 N8DT基因优化对8DN产量的提高根据相关调研和Yazaki等进行的柚皮素饲喂实验结果可知,在保证足量柚皮素供应的条件下,柚皮素的转化率仅为1.9%,这说明大部分柚皮素并没有被N8DT酶催化生成8DN,由于该酶的低催化活性使得增加柚皮素的前体供应并不能很好的提高终产物产量。而通过对N8DT基因进行优化改造,改善其活性,有望大幅提高8DN的产量。
我们对N8DT蛋白的二级结构进行了预测(图 4a),结果显示:① 野生型的N8DT酶的N端1~40位氨基酸是信号肽序列。结合相关调研,我们发现该蛋白质的信号肽在植物体内定位在叶绿体膜上,而本研究中的底盘酿酒酵母中无叶绿体,所以将会产生错误定位,甚至影响异源蛋白N8DT的正确表达折叠。另外,N8DT的作用底物柚皮素主要存在于细胞质内,在酵母细胞中异源表达带有固有信号肽的N8DT可能不利于将柚皮素转化为8DN。② 野生型的N8DT酶的N端存在冗长的不稳定结构序列(红框所示的disordered氨基酸序列)。另外,考虑到85~86位氨基酸Val和Asn可折叠成稳定的sheet结构,所以截断N端前段明显的不稳定结构序列(1~84位氨基酸)可能有助于改善N8DT在异源表达体系中的折叠稳定性。根据以上的结果分析:我们设计了两种序列截断方案,一是敲除第1~40位氨基酸对应的信号肽序列,二是敲除第1~84位氨基酸对应的结构不稳定序列。为了进一步验证设计方案的可靠性,我们对1~40位氨基酸和1~84位氨基酸两种截断后的N8DT变异体序列进行了二级结构预测。如图 4(b)所示,N8DT蛋白N端1~40位氨基酸截断后的二级结构预测结果显示,未检测到定位的信号肽结构,说明该截断方案初步达到了设计效果。另外如图 4(c)所示,N8DT蛋白N端1~84位氨基酸截断后的二级结构预测结果显示,N8DT蛋白N端冗长的不稳定序列结构被明显去除,这可能对蛋白质在异源表达体系中的稳定折叠有利,因此我们也初步确立了该方案进行后续的研究。
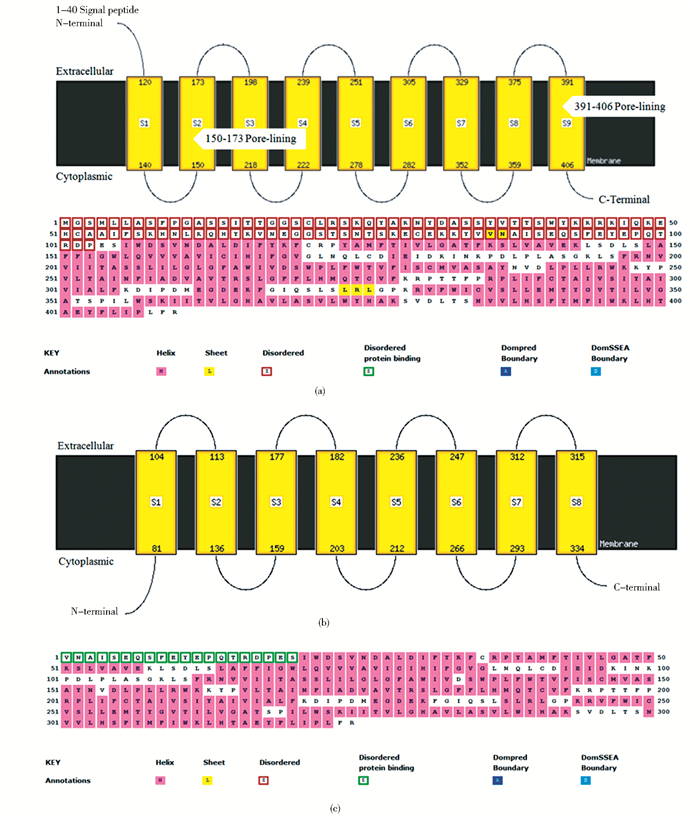
|
| 图 4 N8DT蛋白的二级结构预测和分析 Figure 4 The forecast and analysis of the secondary structure of N8DT (a) The prediction of the signal peptide for sublocalization and strutural stability of N8DT (b) The prediction of structural information for sublocalization of the N-terminal truncated N8DT at Ser40 (c) The analysis of the structural stability of the N-terminal truncated N8DT at Val84 |
我们设计的第一种优化策略是截断N8DT的N端信号肽序列,即1~40位氨基酸序列(图 4a、b),希望优化N8DT在酵母中的定位和表达,以此达到提高该步转化效率的目的。通过终产物8DN的产量变化对N8DT的N端信号肽截断策略进行了验证,结果显示,载入N端信号肽截断的N8DT(N8DTjie1~40) 表达质粒的8DN生产菌SyBe_sc02050033,经摇瓶发酵,8DN的产量提高到52.6μg/L,与载入野生型N8DT表达质粒的生产菌SyBe_sc02050032的8DN产量相比,提高了44.2%(图 5)。第二种优化策略根据蛋白质二级结构预测的稳定性分析,截断了N8DT蛋白N端不稳定的1~84位氨基酸序列(N8DTjie1~84)(图 4a、c)。与最初的8DN生产菌株SyBe_sc02050032的8DN产量相比,包含N8DTjie1-84表达质粒的8DN生产菌SyBe_sc02050034的8DN产量提高了19.1%,达到43.5μg/L(图 5)。
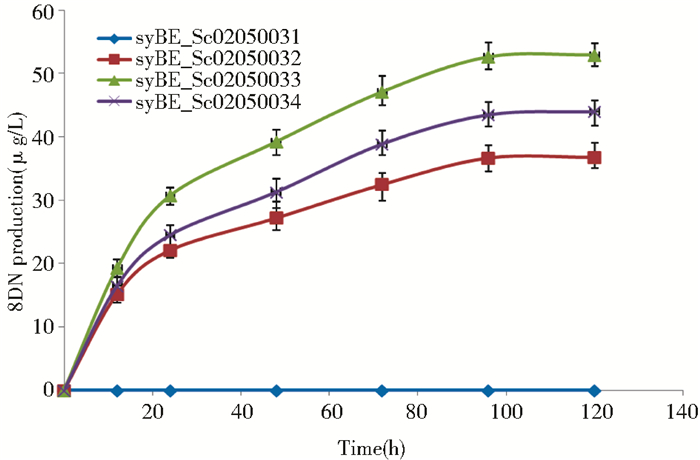
|
| 图 5 N8DT蛋白N端截断后的摇瓶发酵验证 Figure 5 8-dimenthylally naringenin production in shake flask after protein truncated test |
以上结果表明,两种N8DT的N端序列截断方案均起到了一定的优化效果,在不同程度上提高了该步催化反应的效率,达到了我们的预期目的,并且8DN的产量对比说明在酿酒酵母中N8DT酶的N端信号肽关联的定位结构是影响该酶活性的关键因素。对于N8DT酶的结构改造与功能的进一步深入分析,包括酶活力考察等工作,将是我们未来的一个研究工作重点。
3 结论本研究首先将不同来源的外源基因模块进行组合筛选,利用Delta位点将其整合至酵母基因组上,克服了多拷贝质粒容易丢失的问题,获得高产柚皮素的稳定的底盘菌。在底盘菌中进一步引入8DN的关键合成酶N8DT,并确认N8DT酶的低催化活性是重要限速步骤,然后基于蛋白质质二级结构分析提出了两种酶的序列优化截断策略:定位信号肽区域(1~40aa)和disorder区域(1~84aa),都取得了正向效果,促使8DN的产量从36.7μg/L分别提高到52.6μg/L(144.2%)和43.5μg/L(119.1%)。该研究通过关键外源基因的优化组合和关键酶的N端结构截断策略首次获得了高产8DN的在酿酒酵母菌株,对其他黄酮类天然产物的微生物合成体系构建具有重要的参照价值和借鉴意义。
| [1] |
Yan Y J, Kohli A, Koffas M A. Biosynthesis of natural flavanones in Saccharomyces cerevisiae. Applied and Environmental Microbiology, 2005, 71(9): 5610-5613. DOI:10.1128/AEM.71.9.5610-5613.2005 |
| [2] |
Chtourou Y, Aouey B, Aroui S, et al. Anti-apoptotic and anti-inflammatory effects of naringin on cisplatin-induced renal injury in the rat. Chemico-biological Interactions, 2016, 243: 1-9. DOI:10.1016/j.cbi.2015.11.019 |
| [3] |
Harborne J B, Williams C A. Advances in flavonoid research since 1992. Phytochemistry, 2000, 55(6): 481-504. DOI:10.1016/S0031-9422(00)00235-1 |
| [4] |
Maitrejean M, Comte G, Barron D, et al. The flavanolignan silybin and its hemisynthetic derivatives, a novel series of potential modulators of glycoprotein. Bioorg Med Chem Lett, 2000, 10(2): 157-160. DOI:10.1016/S0960-894X(99)00636-8 |
| [5] |
Sasaki K, Tsurumaru Y, Yazaki K. Prenylation of flavonoids by biotransformation of yeast expressing plant membrane-bound prenyltransferase SfN8DT-1. Biosci Biotechnol Biochem, 2009, 73(3): 759-761. DOI:10.1271/bbb.80729 |
| [6] |
Wu J, Du G, Chen J, et al. Enhancing flavonoid production by systematically tuning the central metabolic pathways based on a CRISPR interference system in Escherichia coli. Scientific Reports, 2015, 5: 13477. DOI:10.1038/srep13477 |
| [7] |
Zhang G, Liu S, Tan W, et al. Synthesis and biological evaluations of chalcones, flavones and chromenes as farnesoid x receptor (FXR) antagonists. Eur J Med Chem, 2017, 129(3): 303-309. |
| [8] |
李毅林. 一种天然产物淫羊藿苷类化合物的全合成方法: 中国, 200610165354. 2008-06-25. Li Y L, A Method of Total Synthesis for the Natural Product Lcariin Compound:Chinese, 200610165354. 2008-06-25. |
| [9] |
Geissman T A, David K F. Flavonones and related compounds. V. the oxidation of 2'-hydroqchalcones with alkaline hydrogen peroxide. J Am Chem Soc, 1948, 50(5): 1686-1689. |
| [10] |
Aurangzeb H, Sadipa A, Abbasa A, et al. Isolation and synthesis of flavonols and comparison of their antioxidant activity. Natural Product Research, 2010, 24(11): 995-1003. DOI:10.1080/14786410902847302 |
| [11] |
Fu M L, Wang W, Chen F, et al. Production of 8-phrenylnaringenin from isoxanthohumol through biotransformation by fungi cells. J Agric Food Chem, 2011, 59(13): 7419-7426. DOI:10.1021/jf2011722 |
| [12] |
Sasaki K, Tsurumaru Y, Yazaki K, et al. Molecular characterization of a membrane-bound prenyltransferase specific for isoflavone from Sophora flavescens. The Journal of Biological Chemistry, 2011, 286(8): 24125-24134. |
| [13] |
傅明亮, 刘婧, 陈苗苗, 等. RP-HPLC法同步检测酒花中黄腐酚、异黄腐酚与8异戊烯基柚皮素. 中国食品学报, 2010, 10(6): 193-198. Fu M L, Liu J, Chen M M, et al. Synchronous detected xanthohumol, isoxanthohumol and 8-prenylnaringenin in Humulus lupulus by RP-HPLC. Journal of Chinese Institute of Food Science and Technology, 2010, 10(6): 193-198. |
| [14] |
Vickers C E, Bongers M, Liu Q, et al. Metabolic engineering of volatile isoprenoids in plants and microbes. Plant, Cell & Environment, 2014, 37(8): 1753-1775. |
| [15] |
Fowler Z L, Koffas M A. Biosynthesis and biotechnological production of flavanones:current state and perspectives. Appl Microbiol Biotechnol, 2009, 83(5): 799-808. DOI:10.1007/s00253-009-2039-z |
| [16] |
Wang Y, Chen S, Yu O. Metabolic engineering of flavonoids in plants and microorganisms. Appl Microbiol Biotechnol, 2011, 91(4): 949-956. DOI:10.1007/s00253-011-3449-2 |
| [17] |
Limem I, Guedonc E, Hehn A, et al. Production of phenylpropanoid compounds by recombinant microorganisms expressing plant-specific biosynthesis genes. Process Biochem, 2008, 43: 463-479. DOI:10.1016/j.procbio.2008.02.001 |
| [18] |
Wilhelm H, Wessjohann L A. An efficient synthesis of the phytoestrogen 8-prenylnaringenin from xanthohumol by a novel demethylation process. Tetrahedron, 2006, 62(29): 6961-6966. DOI:10.1016/j.tet.2006.04.060 |
| [19] |
MacDonald I C, Deans T L. Tools and applications in synthetic biology. Adv Drug Deliv Rev, 2016, 105(A): 20-34. |
| [20] |
Gibson D G, Venter J C. Construction of a yeast chromosome. Nature, 2014, 509(5): 168-169. |
| [21] |
Sun J, Shao Z, Zhao H, et al. Cloning and characterization of a panel of constitutive promoters for applications in pathway engineering in Saccharomyces cerevisiae. Biotechnol Bioeng, 2012, 109(8): 2082-2092. DOI:10.1002/bit.24481 |
| [22] |
Fink G, Farabaugh P, Roeder G, et al. Transposable elements (Ty) in yeast. Cold Spring Harb Symp Quant Biol, 1981, 45(2): 575-580. |
| [23] |
Siddiqui M S, Thodey K, Trenchard I, et al. Advancing secondary metabolite biosynthesis in yeast with synthetic biology tools. Fems Yeast Res, 2012, 12(2): 144-170. DOI:10.1111/j.1567-1364.2011.00774.x |
| [24] |
Jiang H X, Karl V, John A. Metabolic engineering of the phenylpropanoid pathway in Saccharomyces cerevisiae. Applied and Environmental Microbiology, 2005, 71(6): 2962-2969. DOI:10.1128/AEM.71.6.2962-2969.2005 |
| [25] |
Lee F W, Da S N. Sequential delta-integration for the regulated insertion of cloned genes in Saccharomyces cerevisiae. Biotechnol Prog, 1997, 13(4): 368-373. DOI:10.1021/bp970055d |
| [26] |
Sasaki K, Tsurumaru Y, Yazaki K, et al. Cloning and characterization of naringenin 8-prenyltransferase, a flavonoid-specific prenyltransferase of Sophora flavescens. Plant Physiology, 2008, 146(3): 1075-1084. DOI:10.1104/pp.107.110544 |
| [27] |
Sugiyama A, Linley P J, Sasaki K, et al. Metabolic engineering for the production of prenylated polyphenols in transgenic legume plants using bacterial and plant prenyltransferases. Metabolic Engineering, 2011, 13(6): 629-637. DOI:10.1016/j.ymben.2011.07.003 |
| [28] |
Kim I K, Roldao A, Siewers V, et al. A systems-level approach for metabolic engineering of yeast cell factories. Fems Yeast Res, 2012, 12(2): 228-248. DOI:10.1111/j.1567-1364.2011.00779.x |
| [29] |
Shao Z, Zhao H, Zhao H. DNA assembler, an in vivo genetic method for rapid construction of biochemical pathways. Nucleic Acids Res, 2008, 37(2): e16. |
 2017, Vol. 37
2017, Vol. 37




