文章信息
- 李继彬, 陈志, 陈华友.
- LI Ji-bin, CHEN Zhi, CHEN Hua-you.
- 腈水解酶克隆表达、固定化及分子改造的研究进展
- Research Progress on Cloning, Expression, Immobilization and Molecular Modification of Nitrilase
- 中国生物工程杂志, 2017, 37(9): 141-147
- China Biotechnology, 2017, 37(9): 141-147
- http://dx.doi.org/DOI:10.13523/j.cb.20170919
-
文章历史
- 收稿日期: 2017-03-08
- 修回日期: 2017-04-19
腈水解酶(Nitrilase; EC 3.5.5.1) 是腈水解酶超级家族中的一种重要催化剂[1],它作为一种重要的工业酶能够直接将有毒腈化物转化为相应的无毒酸和氨,应用广泛[2-3]。但是,腈水解酶的应用开发仍然受到诸多不利因素的限制,如酶催化活力不够高、品种较少、稳定性较差、底物谱窄等问题。随着分子生物学的不断发展,通过对不同菌株腈水解酶基因进行克隆及表达,构建高效并且稳定的基因工程菌,并从分子水平上对酶分子进行改造以及酶的固定化,已成为了目前的研究热点。
1 腈水解酶的简介及来源腈水解酶广泛存在于自然界中,尤其是在丝状真菌、细菌、植物体中[4-6],在有机酸、氨基酸、维生素等化工产品和医药中间体的生物合成中具有重要应用[7-8]。在20世纪60年代,Thimann和Mahadevan[9]发现了第一个腈水解酶,在那之后科学家们发现了各种各样的腈水解酶并从各方面对其进行了研究,如利用芽孢表面展示技术对腈水解酶进行酶的固定化[10]、应用于多种化学品及医药中间体的合成等[11]。成功的案例包括德国巴斯夫公司生产的(R)-(-)-扁桃酸以及中国广州龙沙公司生产的烟酸,都达到了工业化的生产规模,由此也反映出腈水解酶的巨大应用前景[12-13]。
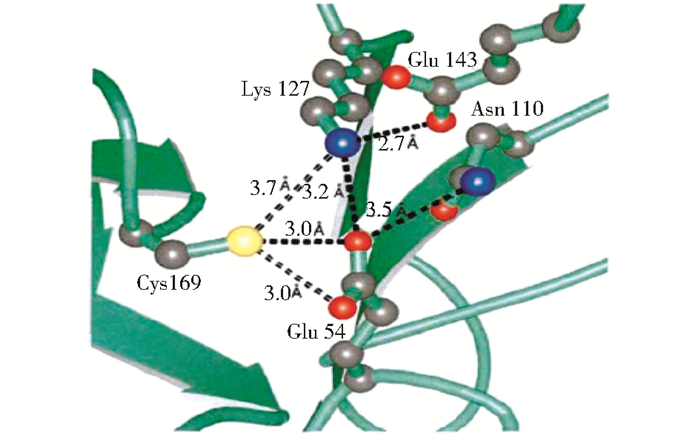
2 腈水解酶的结构及催化机制 2.1 腈水解酶的结构根据基因序列腈水解酶超家族可分为13个分支,其中有9个分支的底物特异性是可以预测的,但只有1个分支具备真正的腈水解酶活力[14]。2011年Raczynska等[15]成功获得了来源于火球菌(Pyrococcus abyssi)的腈水解酶PaNit的结晶体,此结构是首个真正具备腈水解酶活力的晶体结构。此外,还有一个最近被解析出来的来源于集胞藻(Synechocystis sp.)PCC6803的腈水解酶NIT6808[16]。Brenner等[1]的研究表明,腈水解酶的蛋白质结构是一种新型的α-β-β-α三明治折叠结构(图 1),在此三明治型酶蛋白折叠结构上有一个Glu-Lys-Cys的催化三联体结构,这个三联体结构在腈水解酶的活性位点中起着关键性作用[17]。另外,Sewell团队的研究表明,源自玫瑰红球菌(Rhodococcus rhodochrous)J1的腈水解酶具有三种同源低聚体结构形式:一个480kDa的复合体、一个二聚体及一个延伸的螺旋结构[18]。作者也建立了该腈水解酶的模型,依据模型可知此酶含有一个延伸的C端和两个重要的插入物,这两个插入物分散在螺旋低聚物之间并且使螺旋延长。同时,采用电子显微镜也观测到了这些螺旋同源低聚体的3D构型,结果表明,该3D构型和来自腈水解酶超级家族一员的P. stutzeri氰基双水合酶的结构基本一致[19]。此外,根据电子显微镜的观测结果发现,来自苍白地芽孢杆菌(Geobacillus pallidus)RAPc8的腈水解酶却展现出了不同的结构形式,包括C形、月牙形、8字形和圆形[20]。另外,远紫外圆二色谱也被运用来检测来自茄病镰刀菌(Fusarium solani)IMI 196840和F. solani O1腈水解酶的蛋白质二级结构[21],结果显示它们的二级结构比较相似,都包括21%的β折叠、16%的转角、30%的α螺旋及33%的其他结构。
尽管对于腈水解酶有关的序列信息、在环境中的分布情况以及底物特异性等知识的了解越来越多,但是目前有关微生物腈水解酶的晶体结构的报道仍然很少。由于缺乏有关这些酶的具体结构信息,故对腈水解酶的蛋白质结构缺乏深入的了解或许是未来分子操作的一个主要障碍。因此,随着分子操作技术的不断成熟,对腈水解酶的蛋白质结构测定变得越来越重要。
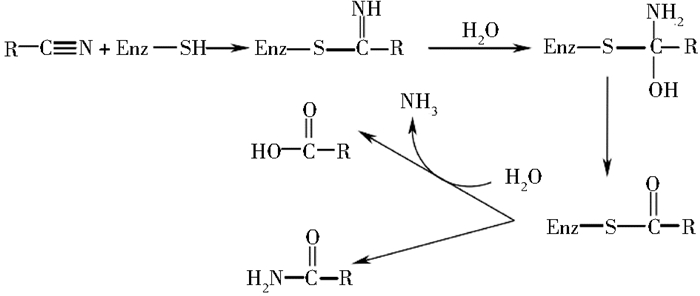
2.2 腈水解酶的催化机制到目前为此,腈水解酶介导的生物催化反应的催化机制已被多次研究与讨论[22]。在1964年,Thimann和Mahadevan[9]发现了第一个腈水解酶,并推测了其可能的催化机制。该团队认为腈化物上的腈基碳原子受到腈水解酶上巯基的亲核攻击,所生成的亚胺接着被水解为相应的酮并产生了酰胺副产物,通过加入1分子H2O,酰基-酶的复合物随后被水解,最终形成了羧酸并重新释放出酶分子。也在同一年科学家Robinson和Hook[23]针对蓖麻碱腈水解酶介导的蓖麻碱的水解反应也提出了相似的机制,并且该机制(图 2)受到了本领域专家学者的广泛认可[24-25]。然而,最近一项研究发现,在腈水解酶介导腈化物水解的过程中也会形成酰胺化合物。德国科学家Piotrowski等[26]研究发现,来自拟南芥的腈水解酶具有较高的腈水合酶的活性,此酶能够将底物β-氰基-L-丙氨酸转化为天冬酰胺和天冬氨酸,并且天冬酰胺的生成率高达60%以上。因此,作者推测这个结果可能是由于此酶具有一种新型的腈水合酶活性所导致的,而这种催化机制也不同于传统的腈水解酶反应机制。Jenrich等[27]发现Poaceae腈水解酶(NIT4) 的活性完全来源于异源复合物,这与其他的植物腈水解酶是显然不同的,这种异源复合物是由两个不同形式的NIT4组成的,分别为NIT4A和NIT4B。Stevenson等[28]还发现在来源于红球菌(Rhodococcus)ATCC 39484的腈水解酶介导的生物催化反应中,底物上的芳香环与之结合会导致腈水解酶的聚集交联,通过47kDa单体的聚集形成560kDa的复合体来激活酶分子。此外,在R. rhodochrous J1腈水解酶当中也观察到了这样的情况,大量的盐溶液和有机溶剂也能够促进这种聚集作用,在腈水解酶聚集过程中酶的存在形式是很不稳定的,只能在二聚体或十倍体状态下才能够检测到腈水解酶的活性[29]。
对腈水解酶的催化机制缺乏足够的认识是进行后续分子改造的主要障碍。测定精确的蛋白质晶体结构,并探究其详尽的催化机制在今后的腈水解酶研究中变得尤为重要[30]。
3 腈水解酶的克隆及其表达到目前为止,已有许多腈水解酶基因被成功克隆出来并且在合适的宿主中进行了表达。例如,在1987年,Stalker和Mcbride[31]克隆到了编码溴苯腈(一种除草剂)的腈水解酶基因bxn,他们将这个基因连接到pUC18载体上并转入到大肠杆菌(Escherichia coli)71-18中成功进行了表达。不久之后,从拟南芥中获取的第一个植物腈水解酶也得到了鉴定,研究人员获得了此酶的cDNA序列,并且进行了测序、表征和功能表达[32]。此外,Pekarsky等[33]也从果蝇和杆状线虫中克隆和表征了肿瘤抑制基因,结果显示其融合蛋白与植物和细菌腈水解酶具有一定的同源性。Kobayashi等[34]克隆了来自粪产碱菌(Alcaligenes faecalis)JM3的腈水解酶基因并在大肠杆菌E. coli中进行了活性表达。同时,该研究也证明Cys163残基对腈水解酶的催化活性起关键作用这一假说。另外,来源于睾丸酮丛毛单胞菌(Comamonas testosteroni sp.)和R. rhodochrous K22的腈水解酶的相关实验结果也验证了这个假说[35-36]。Zhu等[37]从一株慢生型大豆根瘤菌(Bradyrhizobium japonicum)USDA 110克隆了一个新的腈水解酶基因blr3397, 并在大肠杆菌中进行了表达,结果显示,该腈水解酶对苯代丙腈表现出较高的特异性。此外,对来源于极端嗜热菌的腈水解酶也进行了克隆和表达。例如,Chen等[38]克隆和表征了来自超嗜热细菌海栖热袍菌(Thermotoga maritima)MSB8的新型腈水解酶基因,实验证明该酶表现出良好的耐热性,在75℃热处理60min后仍然具有40%的残留活性。除此之外,Mueller及其研究团队[39]也克隆及表达了来自P. abyssi的腈水解酶,结果表明该重组酶具有极高的高温耐受性,90℃的半衰期为6h。除对细菌和植物的研究之外,科学家们也对来自真菌的腈水解酶基因进行了相关研究。最近,Kaplan等[40]从黑曲霉(Aspergillus niger)K10 cDNA文库中克隆到了一株野生型的A. niger K10真菌腈水解酶基因,接着把该基因连接到pRSET B和pET-30a(+)表达载体上,最后进行E. coli BL21-Gold(DE3) (pOK101/pTf16) 原核表达及酶活测试,结果显示该重组腈水解酶的比活力提高了2倍。另外,Yusuf等[41]通过逆转录PCR成功克隆了来自镰刀菌(Fusarium proliferatum)AUF-2的真菌腈水解酶基因,这也是对来自镰刀菌的腈水解酶进行克隆及异源表达的第一次报道。
在众多种类腈水解酶的研究中,催化二腈转化的腈水解酶研究是非常重要的。因为这些反应的产物通常是氰基羧酸,它是用于合成药物和化学品的重要的结构单元[42]。目前,利用真菌腈水解酶选择性水解二腈的潜力还有待进一步挖掘。所以,对真菌腈水解酶基因进行表达及克隆是今后科学家对腈水解酶进行研究的热点之一。
4 腈水解酶的分子改造随着基因工程技术的迅猛发展,通过对腈水解酶进行分子改造可以显著提高酶的活性、稳定性及底物特异性等性能。酶分子改造的常用方法主要有理性的定点突变技术及非理性的定向诱变技术。理性的改造技术首先需要弄清楚腈水解酶的氨基酸序列、空间结构和催化机制等,然后对酶分子进行合理设计和改造,最终获得性能显著提升的突变体;而非理性的改造技术并不需要事先清楚腈水解酶的空间结构和催化机制,只需要进行体外的生物酶基因改造,之后再设定某种进化条件,最终再利用高通量的筛选方法获取性能提升的突变体。
4.1 定向进化定向进化是近年来快速发展的一种有效的分子生物学技术,它在修饰和改造如酶的热稳定性、底物耐受性等方面取得了一定效果。其主要包括两个步骤:① 通过DNA重组或EP-PCR等手段建立随机突变文库;② 利用各种化学检测方法筛选以获取阳性突变株。例如,Schreiner等[43]利用EP-PCR方法获取了一个A. faecalis JM3腈水解酶优良的突变株,其在pH 7.5的条件下对苯丙腈比酶活提高了8倍,序列分析显示该突变体共包含7处突变。除此之外,他们也获得了另一个突变体,该突变体在pH为4.5的条件下仍具有较高的活性,10min即能够完全将(R)-2-氯-扁桃腈转化为(R)-2-氯-扁桃酸,其对映体过量值超过99%。
4.2 定点突变在近期的一些研究中,利用定点突变方法从基因水平修饰腈水解酶受到了许多科学家的关注[22]。点突变,甚至是单个氨基酸的突变,都有可能会对底物特异性、选择性、比酶活及酰胺的形成等产生极大的影响。例如,在玫瑰红球菌(R. Rhodochrous)ATCC 33278腈水解酶中有一个对底物特异性起决定作用的氨基酸Tyr142,该位置的氨基酸残基突变成为非极性的脂肪族氨基酸之后,此突变体只对芳香族的腈化物有作用,而对脂肪族的腈化物没有作用,这可能是由于底物的水解需要腈化物或Tyr142残基上共轭的π-电子云系统所致[44]。荧光假单胞菌(Pseudomonas fluorescens)EBC191腈水解酶中的氨基酸Ala165和Cys163位于活性中心半胱氨酸残基的附近,经证实它们对腈水解酶的底物对映选择性及酰胺的形成起决定作用。特别是突变C163Q明显增加了酰胺的形成,并且降低了对(R)-扁桃腈的水解活力。除此之外,47~67C端氨基酸的缺失也导致了酶活力的降低、酰胺生成量的增加及对映选择性的改变[19]。因此,研究人员整合了这些突变并构建了组合突变体,研究结果显示此举可以使扁桃酰胺的形成量提高1.5倍,并且组合突变体在所有的P. fluorescens EBC191腈水解酶突变体中显示出了最高的水合酶活性[45]。另外,Petǐíková等[46]最近也鉴定了粗糙脉胞菌(Neurospora crassa)的一株突变体W168A,与野生型相比,突变株的酰胺生成量较高,对映体过量值却相对降低。
目前,虽然利用定向进化或定点突变等技术对腈水解酶进行分子改造已经取得了一些重要成果,但应用于产业化的腈水解酶还相对较少,所以还需要不断努力利用现有技术如用蛋白质工程改善酶或者开发新技术,以获取更多优秀的腈水解酶。
5 腈水解酶的固定化固定化技术主要包括细胞的固定化及游离酶的固定化。据目前的研究报道,绝大多数腈类化合物生物催化都是采用固定化后的生物催化剂进行的,直接采用游离细胞进行生物催化的工艺相对较少。例如,Kumar等[47]利用共价交联聚合的方式对恶臭假单胞菌(Pseudomonas putida)腈水解酶进行了固定化,研究表明固定化后的P. Putida腈水解酶能够在4℃的条件下稳定存在75h, 然而游离酶在此温度条件下保存75h活力却降低了90%。另外,Nigam等[48]以琼脂-琼脂作为基质固定化链霉菌(Streptomyces)腈水解酶用来进行丙烯腈的生物催化反应,利用该方法固定化后的腈水解酶酶活力丧失很少,仅丧失约20%,并可以重复使用25次。利用海藻酸钙对A. faecalis腈水解酶进行固定化也可以提高其循环利用率,该方法固定后的酶在连续使用30个循环后酶活力几乎没有丧失,而游离细胞却仅能够使用9个循环;除此之外,利用聚乙烯亚胺和戊二醛交联藻酸钙微珠能够使循环利用效率进一步提高到40次反应[49]。
各种各样的腈水解酶固定化材料和固定方法的报道较多,但是这些固定化方式普遍成本较高,工艺操作烦琐,因此急需新型的酶固定化方式。近年来,表面展示技术作为一种新型且有效的固定化方式已经得到了科学家们的广泛关注,现已广泛应用于生物催化剂、疫苗和生物传感等方面。在众多的展示系统中,细菌芽孢表面展示系统关注度最高而且应用也最广泛[50]。枯草杆菌芽孢具有较高的稳定性和抗逆性,可以使芽孢成为一个优秀的固定化载体并在其表面展示耐逆化工酶,这也为以后耐逆酶固定化研究指明了方向。
6 展望生物催化途径具有反应条件温和、催化效率高、能耗低、整体工艺绿色等优点,符合我国绿色发展、和谐发展的要求。因此,实现“酶尽其用”,对优良腈水解酶进行开发符合国家的战略需求。目前,虽然对于腈水解酶的研究取得了一些成果。但是,对于腈水解酶的应用仍然受到许多因素的制约,如腈水解酶的种类较少、稳定性较差、比酶活不够高等问题。所以,深入挖掘腈水解酶的应用能力迫在眉睫。目前,腈水解酶未来应有如下几个研究发展方向:① 新酶的筛选:数据库基因组挖掘;环境宏基因组库的筛选;② 酶分子的改造:定向进化,定点突变;③ 酶的固定化:最值得一提的是以芽孢衣壳蛋白作为融合伴侣,表面展示不同的耐逆酶来进行酶的固定化研究。例如,通过优化衣壳蛋白CotG与腈水解酶基因nit之间的连接肽选出较合适的连接肽,这样可以得到活性或耐逆性较好的腈水解酶,为腈水解酶的大规模产业化应用打下基础。
| [1] |
Brenner C. Catalysis in the nitrilase superfamily. Current Opinion in Structural Biology, 2002, 12(6): 775-782. DOI:10.1016/S0959-440X(02)00387-1 |
| [2] |
Mathew S, Nadarajan S P, Sundaramoorthy U, et al. Biotransformation of β-keto nitriles to chiral (S)-β-amino acids using nitrilase and ω-transaminase. Biotechnol Lett, 2017, 39(4): 534-543. |
| [3] |
Luo H, Ma J, Chang Y, et al. Directed evolution and mutant characterization of nitrilase from Rhodococcus rhodochrous, tg1-A6. Applied Biochemistry and Biotechnology, 2016, 178(8): 1510-1521. DOI:10.1007/s12010-015-1964-9 |
| [4] |
Banerjee A, Sharma R, Banerjee U C. The nitrile-degrading enzymes:current status and future prospects. Applied Microbiology and Biotechnology, 2002, 60(1-2): 33-44. DOI:10.1007/s00253-002-1062-0 |
| [5] |
Martinkova L, Vejvoda V, Kaplan O, et al. Fungal nitrilases as biocatalysts:Recent developments. Biotechnology Advances, 2009, 27(6): 661-670. DOI:10.1016/j.biotechadv.2009.04.027 |
| [6] |
Lehmann T, Janowitz T, Sánchez-Parra B, et al. Arabidopsis nitrilase I contributes to the regulation of root growth and development through modulation of auxin biosynthesis in seedlings. Frontiers in Plant Science, 2017, 8(36): 1-15. |
| [7] |
龚劲松, 李恒, 谢旻峰, 等. 重组真菌腈水解酶的发酵工艺条件及生物催化特性初探. 精细化工, 2015, 32(10): 1112-1119. Gong J S, Li H, Xie M F, et al. Studies on fermentation conditions and biocatalytic properties of recombinant fungi nitrilase. Fine Chemicals, 2015, 32(10): 1112-1119. |
| [8] |
薛媛, 薛亚平, 郑裕国. 区域选择性腈水解酶在化学品合成中的应用. 精细与专用化学品, 2016, 24(5): 25-31. Xue Y, Xue Y P, Zheng Y G. Application of regioselectivity nitrilase in synthesis of chemicals. Fine & Specialty Chemicals, 2016, 24(5): 25-31. |
| [9] |
Thimann K V, Mahadevan S. Nitrilase.I.occurrence, preparation, and general properties of the enzyme. Archives of Biochemistry & Biophysics, 1964, 105(1): 133-141. |
| [10] |
Chen H, Chen Z, Ni Z, et al. Display of Thermotoga maritima, MSB8 nitrilase on the spore surface of Bacillus subtilis, using out coat protein CotG as the fusion partner. Journal of Molecular Catalysis B Enzymatic, 2016, 123(JAN): 73-80. |
| [11] |
Dennett G V, Blamey J M. A New Thermophilic nitrilase from an antarctic hyperthermophilic microorganism. Frontiers in Bioengineering & Biotechnology, 2016, 4(5): 1-9. |
| [12] |
Nicholas M S, Karen T R, Andreas K. Lonza:20 years of biotransformations. Cheminform, 2003, 34(27): 425-435. |
| [13] |
Schmid A, Dordick J S, Hauer B, et al. Industrial biocatalysis today and tomorrow. Nature, 2001, 409(6817): 258-268. DOI:10.1038/35051736 |
| [14] |
Pace H C, Brenner C. The nitrilase superfamily:classification, structure and function. Genome Biology, 2001, 2(1): 1-9. |
| [15] |
Raczynska J E, Vorgias C E, Antranikian G, et al. Crystallographic analysis of a thermoactive nitrilase. Journal of Structural Biology, 2011, 173(2): 294-302. DOI:10.1016/j.jsb.2010.11.017 |
| [16] |
Zhang L, Yin B, Wang C, et al. Structural insights into enzymatic activity and substrate specificity determination by a single amino acid in nitrilase from Syechocystis sp. PCC6803. Journal of Structural Biology, 2014, 188(2): 93-101. DOI:10.1016/j.jsb.2014.10.003 |
| [17] |
Banerjee A, Sharma R, Banerjee U C. A rapid and sensitive fluorometric assay method for the determination of nitrilase activity. Biotechnology & Applied Biochemistry, 2003, 37(3): 289-293. |
| [18] |
Pace H C, Hodawadekar S C, Draganescu A, et al. Crystal structure of the worm NitFhit Rosetta Stone protein reveals a Nit tetramer binding two Fhit dimers. Current Biology, 2000, 10(15): 907-917. DOI:10.1016/S0960-9822(00)00621-7 |
| [19] |
Kiziak C, Klein J, Stolz A. Influence of different carboxy-terminal mutations on the substrate, reaction-and enantiospecificity of the arylacetonitrilase from Pseudomonas fluorescens EBC191. Protein Engineering Design & Selection Peds, 2007, 20(8): 385-396. |
| [20] |
Williamson D S, Dent K C, Weber B W, et al. Structural and biochemical characterization of a nitrilase from the thermophilic bacterium, Geobacillus pallidus, RAPc8. Applied Microbiology and Biotechnology, 2010, 88(1): 143-153. DOI:10.1007/s00253-010-2734-9 |
| [21] |
Vejvoda V, Kubac D, Davidova A, et al. Purification and characterization of nitrilase from Fusarium solani IMI196840. Process Biochem, 2010, 45(7): 1115-1120. DOI:10.1016/j.procbio.2010.03.033 |
| [22] |
陈志. 海栖热袍菌腈水解酶表达鉴定及在枯草杆菌芽孢表面展示. 镇江: 江苏大学, 生命科学研究院, 2016. Chen Z. Expression and Characterization of Thermotoga maritima MSB8 Nitrilase and Its Surface Display on Bacillus subtilis spore. Zhen Jiang:Life Science Research Institute, Jiangsu University, 2016. http://cdmd.cnki.com.cn/Article/CDMD-10299-1016727716.htm |
| [23] |
Hook R H, Robinson W G. Ricinine nitrilase.purification and properties. Journal of Biological Chemistry, 1965, 239(12): 4263-4267. |
| [24] |
Banerjee A, Sharma R, Banerjee U C. The nitrile-degrading enzymes:current status and future prospects. Applied Microbiology and Biotechnology, 2002, 60(1-2): 33-44. DOI:10.1007/s00253-002-1062-0 |
| [25] |
Chen J, Zheng R C, Zheng Y G, et al. Microbial transformation of nitriles to high-value acids or amides. Advances in Biochemical Engineering/Biotechnology, 2009, 113(35): 33-77. |
| [26] |
Piotrowski M, Schonfelder S, Weiler E W. The Arabidopsis thaliana isogene NIT4 and its orthologs in tobacco encode beta-cyano-L-alanine hydratase/nitrilase. Journal of Biological Chemistry, 2001, 276(4): 2616-2621. DOI:10.1074/jbc.M007890200 |
| [27] |
Jenrich R, Trompetter I, Bak S, et al. Evolution of heteromeric nitrilase complexes in Poaceae with new functions in nitrile metabolism. Proceedings of the National Academy of Sciences of the United States of America, 2007, 104(47): 18848-18853. DOI:10.1073/pnas.0709315104 |
| [28] |
Stevenson D E, Feng R, Dumas F, et al. Mechanistic and structural studies on Rhodococcus ATCC 39484 nitrilase. Biotechnology & Applied Biochemistry, 1992, 15(3): 283-302. |
| [29] |
Nagasawa T, Wieser M, Nakamura T, et al. Nitrilase of Rhodococcus rhodochrous J1-Conversion into the active form by subunit association. Eur J Biochem, 2000, 267(1): 138-144. DOI:10.1046/j.1432-1327.2000.00983.x |
| [30] |
郝劲松. Gibberella intermedia腈水解酶的克隆、鉴定及分子改造. 无锡: 江南大学, 生物工程学院, 2013. Hao J S. Cloning, Identification and Molecular Modification of Gibberella intermedia Nitrilase. Wuxi:Jiangnan University, College of Bioengineering, 2013. http://cdmd.cnki.com.cn/Article/CDMD-10295-1014148906.htm |
| [31] |
Stalker D M, Mcbride K E. Cloning and expression in Escherichia coli of a Klebsiella ozaenae plasmid-borne gene encoding a nitrilase specific for the herbicide bromoxynil. Journal of Bacteriology, 1987, 169(3): 955-960. DOI:10.1128/jb.169.3.955-960.1987 |
| [32] |
Bartling D, Seedorf M, MithÖFer A, et al. Cloning and expression of an Arabidopsis nitrilase which can convert indole-3-acetonitrile to the plant hormone, indole-3-acetic acid. European Journal of Biochemistry, 1992, 205(1): 417-424. DOI:10.1111/ejb.1992.205.issue-1 |
| [33] |
Pekarsky Y, Campiglio M, Siprashvili Z, et al. Nitrilase and fhit homologs are encoded as fusion proteins in Drosophila melanogaster and Caenorhabditis elegans. Proceedings of the National Academy of Sciences of the United States of America, 1998, 95(15): 8744-8749. DOI:10.1073/pnas.95.15.8744 |
| [34] |
Kobayashi M, Izui H, Nagasawa T, et al. Nitrilase in biosynthesis of the plant hormone indole-3-acetic acid from indole-3-acetonitrile:cloning of the Alcaligenes gene and site-directed mutagenesis of cysteine residues. Proceedings of the National Academy of Sciences of the United States of America, 1993, 90(1): 247-251. DOI:10.1073/pnas.90.1.247 |
| [35] |
LevySchil S, Soubrier F, Crutz-Le Coq A M, et al. Aliphatic nitrilase from a soil-isolated Comamonas testosteroni sp.:gene cloning and overexpression, purification and primary structure. Gene, 1995, 161(1): 15-20. DOI:10.1016/0378-1119(95)00242-X |
| [36] |
Kobayashi M, Yanaka N, Nagasawa T, et al. Primary structure of an aliphatic nitrile-degrading enzyme, aliphatic nitrilase, from Rhodococcus rhodochrous K22 and expression of its gene and identification of its active site residue. Biochemistry, 1992, 31(37): 9000-9007. DOI:10.1021/bi00152a042 |
| [37] |
Zhu D, Mukherjee C, Yang Y, et al. A new nitrilase from Bradyrhizobium japonicum USDA 110. Gene cloning, biochemical characterization and substrate specificity. Journal of Biotechnology, 2008, 133(3): 327-333. DOI:10.1016/j.jbiotec.2007.10.001 |
| [38] |
Chen Z, Chen H, Ni Z, et al. Expression and characterization of a novel nitrilase from hyperthermophilic bacterium Thermotoga maritima MSB8. Journal of Microbiology & Biotechnology, 2015, 25(10): 1660-1669. |
| [39] |
Mueller P, Egorova K, Vorgias C E, et al. Cloning, overexpression, and characterization of a thermoactive nitrilase from the hyperthermophilic archaeon Pyrococcus abyssi. Protein Expression & Purification, 2006, 47(2): 672-681. |
| [40] |
Kaplan O, Bezouska K, Plihal O, et al. Heterologous expression, purification and characterization of nitrilase from Aspergillus niger K10. Bmc Biotechnol, 2011, 11(32): 1-15. |
| [41] |
Yusuf F, Jamwal U, Chaubey A, et al. Cloning and functional characterization of nitrilase from Fusarium proliferatum AUF-2 for detoxification of nitriles. Functional & Integrative Genomics, 2015, 15(4): 413-424. |
| [42] |
Veselá A B, Rucká L, Kaplan O, et al. Bringing nitrilase sequences from databases to life:the search for novel substrate specificities with a focus on dinitriles. Applied Microbiology and Biotechnology, 2016, 100(5): 1-10. |
| [43] |
Schreiner U, Hecher B, Obrowsky S, et al. Directed evolution of Alcaligenes faecalis nitrilase. Enzyme & Microbial Technology, 2010, 47(4): 140-146. |
| [44] |
Yeom S J, Kim H J, Lee J K, et al. An amino acid at position 142 in nitrilase from Rhodococcus rhodochrous ATCC 33278 determines the substrate specificity for aliphatic and aromatic nitriles. Biochemical Journal, 2008, 415(3): 401-407. DOI:10.1042/BJ20080440 |
| [45] |
Sosedov O, Baum S, Burger S, et al. Construction and application of variants of the Pseudomonas fluorescens EBC191 arylacetonitrilase for increased production of acids or amides. Applied & Environmental Microbiology, 2010, 76(11): 3668-3674. |
| [46] |
Petǐíčková A, Sosedov O, Baum S, et al. Influence of point mutations near the active site on the catalytic properties of fungal arylacetonitrilases from Aspergillus niger, and Neurospora crassa. Journal of Molecular Catalysis B Enzymatic, 2012, 77(9): 74-80. |
| [47] |
Kumar S, Mohan U, Kamble A L, et al. Cross-linked enzyme aggregates of recombinant Pseudomonas putida nitrilase for enantioselective nitrile hydrolysis. Bioresource Technology, 2010, 101(17): 6856-6858. DOI:10.1016/j.biortech.2010.03.084 |
| [48] |
Nigam V K, Khandelwal A K, Gothwal R K, et al. Nitrilase-catalysed conversion of acrylonitrile by free and immobilized cells of Streptomyces sp. Journal of Biosciences, 2009, 34(1): 21-26. DOI:10.1007/s12038-009-0005-7 |
| [49] |
Kaul P, Banerjee A, Banerjee U C. Stereoselective nitrile hydrolysis by immobilized whole-cell biocatalyst. Biomacromolecules, 2006, 7(5): 1536-1541. DOI:10.1021/bm0507913 |
| [50] |
Chen H, Chen Z, Wu B, et al. Influences of various peptide linkers on the Thermotoga maritima MSB8 nitrilase displayed on the spore surface of Bacillus subtilis. Journal of Molecular Microbiology & Biotechnology, 2017, 27(1): 64-71. |
 2017, Vol. 37
2017, Vol. 37