文章信息
- 宋佳雯, 田苏, 张玉如, 王志珍, 常忠义, 高红亮, 步国建, 金明飞.
- SONG Jia-wen, TIAN Su, ZHANG Yu-ru, WANG Zhi-zhen, CHANG Zhong-yi, GAO Hong-liang, BU Guo-jian, JIN Ming-fei.
- 基因组重排筛选高产谷氨酰胺转胺酶菌株
- Genome Shuffling Enhances Transglutaminase Production of Streptomyces mobaraensis
- 中国生物工程杂志, 2017, 37(9): 105-111
- China Biotechnology, 2017, 37(9): 105-111
- http://dx.doi.org/DOI:10.13523/j.cb.20170914
-
文章历史
- 收稿日期: 2017-02-28
- 修回日期: 2017-05-18
2. 泰兴市东圣食品科技有限公司 泰兴 225411
2. Taixing Dongsheng Food Co. Ltd, Taixing 225411, China
谷氨酰胺转胺酶(transglutaminase,TGase,EC2.3.2.13) 是一种催化酰基转移反应的转移酶,能够将蛋白质中谷氨酰胺残基的γ-酰胺基转移到伯胺基团、赖氨酸残基的ε-氨基或者水分子上,使蛋白质发生交联,改变蛋白质的乳化性、溶解性、稳定性、弹性、凝胶强度、黏性等性质[1]。
1989年,Ando等[2]筛选到一株茂源链霉菌(Streptomyces mobaraensis)并发现其能胞外分泌TGase。由于微生物发酵产酶的原料便宜、生产周期短、纯化工艺简捷、得率高,且得到的TGase无Ca2+依赖性,目前成为市场的主流。中国、日本、荷兰等国都对微生物发酵产TGase的技术进行改善,但是目前的核心问题是菌株普遍产酶量较低,需要更加高产的菌株,因此对茂源链霉菌高产菌株的选育非常重要[3]。
诱变育种是目前国内外工业微生物育种中最常使用的育种技术[4]。对茂源链霉菌的诱变育种,目前已有紫外诱变(UV)[5-6]、亚硝基胍诱变(NTG)[7]、硫酸二乙酯(DES)[8]、大气压辉光放电低温等离子体诱变[9]以及最新的常压室温等离子体诱变(ARTP)[10]等技术被应用,并对谷氨酰胺转胺酶的产量有一定提升。但是也存在一些问题,首先,物理诱变和化学诱变会杀死细胞,诱导癌变对人体有一定危害[10]。其次,诱变育种为非定向性育种,一次需筛选上万菌株才能提高产量[6]。最后,由于诱变后只选择酶活最高的一个突变株,大量酶活提高的突变菌株的诱变突变得不到利用,非常浪费,多个诱变综合作用下酶活可能会有更大的提高[11]。
2002年,Zhang等[12]的研究显示基因组重排育种相比随机诱变能够大幅提高筛选效率。通过建立突变子库,将突变菌株制备原生质体并融合,使基因发生重组从而提高酶活,再不断选择酶活提高的菌株作为下一轮的突变子库,减小发生负突变的可能性[13]。这种方法目前已经应用于吸水链霉菌(Streptomyces hygroscopicus)产雷帕霉素[11]、褐黄孢链霉菌(Streptomyces gilvosporeus)产那他霉素[14]、绿色产生链霉菌(Streptomyces viridochromogenes)产阿维霉素[15]等生产菌株的育种中,并成功提高了产量。赵文超[16]曾尝试将该方法应用在产谷氨酰胺转胺酶的茂源链霉菌育种领域,但是获得的高产菌株只比亲本菌株的产量提高5%,并没体现出该方法相对传统诱变的优势。
本实验室通过多种诱变方法,获得了大量的突变子,也对茂源链霉菌的原生质体制备和融合[17]的方法进行了一定探究,为本文的研究建立基础。本文以这些突变子构建突变子库,对原有的方法进行改进,并应用在筛选高产谷氨酰胺转胺酶的茂源链霉菌中,提高谷氨酰胺转胺酶的产量。
1 材料与方法 1.1 材料菌株:茂源链霉菌(Streptomyces mobaraensis),由华东师范大学生命科学学院微生物实验室分离保存。
试剂:溶菌酶、PEG-2000购自美国Sigma公司,蛋白胨购自美国BD公司,其余药品均购自国药集团化学试剂有限公司,且都为分析纯。
培养基:高氏一号培养基、YEME培养基,再生培养基,种子、发酵培养基参考侯孝仑等[18]的配方。
仪器:冷冻离心机购自美国Sigma公司;酶标仪购自美国Biotek公司;相差显微镜购自日本Olympus公司;恒温水浴锅、恒温培养箱购自上海精宏实验设备有限公司;超净工作台购自上海净化设备有限公司;分光光度计购自上海元析仪器有限公司;恒温调速摇床购自上海知楚仪器有限公司。
1.2 原生质体制备条件优化 1.2.1 原生质体制备方法将在高氏一号培养基上生长的茂源链霉菌刮下孢子,接入YEME培养基中,接种量约为108,于30℃、200r/min条件下培养36h。取培养的菌丝体,用高渗溶液配制的溶菌酶酶解菌丝体,30℃水浴,取样于相差显微镜下观察,待酶解完全,用棉花过滤去除少量的菌丝,离心去除溶菌酶液,加入高渗溶液重悬原生质体。
1.2.2 原生质体制备酶浓度优化用高渗溶液配制2mg/ml、6mg/ml、10mg/ml的溶菌酶处理菌丝体,计算不同浓度溶菌酶酶解后得到的原生质体纯度和形成率。
1.2.3 原生质体制备酶解时间优化酶解时每隔30min取出1ml,计算不同酶解时间下得到的原生质体纯度和形成率。
1.2.4 原生质体纯度和制备率计算原生质体纯度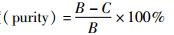
原生质体形成率
A为酶解前的菌丝体数量;B为酶解后高渗溶液下原生质体和菌丝体数量;C为酶解后水溶液下菌丝体数量。
1.3 原生质体融合条件优化 1.3.1 热灭活条件优化取制备好的原生质体悬液,于60℃水浴0min、10min、20min、30min、40min、50min,使菌株的蛋白质变性灭活,计算致死率。
1.3.2 紫外灭活条件优化取制备好的原生质体悬液,在距15W紫外灯下30cm的位置照射0min、1min、2min、3min、4min、5min,使核酸变性灭活,计算致死率。
1.3.3 原生质体融合方法采用亲本灭活法获得融合子[11]。将灭活后的菌体混合,加入用高渗溶液配制的50%PEG-2000室温反应2min,稀释涂布在再生培养基上。
1.4 基因组重排通过随机诱变获得高产菌株HSU7、HSU9、HSN11、HSN12,制备其原生质体,将HSU7、HSU9两株菌进行热灭活,HSN11、HSN12两株菌进行紫外灭活,将灭活后的原生质体融合获得R1代融合子。用96孔板发酵高通量初筛,获得高产菌株后,用摇瓶发酵验证。将得到的高产菌株,构建下一轮重排的突变子库,反复进行多轮融合筛选后,获得高产菌株。
1.5 茂源链霉菌的发酵 1.5.1 96孔板发酵将再生融合子挑入含有发酵培养基的微孔中,并在另一块96孔板中相同位置拷贝。将接种好的96孔板放入恒温摇床于30℃、200r/min的条件下培养4d。
1.5.2 摇瓶发酵用无菌水刮下菌株孢子,接入种子培养基中,于30℃,200r/min条件下培养28~36h。以10%的接种量转接到发酵培养基中,于30℃、200r/min条件下培养36h。
1.6 谷氨酰胺转胺酶的酶活力检测采用氧肟酸比色法[18]。
2 结果与分析 2.1 原生质体制备条件优化通过对野生菌株HS47进行紫外诱变和NTG诱变,获得的4株高产菌株HSU7、HSU9、HSN11、HSN12,平均酶活4.3U/ml,以这4个菌株构建第一轮重排的突变子库。首先需要制备这四个菌株的原生质体。以初始菌株HS47为模板对原生质体制备的条件进行优化,结果如图 1(a)、(b)所示。通过比较得到的原生质体纯度和形成率,酶解最优时间为2h,最优溶菌酶浓度为6mg/ml。通过优化后的条件,制备得到HS47的原生质体。由于细胞壁被酶解,细胞不能维持原本的丝状形态,在高渗溶液中,呈现圆球形,如图 1(d)所示,酶解效果良好。以同样的方法制备后续使用菌株的原生质体。
|
| 图 1 HS47原生质体制备条件优化 Figure 1 Optimization of protoplasts presentation conditions (a)Optimization of time treating with lysozyme (b)Optimization of lysozyme concentration (c)Mycelium before treating with lysozyme (d)Protoplasts after treating with lysozyme |
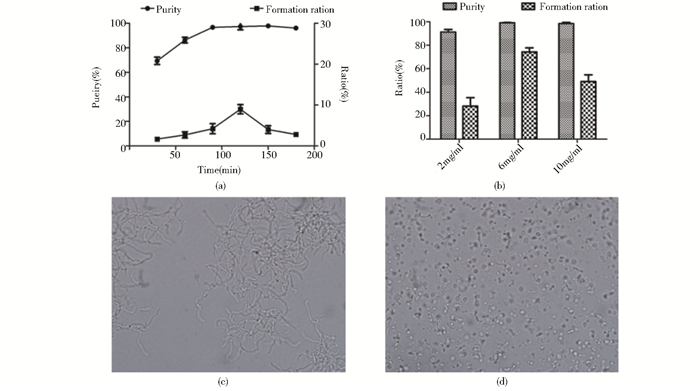
由于茂源链霉菌没有特殊的如抗生素抗性、营养缺陷型等遗传特征作为标记,为更方便地筛选融合子,选用灭活法处理亲本原生质体。其原理为热灭活和紫外灭活对细胞造成的损伤不同,经受不同损伤的原生质体在融合后可能互补,能够再生生长,而没有发生融合的原生质体则被灭活死亡,该方法能快速筛选出融合子[19]。首先对热灭活和紫外灭活的时间优化,结果如图 2(a)所示。紫外灭活的最适时间为2min,热灭活的最适时间为40min,致死率都达到99.99%。将灭活后的原生质体融合,涂布于再生培养基上,再生出融合子,如图 2(b)所示。
|
| 图 2 原生质体的灭活条件优化及融合子的再生 Figure 2 Optimization of protoplasts inactivation and regeneration of fusants (a)Optimization of inactivation time (b)Regeneration of protoplasts |
基因组重排可以让诱变产生的能够提高菌株产量的突变,通过原生质体融合,发生重组,从而大幅提高产酶,并可以以每一轮重排得到的高产菌株构建新的突变子库,极大缩短育种时间[13]。将融合得到的融合子进行96孔板发酵初筛,该发酵体系的变异系数范围为10%左右,故设定初筛时融合子酶活相比对照亲本菌株提高20%为高产菌株。对每轮基因组重排后筛选的约1 000个融合子统计结果显示,第一轮高产菌株所占比例为则为21.11%,第二轮为25.17%。选取每轮中最高的20株进行复筛验证,第二轮复筛得到结果如图 3所示。其中酶活最高的F303酶活达到7.12U/ml,相比第一轮突变子库平均酶活提高65.5%,比第二轮突变子库平均酶活提高30.4%。平均每一轮基因组重排提高酶活约30%。
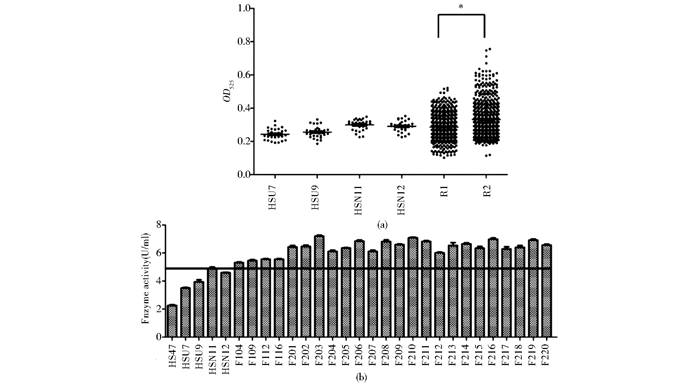
|
| 图 3 基因组重排初筛和验证结果 Figure 3 Screening of strains after genome shuffling (a)96-well plate screening of strains from genome shuffling * P < 0.01, signification difference (b)Enzyme activity of strains screening from genome shuffling |
选取通过基因组重排获得的酶活最高的菌株F303与原始菌株中酶活最高的HSN11一起发酵,发酵曲线和电泳结果如图 4所示。菌株F303在发酵后36h时到达产酶的最高点,相比HSN11的酶活有显著提高,并达到产酶最高点时间提前。TGase在分泌到胞外时带有前体即Pro-TG,需要切割金属蛋白酶切割后形成有活性的TGase[20]。电泳图中可以看出,高产菌株发酵液中的pro-TG明显较少,故酶活提高的原因可能是在重组后,激活了与酶成熟相关的沉默基因[21],使酶成熟更快、更彻底。
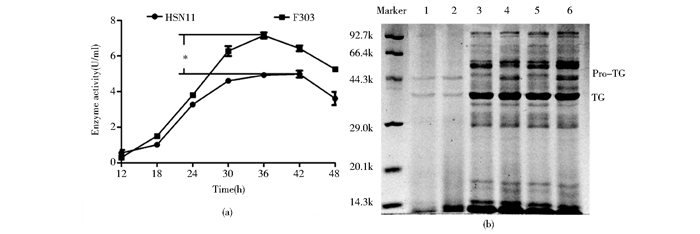
|
| 图 4 高产菌株和原始菌株发酵比较 Figure 4 Comparison of fermentation between high TGase producing strain and incipient strain (a)fermentation curve (b)SDS-PAGE of fermentation broth * P < 0.01, signification difference; Line 1, 3, 5:Fermentation broth of strain F303 after fermenting for 12h, 36h, 48h; Line 2, 4, 6:Fermentation broth of strain HSN11 after fermenting for 12h, 36h, 48h. |
将高产菌株F303在高氏一号培养基上连续转接5代,进行摇瓶发酵检测菌株的稳定性,结果如图 5所示。经过5次传代,菌株产酶情况稳定,每代之间的遗传稳定性非常好,说明由基因组重排得到的菌株,在遗传稳定性上没有问题。
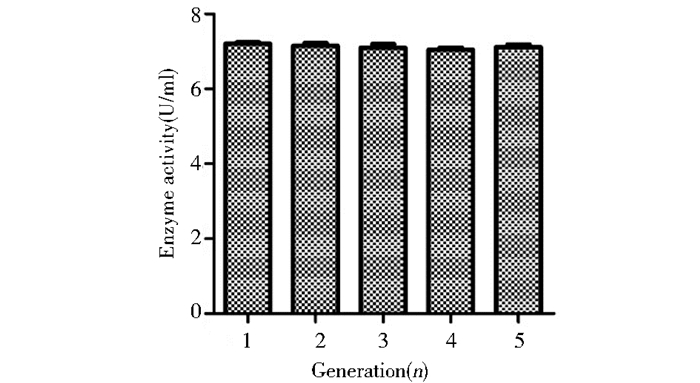
|
| 图 5 F303的传代发酵 Figure 5 Enzyme activity of different generation of F303 |
谷氨酰胺转胺酶有着非常广泛的应用[22],目前普遍使用的生产菌株为茂源链霉菌,但是国内使用的菌株生产力远低于味之素公司代表的世界水平。基因工程育种方法能够提高谷氨酰胺转胺酶的产量[23],但是由于转基因食品的安全性存在很大的社会争议,研究者们通常使用随机诱变的方法获得能应用于工业生产的优良菌株[5-10]。
相比传统诱变技术,基因组重排技术对人体危害性小,能够利用诱变获得的大量突变,构建突变子库[13],解决了大量酶活提高的突变菌株得不到利用的问题[11]。且大量实验证明,通过原生质体融合使这些突变子细胞融合,发生基因重组,将多个正突变基因重组到一个菌株中,能够提高育种效率,相比随机诱变,缩短育种周期[24]。基因组重排技术目前已经在一些链霉菌的育种中得到应用[11, 14-15]。
将文献报道的基因组重排方法中的一些条件进行优化,使其能够适用于茂源链霉菌菌株HS47的高产谷氨酰胺转胺酶的菌株选育。首先对原生质体制备条件进行优化,得到最优条件为以6mg/ml的溶菌酶浓度进行酶解,酶解时间2h,提高得到的亲本菌株的原生质体纯度和形成率,使制备得到的原生质体纯度达到99.62%,形成率达到72.46%。由于菌株HS47没有特殊的如抗生素抗性、营养缺陷型等遗传特征作为标记,为更方便地筛选融合子,选用亲本灭活法筛选融合子。对亲本菌株原生质体灭活条件优化的结果显示,紫外灭活的最适时间为2min,热灭活的最适时间为40min,灭活后的原生质体经融合能够互补不同灭活方法造成的损伤,在平板上再生,以此筛选融合子提高效率。
将不同诱变方法下得到的突变子构建突变子库,制备原生质体并融合后,将获得的融合子进行初筛和复筛验证。结果显示,通过两轮基因组重排,每轮初筛有20%以上的融合子酶活提高20%,摇瓶验证每轮最高产菌株酶活提高约30%,经过筛选获得了一株产酶高达7.12U/ml的茂源链霉菌,相比最初突变子库中4株菌的平均酶活提高65.5%,相比其中酶活最高的HSN11提高42%。相比赵文超的研究成果,通过本文的方法改进大幅提高了基因组重排育种的效果,菌株产酶的提高率远远高于5%,证明了基因组重排提高茂源链霉菌产谷氨酰胺转胺酶产量的可能性。
最近的研究报道,通过紫外诱变,臧学丽等[8]将菌株产量提高36.8%。通过ARTP诱变,田淑翠等[13]将酶活提升至5.1U/ml。与本文研究的结果对比,基因组重排育种的提高比率和酶活性都处于比较高的水平。结合96孔板高通量筛选的方法,提高了筛选效率,节省了人力、物力和时间。在后续的研究中,可以在现有条件下,以第二轮基因组重排得到的高产菌株构建的突变子库为基础,继续扩大突变子库,收纳更多方式诱变得到的突变,丰富突变的多样性,继续提高育种效率和酶活。
对其的发酵研究显示,它于发酵18h时开始产酶,发酵36h时达到最高酶活,时间有所提前。此外,在研究高产菌株的发酵产酶时,我们证实高产菌株发酵液中的pro-TG相对亲本对照菌株较少。分析可能是在基因重组后,与原酶成熟相关的蛋白质发生了改变,提高了成熟酶的产量。
通过对高产菌株的稳定性验证,证明经过基因组重排获得的高产菌株,遗传稳定性良好,能够应用于工业生产。为以后高产谷氨酰胺转胺酶茂源链霉菌菌株的选育提供一个良好的方法。
| [1] |
常中义, 江波, 王璋. 微生物谷氨酰胺转胺酶的应用进展. 食品科学, 2000, 21(9): 6-8. Chang Z Y, Jiang B, Wang Z. Application progress of microbial transglutaminase. Food Science, 2000, 21(9): 6-8. |
| [2] |
Ando H, Adachi M, Umeda K, et al. Purification and characteristics of a novel transglutaminase derived from microorganisms. Agricultural and Biological Chemistry, 1989, 53(10): 2613-2617. |
| [3] |
陶红军, 邵虎, 黄亚东, 等. 谷氨酰胺转胺酶的研究进展. 中国酿造, 2010(6): 9-12. Tao H J, Shao H, Hang Y D, et al. Research advance of transglutaminase. China Brewing, 2010(6): 9-12. |
| [4] |
王璋, 王灼维, 莫湘筠. 微生物谷氨酰胺转胺酶生产菌株的育种研究. 中国生物工程杂志, 2003, 23(6): 1-5. Wang Z, Wang Z W, Mo X Y. New advances of screening and breeding of efficient microbial strains producing transglutaminase. China Biotechnology, 2003, 23(6): 1-5. |
| [5] |
臧学丽, 陈光. 产转谷氨酰胺酶茂源链霉菌的诱变育种及发酵培养基优化. 吉林农业大学学报, 2016, 38(5): 548-551. Zang X L, Chen G. Mutation breeding and fermentation medium optimization of transglutaminase production of Streptoverticillium mobaraense. Journal of Jilin Agricultural University, 2016, 38(5): 548-551. |
| [6] |
Yokoyama K, Ustumi H, Nakamura T, et al. Screening for improved activity of a transglutaminase Streptomyces mobaraensis created by a novel rational mutagenesis and random mutagenesis. Applied Microbiology and Biotechnology, 2010, 86(6): 2087-2096. |
| [7] |
王璋, 王灼维. 微生物谷氨酰胺转胺酶高产菌株的诱变选育. 食品科学, 2003, 24: 62-67. Wang Z, Wang Z W. Mutation of microbial transglutaminase high production strains. Food Science, 2003, 24: 62-67. DOI:10.3321/j.issn:1002-6630.2003.04.013 |
| [8] |
张莹, 郭琛琛, 黑东燕, 等. 复合诱变法选育谷氨酰胺转胺酶高产菌株. 食品科学, 2014, 35(11): 139-142. Zhang Y, Guo C C, Hei D Y, et al. Screening and characterization of high-yield microbial transglutaminase-producing mutants induced by a novel method. Food Science, 2014, 35(11): 139-142. DOI:10.7506/spkx1002-6630-201411028 |
| [9] |
夏书琴, 刘龙, 张东旭, 等. 大气压辉光放电低温等离子体诱变选育谷氨酰胺转胺酶高产菌株. 微生物学通报, 2010, 37(11): 1642-1649. Xia S Q, Liu L, Zhang D X, et al. Mutation and selection of transglutaminase producing strain by atmospheric pressure glow discharge plasma. Microbiology China, 2010, 37(11): 1642-1649. |
| [10] |
田淑翠, 牛延宁, 常忠义, 等. 常压室温等离子体(ARTP)诱变茂源链霉菌菌株. 中国生物工程杂志, 2016, 36(9): 47-53. Tian S C, Niu Y N, Chang Z Y, et al. The Streptoverticillium mobaraense mutagenesis using atmospheric pressure plasma at room temperature (ARTP) method. China Biotechnology, 2016, 36(9): 47-53. |
| [11] |
Chen X, Wei P, Fan L, et al. Generation of high-yield rapamycin-producing strains through protoplasts-related techniques J. Applied Microbiology and Biotechnology, 2009, 83(3): 507-512. DOI:10.1007/s00253-009-1918-7 |
| [12] |
Zhang Y X, Perry K, Vinci V A, et al. Genome shuffling leads to rapid phenotypic improvement in bacteria. Nature, 2002, 415(6872): 644-646. DOI:10.1038/415644a |
| [13] |
秦芳芳, 龚雪萍, 赵文超, 等. 基因组重排技术在链霉菌育种应用的研究进展. 安徽农业科学, 2012, 40(34): 16529-16530. Qin F F, Gong X P, Zhao W C, et al. Recent advances of genome shuffling in the application of Streptomyces breeding. Journal of Anhui Agricultural Science, 2012, 40(34): 16529-16530. DOI:10.3969/j.issn.0517-6611.2012.34.010 |
| [14] |
Luo J M, Li J S, Liu D, et al. Genome shuffling of Streptomyces gilvosporeus for improving natamycin production. Journal of Agricultural and Food Chemistry, 2012, 60(23): 6026-6036. DOI:10.1021/jf300663w |
| [15] |
Lv X A, Jin Y Y, Li Y D, et al. Genome shuffling of Streptomyces viridochromogenes for improved production of avilamycin. Applied Microbiology and Biotechnology, 2013, 97(2): 641-648. DOI:10.1007/s00253-012-4322-7 |
| [16] |
赵文超. 茂源链霉菌基因组重排的初步探究. 上海: 华东师范大学, 生命科学学院, 2014. Zhao W C. Genome shuffling of Streptoverticillium mobaraensis. Shanghai:College of Life Science, East China Normal University, 2014. http://cdmd.cnki.com.cn/Article/CDMD-10269-1014322473.htm |
| [17] |
侯孝仑, 刘雅清, 郭玮婷, 等. 原生质体融合提高谷氨酰胺转氨酶菌株产量. 食品科学, 2016, 37(3): 145-150. Hou X L, Liu Y Q, Guo W T, et al. Strain improvement by protoplast fusion for enhanced transglutaminase production. Food Science, 2016, 37(3): 145-150. DOI:10.7506/spkx1002-6630-201603027 |
| [18] |
包莹玲, 潘力. 微生物谷氨酰胺转胺酶作用机制及检测方法研究进展. 食品工业科技, 2008, 29(7): 265-269. Bao L L, Pan L. Research progress of mechanism and activity assay of microbial transglutaminase. Science and Technology of Food Industry, 2008, 29(7): 265-269. |
| [19] |
赵凯, 段巍, 孙立新, 等. 用基因组重排技术选育赖氨酸高产菌株. 微生物学报, 2009, 49(8): 1075-1080. Zhao K, Duan W, Sun L X, et al. Screening and breeding of high lysine-producing strains by genome shuffling. Acta Microbiologica Sinica, 2009, 49(8): 1075-1080. |
| [20] |
Zotzel J, Pasternack R, Pelzer C, et al. Activated transglutaminase from Streptomyces mobaraensis is processed by a tripeptidyl aminopeptidase in the final step. European Journal of Biochemistry, 2003, 270(20): 4149-4155. DOI:10.1046/j.1432-1033.2003.03809.x |
| [21] |
连宝, 裘娟萍. 基因组重排技术在开发新代谢产物中的应用. 中国生物工程杂志, 2012, 32(3): 100-105. Lian B, Qiu J P. The Application of genome shuffling in developing new metabolites. China Biotechnology, 2012, 32(3): 100-105. |
| [22] |
Gaspar A L C, Góes-Favoni S P D. Action of microbial transglutaminase (MTGase) in the modification of food proteins:A review. Food Chemistry, 2015, 171: 315-322. DOI:10.1016/j.foodchem.2014.09.019 |
| [23] |
Yokoyama K, Nakamura N, Seguro K, et al. Overproduction of microbial transglutaminase in Escherichia coli, in vitro refolding, and characterization of the refolded form. Bioscience Biotechnology & Biochemistry, 2014, 64(6): 1263-1270. |
| [24] |
卢圣国, 李霜, 朱建国, 等. 基因组重排技术应用与进展. 中国生物工程杂志, 2010, 30(7): 108-111. Lu S G, Li S, Zhu J G, et al. The applications and progress of genome shuffling. China Biotechnology, 2010, 30(7): 108-111. |
 2017, Vol. 37
2017, Vol. 37




