文章信息
- 崔庆宇, 王明钰, 徐海.
- CUI Qing-yu, WANG Ming-yu, XU Hai.
- 细菌lux荧光报告系统的研究进展
- Research Progress of Bacterial Lux Bioluminescence Reporter System
- 中国生物工程杂志, 2017, 37(8): 66-71
- China Biotechnology, 2017, 37(8): 66-71
- http://dx.doi.org/DOI:10.13523/j.cb.20170810
-
文章历史
- 收稿日期: 2017-03-03
- 修回日期: 2017-03-24
“生物发光”现象很久以来不断地引起科学家们的好奇,它奇特瑰丽,却也相当普遍。在生物界里提到发光,人们首先想到的便是萤火虫,夏夜里星星点点的淡绿色光芒,令人心驰神往。深海中大约有90%的生物都有发光能力[1], 如某些鱼类、藻类、贝类、珊瑚、蠕虫、水母以及细菌等。这种能力并非“花拳绣腿”,在阳光终年无法穿透的深海,一盏盏色彩各异的明灯在求偶、捕食、逃避敌害、信息交流等方面发挥着重要的作用。海洋以其稳定而广阔的生境和缺少光能的自然条件,为生物发光多样性的进化提供了可能[2]。
生物发光是一种由荧光素酶催化的、以荧光素为底物的酶促反应。由于不同种类生物的发光系统在进化上有着不同的起源,因此编码荧光素酶的基因并不是同源的,荧光素也是一些不相关联的化合物[3]。现阶段广泛应用的生物发光系统/基因主要是来源于水母的绿色荧光蛋白(GFP)、来源于昆虫的luc基因以及来源于发光细菌的lux基因。
几乎所有的发光细菌可归为三个属(发光杆菌属(Photobacterium); 弧菌属(Vibrio); 异短杆菌属(Xenorhabdus)[4],lux基因簇编码和调控细菌生物发光,利用这类基因构建的报告系统作为一种快速而经济的工具,在特定条件下不仅可以精确反映基因的实时表达量和细菌的生长速率[5],还可在基因组范围内检测启动子的活性[6]。因其具有即时性和高灵敏度,在分子生物学、临床微生物及生化检测中呈现出潜在的应用价值。本文主要介绍了lux报告系统的分类及其各自的优缺点,综述了近年来该报告系统在环境监测、食品安全、新药发现等领域的部分研究成果,并对其应用前景进行了展望。
1 细菌生物发光的反应机制以及lux基因簇的组成所有的发光细菌都具有生化上相似的发光系统,其反应机制为:在分子氧和荧光酶的作用下,将还原态的黄素单核苷酸(FMNH2)及长链脂肪醛(RCHO)氧化为FMN及长链脂肪酸(RCOOH),并发射出波长为490nm的蓝绿光[7]。细菌荧光素酶都由包含有α亚基(分子量为42kDa)和β亚基(分子量为37kDa)的异二聚体构成[8],仅当两亚基共存时荧光素酶才有活性。该酶反应的最佳温度为18℃,超过25℃即迅速失活[9]。脂肪醛底物的供应是由脂肪酸还原酶作用于相应的脂肪酸,而还原态黄素单核苷酸可能是由依赖于NAD或NADP的氧化还原酶催化得来[10]。
lux基因的表达发生在细菌生长的对数末期,并被生长过程中积累的一种物质(自诱导物)所诱导。该基因可以分成3部分:核心元件、附属元件以及调节元件。核心元件包括lux C、D、A、B、E(图 1),其中luxA、luxB分别编码荧光酶的α和β亚基, 而luxC、luxD、luxE分别编码依赖NADPH的脂肪酸还原酶(54 kDa)、酰基转移酶(33 kDa)和ATP合成酶(42 kDa), 三者共同构成脂肪酸还原酶复合体, 产生长链脂肪醛作为发光反应的电子供体。lux基因的组成具有种属特异性,同一属间会因为菌株来源不同而造成基因组成上的差异。例如,大多数发光杆菌属在基因luxB、luxE之间存在附属基因luxF,但luxF并不存在于弧菌属和异短杆菌属的lux系统中。此外,所有海洋环境中分离得到的菌属(即:弧菌属、发光杆菌属)的lux操纵子下游均含有luxG基因,luxG在弧菌中编码一种在发光过程中产生的mRNA, 然而这类mRNA的功能未知,仍需进一步研究。哈氏弧菌(V. harveyi)中,基因luxG的下游还存在luxH。对luxG、luxH进行转座子插入致突变实验后,发现细菌的发光不受影响,由此推测这些基因可能与luxF相似,在细菌对海洋环境的适应性以及生存/共生等方面发挥着重要作用[11]。

|
| 图 1 lux操纵子的结构简式 Figure 1 The structure of the lux operon |
费氏弧菌中,luxC基因的上游含有两个调节基因:luxI和luxR。luxI与luxC、D、A、B、E同属于右操纵子,luxI的编码产物被证实是发光细菌的自诱导物因子合成酶。基因luxR属于左操纵子,其转录方向与luxI相反。luxR的编码产物被认为是自诱导物的受体[12],自诱导物-受体复合物随后会激发右操纵子的转录。
2 原核生物lux报告系统的分类lux报告系统可应用于原核和真核生物中来得到报告菌株,本文主要介绍其在原核生物中的应用。大多数微生物的染色体上并不存在合成荧光素酶的基因,因此若想得到可发光微生物,必须转入控制合成荧光素酶的基因(luxA, luxB)和控制合成长链脂肪醛酸还原酶的基因(luxC, luxD, luxE),这可以通过基因工程的手段来得到。由于某些细菌的基因会受到外界因素如抗生素、重金属、温度等影响而改变转录水平,因此,构建lux报告系统最常用的方法是将这些特定基因的启动子与lux基因相融合。目前,可以在原核生物中表达的lux报告系统主要有两种类型:以基因组为载体的lux报告系统和以质粒为载体的lux报告系统。
2.1 以基因组为载体的lux报告系统这种报告系统可通过利用含有lux基因的转座子载体得到。缺少启动子的lux基因位于载体一端,只有转座子插入在染色体某个基因的启动子的下游时,lux基因方可表达。目前,已构建的此类载体有mini-Mu lux, Tn5 lux, mini-Tn5 luxAB, mini-Tn7 lux,由mini-Tn7衍生出的具有高发光强度、可应用于多种革兰氏阴性菌的载体pAG4[13]、pUC18T-mini-Tn7T-lux-Tp[14],以及可用于捕捉和测定启动子及其表达强度的载体Tn5/7-lux[15]。将lux基因整合到染色体上可保证其稳定性和可遗传性,然而这种方法技术难度较大,需要耗用大量时间并可能对细菌的表型产生一定影响。
2.2 以质粒为载体的lux报告系统这种类型的lux报告系统由于存在基因的剂量效应,有着更高的发光强度,与转座子载体相比更容易构建、使用更方便。其构建方法通常为:首先将lux基因整合到载体上,随后通过酶切连接等方式将特定基因的启动子片段插入到lux基因的上游,最后将重组质粒导入细菌中。然而,有文献报道lux基因的插入会降低质粒的稳定性。为了防止质粒丢失,人们通常使用抗生素抗性基因作为筛选标记,然而此举会限制lux报告株在活体动物内的应用。当研究动物体内病原菌定殖状况时不宜施用抗生素,因而无法判断荧光强度的减弱是由于无抗生素条件下造成的质粒丢失还是由于免疫系统对病原菌的吞噬。为了解决以上问题,Bacconi等[16]在原有载体骨架的基础上加入了毒素/抗毒素系统(T/A)和分离系统(Par)的基因序列,最终得到可在子代中稳定复制的质粒pMABA-Par/TA-lux。用含有这种质粒的金黄色葡萄球菌经由静脉注射感染小鼠,在无抗生素选择压力下,7天后该质粒仍有70%~100%的保留率。
由于lux基因在革兰氏阳性菌中表达量较低,因此有人通过变更lux基因的核糖体结合位点以及重排lux基因簇来提高其表达量[17]。pAL2是首个应用于革兰氏阳性菌的lux报告系统[18],由此衍生出的pGYlux、pAmilux(图 2)有着更高的稳定性和发光强度。pGYlux更适用于检测强启动子,而pAmilux可用于检测不同强度的启动子或是假定启动子[19]。
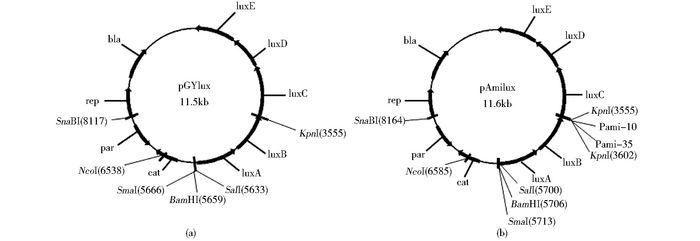
|
| 图 2 pGYlux、pAmiLux载体结构示意 Figure 2 The structures of pGYlux and pAmiLux Restriction enzymes and genes are indicated. bla: β-lactamase gene; rep: Replication initiation proteins gene; cat: Chloramphenical resistant gene; par: Partition sequence |
抗生素的滥用会使一些经常接触抗生素而未被杀死的细菌对其产生耐药性或发生变异, 从而导致抗生素失效。同时,新的抗菌药物的开发成本高、难度大、速度慢。为了防止细菌感染陷入“无药可医”的困境,我们需要一种直观、高效的方法从环境中筛选新型抗生素[20-21]。
几乎所有的抗生素在亚抑菌浓度下可使细菌中5%以上的基因发生转录水平上的改变[22],这种特性为在环境中检测微量的生物活性物质提供了一个新的思路。亚致死量的抗生素对某些基因(如MMR系统及SOS系统基因)的启动子有一定的上调作用,由这些特殊基因的启动子与lux基因构成荧光报告系统,在细菌中受到抗生素刺激而发出荧光。Mesak等[22]将tet、recA等基因的启动子与pAmilux或pGYlux融合后导入金黄色葡萄球菌中。采用双层平板法,将含有抗生素的滤纸片贴在平板上,借助荧光成像系统观察到同一种抗生素可以引起不同基因的不同反应,而同一基因对不同的抗生素反应不同。借助这种报告系统,证实具有抗金黄色葡萄球菌作用的一种北里孢菌(Kitosatospora)发酵液对各启动子的激发作用不同于现有已知抗生素,随后通过薄层色谱、核磁共振和质谱鉴定证实这些抗菌物质为链黑菌素、氧代脯氨酸D、氧代脯氨酸G。
赵冰等[23]以含lux基因的质粒pCS26、pMS402为载体,构建大肠杆菌重组质粒、筛选铜绿假单胞菌以及沙门氏菌的随机启动子库,获得15个对抗生素有反应的启动子-报告子融合体从而构成抗生素筛选体系。利用这种体系检测一种未知的抗生素样品,经对比发现该样品对同种启动子的促进作用与四环素、庆大霉素、壮观霉素的相似性较高,后经鉴定证实该抗生素为氨基糖苷类的硫酸新霉素,说明这种筛选体系可进行初步的抗生素类别鉴定。
此外,Fenton等[24]利用活体荧光成像系统(IVIS),给鼻部感染了金黄色葡萄球菌-lux报告株(S. aureus Xen29)的小鼠注射一种经过改造的由金黄色葡萄球菌噬菌体K表达的细胞溶解酶(CHAPk),图像显示注射之前小鼠鼻部有明亮的荧光斑块,注射1h后小鼠鼻部的荧光完全消失,因而证实了CHAPk对感染金黄色葡萄球菌的清除作用。与抗生素不同的是,噬菌体细胞溶解酶可定向作用于病原菌从而免于影响周围的共生微生物群落,且不会使细菌产生耐药性。这种类型的报告菌株为新型抗菌物质杀菌效力的检测提供了一种直观、便捷的方式。本实验室通过与加拿大英属哥伦比亚大学(UBC)的Davies实验室合作,利用Mesak等人构建的荧光报告系统PAmiLux,与编码金黄色葡萄球菌热休克蛋白的基因(dnaK, dnaJ, groES-groEL, clpB, clpC, clpP, clpX)的启动子融合后导入金黄色葡萄球菌中得到金黄色葡萄球菌启动子-lux报告株, 并研究了部分抗生素对这些启动子的激发作用。后续工作的重点是利用该报告系统建立一套能够快速判断环境中微量抗生素的类型的方法,为新药发现提供一个可行的思路。
3.2 应用于动物体内细菌定殖的研究及肿瘤定位Francis等[17]通过改进lux操纵子的核糖体结合位点得到了新的荧光表达载体pMK4luxABCDE, 并将其导入到金黄色葡萄球菌中。改造后的lux报告株在特定启动子序列的激发下可以在37摄氏度时稳定发光,并且无需添加外源醛类底物。利用活体荧光成像系统可通过观察发光区域的大小、发光的强弱来实时监测金黄色葡萄球菌在小鼠体内的感染进程。鼻疽假单胞菌(Burkholderia mallei)是鼻疽病的病原菌,人们对它的发病机理及治疗方式所知甚少,Massey等[25]利用载体pUTmini-Tn5∷luxKm2将lux基因整合到鼻疽假单胞菌的基因组上,并使小鼠经由鼻腔感染这种报告菌株。感染后的24h、48h、72h时借助荧光成像检测小鼠身体的发光区域,结果表明,在整个感染进程中病灶最先出现在肺部,而后逐渐扩散至肝脏和脾脏,感染24h后对小鼠施用抗生素则会减少发光区域的面积。利用lux报告株检测细菌定殖情况的方法具有灵敏无创伤的优点,与传统检测方法(摘取器官进行细菌培养)相比可明显减少实验动物的用量。近年来,关于利用荧光报告菌株进行癌症研究的报道层出不穷,人们发现细菌在小鼠体内随着循环系统传播时,会优先定殖于肿瘤组织中。Cronin等[26]用含有lux基因的大肠杆菌和短双歧杆菌(Bifidobacterium breve)来感染小鼠,借助活体成像得到了发光区域的2D和3D图像,这为肿瘤的精确定位提供了一个可行的思路,Leschner等[27]也做了相关研究。
此外,lux报告系统还可用于研究基因间的相互作用。Steinhuber等[28]将编码α毒素的基因(hla)的启动子整合到含有lux基因的载体pAS1上得到表达载体pAS2。将pAS2分别导入金黄色葡萄球菌Newman Xen29的sae和sigB的等位基因突变株,通过IVIS系统比较小鼠体内各个菌株的发光强度来检测sae、sigB对hla启动子活性的影响。结果显示,菌株Newman Xen29 pAS2的发光强度高于它的sae同源突变株,并弱于它的sigB同源突变株,说明hla的基因表达量可被sae提高,同时被sigB所抑制。
3.3 应用于检测环境污染物及其毒性一些细菌由于长期在含有某种化学物质的环境中而产生了适应现象,细菌基因组中含有对该种物质具有特异性的诱导基因和降解基因。将这些基因的启动子与含有lux基因的载体融合并导入宿主细菌,在特定的化学物质存在时便可被诱导发光。二氯甲烷(DCM)是一种具有挥发性的有机化合物,在工业生产中常用作卤化溶剂,但同时也是潜在的致癌物。环境中DCM的检测主要有固相微萃取技术、毛细管气相色谱法及光声检测法,然而这些传统方法不仅需要专业人员,还需要昂贵的设备。Nicholas等[29]设计了一种用于DCM检测的生物感应器,原理是:将编码DCM脱卤素酶的基因的启动子整合到载体pCM66中lux基因的上游,随后将重组载体导入甲基杆菌中。当报告菌株暴露于DCM中时便会发光,对气相及液相的DCM,检测限分别低至1.0×10-6和0.1×10-6。Sandra等[30]构建了载体pRpoD-lux、pArs-lux并导入大肠杆菌Dh10beta中,分别用于检测环境水样本对细胞的整体毒性以及是否存在亚砷酸盐。报告菌株在4度储存7个月后仍能用于样本检测,对亚砷酸盐的检测限低至0.2 μM,具有高灵敏度、高重现性等优点。King等[31]构建了一种含lux基因的质粒pUTK21,由转座子Tn4431介导,插入到荧光假单胞菌(Pseudomonas fluorescens)基因组的特定位点,用以检测土壤中的萘。随后,Heitzer等[32]对此进行了更为深入的研究,他们将这类报告菌株固定在光纤上制成传感器,发现萘的含量和菌体发光强度有良好的对应关系,为利用lux报告系统进行化学物质的定量检测提供了新的思路。随着工业化的发展,环境中存在的潜在致突变物质成了一个日益加剧的问题。那么,如何甄别大批量的环境样品的遗传毒性和致癌性?传统策略是选用Ames实验,但是这种方法耗时较长,需要大量的人力物力。针对这个问题,Alhadrami等[33]借助SOS-lux标记的报告菌株:E.coli DPD1718和E.coli K12C600,检测吖啶、叠氮化钠等6种化学物质对报告株发光的诱导作用,若化学物质能破坏细菌的基因组,SOS修复系统便会启动,从而导致lux基因的表达。与Ames实验的结果进行对比,证实这两种方法之间有着良好的相关性。
3.4 应用于食品安全监测和质控民以食为天,食品安全是关乎民生的重要问题。传统的检测食品中微生物的方法是将样品研磨后进行菌落培养计数,耗时较长。Loessner等[34]利用经过重组的李斯特菌(Listeria monocytogenes)专性噬菌体A511∷luxAB来检测食物样本中的李斯特菌,在意大利干酪、巧克力布丁、甘蓝中,即使每克食物样品中只含有一个李斯特菌也会被检测到;而在微生物区系较为复杂的肉馅、软奶酪中,每克食物至少含有10个李斯特菌才会有荧光信号。与传统的平板计数法相比,这种利用lux报告系统检测食物中李斯特菌的方法将检测时间由4天缩短到24h,并具有较高的灵敏度。Kricka[35]将lux基因转入P22噬菌体,用这种噬菌体感染所需检测的样品后,1h之内可以检测1×102数量级的沙门氏菌,可用于乳制品质量的在线控制。引入lux基因的噬菌体由于不具备代谢系统,缺乏发光反应所必需的荧光素酶、底物和能量,因此这种噬菌体在体外不发光,当宿主细菌被这种专一性的噬菌体感染后,方可发生发光反应。由于此方法不依赖于细菌细胞的增殖,一般在20~30分钟内就能够检测到特定细菌的存在,且发光量与宿主细菌的量正相关,可通过测定发光量来检测相应微生物的量。
4 小结与展望以lux报告系统为代表的生物发光检测技术与常规检测方法比较,有一些明显的优点,如具有高灵敏度、高特异性以及迅速、方便等。随着lux报告系统稳定性的提高和功能上的改进,其在新药发现、动物体内细菌定殖情况的追踪、肿瘤定位、环境污染物毒性检测、食品安全监测与质控等领域中逐渐显现出独特的应用价值。同时,该技术仍有需要完善的方面:其一,可通过改进检测设备来捕捉更加微弱的发光信号;其二,可借助蛋白质工程技术对荧光素酶进行改造,使发光波长红移或蓝移从而提高生物发光在不同组织中的穿透力。如今,lux报告系统正与其他高新技术如细胞固定化技术、生物传感器技术、计算机技术、低光影像技术、CCD(Charge-coupled Device)相机等紧密结合在一起,使得生物发光检测技术的应用范围愈加宽广。目前,人们正努力发展多颜色的lux基因,使同步检测多个转录事件成为可能。可以预见随着研究的深入,这类报告系统的功能将会更加丰富和完善。
| [1] |
Smith D C, Douglas A E. The Biology of Symbiosis. London: Edward Arnold, 1987.
|
| [2] |
林济民, 王江云. 有趣的发光现象与奇妙的发光蛋白. 自然杂志, 2011, 33(5): 286-290. Lin J M, Wang J Y. Interesting bioluminescent creatures and amazing fluorescent proteins. Chinese Journal of Nature, 2011, 33(5): 286-290. |
| [3] |
Hastings J W. Chemistries and colors of bioluminescent reactions:a review. Gene, 1996, 173(1): 5-11. DOI:10.1016/0378-1119(95)00676-1 |
| [4] |
茆灿泉, 杨树德. 发光细菌lux报告基因系统的评价及应用. 微生物学通报, 2000, 27(4): 297-299. Mao C Q, Yang S D. Evaluation and application of lux reporter system in bioluminous bacteria. Microbiology, 2000, 27(4): 297-299. |
| [5] |
Shah N, Naseby D C. Bioluminescence-based measurement of viability of Pseudomonas aeruginosa ATCC 9027 harbouring plasmid-based lux genes under the control of constitutive promoters. Journal of Applied Microbiology, 2014, 117(5): 1373-1387. DOI:10.1111/jam.2014.117.issue-5 |
| [6] |
Van Dyk T K, Derose E J, Gonye G E. LuxArray, a high-density, genomewide transcription analysis of Escherichia coli using bioluminescent reporter strains. Journal of Bacteriology, 2001, 183(19): 5496-5505. DOI:10.1128/JB.183.19.5496-5505.2001 |
| [7] |
Greer L F, Szalay A A. Imaging of light emission from the expression of luciferases in living cells and organisms:a review. Luminescence, 2002, 17(1): 43-74. DOI:10.1002/(ISSN)1522-7243 |
| [8] |
Belas R, Mileham A, Cohn D, et al. Bacterial bioluminescence:isolation and expression of the luciferase genes from Vibrio harveyi. Science, 1982, 218(4574): 791-793. DOI:10.1126/science.10636771 |
| [9] |
杜宗军, 王祥红, 李海峰, 等. 发光细菌的研究和应用. 高技术通讯, 2003, 13(12): 103-106. Du Z J, Wang X H, Li H F, et al. The study and application of luminescent bacteria. Chinese High Technology Letters, 2003, 13(12): 103-106. DOI:10.3321/j.issn:1002-0470.2003.12.022 |
| [10] |
Riendeau D, Meighen E. Co-induction of fatty acid reductase and luciferase during development of bacterial bioluminescence. J Biol Chem, 1980, 255(24): 12060-12065. |
| [11] |
Meighen E A. Molecular biology of bacterial bioluminescence. Microbiol Rev, 1991, 55(1): 123-142. |
| [12] |
Engebrecht J A, Silverman M. Regulation of expression of bacterial genes for bioluminescence. Genet, 1986, 8(1): 31-44. |
| [13] |
Glassing A, Lewis T A. An improved Tn7-lux reporter for broad host range, chromosomally-integrated promoter fusions in Gram-negative bacteria. J Microbiol Methods, 2015, 118(11): 75-77. |
| [14] |
Damron F H, McKenney E S, Barbier M, et al. Construction of mobilizable mini-Tn7 vectors for bioluminescent detection of gram-negative bacteria and single-copy promoter lux reporter analysis. Appl Environ Microbiol, 2013, 79(13): 4149-4153. DOI:10.1128/AEM.00640-13 |
| [15] |
Bruckbauer S T, Kvitko B H, Karkhoff-Schweizer R R, et al. Tn5/7-lux:a versatile tool for the identification and capture of promoters in gram-negative bacteria. BMC Microbiol, 2015, 15(1): 1-16. DOI:10.1186/s12866-014-0320-5 |
| [16] |
Bacconi M, Haag A F, Torre A, et al. A stable luciferase reporter plasmid for in vivo imaging in murine models of Staphylococcus aureus infections. Applied Microbiology and Biotechnology, 100(7): 3197-3206. DOI:10.1007/s00253-015-7229-2 |
| [17] |
Francis K P, Joh D, Bellinger-Kawahara C, et al. Monitoring bioluminescent Staphylococcus aureus infections in living mice using a novel luxABCDE construct. Infection and Immunity, 2000, 68(6): 3594-3600. DOI:10.1128/IAI.68.6.3594-3600.2000 |
| [18] |
Beard S J, Salisbury V, Lewis R J, et al. Expression of lux genes in a clinical isolate of Streptococcus pneumoniae:using bioluminescence to monitor gemifloxacin activity. Antimicrobial Agents and Chemotherapy, 2002, 46(2): 538-542. DOI:10.1128/AAC.46.2.538-542.2002 |
| [19] |
Mesak L R, Yim G, Davies J. Improved lux reporters for use in Staphylococcus aureus. Plasmid, 2009, 61(3): 182-187. DOI:10.1016/j.plasmid.2009.01.003 |
| [20] |
Fischbach M A, Walsh C T. Antibiotics for emerging pathogens. Science, 2009, 325(5944): 1089-1093. DOI:10.1126/science.1176667 |
| [21] |
Projan S J, Youngman P J. Antimicrobials:new solutions badly needed. Current Opinion in Microbiology, 2002, 5(5): 463-465. DOI:10.1016/S1369-5274(02)00364-8 |
| [22] |
Mesak L R, Qi S, Villanueva I, et al. Staphylococcus aureus promoter-lux reporters for drug discovery. Journal of Antibiotics (Tokyo), 2010, 63(8): 492-498. DOI:10.1038/ja.2010.74 |
| [23] |
赵冰, 孙赵麟, 杨亮, 等. 一种利用基因表达变化检测、鉴别抗生素的新方法. 生物工程学报, 2010, 26(1): 93-99. Zhao B, Sun Z L, Yang L, et al. Construction of a promoter reporter array for antibiotic screening. Chinese Journal of Biotechnology, 2010, 26(1): 93-99. |
| [24] |
Fenton M, Casey P G, Hill C, et al. The truncated phage lysin CHAP(k) eliminates Staphylococcus aureus in the nares of mice. Bioengineered Bugs, 2010, 1(6): 404-407. DOI:10.4161/bbug.1.6.13422 |
| [25] |
Massey S, Johnston K, Mott T M, et al. In vivo bioluminescence imaging of Burkholderia mallei respiratory infection and treatment in the mouse model. Front Microbiol, 2011, 2(4): 174-183. |
| [26] |
Cronin M, Akin A R, Francis K P. In vivo bioluminescence imaging of intratumoral bacteria. Methods Mol Biol, 2016, 1409(2): 69-77. |
| [27] |
Leschner S, Weiss S. Noninvasive in vivo imaging to follow bacteria engaged in cancer therapy. Methods Mol Biol, 2016, 1409(2): 61-68. |
| [28] |
Steinhuber A, Landmann R, Goerke C, et al. Bioluminescence imaging to study the promoter activity of hla of Staphylococcus aureus in vitro and in vivo. International Journal of Medical Microbiology, 2008, 298(7-8): 599-605. DOI:10.1016/j.ijmm.2007.09.008 |
| [29] |
Nicholas Lopes, Shawn A Hawkins, Patricia Jegier, et al. Detection of dichloromethane with a bioluminescent (lux) bacterial bioreporter. J Ind Microbiol Biotechnol, 2012, 39(1): 45-53. DOI:10.1007/s10295-011-0997-5 |
| [30] |
Prévéral S, Brutesco C, Descamps E C, et al. A bioluminescent arsenite biosensor designed for inline water analyzer. Environ Sci Pollut Res Int, 2017, 24(1): 25-32. DOI:10.1007/s11356-015-6000-7 |
| [31] |
King J M, Digrazia P M, Applegate B, et al. Rapid, sensitive bioluminescent reporter technology for naphthalene exposure and biodegradation. Science, 1990, 249(4970): 778-781. DOI:10.1126/science.249.4970.778 |
| [32] |
Heitzer A, Malachowsky K, Thonnard J E. Optical biosensor for environmental on-line monitoring of naphthalene and salicylate bioavailability with an immobilized bioluminescent catabolic reporter bacterium. Appl Environ Microbiol, 1994, 60(5): 1487-1494. |
| [33] |
Alhadrami H A, Paton G I. The potential applications of SOS-lux biosensors for rapid screening of mutagenic chemicals. FEMS Microbiol Lett, 2013, 344(1): 69-76. DOI:10.1111/femsle.2013.344.issue-1 |
| [34] |
Loessner M J, Rudolf M, Scherer S. Evaluation of luciferase reporter bacteriophage A511::luxAB for detection of Listeria monocytogenes in contaminated foods. Appl Environ Microbiol, 1997, 63(8): 2961-2965. |
| [35] |
Kricka L J. Clinical and biochemical applications of luciferases and luciferins. Anal Biochem, 1988, 175(1): 14-21. DOI:10.1016/0003-2697(88)90354-5 |
 2017, Vol. 37
2017, Vol. 37




