文章信息
- 王佩, 陈凯, 高嵩.
- WANG Pei, CHEN Kai, GAO Song.
- 利用CpG DNA甲基化酶M.Sss Ⅰ共表达载体制备限制性内切酶Not Ⅰ
- Production of Restriction Endonuclease Not Ⅰ Utilizing CpG DNA Methylase M.Sss Ⅰ Co-expression Vector
- 中国生物工程杂志, 2017, 37(8): 51-58
- China Biotechnology, 2017, 37(8): 51-58
- http://dx.doi.org/DOI:10.13523/j.cb.20170808
-
文章历史
- 收稿日期: 2017-01-16
- 修回日期: 2017-04-15
2. 淮海工学院江苏省海洋生物产业技术协同创新中心 连云港 222005;
3. 江苏省海洋资源开发研究院 连云港 222005
2. Co-Innovation Center of Jiangsu Marine Bio-industry Technology, Huaihai Institute of Technology, Lianyungang 222005, China;
3. Jiangsu Marine Resources Development Research Institute, Lianyungang 222005, China
依赖于核酸序列识别并特异性切割DNA的限制性内切酶是现代分子生物学和基因工程领域研究基因组成、功能和表达不可或缺的重要工具。随着这类酶的广泛应用,近年来其商业化生产和重组表达日益增长[1-3]。然而,由于限制酶具有切割DNA的特性,在对其进行重组表达时往往会使宿主DNA被切割,造成宿主菌的死亡[4-7]。迄今为止,限制性内切酶的重组表达在技术手段上仍然存在极大困难。根据最早从事限制性内切酶商业化生产的美国New England Biolabs公司(NEB)的公开专利可知,NEB公司推出的多种限制性内切酶产品都是利用该限制性内切酶对应的专一型甲基化酶对宿主DNA进行保护,从而获得重组表达的限制性内切酶[8-9]。此方法的原理是,产生限制酶的天然菌株会同时产生相应的专一型甲基化酶,将限制酶的识别位点甲基化,保护自身的DNA免受限制酶的切割[1, 10]。但是这种利用专一型甲基化酶的传统限制酶表达技术十分繁琐、应用范围较为狭窄。
DNA甲基化酶M.Sss Ⅰ是一种来源于Spiroplasma sp. MQ1的甲基化酶[11]。这种甲基化酶可以特异性地将DNA中的CpG序列甲基化,而由于序列CpG是许多限制性内切酶酶切位点的子集,对CpG的甲基化能使DNA免受识别位点中含有CpG序列的限制性内切酶的切割(www.neb.com)。若利用甲基化酶M.Sss Ⅰ的这一特性对宿主细胞的DNA进行甲基化,使其形成CpG甲基化样式,该宿主细胞的DNA将会免受许多限制性内切酶的切割,并能支持这些酶的表达。
本研究将DNA甲基化酶M.Sss Ⅰ导入大肠杆菌表达宿主ER2566中,使M.Sss Ⅰ基因在宿主中持续表达,并甲基化宿主DNA形成CpG甲基化样式。利用这一表达宿主进行常用限制性内切酶Not Ⅰ的重组表达获得成功。同时,表达产物末端添加了多聚组氨酸标签,仅通过简便的Ni亲和层析和阴离子交换层析2步柱洗脱纯化步骤即获得了性能优、活力高且纯度好的重组限制酶Not Ⅰ。
1 材料与方法 1.1 材料 1.1.1 菌种和质粒大肠杆菌DH5α和ER2566购自美国菌种保藏中心(ATCC);质粒pACYC184购自武汉淼灵生物科技有限公司;质粒pUC19、pET-28b及其改造的衍生质粒由本实验室提供。
1.1.2 仪器和试剂引物合成、基因合成及测序由苏州金唯智生物科技有限公司完成;细胞高压破碎仪购自广州聚能生物科技有限公司;Ni亲和层析基质和阴离子交换层析基质(Toyopearl)购自东曹(上海)生物科技有限公司;限制性内切酶、CutOne Buffer、T4 DNA Ligase、抗生素等生化试剂购自江苏愚公生命科技有限公司;B-PER细菌蛋白提取试剂购自美国Thermo Fisher Scientific公司;蛋白分子量标准购自金斯瑞生物科技有限公司;胶回收试剂盒购自广州飞扬生物工程有限公司(Omega Bio-Tek);质粒提取试剂盒(Axygen)购自美国Corning公司;凝胶成像仪(Clinx)购自上海勤翔科学仪器有限公司。
1.2 方法 1.2.1 甲基化酶M.Sss Ⅰ组成型表达菌株的构建甲基化酶M.Sss Ⅰ的DNA序列(GenBank:X17195.1)通过基因合成获得,连接至克隆载体pUC57上,构建pUC57-M.Sss Ⅰ质粒。合成序列全长1 360 bp,5′-端的增添序列包括:Hind Ⅲ酶切位点和E. coli pltl promoter序列(GenBank:M88256.1),3′-端的增添序列包括:TAA终止密码子和E. coli rrnB T1 terminator序列(GenBank:CP010229.1),以及Hind Ⅲ酶切位点。pUC57-M.Sss Ⅰ质粒经Hind Ⅲ单酶切后,含有M.Sss Ⅰ基因的片段以T4 DNA Ligase连接至相同限制性内切酶酶切后的pACYC184载体上,并转化大肠杆菌DH5α感受态细胞进行质粒扩增,然后提取重组质粒pACYC184-M.Sss Ⅰ。此质粒经测序正确后,转化进入感受态细胞ER2566,并于含35 mg/L氯霉素的LB平板上生长,获得甲基化酶M.Sss Ⅰ组成型表达菌株ER2566 [pACYC184-M.Sss Ⅰ],利用大肠杆菌pltl启动子持续表达甲基化酶M.Sss Ⅰ。
1.2.2 限制性内切酶Not Ⅰ重组表达菌株的构建限制性内切酶Not Ⅰ的DNA序列(GenBank:ABC87270.1)通过基因合成获得,连接至克隆载体pUC57上,构建pUC57-Not Ⅰ质粒。合成序列全长1 164 bp,包括5′-端添加的BamH Ⅰ酶切位点和3′-端添加的TAA终止密码子以及Xho Ⅰ酶切位点。pUC57-Not Ⅰ质粒经BamH Ⅰ和Xho Ⅰ双酶切后,带有Not Ⅰ基因的片段以T4 DNA Ligase连接至相同限制性内切酶酶切后的pET-28b载体上,使Not Ⅰ基因的表达框与载体上的多聚组氨酸纯化标签6×His的表达框一致。连接产物转化进入甲基化酶M.Sss Ⅰ组成型表达菌株ER2566 [pACYC184-M.Sss Ⅰ]感受态细胞,并于含75mg/L卡那霉素和35mg/L氯霉素的LB平板上生长,获得限制性内切酶Not Ⅰ重组表达菌株ER2566 [pACYC184-M.Sss Ⅰ, pET-Not Ⅰ]。限制性内切酶Not Ⅰ的基因序列经测序确认正确,诱导后表达为N-端融合6×His纯化标签的重组限制酶Not Ⅰ。
1.2.3 重组限制酶Not Ⅰ的诱导及活性检测将限制性内切酶Not Ⅰ重组表达菌株ER2566 [pACYC184-M.Sss Ⅰ, pET-Not Ⅰ]接种于含75 mg/L卡那霉素和35 mg/L氯霉素的液体LB培养基中,在37℃以200~250r/min振荡培养至OD600值为0.6~0.8,然后加入终浓度为0.5 mmol/L的IPTG,在16℃以200~250 r/min振荡培养16 h。以8 000g离心10 min收集菌体后,使用B-PER细菌蛋白提取试剂对菌体总蛋白进行提取。取1~3 μl细菌蛋白粗提液,在1X CutOne Buffer(51 mmol/L乙酸钾, 22 mmol/L Tris-Ac, 10 mmol/L乙酸镁, 1 mg/ml BSA, pH 8.0)缓冲条件下的10 μl反应体系中,与Not Ⅰ的酶切底物(control plasmid,改造的pET-28b质粒,带有2个Not Ⅰ酶切位点)在37℃温育15 min,然后使用1%琼脂糖凝胶电泳检测底物被细菌蛋白粗提液消化的情况。
1.2.4 重组限制酶Not Ⅰ的纯化重组限制酶Not Ⅰ经IPTG诱导后,培养液以8 000g离心10 min收集菌体。菌体以缓冲液(10 mmol/L Tris-HCl, pH 7.4, 250 mmol/L NaCl, 5%甘油, 0.15% Triton X-100, 5mmol/L咪唑, 1 mmol/L苯甲基磺酰氟)充分重悬后,以细胞高压破碎仪裂解。不可溶组分以43 000g低温离心30 min去除,含有重组限制酶Not Ⅰ的上清可溶组分经过Ni亲和层析初步纯化(Toyopearl AF-Chelate-650M)和阴离子交换层析精细纯化(Toyopearl GigaCap Q-650M)。Ni亲和层析过程中使用的缓冲液成分如下:平衡Buffer -10 mmol/L Tris-HCl, pH 7.4, 250 mmol/L NaCl, 5%甘油, 0.15% Triton X-100, 5 mmol/L咪唑;漂洗Buffer -10 mmol/L Tris-HCl, pH7.4, 250 mmol/L NaCl, 5%甘油, 0.15% Triton X-100, 20 mmol/L咪唑;洗脱Buffer -10 mmol/L Tris-HCl, pH7.4, 250 mmol/L NaCl, 5%甘油, 0.15% Triton X-100, 400 mmol/L咪唑。阴离子交换层析使用的缓冲液成分如下:平衡Buffer -10 mmol/L Tris-HCl, pH 7.4, 250 mmol/L NaCl, 5%甘油, 0.15% Triton X-100, 0.1 mmol/L EDTA, 1 mmol/L DTT;梯度洗脱分别为250 mmol/L、300 mmol/L、500 mmol/L、700 mmol/L、1 mol/L NaCl。含有重组限制酶Not Ⅰ的洗脱峰合并后,透析至贮存缓冲液(10 mmol/L Tris-HCl, pH 7.4, 250 mmol/L NaCl, 50%甘油, 0.15% Triton X-100, 0.1 mmol/L EDTA, 1 mmol/L DTT)中,并添加BSA至终浓度200 μg/ml,长期保存于-20℃。整个纯化过程在4℃或冰上进行。
1.2.5 重组限制酶Not Ⅰ的活力测定将重组限制酶Not Ⅰ进行梯度稀释后,在1X CutOne Buffer缓冲条件下的20 μl反应体系中,与Not Ⅰ的酶切底物(control plasmid)在37℃温育15 min,将能够把底物完全酶切的酶量定义为1 U。使用1%琼脂糖凝胶电泳检测底物的酶切情况,根据酶切完全时对应的稀释度计算重组限制酶Not Ⅰ的活力。
1.2.6 星号活性及非特异性DNA酶污染检测将重组限制酶Not Ⅰ进行梯度稀释后,在1X CutOne Buffer缓冲条件下的20 μl反应体系中,与Not Ⅰ的酶切底物(control plasmid)在37℃温育60 min或16 h,然后使用1%琼脂糖凝胶电泳检测底物的酶切情况。
1.2.7 酶切再连检测使用20 U重组限制酶Not Ⅰ在1X CutOne Buffer缓冲条件下的20 μl反应体系中,与0.2 μg底物DNA分子(Common Fragment,894 bp,带有1个Not Ⅰ酶切位点,位于413 bp处)在37℃温育15 min。酶切完成后,在80℃温育20 min使限制酶Not Ⅰ失活,然后在反应液中补充ATP至终浓度0.5 mmol/L,并使用T4 DNA Ligase在22℃连接1 h。连接产物在65℃温育10 min失活后,加入20 U重组限制酶Not Ⅰ进行再次酶切(37℃温育15 min)。酶切、连接及再次酶切的效率以1%琼脂糖凝胶电泳检测,并以Clinx GenoSens凝胶成像分析系统进行量化分析。
1.2.8 蓝白斑检测使用10 U重组限制酶Not Ⅰ在1X CutOne Buffer缓冲条件下的20 μl反应体系中,与1 μg pUC19质粒DNA在37℃温育15 min。酶切完成后,在80℃温育20 min使限制酶Not Ⅰ失活,然后在反应液中补充ATP至终浓度0.5 mmol/L,并使用T4 DNA Ligase在22℃连接1 h。连接产物转化进入DH5α大肠杆菌感受态细胞中,并涂布含氨苄抗性的IPTG/X-gal平板,于37℃培养过夜。统计所得白斑(白色菌落)与蓝斑(蓝色菌落)的数目并计算白斑比例。将重组限制酶Not Ⅰ酶切线性化后的pUC19质粒(未进行连接反应)作为对照,同样转化DH5α大肠杆菌感受态细胞中,并在含氨苄抗性的IPTG/X-gal平板上培养过夜,用于蓝斑背景的统计。
2 结果 2.1 限制性内切酶Not Ⅰ重组表达菌株的构建首先构建了用于保护宿主DNA的甲基化酶组成型表达质粒pACYC184-M.Sss Ⅰ(图 1a)。该质粒含有甲基化酶M.Sss Ⅰ的基因,并且M.Sss Ⅰ基因被大肠杆菌组成型表达元件pltl promoter和rrnB T1 terminator所调控。带有此质粒的大肠杆菌宿主细胞能够持续表达甲基化酶M.Sss Ⅰ,在宿主DNA上的CpG位点形成甲基化。而这种CpG甲基化样式能够保护宿主DNA免受限制性内切酶Not Ⅰ的切割(www.neb.com)。大肠杆菌ER2566细胞株经转化质粒pACYC184-M.Sss Ⅰ,形成了适合于表达限制性内切酶Not Ⅰ的细胞株ER2566 [pACYC184-M.Sss Ⅰ]。此细胞株能够持续表达甲基化酶M.Sss Ⅰ,宿主DNA受到CpG甲基化样式的保护,不会由于限制性内切酶Not Ⅰ的表达而死亡。
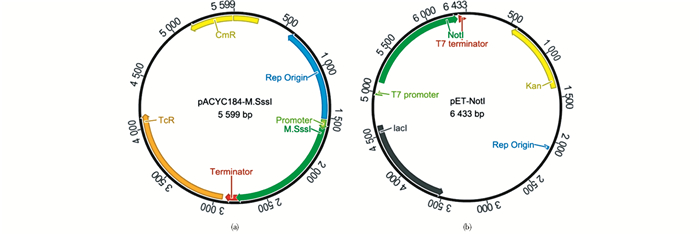
|
| 图 1 pACYC184-M.Sss Ⅰ和pET-Not Ⅰ表达载体的构建 Figure 1 Schematic of the pACYC184-M.Sss Ⅰ and pET-Not Ⅰ constructs (a) Schematic of pACYC184-M.Sss Ⅰ(b) Schematic of pET-Not ⅠThe circular plasmid DNA molecules are represented by circles, with numbers indicating the lengths of the molecules (in bp). The M.Sss Ⅰ and Not Ⅰ genes are represented by green ribbons. Promoters, terminators, antibiotic resistance genes, the lacI gene, and the replication origins are indicated |
然后构建了限制性内切酶Not Ⅰ的重组表达质粒pET-Not Ⅰ(图 1b)。限制酶Not Ⅰ的基因被插入到pET-28b载体的表达区域,置于强诱导型启动子T7 promoter的调控之下,并与载体上的多聚组氨酸纯化标签6×His融合,形成了带有N-端融合6×His纯化标签的重组Not Ⅰ基因。将插入Not Ⅰ基因的环状DNA分子pET-Not Ⅰ转化进入ER2566 [pACYC184-M.Sss Ⅰ]感受态细胞中,成功获得了限制性内切酶Not Ⅰ重组表达菌株ER2566 [pACYC184-M.Sss Ⅰ, pET-Not Ⅰ]。由于pACYC184和pET质粒的复制子来源不同,可相容共存于同一个细胞株系中。值得一提的是,带有Not Ⅰ基因的环状DNA分子pET-Not Ⅰ只能转化进入宿主DNA受到CpG甲基化样式保护的ER2566 [pACYC184-M.Sss Ⅰ]菌株,而转化其它宿主DNA未受CpG甲基化样式保护的菌株(如DH5α等)则不能获得转化子。此现象表明,T7 promoter调控下的Not Ⅰ基因在非诱导条件下的微量本底表达即足以杀死DNA未受甲基化保护的宿主细胞。
2.2 重组表达的限制酶Not Ⅰ具有功能活性为检测限制酶Not Ⅰ的酶切活性,本研究改造了pET-28b质粒,插入一段带有一个Not Ⅰ酶切位点的DNA,新插入的Not Ⅰ酶切位点与pET-28b质粒上原有的Not Ⅰ酶切位点相距860 bp。此质粒作为本研究中Not Ⅰ的酶切底物(control plasmid),酶切后会形成大小分别为5 467 bp和860 bp的2条DNA条带。在对Not Ⅰ重组表达菌株ER2566[pACYC184-M.Sss Ⅰ, pET-Not Ⅰ]以IPTG进行小量诱导后,通过差速离心收集菌体,破碎细胞并粗提总蛋白。对蛋白粗提液使用上述改造的pET-28b质粒进行Not Ⅰ酶切活性检测,结果显示,改造的pET-28b质粒被切割成大小分别为5 467 bp和860 bp的2条特异性条带,表明重组表达的限制酶Not Ⅰ具有功能活性(图 2)。
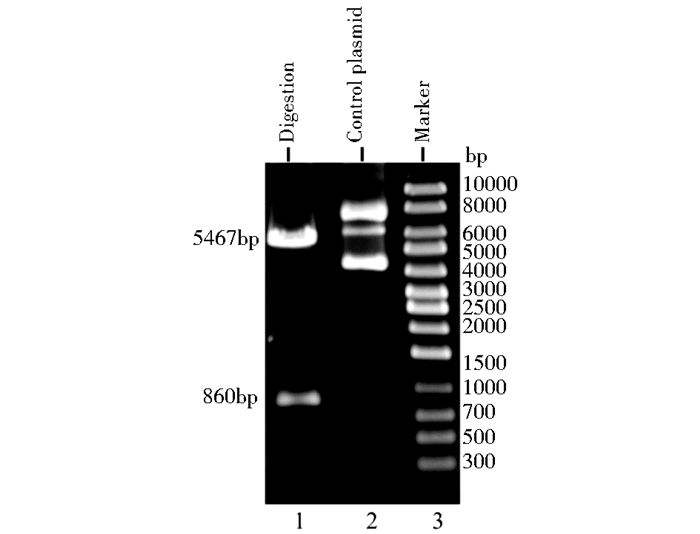
|
| 图 2 细胞总蛋白粗提液具有限制酶Not Ⅰ的酶切活性 Figure 2 The total protein extract from the induced cells showed restriction activity of Not Ⅰ Agarose gel shows the digested specific DNA fragments (lane 1, 5467 bp and 860 bp) of the control plasmid (lane 2) by 1 μl of the total protein extract from the induced cells |
在验证了重组表达的限制酶Not Ⅰ具有功能活性后,对ER2566[pACYC184-M.Sss Ⅰ, pET-Not Ⅰ]菌株进行了大规模诱导,大量表达重组限制酶Not Ⅰ。诱导后的菌体使用差速离心收集并破碎,然后对细胞裂解液进行纯化。纯化分为利用N-端融合的6×His纯化标签的Ni亲和层析和阴离子交换层析2个步骤。在Ni亲和层析步骤,重组限制酶Not Ⅰ被Ni基质捕获,使用咪唑进行特异性洗脱后,获得纯度约为50%的纯化效果。在SDS-PAGE胶图上,大小为46 kDa的重组限制酶Not Ⅰ目的条带十分明显(图 3a)。然后使用阴离子交换层析进行精细纯化,将重组限制酶Not Ⅰ蛋白的纯度提升至75%以上(图 3b)。阴离子交换层析不仅进一步提高了蛋白纯度,而且去除了蛋白样品中的宿主DNA残留。

|
| 图 3 重组限制酶Not Ⅰ的纯化 Figure 3 Purification of the recombinant Not Ⅰ (a) SDS-PAGE analysis of the purification of recombinant Not Ⅰ by Ni-affinity chromatographySamples of flow-through after capture (lane 2), wash (lane 3), and imidazole elution (lanes 4~9) were electrophoresed(b) SDS-PAGE analysis of the purification of recombinant Not Ⅰ by ion-exchange chromatographySamples of flow-through (lane 2, in 250 mmol/L NaCl) and step gradient elution (lanes 3~7, 250 mmol/L to 1mol/L NaCl) were electrophoresed. 12% SDS-PAGE gels were used and the protein bands were stained with Coomassie Brilliant Blue. The arrows indicate the band position of recombinant NotⅠ |
纯化完成后,对重组限制酶Not Ⅰ进行了酶活力检测,采用了快速酶切的检测标准[12]:37℃下,在20 μl反应体系中,1 μl(1 U)酶能够在15 min内完全消化1 μg底物质粒DNA。按照这一活性标准,将纯化后的重组限制酶Not Ⅰ进行了梯度稀释并检测活力。结果表明,纯化后的重组限制酶Not Ⅰ样本在稀释1 920倍后仍能达到1 U/μl快速酶切活力(图 4a)。经换算,重组限制酶Not Ⅰ比活力为872 000 U/mg,产率为1 152 000 U/L诱导菌液(图 4b)。与以往的限制酶Not Ⅰ制备方法相比,本研究制备的重组限制酶Not Ⅰ具有快速酶切性能,比活较好[3, 13]。
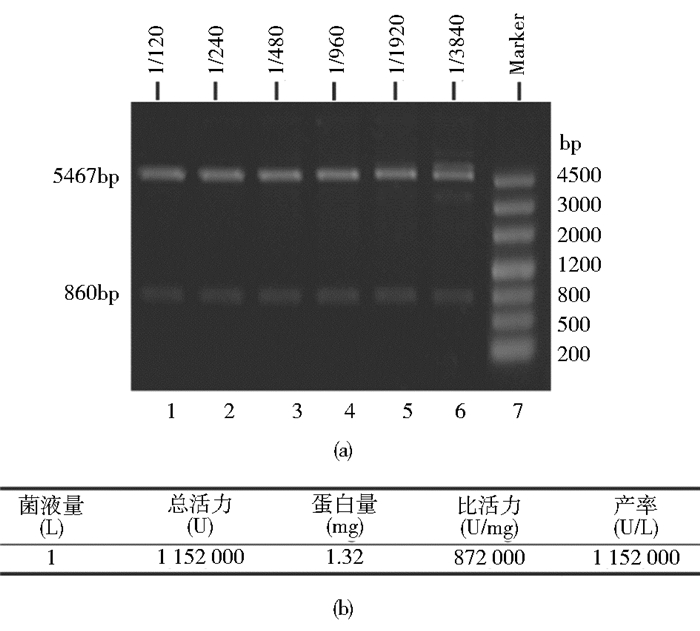
|
| 图 4 重组限制酶Not Ⅰ的活力及产率 Figure 4 Determination of enzymatic activity and yield of the recombinant Not Ⅰ (a) Agarose gel showing the digestion of 1 μg of control plasmid by 1 μl of the serial diluted recombinant Not Ⅰ sampleLanes 1~6 show dilution factors 1/120, 1/240, 1/480, 1/960, 1/1920 and 1/3840, respectively(b) Purification summary of recombinant Not Ⅰ Results from a typical purification of 1 L of induced culture are presented |
依据限制酶生产的行业标准,对限制酶的提纯是以去除干扰限制酶功能的其它活性为目的,对其纯度的要求为无干扰活性,而提纯后的蛋白纯度并不是评估限制酶品质的必需标准[1]。星号活性及非特异性DNA酶污染检测、酶切再连检测、蓝白斑检测是对限制性内切酶纯度和质量进行检测的经典项目,一直为国际知名的限制性内切酶生产企业(如美国NEB公司等)所使用。本研究参照美国NEB公司的限制酶Not Ⅰ质控方法,在这3个检测项目上对重组限制酶Not Ⅰ的纯度和质量进行了检测( www.neb.com)。
在星号活性及非特异性DNA酶污染检测中,将纯化的重组限制酶Not Ⅰ进行梯度稀释,并与1 μg底物质粒DNA在37℃温育60 min或16 h(图 5a)。结果显示,在温育60 min后,超过量(16 U,对应稀释度1/120)的酶切并未造成底物DNA产生非特异性降解条带。在超长时间(16 h)温育后,4 U(对应稀释度1/480)酶量的酶切未产生非特异性降解条带,而高达8 U酶量的酶切仅造成了轻微的非特异性降解。此结果表明重组限制酶Not Ⅰ在超过量酶切和超长时间酶切条件下,均不会产生星号活性及非特异性DNA降解。
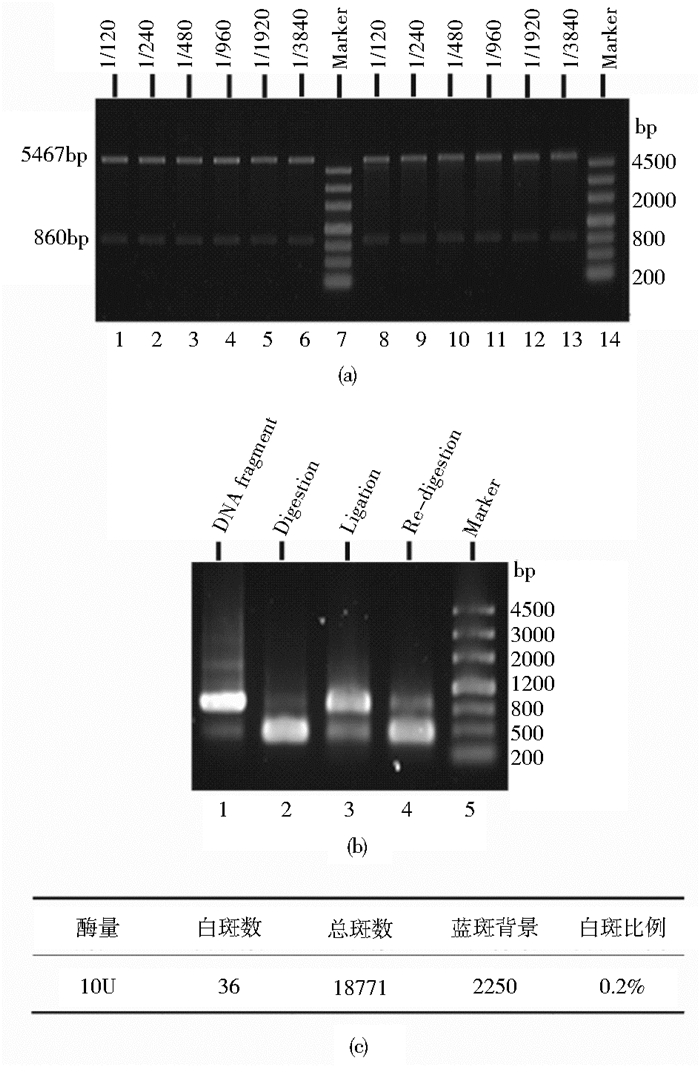
|
| 图 5 重组限制酶Not Ⅰ的质量控制检测显示高纯度及高质量 Figure 5 Quality control of the recombinant Not Ⅰ suggested high purity and quality (a) Prolonged incubation and star activity assayAgarose gel shows the digestion of 1 μg of control plasmid by 1 μl of the serial diluted recombinant Not Ⅰ sample that incubated at 37℃ for 60 min (lanes 1~6) or 16 h (lanes 8~13). The dilution factors are indicated at the top of each lane(b) Ligation and recut assayThe DNA fragment and samples after digestion, ligation and re-digestion were analyzed by agarose gel electrophoresis(c) Result of the blue/white screening assay |
在酶切再连检测中,使用20 U重组限制酶Not Ⅰ与底物DNA分子(Common Fragment)在37℃温育15 min后,所得的酶切片段使用T4 DNA连接酶进行连接,获得了> 95%的重新连接。所得的连接产物> 95%可以被重组限制酶Not Ⅰ再次切开(图 5b)。此结果表明,重组限制酶Not Ⅰ切割DNA形成的粘性末端准确并且完整。
在蓝白斑检测中,将含有单一lacZα基因的pUC19载体以10 U酶量消化,重新连接后转化入大肠杆菌感受态细胞,涂布在含有IPTG和X-gal的LB培养基平板上。连接正确的产物会生长出蓝色菌落,而连接错误(即DNA末端切口不完整)的产物将得到白色菌落,以此检测重组限制酶Not Ⅰ切割DNA形成的粘性末端的完整性。结果显示,所得白斑(白色菌落)比例仅为0.2%,表明重组限制酶Not Ⅰ切割DNA形成的粘性末端十分完整(图 5c)。
以上3项重组限制酶Not Ⅰ的检测结果与NEB公司公布的限制酶Not Ⅰ质控标准(Certificate of Analysis)比较可知,本研究获得的重组限制酶Not Ⅰ在这3个指标上达到了的NEB公司的商业化限制酶标准,具有高质量(www.neb.com)。
3 讨论限制性核酸内切酶作为分子生物学最重要的基础工具酶,其发现、应用开发和商业化生产奠定了重组DNA技术的基础,并在很大程度上推动了基因工程和现代分子生物学的发展[1-2]。然而,许多常用限制性内切酶目前仍面临重组表达产率低、制备工艺繁琐等难题,尤其是由于限制酶能降解宿主DNA,目前的制备工艺对于每个限制酶都必须筛选出专一型的甲基化酶基因并克隆至宿主细胞中,用于保护宿主DNA,因此增加了制备限制酶的繁琐程度。本研究利用甲基化酶M.Sss Ⅰ的甲基化位点CpG是多种限制酶识别位点的子集这一特点,尝试构建一种宿主DNA具有CpG甲基化样式的表达菌株,可用于多种限制酶的重组表达。
在构建这一表达菌株时,使用了E. coli pltl promoter和rrnB T1 terminator作为甲基化酶M.Sss Ⅰ的表达调控元件。这一对表达元件调控下的基因在大肠杆菌细胞中能持续稳定地表达,已在多种大肠杆菌工程菌株的改造中用于实现外源蛋白的组成型表达[14-16]。对于表达宿主菌株的选择,由于大部分大肠杆菌工程菌株都具备甲基化限制基因(methylation requiring restriction,mrr),能够抵制具有甲基化活性的外源基因的转入,因此选择了大肠杆菌突变株ER2566(mcrC-mrr)[17]。菌株ER2566中的甲基化限制基因mrr是缺失的,外源甲基化酶M.Sss Ⅰ的基因能够顺利转入。在转入了含甲基化酶M.Sss Ⅰ基因的质粒pACYC184-M.Sss Ⅰ后,菌株ER2566 [pACYC184-M.Sss Ⅰ]被用于转化限制酶Not Ⅰ的表达质粒pET-Not Ⅰ,并成功获得了转化子。而其它菌株(如DH5α等)由于DNA未受CpG甲基化样式的保护,转化限制酶Not Ⅰ的表达质粒pET-Not Ⅰ未能产生转化子,这与以前的研究中观察到的现象一致[3, 13]。以上结果表明,本研究构建的菌株ER2566 [pACYC184-M.Sss Ⅰ]实现了甲基化酶M.Sss Ⅰ的组成型表达,其宿主DNA受到了CpG甲基化样式的保护,在转化限制酶Not Ⅰ的表达质粒pET-Not Ⅰ后不会受到Not Ⅰ降解宿主DNA的毒性影响,能够正常生长。而在DNA未受甲基化保护的菌株中,T7 promoter调控下的Not Ⅰ基因在非诱导条件下的微量本底表达即可将宿主细胞杀死。
快速酶切已成为现代生物医学研究对于限制性内切酶酶切属性的一项基本要求,国际知名的限制酶生产商美国NEB公司(HF及Time-Saver系列)、美国Thermo Fisher Scientific公司(FastDigest系列)和日本TaKaRa Bio公司(QuickCut系列)都开发出了能够在5~15 min内快速酶切的限制酶产品。本研究利用宿主ER2566 [pACYC184-M.Sss Ⅰ]重组表达的限制酶Not Ⅰ也具有快速酶切的性能,1 U重组限制酶Not Ⅰ能在15 min内完全消化1 μg底物质粒DNA。以这样的活性标准,重组限制酶Not Ⅰ的比活力仍达到了十分优良的872 000 U/mg,产率也达到了1 152 000 U/L诱导菌液。在以前的报道中,研究人员使用Eag Ⅰ的甲基化酶对宿主DNA进行保护,开发出优化的限制酶Not Ⅰ制备工艺,大大提升了Not Ⅰ的表达效率[3]。这一经过优化的工艺制备的限制酶Not Ⅰ仍属于慢速酶(在1 h内完成1 μg底物的消化),其比活力与本研究制备的能快速酶切的重组限制酶Not Ⅰ处于同一水平,说明本研究构建的限制酶表达宿主ER2566 [pACYC184-M.Sss Ⅰ]具有更高的表达效率,能产生具有更高活力的限制酶。
本研究对于重组限制酶Not Ⅰ的纯化步骤包括Ni亲和层析和阴离子交换层析这2个上柱洗脱过程,与包含磷酸纤维素阴离子交换柱、肝素琼脂糖亲和层析柱、DEAE-琼脂糖柱、PEI柱四步上柱洗脱过程的传统限制酶Not Ⅰ纯化流程相比,大大简化了步骤;也比近年报道的优化的Not Ⅰ制备工艺更为简便[3]。简化的制备工艺不仅缩短了纯化时间,也有利于保持酶活力及避免蛋白损失。本研究制备的重组限制酶Not Ⅰ的蛋白纯度达到了75%;更重要的是,3项经典限制酶质检项目的检测结果证明,这3项指标均达到美国NEB公司限制酶Not Ⅰ的质量标准,污染物水平极低,无干扰活性,具有高纯度及高质量。
甲基化酶M.Sss Ⅰ的发现和研究已有较长的历史,其甲基化作用产生的CpG甲基化样式能使DNA免于多种限制性内切酶的切割。现已证明美国NEB公司Time-Saver系列185种(2016年12月份数据)商业化限制酶中,包括但不限于Aat Ⅱ、Asc Ⅰ、Cla Ⅰ、Eag Ⅰ、Fsp Ⅰ、Mlu Ⅰ、Not Ⅰ、Pvu Ⅰ、Sal Ⅰ、Sma Ⅰ、Xho Ⅰ、Xma Ⅰ等在内的59种常用限制酶会被CpG甲基化样式阻断切割作用( www.neb.com)。因此,本研究构建的表达宿主ER2566 [pACYC184-M.Sss Ⅰ]可应用于一大批限制酶的高效表达。重组限制酶Not Ⅰ仅经过2步上柱洗脱的简便纯化流程即达到高品质的标准,说明由于菌株ER2566 [pACYC184-M.Sss Ⅰ]对限制酶Not Ⅰ的高效表达,下游的提纯工艺可以简化。本研究不仅开发出一种更为简便的限制酶Not Ⅰ制备工艺,成功制备了达到商业化品质标准、具有快速酶切性能的重组限制酶Not Ⅰ,而且构建了基于甲基化酶M.Sss Ⅰ的甲基化保护菌株,可应用于一大批酶切位点中包含CpG序列的限制酶的高效表达,为进一步提升这些限制酶的制备工艺提供了参考。
| [1] |
Pingoud A, Wilson G G, Wende W. Type Ⅱ restriction endonucleases——a historical perspective and more. Nucleic Acids Research, 2014, 42(12): 7489-7527. DOI:10.1093/nar/gku447 |
| [2] |
Williams R J. Restriction endonucleases:classification, properties, and applications. Molecular Biotechnology, 2003, 23(3): 225-243. DOI:10.1385/MB:23:3 |
| [3] |
张巧, 叶贤龙, 任桂萍, 等. 限制性内切酶Not Ⅰ提纯的新工艺. 中国生物工程杂志, 2011, 31(8): 102-109. Zhang Q, Ye X L, Ren G P, et al. A new method for the purification of restriction enzyme Not Ⅰ. China Biotechnology, 2011, 31(8): 102-109. |
| [4] |
Naito T, Kusano K, Kobayashi I. Selfish behavior of restriction-modification systems. Science, 1995, 267(5199): 897-899. DOI:10.1126/science.7846533 |
| [5] |
Smith H O, Nathans D. A suggested nomenclature for bacterial host modification and restriction systems and their enzymes. Journal of Molecular Biology, 1973, 81(3): 419-423. DOI:10.1016/0022-2836(73)90152-6 |
| [6] |
Haberman A, Heywood J, Meselson M. DNA modification methylase activity of Escherichia coli restriction endonucleases K and P. Proceedings of the National Academy of Sciences of the United States of America, 1972, 69(11): 3138-3141. DOI:10.1073/pnas.69.11.3138 |
| [7] |
Lunnen K D, Barsomian J M, Camp R R, et al. Cloning type-Ⅱ restriction and modification genes. Gene, 1988, 74(1): 25-32. DOI:10.1016/0378-1119(88)90242-9 |
| [8] |
Zhu Z Y, Quimby A, Guan S X, et al. High fidelity restriction endonucleases. US Patent, US20110151450A1, 2011. |
| [9] |
Zhu Z Y, Blanchard A, Xu S X, et al. High fidelity restriction endonucleases. US Patent, US20090029376A1, 2009. |
| [10] |
Morgan R D, Camp R R, Wilson G G, et al. Molecular cloning and expression of NlaⅢ restriction-modification system in E. coli. Gene, 1996, 183(1-2): 215-218. DOI:10.1016/S0378-1119(96)00561-6 |
| [11] |
Renbaum P, Abrahamove D, Fainsod A, et al. Cloning, characterization, and expression in Escherichia coli of the gene coding for the CpG DNA methylase from Spiroplasma sp. strain MQ1(M SssI).. Nucleic Acids Research, 1990, 18(5): 1145-1152. DOI:10.1093/nar/18.5.1145 |
| [12] |
Thermo Fisher Scientific Inc. Thermo Scientific Molecular Biology Solutions. Thermo Fisher Scientific Inc., MA, USA, 2012-2013. |
| [13] |
Richard D M, Middleton, Jack S B, et al. Isolation DNA encoding the Not Ⅰ restriction endonuclease and relation methods for producing the same. US Patent, 1994, 5371006. |
| [14] |
Konstantinovic M, Maksimovic V, Nikcevic G, et al. Hybrid PLtl promoter with dual regulation control. DNA and Cell Biology, 1991, 10(5): 389-395. DOI:10.1089/dna.1991.10.389 |
| [15] |
Cheong D E, Choi J H, Song J J, et al. Construction of non-invasively constitutive expression vectors using a metagenome-derived promoter for soluble expression of proteins. Bioprocess and Biosystems Engineering, 2013, 36(6): 667-676. DOI:10.1007/s00449-013-0890-x |
| [16] |
Sabido A, Martínez L M, de Anda R, et al. A novel plasmid vector designed for chromosomal gene integration and expression:use for developing a genetically stable Escherichia coli melanin production strain. Plasmid, 2013, 69(1): 16-23. DOI:10.1016/j.plasmid.2012.04.005 |
| [17] |
Waite-Rees P A, Keating C J, Moran L S, et al. Characterization and expression of the Escherichia coli Mrr restriction system. Journal of Bacteriology, 1991, 173(16): 5207-5219. DOI:10.1128/jb.173.16.5207-5219.1991 |
 2017, Vol. 37
2017, Vol. 37




