文章信息
- 胡春辉, 徐青, 于浩.
- HU Chun-Hui, XU Qing, YU Hao.
- Arthrobacter sp.2PR降解2-羟基吡啶动力学及降解特性研究
- Characteristics and Kinetic Study of 2-Hydroxypyridine Degradation by a Novel Bacterium Arthrobacter sp. 2PR
- 中国生物工程杂志, 2017, 37(8): 31-38
- China Biotechnology, 2017, 37(8): 31-38
- http://dx.doi.org/DOI:10.13523/j.cb.20170805
-
文章历史
- 收稿日期: 2016-11-26
- 修回日期: 2016-12-14
2. 中国海洋大学环境科学与工程学院 青岛 266100
2. College of Environmental Science and Engineering, Ocean University of China, Qingdao 266100, China
吡啶及其衍生物是一类常见的氮杂环有机污染物,主要来自石油冶炼,药物、杀虫剂、除草剂的有机合成等过程[1]。吡啶类污染物具有较高的生物学毒性,由于氮原子的存在,使得吡啶类化合物催化特异性强,因此一定程度上吡啶环降解相对于芳香环更加复杂[2]。环境中吡啶类化合物代谢微生物数量远远小于芳香族污染物代谢微生物的数目。羟基吡啶是吡啶类污染物中的重要的一类,也是微生物降解吡啶类化合物的核心中间代谢产物[3]。吡啶氮氧化物、尼古丁、吡啶羧酸、吡啶等吡啶类化合物代谢过程中均会产生羟基吡啶类中间代谢产物。因此羟基吡啶类化合物的微生物代谢研究对于吡啶类污染物的生物修复研究具有十分重要的意义[1]。
2-羟基吡啶(2HP)是羟基吡啶污染物的一种,降解菌株在自然界中分布很多。从上世纪70年代开始就有研究者研究2-羟基吡啶降解菌株[4-5]。他们陆续分离得到了多株能够以2-羟基吡啶作为唯一碳氮源进行生长的菌株,它们分别属于Arthrobacter[6-7]、Rhodococcus[8]、Achromobacter[9]、Nocardia[10]等属。同时研究者对分离到的菌株的代谢产物进行了研究,大部分菌株在代谢2-羟基吡啶的过程中会产生一种蓝色物质,据文献报道这种蓝色物质为4, 5, 4′, 5′, -tetrahydroxy-3, 3′, -diazadiphenoquinone-(2, 2′)(blue pigment),是由2, 3, 6-三羟基吡啶自动氧化生成[11]。这表明大部分菌株是通过在吡啶环上进行单加氧或者双加氧反应生成2, 3, 6-三羟基吡啶再进行后续降解的。这一点与酶学研究结果一致,有报道表明2-羟基吡啶的第一阶段的反应涉及NADH依赖的黄素(FMN)氧化还原酶[3]。在菌株Achromobacter sp.中[9]2-羟基吡啶和3-羟基吡啶首先转化为2, 5-二羟基吡啶,随后吡啶环发生双加氧反应,断裂生成马来酰胺酸、马来酸,并最终生成富马酸进入三羧酸循环,完成整个降解过程[12]。这些都表明微生物对2-羟基吡啶的代谢具有多样性,但是完整的2-羟基吡啶的代谢途径并没有报道。关于微生物对2-羟基吡啶的降解效率和降解动力学的研究到目前也未见相关报道。
微生物代谢2-羟基吡啶分子机理研究较少,有研究表明部分菌株的2-羟基吡啶降解基因位于质粒上面,如菌株Arthrobacter crystallopoietes,该菌株经过传代后会丧失降解2HP的能力[7]。大部分菌株的降解基因位于基因组上,如菌株A.pyridinolis和A.viridescens[6]。菌株Rhodococcus rhodochrous PY11降解2-羟基吡啶的分子机理研究得比较清楚,该菌株中2-羟基吡啶首先经由一个四组分的双加氧酶(HpoBCDF)氧化为3, 6-dihydroxy-1, 2, 3, 6-tetrahydropyridin-2-one,进一步由HpoE氧化成2, 3, 6-trihydroxypyridine三羟基吡啶,三羟基吡啶被HpoH进一步转化为2-酮戊二酸单酰胺,2-酮戊二酸单酰胺被HpoI转化为α-酮戊二酸进入三羧酸循环[8]。但是相关酶的酶学性质还没有具体研究,另外节杆菌降解2-羟基吡啶的分子机理尚不清楚。
本研究筛选到一株能够高效降解2-羟基吡啶的菌株Arthrobacter sp. 2PR,对其降解特性和降解动力学进行了研究。菌株2PR对底物的耐受能力强,在2-羟基吡啶浓度为8mg/ml下仍然能够正常生长,并且2-羟基吡啶降解效率接近90%。本研究详细研究了菌株2PR在不同2-羟基吡啶浓度下生长和降解过程,并建立了菌株生长和降解动力学模型。利用HPLC和LC-MS等手段对其中间代谢产物进行检测,推测菌株2PR首先将2-羟基吡啶氧化生成2, 3, 6-三羟基吡啶,后者自动生成4, 5, 4′, 5′-tetrahydroxy-3, 3′-diazadiphenoquinone-(2, 2′)使得培养物产生蓝色;生成的2, 3, 6-三羟基吡啶发生吡啶环开环反应最终被逐步降解进入三羧酸循环。菌株2PR能够在较短的时间内将高浓度的2-羟基吡啶完全降解,表明该菌株在生物修复2-羟基吡啶污染物方面具有潜在应用价值。
1 材料与方法 1.1 样品和培养基本研究的样品取自辽河口湿地石油污染土壤。
无机盐(MSM)培养基(去离子水1 000ml):12.6g K2HPO4·3H2O,3.4g KH2PO4,1.0g Na2SO4,0.2g MgSO4·7H2O,微量金属盐溶液1ml;pH7.0,121℃灭菌20min。金属盐溶液(去离子水1 000ml):0.05g CaCl2·2H2O,0.05g CuCl2·2H2O,0.008g MnSO4·H2O,0.04g FeSO4·7H2O,0.05g ZnSO4,0.1g Na2MoO4·2H2O,0.05g Na2WO4·2H2O,0.038g CoCl2·6H2O,0.02g MnCl2·4H2O,0.0124g H3BO3。
富集培养基:无机盐培养基中加入1mg/ml的2-羟基吡啶、0.5g/L酵母粉和0.5g/L蛋白胨,121℃高压蒸汽灭菌20min。
2-羟基吡啶无机盐(MSM-2HP)液体培养基:MSM无机盐培养基中加入1mg/ml的2-羟基吡啶,121℃高压蒸汽灭菌20min。
MSM-2HP固体培养基:MSM-2HP液体培养基中加入1.5%琼脂粉。
1.2 试剂2-羟基吡啶(上海阿拉丁生化科技股份有限公司),基因组提取试剂盒[生工生物工程(上海)股份有限公司];2×Pfu Mix(天根生物科技有限公司),甲醇(上海安谱实验科技股份有限公司)。
1.3 仪器超净工作台(SW-CJ-2FD,苏州净化设备有限公司);恒温摇床(QYC 2112,上海福玛实验设备有限公司);PCR仪(T100 Thermal Cycler,Bio-Rad);离心机(GTR 22-1,北京时代北利离心机有限公司);紫外分光光度计(UV2310Ⅱ,上海天美公司);立式压力蒸汽灭菌锅(MLS-3750,SANYO);HPLC(Agilent 1100,美国Agilent公司);LC-MS(1290 Infinity /6460 UHPLC /MS/MS,美国Agilent公司)。
1.4 菌株2PR的16S rRNA基因序列分析用基因组提取试剂盒提取菌株2PR的全基因组,以其为模板,采用通用引物27F(5′-GAGTTTGATCATGGCTCAG-3′)和1492R(5′-GGTTACCTTGTTACGATC-3′),扩增菌株的16S rRNA基因序列。PCR反应体系:基因组DNA为模板1μl,2×Pfu Mix 12.5μl,引物27F 1μl,引物1 492 R 1μl,用超纯水补足至25μl。PCR反应条件:94℃预变性5min,94℃变性1min,55℃退火1min,72℃延伸2min,共30个循环;最后72℃延伸5min,4℃保温。
将PCR产物送至擎科新业生物技术有限公司进行测序。将获得的序列与其他模式微生物的16S rRNA基因序列利用MEGA 6.0软件Neighbour-Joining法构建系统发育进化树[13]。
1.5 菌株2PR生长条件优化菌株生长最适pH测定:分别配制不同pH值(5.0、6.0、7.0、8.0、9.0)的MSM-2HP培养基,按照1%的接种量,置于30℃,180r/min恒温振荡培养箱中培养,每隔6h取培养物1ml,9 000r/min离心1min;将沉淀用1ml蒸馏水重悬,测定600nm处的吸光值(OD600nm),以确定菌株Arthrobacter sp. 2PR生长的最适pH值,每组试验做3个重复。
菌株生长最适温度测定:选取最适pH值条件,按照1%的接种量,将其置于25℃、30℃、35℃和40℃恒温振荡培养箱(180r/min)中培养,每隔6h取样,测定OD600nm值,确定菌株Arthrobacter sp. 2PR生长的最适温度,每组试验做3个重复。
1.6 菌株2PR降解2-羟基吡啶特性研究将Arthrobacter sp. 2PR接种到MSM-2HP培养基中培养至对数后期,8 000r/min离心10min收集菌体,用PBS缓冲液重悬并洗涤菌体两次,最终菌体用PBS缓冲液重悬至OD600nm为6.0,作为休止细胞。加入终浓度为2mg/ml的2-羟基吡啶,在30℃ 120r/min下进行反应,每隔2h取样,用紫外可见光扫描200nm~400nm范围内的吸光值,研究菌株Arthrobacter sp. 2PR对2-羟基吡啶的降解情况。
1.7 菌株2PR降解2-羟基吡啶动力学分析按照1%的接种量将菌株2PR接种到不同浓度(0.4、0.8、1.2、1.6、2.0、3.0、4.0、6.0和8.0mg/ml)的MSM-2HP培养基中,30℃ 180r/min振荡培养,每12h取样测定菌株OD600nm值和2-羟基吡啶的浓度,构建菌株2PR降解2-羟基吡啶的动力学模型,每组试验做3个重复。利用Origin 8.0软件对菌株的生长动力学和底物降解动力学进行模拟。
1.8 2-羟基吡啶测定方法高效液相色谱仪(HPLC)检测法:色谱柱为Agilent Eclipse XDB-C18反相柱(5μm×4.6mm×250mm),流动相为10%甲醇和90% 1mmol/L H2SO4,进样量为2μl,柱温30℃,流速为1ml/min,检测器为DAD检测器,检测波长范围为200nm~400nm。
紫外分光光度计检测法:用紫外可见光扫描200nm~400nm范围内的吸光值。HPLC结果表明2-羟基吡啶在340nm处有特征吸收峰,且峰值大小跟2-羟基吡啶的含量存在正相关,因此可以用340nm处的吸光值变化表征2-羟基吡啶浓度的变化。
1.9 菌株2PR降解2-羟基吡啶代谢中间化合物检测利用安捷伦高效液相色谱仪检测底物的消耗和产物的生成。样品处理方法:将步骤1.6中取得的样品,加入2倍体积甲醇,4℃静置10min,10 000r/min离心2min,取上清液用0.22μm滤膜过滤后上机检测。
用高效液相色谱-质谱(LC-MS)对底物和产物进行分析。色谱柱为Agilent Eclipse XDB-C18反相柱(5μm×4.6mm×250mm),检测器为PDA,离子源为EIS。用水相的C18柱对样品进行分离,柱温为40℃,流动相A为超纯水,流动相B为甲醇溶液,流速为0.3ml/min。洗脱程序如下:0min到1min为15%流动相B,1min到4min为100%流动相B,4min到6min为100%流动相B,6min到6.01min为15%流动相B,6.01min到8min为15%流动相B。质谱扫描范围为m/z从50~500,氮气为雾化和干燥气体,分别收集阳离子模式和阴离子模式下的质谱信号。收集到的数据用Qualitative Analysis软件进行分析。
2 结果与分析 2.1 菌株2PR鉴定与形态学观察将菌株2PR在MSM-2HP固体培养基上进行划线,观察菌落形态为白色、圆形、边缘规则、湿润呈半透明状。菌株Arthrobacter sp. 2PR经碘液染色后用油镜观察不同生长时期均为短杆状。扫描电镜结果显示菌株为短杆状,长约为0.5μm~1μm,直径约为0.4μm~0.6μm,呈圆柱形且末端有辐射状花纹。
将菌株2PR的16S rRNA基因序列提交到NCBI(菌株2PR的序列注册号为KY203652)上进行BLAST比对,结果表明该菌株跟节杆菌属的亲缘关系最近。与Arthrobacter globiformis的序列相似性最高,在99%以上。综上所述,菌株2PR属于节杆菌属,将其命名为Arthrobacter sp. 2PR。
菌株2-PR能够以2-羟基吡啶为唯一碳源、氮源和能源进行生长,不能够利用4-羟基吡啶、烟酰胺、烟酸、2-氨基吡啶、2, 3-二羟基吡啶、2-吡啶甲酸、2, 4-二羟基吡啶、2, 5-二羟基吡啶、2, 6-二羟基吡啶等吡啶类化合物作为底物进行生长。
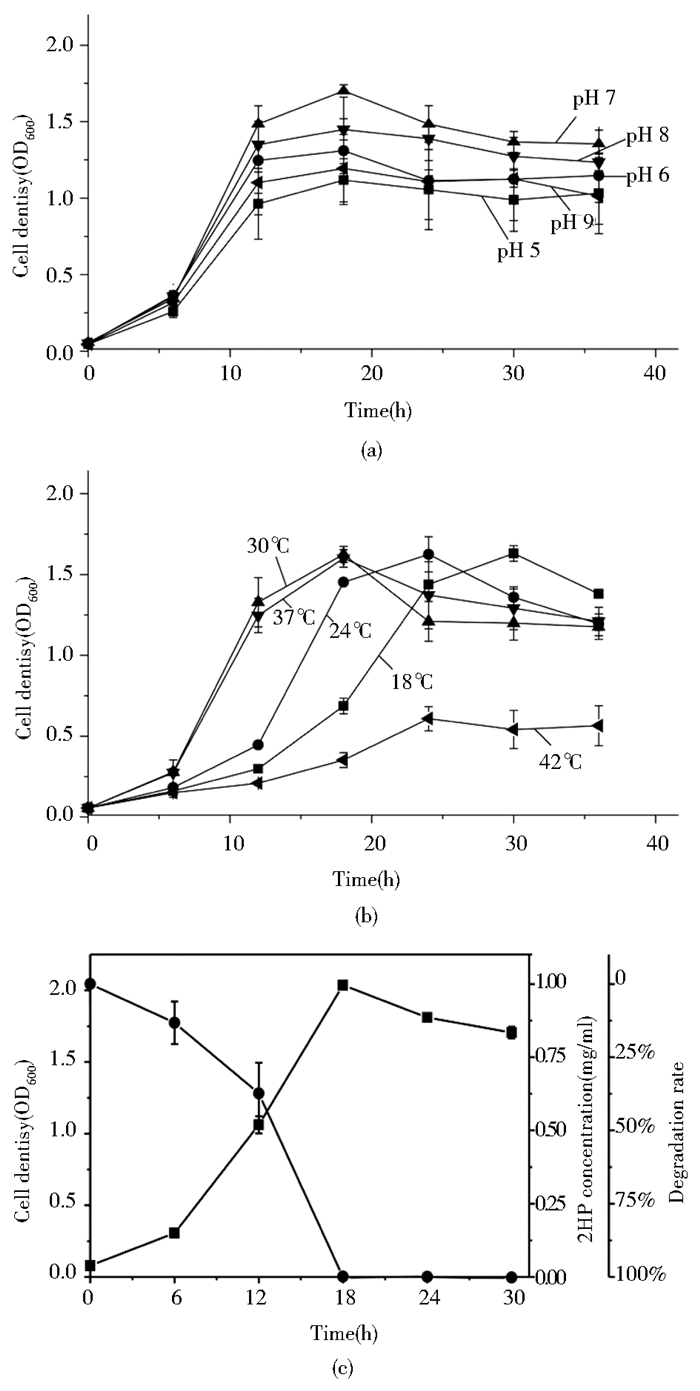
2.2 节杆菌2PR生长条件优化按照1%接种量,初始2-羟基吡啶浓度为1mg/ml,在不同pH的MSM-2HP培养基中培养菌株2PR,观察菌株2PR在不同条件下的生长情况。图 1a结果表明,节杆菌2PR在较宽的pH范围内都可以生长,在pH为7时菌株生长最好,其次是pH 8和pH 6,因此选择pH 7的培养基进行后续试验。
|
| 图 1 pH(a)和温度(b)对Arthrobacter sp. 2PR生长的影响以及在最适生长条件下的生长曲线和2HP降解曲线(c) Figure 1 Effect of pH (a) and temperature (b) on growth, and degradation curves (c) of Arthrobacter sp. 2PR under optimum growth conditions |
培养基pH值为7,分别将菌株2PR放置于不同温度18℃、24℃、30℃、37℃和42℃的恒温振荡培养箱中培养,转速为180r/min,研究温度对菌株2PR生长情况的影响。如图 1b所示,在30℃和37℃下菌株2PR生长速率最快,生长曲线基本重合。在18℃和23℃下生长较慢,但是也可以达到最大生长量,在42℃菌株生长十分微弱,本研究中选择30℃作为后续试验的培养温度。
在最优生长条件下,将节杆菌2PR按照1%接种量接种至MSM-2HP培养基,测定菌株2PR的生长量和2-羟基吡啶的降解效率。如图 1c所示,2-羟基吡啶随着菌体的生长被逐渐降解,在培养18h时,菌体浓度达到最大值,同时培养基中2-羟基吡啶被完全降解,降解效率达到100%。
2.3 菌株2PR降解2-羟基吡啶动力学分析为研究菌株2PR对于底物2-羟基吡啶的降解动力学,将菌株2PR接种至不同2-羟基吡啶浓度(0.4、0.8、1.2、1.6、2.0、3.0、4.0、6.0、8.0mg/ml)的MSM-2HP培养基中,将接种后的菌株置于30℃,180r/min振荡培养,每隔一段时间取样测定菌株浓度和2-羟基吡啶浓度。
如图 2a所示,菌株2PR在2-羟基吡啶浓度从0.4mg/ml~8.0mg/ml的无机盐培养基中均能较好地生长。一定范围之内2-羟基吡啶的浓度越高,细菌所能达到的最大生长浓度越高。菌株2PR在2-羟基吡啶浓度为6.0mg/ml的无机盐培养基中培养120h后,OD600nm值达到最高8.77。同时随着2-羟基吡啶浓度生长,细菌生长的延迟期越长,当2-羟基吡啶浓度为8.0mg/ml时,菌株最高生长浓度为OD600nm=7.77,小于6.0mg/ml时。这说明了2-羟基吡啶对菌株2PR具有一定的生物学毒性,菌株需要经过一段时间适应才能利用2-羟基吡啶进行生长并对其进行降解。

|
| 图 2 Arthrobacter sp. 2PR在不同初始2HP浓度的MSM-2HP培养基中的生长曲线(a)和降解曲线(b) Figure 2 Growth (a) and biodegradation curve (b) of strain Arthrobacter sp. 2PR under different 2HP concentration (0.4 mg/ml~8.0 mg/ml) |
描述微生物在限制条件下生长的模型主要是Mond方程和Logistic方程[14],这两个方程均不能适用于分析延迟期较长的菌体生长情况[15],从图 2可以看出,菌株2PR在MSM-2HP培养基中培养时延迟期较长,且随着底物浓度的提高,延迟期不断延长。在分析菌株生长动力学时,将延迟期的数据剔除,生长期和稳定期生长和降解的数据均符合Logistic方程(式中相关性参数值见表 1):
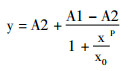 |
| Initial 2HP concentration/(mg/ml) | Lag time/h | Growth Parameters | Degradation Parameters | |||||||||
| A1 | A2 | X0 | P | R2 | A1 | A2 | X0 | P | R2 | |||
| 1 | 0 | 0.1 | 1.82 | 12.61 | 29.13 | 0.95 | 48.65 | 2.04 | 14.71 | 8.43 | 1.00 | |
| 2 | 0 | 0.23 | 4.11 | 35.70 | 115.05 | 0.90 | 89.65 | 1.91 | 35.6 | 30.59 | 0.99 | |
| 3 | 12 | 0.33 | 5.35 | 65.48 | 18.79 | 0.96 | 127.19 | -6.71 | 70.50 | 11.34 | 0.98 | |
| 4 | 48 | 0.43 | 6.00 | 73.17 | 41.27 | 0.88 | 184.47 | 3.39 | 77.54 | 14.45 | 1.00 | |
| 6 | 66 | 0.37 | 7.46 | 92.18 | 22.27 | 0.90 | 287.00 | -89.03 | 107.91 | 8.68 | 0.99 | |
| 8 | 84 | 0.23 | 6.86 | 102.45 | 31.3 | 0.97 | 365.71 | 29.09 | 120.88 | 15.79 | 0.99 | |
菌株2PR在不同浓度2-羟基吡啶的降解效率如图 3所示。在2-羟基吡啶浓度为1mg/ml时,在24h降解效率为100%;在2-羟基吡啶浓度为2mg/ml时,在42h降解效率为97.34%;在2-羟基吡啶浓度为3mg/ml时,在84h降解效率为96.75%;在2-羟基吡啶浓度为4mg/ml时,在96h降解效率为94.95%;在2-羟基吡啶浓度为6mg/ml时,在120h降解效率为94.48%;在2-羟基吡啶浓度为8mg/ml时,在156h降解效率为89.21%。随着底物浓度的提高,菌株2PR对2-羟基吡啶的降解效率没有下降。显示出了菌株2PR对2-羟基吡啶强大的耐受能力和降解能力,表明该菌株在2-羟基吡啶污染生物修复应用方面具有较大潜力。
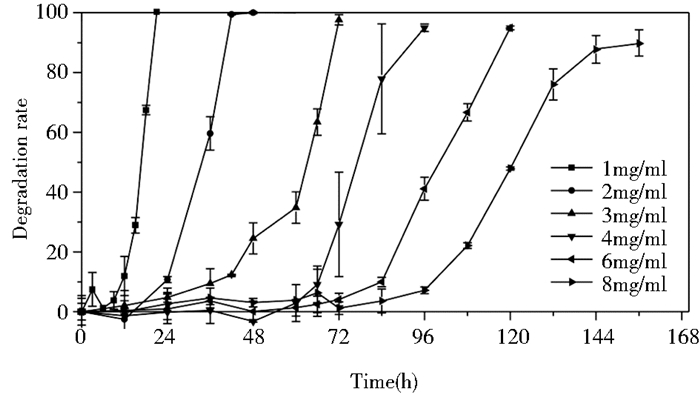
|
| 图 3 不同浓度2HP下Arthrobacter sp. 2PR的降解效率 Figure 3 Degradation efficiency of Arthrobacter sp. 2PR under different concentrations of 2HP |
利用MSM-2HP培养基培养菌株2PR,培养物的颜色随时间不同依次呈现淡紫色、蓝紫色和棕黄色如图 4所示,这表明2-羟基吡啶在代谢过程中产生了其他的吡啶类中间代谢产物。该蓝色物质在吡啶类化合物代谢中十分常见,推测该蓝色中间代谢产物可能是4, 5, 4′, 5′-tetrahydroxy-3, 3′-diazadiphenoquinone-(2, 2′)[11]。

|
| 图 4 菌株2PR在MSM-2HP培养基中随时间产生不同颜色变化 Figure 4 The colour of strain 2PR culture changed at different time in MSM-2HP medium |
利用菌株2PR的休止细胞对2HP进行降解,每隔2h取样进行紫外全波长扫描,结果如图 5a所示,0h的样品在220nm和295nm处有吸收峰,随着反应时间的延长,这两个吸收峰的峰值不断变小,这表明2HP被不断降解。但是并没有其他新峰出现,这表明2HP第一步反应是开环反应或者最初反应生成的吡啶环中间代谢产物迅速发生开环反应,因此从全波长扫描图谱中检测不到。
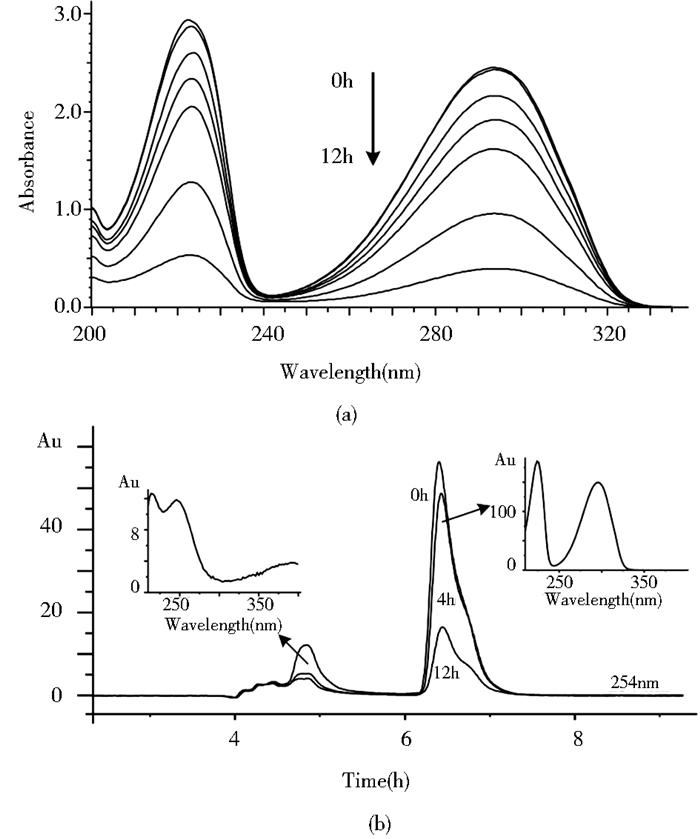
|
| 图 5 全波长扫描(a)和HPLC(b)检测2HP降解休止细胞反应 Figure 5 UV absorption spectra (a) and HPLC (b) analysis of 2HP degraded by resting cells of Arthrobacter sp. 2PR 全波长扫描图谱从上到下依此是0h、2h、4h、6h、8h、10h、12h取样样品,HPLC检测0h、4h、12h的样品 |
将休止细胞反应样品用HPLC分析底物变化和产物生成情况,0h样品在6.5min左右有一个特征峰(图 5b),光谱信号显示该化合物在220nm和295nm处有特征吸收峰,与标准品比较可以确定该峰为底物2HP。休止细胞反应4h和12h样品的HPLC信号可以看出,底物2-羟基吡啶的信号峰下降,在4min 55s出现一个新的吸收峰,光谱学信号显示该产物在245nm处有一个特征吸收峰,另外在大于400nm处有吸收峰,推测可能是2-羟基吡啶降解的中间代谢产物。
将休止细胞反应0h和12h的样品利用LC-MS进行检测,结果(图 6)表明0h的样品在阳离子模式下有信号峰(m/z=96.03),跟2HP标准品的保留时间和分子量一致确定该峰为底物2HP;休止细胞反应12h样品,在阳离子模式下仍有m/z=96.03的信号峰,另外在阴离子模式下出现一个新的信号峰(m/z=249)峰出现,结合培养基反应颜色变化、HPLC光谱学信号可以确定该信号峰属于4, 5, 4′, 5′-tetrahydroxy-3, 3′-diazadiphenoquinone-(2, 2′),该化合物是由2, 3, 6-三羟基吡啶自动氧化生成。
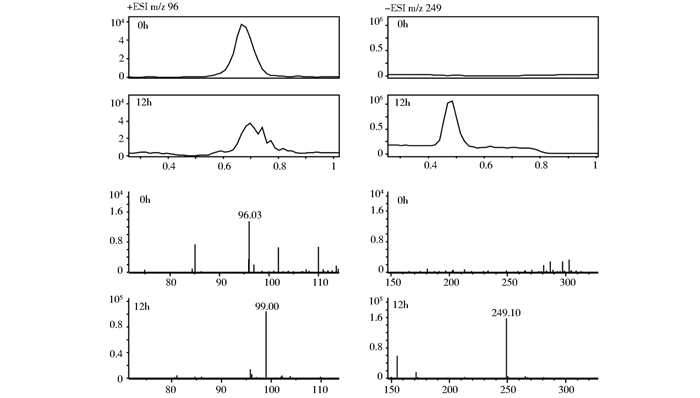
|
| 图 6 菌株Arthrobacter sp. 2PR降解2HP质谱图 Figure 6 LC-MS results of 2-hydroxypyridine degradation by Arthrobacter sp. 2PR |
本研究从辽河口湿地石油污染土壤中筛选到一株能够以2-羟基吡啶为唯一碳源、氮源和能源进行生长的菌株,经形态学和16S rRNA基因进化分析表明该菌株属于节杆菌属,命名为Arthrobacter sp. 2PR。菌株2PR的最适生长温度为30℃和最适pH为7.0。菌株2PR对于2-羟基吡啶的耐受性很高,能够在高达8mg/ml的2-羟基吡啶浓度下生长,并且降解效率接近90%。当浓度小于8mg/ml的时候,菌株2PR对2-羟基吡啶的降解效率可以达到95%。在2-羟基吡啶浓度为0.4~8.0mg/ml范围内菌株2PR的生长和降解过程均符合Logisitic模型。随着底物2-羟基吡啶浓度的升高,菌体生长的延迟期也随着增长,这可能是高浓度的2-羟基吡啶对细胞生长有一定的抑制作用,通过一段时间的适应细胞能够消除这种抑制作用,因此虽然菌体生长表现出一定时间的延迟但是降解效率并没有降低。
在MSM-2HP培养基中菌株2PR的培养物随着时间颜色不断变化,首先产生蓝色物质随后培养物变成棕色。通过HPLC和LC-MS可以确定该蓝色物质为4, 5, 4′, 5′-tetrahydroxy-3, 3′-diazadiphenoquinone-(2, 2′),推测是由2, 3, 6-三羟基吡啶自动氧化生成。催化2-羟基吡啶第一步反应的酶,在菌株Arthrobacter spp.中报道为单加氧酶[7],在菌株Achromobacter sp. [9]和Rhodococcus rhodochrous PY11[8]中为双加氧酶。考虑到在休止细胞反应中并没有检测到其他吡啶环中间代谢产物,因此可以推测菌株2PR第一步催化的酶也是双加氧酶,经过该酶催化生成了2, 3, 6-三羟基吡啶,经过进一步的开环反应,最终氧化成α-酮戊二酸进入三羧酸循环彻底降解[8]。Gasparaviciute等[16]筛选到的另外一株节杆菌菌株Arthrobacter sp. IN13能够同时利用4-羟基吡啶和2-羟基吡啶,但是诱导顺序对降解能力有很大影响,菌株Arthrobacter sp. IN13在2-羟基吡啶的培养基中培养后会丧失利用4-羟基吡啶的能力。而菌株2PR不能利用4-羟基吡啶,因此推测菌株2PR的吡啶化合物的降解基因与菌株IN13不同。菌株2PR降解2-羟基吡啶的分子机理还不清楚,需要后续实验继续研究。
随着人们对环境问题的关注,高效、温和处理环境中有机污染物的研究更具有重要意义。本研究分离获得了一株高效降解2-羟基吡啶的菌株,该菌株是目前已报道降解2-羟基吡啶能力最强的菌株,此外本研究对其降解动力学和降解特性也进行了研究。这将为该类污染物的处理和污染环境修复提供理论基础。
致谢 本项目获得了青岛农业大学人才科研启动基金(No. 6631115052) 和青岛农业大学大学生创新立项项目的资助,特此致谢。| [1] |
Kaiser J P, Feng Y, Bollag J M. Microbial metabolism of pyridine, quinoline, acridine, and their derivatives under aerobic and anaerobic conditions. Microbiology Reviews, 1996, 60(3): 483-498. |
| [2] |
Yu H, Hausinger R, Tang H, et al. Mechanism of the 6-hydroxy-3-succinoyl-pyridine 3-monooxygenase flavoprotein from Pseudomonas putida S16. Journal of Biological Chemistry, 2014(289): 29158-29170. |
| [3] |
Yao Y, Tang H, Ren H, et al. Iron(Ⅱ)-dependent dioxygenase and N-formylamide deformylase catalyze the reactions from 5-hydroxy-2-pyridone to maleamate. Scientific Report, 2013, 3(11): 3235. |
| [4] |
Semenaite R, Gasparaviciute R, Duran R, et al. Genetic diversity of 2-hydroxypyridine-degrading soil bacteria. Biologija, 2003, 2: 27-30. |
| [5] |
Sun J Q, Xu L, Tang Y Q, et al. Bacterial pyridine hydroxylation is ubiquitous in environment. Applied Microbiology and Biotechnology, 2014, 98(1): 455-464. DOI:10.1007/s00253-013-4818-9 |
| [6] |
Kolenbrander P E, Lotong N, Ensign J C. Growth and pigment production by Arthrobacter pyridinolis n. sp. Archives of Microbiology, 1976, 110(2-3): 239-245. DOI:10.1007/BF00690233 |
| [7] |
Kolenbrander P E, Weinberger M. 2-Hydroxypyridine metabolism and pigment formation in three Arthrobacter species. Journal of Bacteriology, 1977, 132(1): 51-59. |
| [8] |
Vaitekunas J, Gasparaviciute R, Rutkiene R, et al. A 2-hydroxypyridine catabolism pathway in Rhodococus rhodochrous strain PY11. Applied and Environmental Microbiology, 2016, 82(4): 1264-1273. DOI:10.1128/AEM.02975-15 |
| [9] |
Cain R B, Houghton C, Wright K A. Microbial metabolism of the puridine ring. Metabolism of 2-and 3-hydroxypyridines by the maleamate pathway in Achromobacter sp.. Biochemical Journal, 1974, 140(2): 293-300. DOI:10.1042/bj1400293 |
| [10] |
Shukla O P, Kaul S M. Microbiological transformation of pyridine N-oxide and pyridine by Nocardia sp.. Canadian Journal of Microbiology, 1986, 32(4): 330-341. DOI:10.1139/m86-065 |
| [11] |
Knackmuss H J, Beckmann W. The structure of nictine blue from Arthrobacter oxidans. Arch. Mikrobiol, 1973, 90(2): 167-169. DOI:10.1007/BF00414521 |
| [12] |
Kaiser J P, Feng Y, Bollag J M. Microbial metabolism of pyridine, quinoline, acridine, and their derivatives under aerobic and anaerobic conditions. American Society for Microbiology, 1996, 60(3): 483-498. |
| [13] |
Pearson W R, Lipman D J. Improved tools for biological sequence comparison. Proceedings of the National Academy of Science of the United States of America, 1988, 85(8): 2444-2448. DOI:10.1073/pnas.85.8.2444 |
| [14] |
Gabriel J P, Saucy F, Bersier L F. Paradoxes in the logisitic equation. Ecological Modelling, 2005, 185(1): 147-151. DOI:10.1016/j.ecolmodel.2004.10.009 |
| [15] |
宋健, 林建群, 金燕, 等. 以比生长速率时间曲线为基础的生物群体生长数学模型. 微生物学通报, 2007, 34(5): 836-838. Song J, Lin J Q, Jin Y, et al. A new population growth model based on the time dependent changes of the specific growth rate. Microbiology China, 2007, 34(5): 836-838. |
| [16] |
Gasparaviciute R, Kropa A, Meskys R. A new Arthrobacter strain utilizing 4-hydroxypyridine. Biologija, 2006, 4: 41-45. |
 2017, Vol. 37
2017, Vol. 37




