文章信息
- 温国霞, 黄子豪, 谭俊杰, 阚乃鹏, 凌婧怡, 张霞, 刘刚, 陈惠鹏.
- WEN Guo-xia, HUANG Zi-hao, TAN Jun-jie, KAN Nai-peng, LING Jing-yi, ZHANG Xia, LIU Gang, CHEN Hui-peng.
- 以大肠杆菌为底盘细胞构建XylR-Pu线路检测2, 4, 6-三硝基甲苯
- Construction of XylR-Pugene Lines in Escherichia coli to Detect 2, 4, 6-trinitrotoluene
- 中国生物工程杂志, 2017, 37(7): 105-114
- China Biotechnology, 2017, 37(7): 105-114
- http://dx.doi.org/DOI:10.13523/j.cb.20170717
-
文章历史
- 收稿日期: 2017-01-23
- 修回日期: 2017-03-07
2. 军事医学科学院 北京 100071;
3. 成都军区总医院 成都 610083;
4. 福建省疾病预防控制中心 福州 350001;
5. 南京军区福州总医院 福州 350001
2. Academy of Military Medical Sciences, Beijing 100071, China;
3. Chengdu Military General Hospital, Chengdu 610083, China;
4. Fujian CenterFor Disease Control & Prevention, Fuzhou 350001, China;
5. Fuzhou General Hospital of Nanjing Military Command, Fuzhou 350001, China
2, 4, 6-三硝基甲苯(TNT)是一种带苯环的有机化合物,因其良好的爆炸性能,常被用来制造地雷及各种炸药。地雷的广泛使用及TNT的长效性,导致战争遗留下来的地雷每年都造成上百人的伤亡,严重威胁着人们的生命安全。同时泄露在环境中的TNT还是一种重要的环境污染物,它可以渗入到土壤及水系统中,长期参与生态循环;而且TNT及其降解物还可以进入食物链进而引发急性中毒,其对生态环境和人类健康造成了极大威胁[1]。研发高效、准确的检测TNT的工具,对战后雷场清除及环境治理都有重要意义。
TNT在工业生产过程中会产生多种不可避免的副产物,如1, 3-二硝基苯(1, 3-DNB)、2, 4-二硝基甲苯(2, 4-DNT)及2, 6-二硝基甲苯(2, 6-DNT),因此在检测TNT时,1, 3-DNB、2, 4-DNT及2, 6-DNT等化合物也可作为检测的靶标[2]。目前传统的TNT检测方法有高效液相色谱(HPLC)、紫外检测(UV)、气相色谱-质谱(GC-MS)、激光表面增强拉曼光谱(SERS)、核磁共振(NMR)和离子迁移法等[3-4],但这些方法大都需要昂贵的仪器和繁复的样品制备方法以及专业的操作流程,在实际应用中会受到很大的限制。目前基于合成生物学思想构建生物传感器成为研究热点[5-6],由于生物传感器具有成本低、敏感度高及易操作等优点,因此为TNT检测提供了新的思路,特别适合于战后雷场的大规模检测,具备良好的发展前景[7-8]。
恶臭假单胞菌MT-2菌株携带的TOL质粒具有经典的甲苯代谢通路,常被广泛研究用来构建生物传感器实现甲苯、苯、乙苯及二甲苯的检测[9-11]。在甲苯类化合物存在时,其中的调控蛋白XylR可以特异性的激活Pu启动子,进而引发甲苯代谢通路的表达(图 1a)。原理大致是:反式作用因子XylR蛋白属于NtrC家族成员,其由4部分组成,A区是诱导物的结合区,结合诱导物后构象改变,通过短的连接区域(B区),激活C区活性,C区结构高度保守,负责协调ATP的水解,并通过与依赖σ54因子的RNA聚合酶相互作用起始转录;D区的螺旋-转角-螺旋结构辅助其结合到Pu启动子上,最终激活Pu活性[12-15](图 1b)。
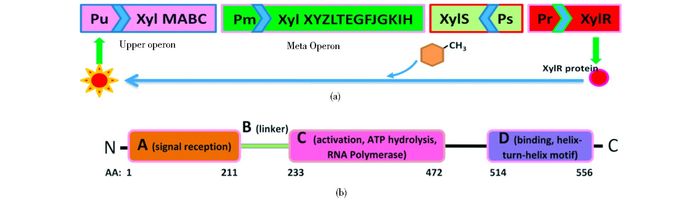
|
| 图 1 TOL质粒甲苯代谢通路(a)及XylR蛋白结构(b) Figure 1 Toluene degradation pathway of TOL plasmid (a)and domain organization of XylR (b) |
由于XylR蛋白的天然诱导剂是甲苯,其对硝基甲苯没有明显感应,研究者[11]通过将XylR蛋白的A区域与其NtrC家族的同源蛋白DmpR(其可结合苯酚类化合物从而激活Po启动子启动苯酚代谢通路的表达)进行DNA shuffling,得到了可感应2, 4-DNT的XylR突变体蛋白XylR5,并且构建XylR5-Pu基因线路在模拟雷场中首次实现了对爆炸物的定位。但XylR5蛋白的感应灵敏度很低,环境中的2, 4-DNT浓度不低于364mg/L时才能激活Pu启动子,而且XylR5蛋白的特异性较差,其对甲苯的感应强度要高于TNT。
为了更好地将XylR5-Pu基因线路应用于TNT的检测,我们以pETDuet-1为载体骨架,构建了XylR-Pu和XylR5-Pu两条基因线路,并以绿色荧光蛋白(GFP)为报告分子,GFP的荧光值可以指征结合诱导剂后的XylR蛋白对Pu启动子的诱导强度,并将其导入遗传背景研究清楚、操作简单的大肠杆菌细胞中构建全细胞生物传感器,用于检测环境中的污染物TNT。通过对比两条线路的荧光值,既可以验证基因线路在大肠杆菌中是否发挥功能,又可以验证XylR5突变体在大肠杆菌中对TNT是否仍具有较好的感应强度。最后通过对基因线路的优化设计,以及对XylR蛋白的A区的随机突变,从中筛选出对靶标化合物TNT有更高感应强度、灵敏度及特异性的蛋白质元件,并构建性能更加优良的基因线路。
1 材料与方法 1.1 材料2, 4, 6-三硝基甲苯(TNT),2-硝基甲苯(2-NT),3-硝基甲苯(3-NT),4-硝基甲苯(4-NT),1, 4-二硝基苯(1, 4-DNB),1, 3-二硝基苯(1, 3-DNB),2, 4-二硝基甲苯(2, 4-DNT),2, 6-二硝基甲苯(2, 6-DNT),甲苯(Toluene),苯(Benzene),硝基苯(Nitrobenzene)均购自Sigma-Aldrich公司;质粒pETDuet-1为本实验室保存;感受态DH5α及BL21(DE3) 购于天根生物科技有限公司;Q5 DNA聚合酶、限制性内切核酸酶及T4 DNA连接酶均购自NEB公司;96孔深孔板、黑色96孔荧光板均购自Thermo公司。
1.2 基因线路的构建 1.2.1 目的基因序列的获得从NCBI网站上获取XylR蛋白(1 701bp)及Pu启动子(234bp)的基因序列,由北京奥科鼎盛生物技术有限公司合成2段序列并分别连接在pMD18-T载体上,分别命名为pMD18T-XylR和pMD18T-Pu。
通过在NCBI上查询XylR与DmpR的序列,得到了XylR5的核酸序列,合成后克隆到pGH载体上,命名为pGH-XylR5。
质粒pPROBE-TT上含有783bp四串联的终止序列4×Terminator,人工合成此四串联序列,并在两端分别加上Hind Ⅲ和Not Ⅰ酶切位点,克隆到pGH载体上,命名为pGH-4Ter。
1.2.2 PCR扩增目的片段实验涉及的引物见表 1。按照常规PCR方法,利用Q5 DNA聚合酶组成PCR体系,以pMD18T-XylR为模板,使用引物X0-B和X0-N进行PCR,扩增目的片段XylR;以pGH-XylR5为模板,使用引物X5-B和X5-N进行PCR,扩增目的片段XylR5;以pMD18T-Pu为模板,使用引物Pu-BsrG Ⅰ和PU-Nde Ⅰ进行PCR,扩增启动子Pu序列;以质粒pPROBE-TT为模板,使用引物GFP-KpnⅠ和GFP-Avr Ⅱ进行PCR,扩增质粒上的GFP基因。
| Primer name | Sequence(5′-3′) | Restriction size |
| X0-B | CAGGATCCAATGTCGCTTACATACAAACCC | BamHⅠ |
| X0-N | TAGCGGCCGCCTATCGGCCCATTG | NotⅠ |
| X5-B | GGGGGGATCCAATGTCGCTTACATACAAA | BamHⅠ |
| X5-N | TAGCGGCCGCCTATCGGCCCATTG | NotⅠ |
| PU-BsrGⅠ | ATTGTACACGGCCGCATAATCGAAATGGAAAGCGCGATGAACCTTTTTTA | BsrGⅠ |
| PU-NdeⅠ | GCCATATGTATATCTCCTTCTTATACTTAACTAATATACTAAGATGGTGAAGGGTCA CCACTATTTTTATTTTAAGTCTCGTATA | NdeⅠ |
| GFP-KpnⅠ | TCGGTACCATGAGTAAAGGAGAAGAA | KpnⅠ |
| GFP-AvrⅡ | GGCCTAGGCTATTTGTATAGTTCATCC | AvrⅡ |
| Error-PstⅠ | GTCTGCAGCTCGTAGCGCTCG | PstⅠ |
1.2.3 基因线路X0-Pu-GFP-Duet及X5-Pu-GFP-Duet的构建
以pETDuet-1为基础载体,用BamH Ⅰ/Not Ⅰ对载体和扩增片段XylR分别进行双酶切,连接得到正确的载体pX0-Duet,其中XylR基因插入在pETDuet-1的第一个T7启动子控制的多克隆位点(MCS)内。
以构建的pX0-Duet为基础载体,用BsrG Ⅰ/Nde Ⅰ对载体和扩增片段Pu进行双酶切,连接得到正确的载体pX0-Pu-Duet,其中Pu启动子替换掉pETDuet-1的第二个T7启动子。
以构建的pX0-Pu-Duet为基础载体,用Kpn Ⅰ/Avr Ⅱ对载体和扩增片段GFP进行双酶切,连接得到正确的载体pX0-Pu-GFP-Duet,其中GFP插入在有Pu启动子控制的第二个MCS内(图 2a)。
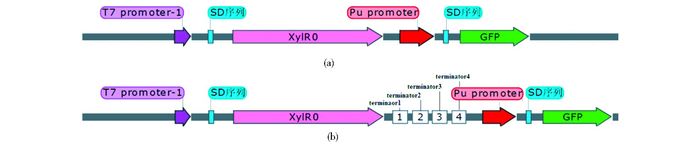
|
| 图 2 基因线路图谱 Figure 2 The map of gene lines (a) pX0-Pu-GFP-Duet (b) pX0-4Ter-Pu-GFP-Duet |
以上述同样的方法,用XylR5片段替代XylR片段,得到载体pX5-Pu-GFP-Duet。
1.2.4 SDS-PAGE检测基因线路的蛋白质表达将质粒pX0-Pu-GFP-Duet或pX5-Pu-GFP-Duet转化大肠杆菌后,37℃、200r/min培养至OD600约0.6时,加入终浓度为1μmol的IPTG,30℃、200r/min培养12h后,取样进行电泳分析。
1.2.5 基因线路X0-4Ter-Pu-GFP-Duet及X5-4Ter-Pu-GFP-Duet的构建分别将pX0-Pu-GFP-Duet及pGH-4Ter载体用Hind Ⅲ/Not Ⅰ双酶切后连接,得到在XylR序列末端带有4串联终止序列的的载体pX0-4Ter-Pu-GFP-Duet(图 2b)。以同样的方法,得到载体pX5-4Ter-Pu-GFP-Duet。
1.3 基因线路检测TNT挑取pX0-Pu-GFP-Duet或pX5-Pu-GFP-Duet单克隆过夜培养后,百倍稀释至新鲜LB培养基,并将其分为三组。37℃、200r/min培养至OD600约0.6时,于第一组加入终浓度为1μmol的IPTG和终浓度为15mg/L的TNT;第二组加入终浓度为1μmol的IPTG;第三组为阴性对照。30℃、200r/min培养12h后,各吸取200μl菌液至黑色96孔荧光板,在PekinElmer Multimode Plate Reader中测定荧光值(检测条件为excitation/emission, 488/507nm)。
基因线路X0-4Ter-Pu-GFP-Duet及X5-4Ter-Pu-GFP-Duet检测TNT的方法与步骤同上。
1.4 易错PCR构建XylR突变体文库以X0-B和Error-Pst Ⅰ为引物,pX0-4Ter-Pu-GFP-Duet为模板,利用GeneMorph Random Mutagenesis Kit (cat:#200550,Agilent Technologies)对XylR基因的A区进行易错PCR。按照说明书配制扩增体系,扩增条件为:95℃预变性2min,然后以95℃变性30s、50℃退火30s、72℃延伸2min扩增35个循环,72℃延伸10min。回收目的产物,以此为模板,进行第二轮易错PCR,PCR体系和程序不变;然后再以第二轮胶回收产物为模板,进行第三轮易错PCR,回收第三轮易错PCR产物。
pX0-4Ter-Pu-GFP-Duet和第三轮易错PCR产物分别用BamH Ⅰ/Pst Ⅰ进行双酶切,连接产物转化大肠杆菌,构建XylR突变体文库。
1.5 文库筛选挑取突变体文库中的单克隆至96孔深孔板进行培养,37℃、200r/min培养至OD600约0.6时每孔加入终浓度为1μmol的IPTG和终浓度为15mg/L的TNT,30℃、200r/min培养12h后,每孔吸取200μl菌液测定荧光值,将荧光值高于XylR5的菌挑选出来。
将挑选出的菌液百倍稀释进行培养,分为三组,第一组在OD600约0.6时加入终浓度为1μmol的IPTG和终浓度为15mg/L的TNT;第二组加入终浓度为1μmol的IPTG;第三组为阴性对照;30℃、200r/min培养12h后,测定荧光值。将三组荧光值有显著差异(P<0.05) 的克隆挑选出来。
1.6 特异性检测将荧光值有显著差异的单克隆接种于LB培养基,37℃、200r/min培养至OD600约0.6时加入终浓度为1μmol的IPTG,按照国际公认的检测方法[16],分别加入终浓度为15mg/L的如下10种目标化合物:2, 4, 6-TNT、2, 4-DNT、2, 6-DNT、1, 3-DNB、1, 4-DNB、2-NT、3-NT、4-NT、Toluene和Nitrobenzene,30℃、200r/min振荡培养12h后,测定荧光值。挑选对TNT或其衍生物2, 4-DNT、2, 6-DNT及1, 3-DNB的反应强度较其他化合物有明显差别(P<0.05) 的克隆。
1.7 灵敏度检测将筛选得到的阳性克隆与pX0-4Ter-Pu-GFP-Duet、pX5-4Ter-Pu-GFP-Duet分别接种至LB培养基,37℃、200r/min培养至OD600约0.6时加入终浓度1μmol的IPTG,并分别加入终浓度为0mg/L、5mg/L、10mg/L、15mg/L、20mg/L、30mg/L、50mg/L、75mg/L、100mg/L、150mg/L、200和300mg/L的TNT,然后30℃、200r/min振荡培养12h后分别测定荧光值,通过计算元件的EC200值,考察元件对TNT的感应灵敏度。
1.8 蛋白质结构分析将筛选得到的阳性克隆送交测序,并将序列提交至PSIPRED(http://bioinf.cs.ucl.ac.uk/psipred/)进行二级结构预测和SWISS-MODEL (http://swissmodel.expasy.org/)进行同源模建,对蛋白质结构进行比对分析。
2 结果 2.1 基因线路X0-Pu-GFP-Duet及X5-Pu-GFP-Duet的鉴定构建的重组质粒pX0-Pu-GFP-Duet和pX5-Pu-GFP-Duet经提取纯化后,分别利用限制性内切核酸酶BamH Ⅰ/Not Ⅰ、BamH Ⅰ/Not Ⅰ、Kpn Ⅰ/Avr Ⅱ进行双酶切,结果均获得了与预期大小相符的目的条带(图 3),说明基因线路X0-Pu-GFP-Duet和X5-Pu-GFP-Duet构建成功。
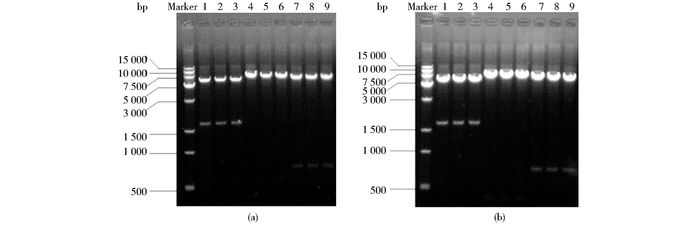
|
| 图 3 重组质粒酶切鉴定 Figure 3 Identification of recombinantplasmidsbyagarose gel electrophoresis (a) pX0-Pu-GFP-Duet (b) pX5-Pu-GFP-Duet 1-3:BamH Ⅰ/Not Ⅰ; 4-6:BamH Ⅰ/Not Ⅰ; 7-9:Kpn Ⅰ/Avr Ⅱ; Marker:Trans 15K DNA marker |
SDS-PAGE结果表明,加入IPTG诱导后,特异性表达的XylR和XylR5蛋白分子质量大小与预期相符(图 4),说明T7启动子可以启动XylR和XylR5蛋白的正常表达。
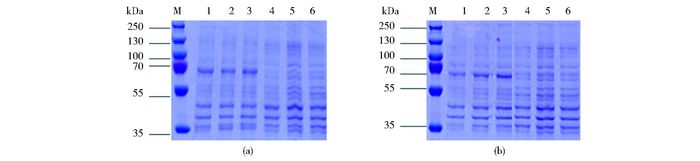
|
| 图 4 SDS-PAGE鉴定XylR/XylR5蛋白表达 Figure 4 Identification of expression of XylR/XylR5 by SDS-PAGE (a) XylR (b) XylR5 M:Protein marker; 1-3:1μmol IPTG; 4-6:Control |
将pX0-Pu-GFP-Duet及pX5-Pu-GFP-Duet分别转化大肠杆菌,构建成全细胞生物传感器,用于检测TNT。结果发现,诱导表达的XylR/XylR5蛋白在无TNT时也能启动下游GFP的表达,而且基因线路的本底表达值很高(图 5)。分析原因可能是由于T7强启动子造成了线路通读,为了抑制通读、降低本底表达,尝试在XylR与Pu之间插入四串联终止序列。
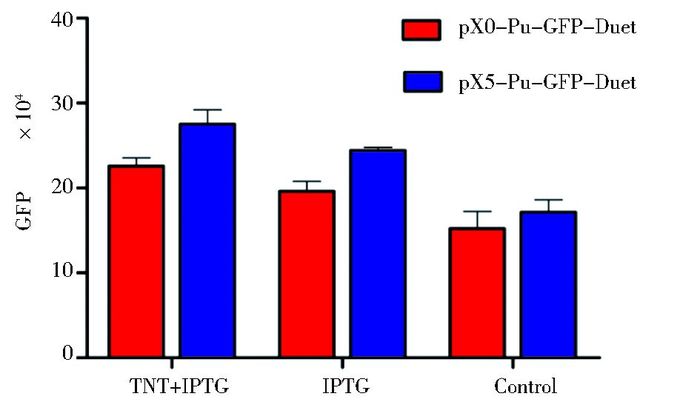
|
| 图 5 pX0-Pu-GFP-Duet及pX5-Pu-GFP-Duet检测TNT Figure 5 Detection of TNT by pX0-Pu-GFP-Duet and pX5-Pu-GFP-Duet |
构建的重组质粒pX0-4Ter-Pu-GFP-Duet和pX5-4Ter-Pu-GFP-Duet分别用Hind Ⅲ/Not Ⅰ进行双酶切,结果表明带有四串联终止序列的基因线路构建成功(图 6)。
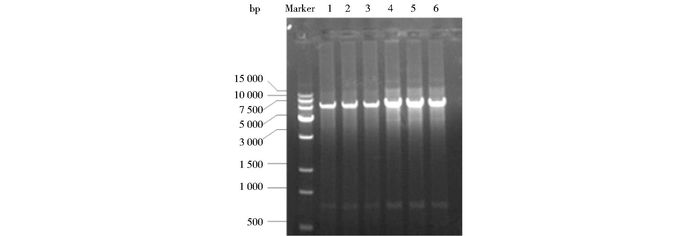
|
| 图 6 重组质粒pX0-4Ter-Pu-GFP-Duet及pX5-4Ter-Pu-GFP-Duet酶切鉴定 Figure 6 Identification of recombinant plasmids by agarose gel electrophoresis 1-3:pX0-4Ter-Pu-GFP-Duet; 4-6:pX5-4Ter-Pu-GFP-Duet |
将基因线路X0-4Ter-Pu-GFP-Duet及X5-4Ter-Pu-GFP-Duet经过相同的步骤检测TNT,结果表明在基因线路中加入四串联终止序列可以有效阻止通读,降低本底表达(图 7a)。XylR5蛋白较XylR蛋白对TNT的感应强度高,但两者对TNT的感应效果都不显著,特异性较差(图 7b)
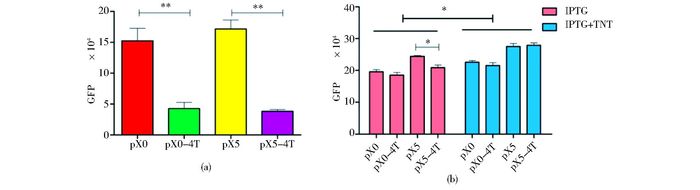
|
| 图 7 pX0-4Ter-Pu-GFP-Duet及pX5-4Ter-Pu-GFP-Due检测TNT Figure 7 Detection of TNT by pX0-4Ter-Pu-GFP-Duet and pX5-4Ter-Pu-GFP-Duet (a) Comparison of background values after optimization (b) Comparison of induction strength of gene lines * P < 0.05 ** P < 0.01 |
通过引物X0-B和Error-Pst Ⅰ,利用Agilent易错试剂盒对XylR基因的A区进行连续三轮易错PCR,BamH Ⅰ/Pst Ⅰ双酶切第三轮PCR产物,然后连入经相同酶切后的载体pX0-4Ter-Pu-GFP-Duet,转化大肠杆菌,构建突变体文库。由于BamH Ⅰ/Pst Ⅰ位于A区边缘,因此可以精确控制只对蛋白质A区进行随机突变。
挑取突变体文库中的单克隆至96孔深孔板,先通过在终浓度为15mg/L的TNT条件下,筛选荧光值高于XylR5(28×104)的克隆。为了去除假阳性,再将筛选出来的克隆经过三组不同处理,第一组加入1μmol的IPTG和终浓度为15mg/L的TNT,第二组只加入1μmol的IPTG,第三组为阴性对照,将在三组中荧光值有显著差异的克隆挑选出来,最后得到11个,其中的克隆eX0-4在TNT中的荧光值较其余两组有极显著性差异(图 8a)。
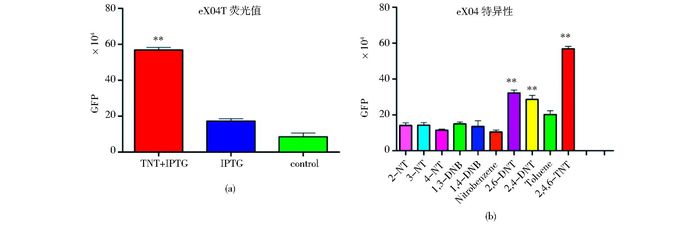
|
| 图 8 基因线路eX0-4检测TNT及其特异性研究 Figure 8 Detection of TNT by peX0-4 and the study of its specificity (a) TNT fluorescence detection (b) Specificity study ** P < 0.01 |
将筛选出来的11个XylR突变体对十种苯类化合物的感应强度进行比较,筛选得到了1株能特异性感知TNT的阳性克隆eX0-4,如图 8(b),其对TNT及其衍生物DNB的感应强度较其余化合物达到了极显著性差异,表现出了对靶标化合物良好的特异性。
2.8 灵敏度检测EC200值是目前构建生物传感器领域比较通用和认可的检测灵敏度的指标,具体是指感应元件引发的荧光值的倍数变化能达到初始荧光值2倍时所需的最低诱导物浓度,EC200值越小,说明感应元件的灵敏度越高。
我们将pX0-4Ter-Pu-GFP-Duet、pX5-4Ter-Pu-GFP-Duet及阳性克隆peX0-4分别接种至LB培养基,培养至OD600约为0.6时加入浓度呈梯度上升的TNT中,振荡培养12h后测定荧光值,如图 9所示,pX0-4Ter-Pu-GFP-Duet对TNT没有明显感应,无法计算EC200;pX5-4Ter-Pu-GFP-Duet的EC200值为61.3±1.56mg/L,peX0-4的值为(12±5.21) mg/L,可见突变体eX0-4对TNT的感应灵敏度大幅度提高,其EC200值达到了国际上报道同类元件的领先水平。并且TNT浓度在15~75mg/L时,pX5-4Ter-Pu-GFP-Duet及peX0-4表现出了浓度依赖性,而随着TNT浓度不断增加荧光值下降,原因是高浓度的TNT严重损害了细菌的正常生长。
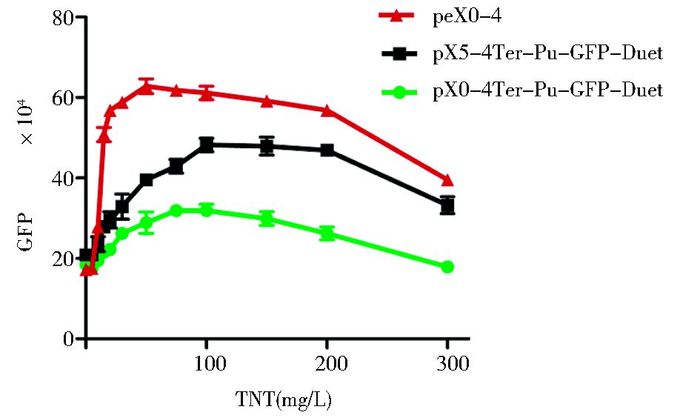
|
| 图 9 基因线路对不同浓度TNT的感应强度 Figure 9 Induction intensity of gene lines to different concentrations of TNT |
通过测序分析,突变体eX0-4较野生型XylR在A区有12个碱基缺失,并且中间有7个碱基替换,造成N端4个氨基酸缺失及5个氨基酸替换(图 10)。
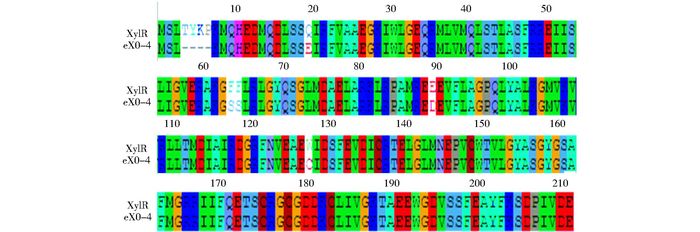
|
| 图 10 蛋白质序列比对 Figure 10 Protein sequence alignment of XylR and eX0-4 |
生物信息学软件分析表明,蛋白质突变前后二级结构及三维空间结构都发生了一定改变(图 11)。我们推测eX0-4突变蛋白A区的氨基酸改变,使其可以产生更合适的取向,与小分子TNT能够充分接近,相互契合产生更稳定的复合物构象,作用在Pu启动子上发挥功能。
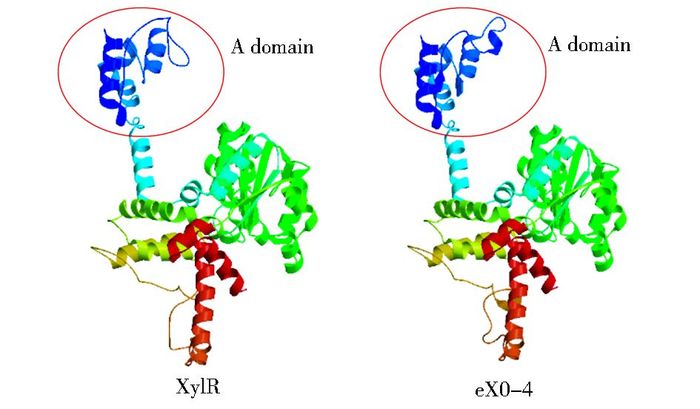
|
| 图 11 蛋白质三维结构模型 Figure 11 The three-dimensional structure models of XylR and eX0-4 |
在本研究中,我们在合成生物学思想的指导下,首先将恶臭假单胞菌内的XylR蛋白、突变体XylR5、Pu等生物元件与绿色荧光蛋白进行逐级组装搭建检测线路,然后选择遗传背景清晰、操作简便的大肠杆菌作为底盘细胞,用来承载设计的生物元件及基因线路,构建成全细胞生物传感器用于检测TNT。由于国际上报道的感应元件对TNT的最高灵敏度达到15mg/L,所以我们采用的TNT筛选终浓度也低至15mg/L,希望获得的蛋白质元件能够对如此低浓度的TNT有明显感应。但两条基因线路X0-Pu-GFP-Duet及X5-Pu-GFP-Duet在检测TNT时,无论是否加TNT,IPTG诱导后表达的XylR/XylR5蛋白都能激活下游的Pu启动子。分析可能是因为T7启动子的启动活性太强,覆盖了Pu启动子的活性,造成了整个基因线路的通读。为了更好地实现预定功能,尝试在XylR与Pu序列之间插入一个终止序列,发现背景值有所降低,直到最终加入四串联终止序列才彻底阻止了T7启动子的通读。
优化的基因线路在检测TNT时,XylR5蛋白对TNT有感应,表明对XylR蛋白的A区进行改造获得感应TNT的蛋白质元件的方法是可行的。但XylR5蛋白对TNT的感应强度及特异性差,分析可能是由于XylR5蛋白的A区结合TNT后引发的构象并不能使之很好的结合到Pu启动子上,最大程度地激活Pu活性,于是进一步对XylR蛋白的A区进行人工改造,通过采用连续易错PCR的方法对其进行随机突变构建文库,从中挖掘出性状更加优良的蛋白质元件。最终筛选得到的蛋白质突变体eX0-4对TNT表现出了高的感应强度和良好的特异性及灵敏度。同源模建结果表明蛋白质eX0-4的三维空间结构发生了改变,这可能有利于其以更合适的取向与TNT相互作用,蛋白质与小分子相互作用时,氨基酸残基的改变会直接影响两者的亲和力,我们目前正在尝试用分子对接的方法进行预测分析,期望从中归纳出XylR蛋白与TNT相互作用的氨基酸残基类型,为未来的突变提供较为明确的筛选方向,也希望通过对元件作用机制以及底盘细胞研究更清楚的基础上,构建的生物传感器可以高灵敏度、高特异性的对环境中的TNT及其靶标化合物进行检测,可为战后雷场或现场爆炸物的大规模排查和清除提供重要的辅助手段。
另外,构建的基因工程菌对人类及环境可能造成的安全问题也一直牵动着科学家的神经,我们实验室也一直致力于实现细菌自裂解方面的研究。我们基于合成生物学的方法尝试构建了可受阿拉伯糖诱导的自杀线路,并取得了初步成效[17]。阿拉伯糖较常用的抗生素具有更好的环境亲和力,加入之后,线路表达的Holin蛋白可以在细菌的细胞膜上打孔,使得细胞内的溶解酶(lysozyme)可以透过小孔溶解细胞壁从而降解细胞,组成型表达的anti-Holin可以抑制本底表达的Holin,保证未诱导时细菌的正常生长。构建的自杀线路单独导入大肠杆菌,加入诱导剂2h后可以实现菌体完全裂解,但其配合peX0-4感应线路共同发挥作用的条件还在摸索,争取尽快做到检测、裂解合二为一。
合成生物学作为一门交叉学科,在21世纪获得了迅猛发展,基于标准化、模块化的生物元件,通过人工设计、化学合成和重新组装基因线路或基因组,合成生物学可以构建具有特定功能的生命系统,甚至是人造生命[18-20]。构建生物传感器是合成生物学研究的重要应用领域之一,通过挖掘自然界中对靶标化合物敏感的生物元件,并将其与报告元件组装成基因线路,报告元件的信号强度指征生物元件对靶标化合物的感应强度[21-22]。
生物元件、装置和系统是合成生物学的三大基本要素。作为要素之一的生物元件是指遗传系统中最简单、最基本的生物积块(BioBrick),是具有特定功能的氨基酸或者核苷酸序列,可以说它是合成生物学的基石[23]。而且合成生物学与基因工程和代谢工程最显著的区别也正在于可以将大量生物元件进行快速、随意组装,以形成新的具有特定生物学功能的装置。而实现这一目标的前提是我们仍需要大量挖掘、鉴定并改造更多的生物元件。我们通过以XylR-Pu为基因线路,以GFP为报告蛋白,以大肠杆菌为底盘细胞,以连续易错PCR构建随机突变体文库,挖掘到了可特异、高效感应TNT的蛋白质元件,为下一步成型的生物传感器提供了合成生物学的元件基础、线路基础以及技术基础。
| [1] |
Tan J, Kan N, Wang W, et al. Construction of 2, 4, 6-trinitrotoluene biosensors with novel sensing elements from Escherichia coli K-12 MG1655. Cell Biochemistry and Biophysics, 2015, 72(2): 417-428. DOI:10.1007/s12013-014-0481-8 |
| [2] |
Habib M K. Controlled biological and biomimetic systems for landmine detection. Biosensors and Bioelectronics, 2007, 23(1): 1-18. DOI:10.1016/j.bios.2007.05.005 |
| [3] |
Ewing R G, Atkinson D A, Eiceman G A, et al. A critical review of ion mobility spectrometry for the detection of explosives and explosive related compounds. Talanta, 2001, 54(3): 515-529. DOI:10.1016/S0039-9140(00)00565-8 |
| [4] |
Armenta S, Alcala M, Blanco M. A review of recent, unconventional applications of ion mobility spectrometry (IMS). Analytica Chimica Acta, 2011, 703(2): 114-123. DOI:10.1016/j.aca.2011.07.021 |
| [5] |
Magrisso S, Erel Y, Belkin S. Microbial reporters of metal bioavailability. Micro Biotechnol, 2008, 1(4): 320-330. DOI:10.1111/j.1751-7915.2008.00022.x |
| [6] |
Robin T, van der Meer J R. Bacterial biosensors for measuring availability of environmental pollutants. Sensors, 2008, 8(7): 4062-4080. DOI:10.3390/s8074062 |
| [7] |
Turner A P F. Biosensors: sense and sensibility. Chemical Society Reviews, 2013, 42(8): 3184-3196. DOI:10.1039/c3cs35528d |
| [8] |
Trang P T K, Berg M, Viet P H, et al. Bacterial bioassay for rapid and accurate analysis of arsenic in highly variable groundwater samples. Environmental Science & Technology, 2005, 39(19): 7625-7630. |
| [9] |
Kim M N, Park H H, Lim W K, et al. Construction and comparison of Escherichia coli whole-cell biosensors capable of detecting aromatic compounds. Journal of Microbiological Methods, 2005, 60(2): 235-245. DOI:10.1016/j.mimet.2004.09.018 |
| [10] |
Pérez-Martín J, De Lorenzo V. Identification of the repressor subdomain within the signal reception module of the prokaryotic enhancer-binding protein XylR of Pseudomonas putida. Journal of Biological Chemistry, 1996, 271(14): 7899-7902. DOI:10.1074/jbc.271.14.7899 |
| [11] |
Delgado A, Ramos J L. Genetic evidence for activation of the positive transcriptional regulator Xy1R, a member of the NtrC family of regulators, by effector binding. Journal of Biological Chemistry, 1994, 269(11): 8059-8062. |
| [12] |
Natasha V, Ann E M, Anisa Y, et al. Structure, function, and tethering of DNA-binding domains in σ54 transcriptional activators. Biopolymers, 2013, 99(12): 1082-1096. |
| [13] |
Galvāo T C, Mencía M, De Lorenzo V. Emergence of novel functions in transcriptional regulators by regression to stem protein types. Molecular Microbiology, 2007, 65(4): 907-919. DOI:10.1111/mmi.2007.65.issue-4 |
| [14] |
Garmendia J, De Las Heras A, Galvāo T C, et al. Tracing explosives in soil with transcriptional regulators of Pseudomonas putida evolved for responding to nitrotoluenes. Microbial Biotechnology, 2008, 1(3): 236-246. DOI:10.1111/j.1751-7915.2008.00027.x |
| [15] |
De Las Heras A, De Lorenzo V. Cooperative amino acid changes shift the response of the σ54-dependent regulator XylR from natural m-xylene towards xenobiotic 2, 4-dinitrotoluene. Molecular Microbiology, 2011, 79(5): 1248-1259. DOI:10.1111/mmi.2011.79.issue-5 |
| [16] |
Yagur-Kroll S, Lalush C, Rosen R, et al. Escherichia coli bioreporters for the detection of 2, 4-dinitrotoluene and 2, 4, 6-trinitrotoluene. Applied Microbiology and Biotechnology, 2014, 98(2): 885-895. DOI:10.1007/s00253-013-4888-8 |
| [17] |
谭俊杰. 基于合成生物学的地雷检测方法研究. 北京: 中国人民解放军军事医学科学院, 2015: 90-96. Tan J J, Research of Landmine Detection Methods Based on Synthetic Biology. Beijing: Academy of Military Medical Sciences, 2015:90-96. http://cdmd.cnki.com.cn/Article/CDMD-90106-1015382217.htm |
| [18] |
Mcdaniel R, Weiss R. Advances in synthetic biology: on the path from prototypes to applications. Current Opinion in Biotechnology, 2005, 16(4): 476-483. DOI:10.1016/j.copbio.2005.07.002 |
| [19] |
Matsumura I. Bacterial cells as model factories. American Journal of Operations Research, 2013, 3(1): 81-86. DOI:10.4236/ajor.2013.31A007 |
| [20] |
Gibbs W W. Synthetic life. Scientific American, 2004, 290(5): 74-81. |
| [21] |
Van Der Meer J R, Belkin S. Where microbiology meets microengineering: design and applications of reporter bacteria. Nat Rev Micro, 2010, 8(7): 511-522. DOI:10.1038/nrmicro2392 |
| [22] |
Marra A, Asundi J, Bartilson M, et al. Differential fluorescence induction analysis of Streptococcus pneumoniae identifies genes involved in pathogenesis. Infection & Immunity, 2002, 70(3): 1422. |
| [23] |
Endy D. Foundations for engineering biology. Nature, 2005, 438(7067): 449-453. DOI:10.1038/nature04342 |
 2017, Vol. 37
2017, Vol. 37




