文章信息
- 罗丹, 王莉娟, 孙秀璇, 张征, 陈志南.
- LUO Dan, WANG Li-juan, SUN Xiu-xuan, ZHANG Zheng, CHEN Zhi-nan.
- 应用CRISPR/Cas9技术构建miR-362敲除的95-D肺癌细胞株
- Construction of miR-362 Knockdown 95-D Cell Line by CRISPR/Cas9 Technology
- 中国生物工程杂志, 2017, 37(11): 94-100
- China Biotechnology, 2017, 37(11): 94-100
- http://dx.doi.org/DOI:10.13523/j.cb.20171113
-
文章历史
- 收稿日期: 2017-06-14
- 修回日期: 2017-08-08
2. 空军军医大学(第四医科大学)细胞工程研究中心 西安 710032
2. Cell Engineering Research Center, The Fourth Military Medical University, Xi'an 710032, China
肺癌是我国常见的、发病率和致死率均居首位的恶性肿瘤[1]。肺癌的发生发展是缓慢而持续的过程,涉及染色体稳定性、表观遗传学改变、基因表达调控等多因素、多信号通路的共同参与。近年来,随着高通量测序技术的发展,尤其是基因组、转录组等研究的不断深入,包括miRNAs在内的非编码RNA在肿瘤发生发展中的作用也受到人们的关注。
MicroRNAs(miRNAs)是一类非编码小分子RNA,通过与沉默复合体(RISC)结合介导靶基因mRNA降解或阻碍其翻译[2]。研究表明miRNAs在肿瘤细胞中广泛存在,并发挥着重要的促癌因子或抑癌因子的作用[3-5]。MiR-362定位于人基因组Xp11.23。研究表明,miR-362在胃癌中可作为促癌因子,通过激活NF-κB通路诱导细胞增殖和凋亡耐受[6];然而,也有研究证明,miR-362在神经母细胞瘤中作为肿瘤抑制子,通过靶向PIK3C2B抑制肿瘤细胞的增殖、迁移[7]。为了明确miR-362在非小细胞肺癌演进中的作用机制,有必要建立完全敲除的细胞株。
基因编辑技术为在基因组水平进行定点操作提供了良好的方法。目前,常用的基因组编辑技术除了锌指蛋白(zinc finger protein, ZFP)技术、转录激活因子样效应物核酸酶(transcription activator-like effector nucleases,TALEN)技术以外,RNA引导的CRISPR/Cas9核酸酶技术在基因编辑中的应用也越来越广泛。Cas9系统是微生物自我保护、防止外来微生物的侵入的一种独特机制,它是继ZFN和TALEN之后出现的更为快捷和有效的基因编辑工具。目前已发现的有三种CRISPR/Cas9系统,其中Ⅱ型系统研究最为深入、应用最为广泛,其可产生抗噬菌体的嗜热链球菌菌株(Streptococcus thermophiles strains)。该系统通过Cas9内切核酸酶在特定的靶向序列处引入双链断裂的(double strand break, DSB) DNA缺口,继而通过细胞内NHEJ途径(non-homologous end joining, NHEJ)和同源重组(homologous recombination,HR)途径两种DNA修复机制完成修复,其中NHEJ途径使基因组在DNA缺口处有碱基的插入或者缺失,造成移码突变,导致基因的敲除。而HR途径则以外源DNA为模板,使基因组DNA得到精确的基因修复或靶向基因的添加。
本研究采用了CRISPR/Cas9介导的基因编辑技术,通过同源重组途径,并结合Cre/LoxP系统构建了miR-362敲除的95-D细胞株;并利用Transwell方法初步证明miR-362可促进非小细胞肺癌的迁移和侵袭,为进一步研究miR-362在非小细胞肺癌中的作用机制和功能奠定了基础。
1 材料与方法 1.1 材料人肺癌细胞系95-D为本实验室冻存。PX330质粒(Addgene公司),donor vector (Mo Yin-yuan惠赠),试剂:限制性内切核酸酶BbsI、T4连接酶(NEB公司),质粒DNA小提试剂盒、DNA片段纯化试剂盒(Omega-biotek公司),转染试剂Lipofectamine 2000(Invitrogen公司),miScript Ⅱ RT kit和miScript SYBR® Green PCR Kit(QIAGEN)。
1.2 方法 1.2.1 质粒构建基于pre-miR-362基因组序列,本文利用在线工具CRISPR design (http://crispr.mit.edu/)查找潜在的sgRNA序列,并在正义链的5′端添加CACC,反义链的5′端添加AAAC(表 1),与BbsI酶切后形成的黏性末端互补。所有寡核苷酸链均由生工合成。将sgRNA寡核苷酸链经退火(95℃,5min后自然降至室温),连入经Bbs I酶切回收的pX330-U6-Chimeric_BB-CBh-hSpCas9表达载体。转化感受态细胞挑取单克隆,小提质粒并测序验证(由华大基因公司完成)。
| Primers | Sequences (5′-3′) |
| gRNA1 F | CACCTGGAACCTAGGTGTGAGTGC |
| gRNA1 R | AAACGCACTCACACCTAGGTTCCA |
| gRNA2 F | CACCCAGTGCAACACACCTATTCA |
| gRNA2 R | AAACTGAATAGGTGTGTTGCACTG |
| gRNA3 F | CACCAGGTGTGAGTGCTATTTCAG |
| gRNA3 R | AAACCTGAAATAGCACTCACACCT |
| gRNA4 F | CACCGGTTCCAAGGATTCAAGAGG |
| gRNA4 R | AAACCCTCTTGAATCCTTGGAACC |
| gRNA5 F | CACCAGGTTCCAAGGATTCAAGAG |
| gRNA5 R | AAACCTCTTGAATCCTTGGAACCT |
| gRNA6 F | CACCTGCTCCCCCTCTTGAATCCT |
| gRNA6 R | AAACAGGATTCAAGAGGGGGAGCA |
| gRNA7 F | CACCGCCTCTTTGAATCCTTGAAT |
| gRNA7 R | AAACATTCAAGGATTCAAAGAGGC |
| D362-F | CCCACAAACATACACAAAAGCAT |
| D362-R | TGGGCACACATGTACATGCAC |
| Cre-5.1-HindIII | AAGCTTGCCACCatgtccaatttactgaccgtac |
| Cre-3.1-XhoI | CTCGAGctaatcgccatcttccagcag |
| 362-seq-5.1 | Agtaagtaccttgtgtgaagaagaatg |
| 362-seq-3.1 | Cacacatgtacatgcacatgtt |
为构建miR-362的donor vector,我们利用pfu DNA聚合酶从基因组DNA中分别扩增了miR-362的上下游同源臂(约800bp),所用引物见表 1,并插入到donor vector中,转化感受态细胞挑取单克隆,小提质粒并测序验证(由华大基因公司完成)。
1.2.2 T7 endonuclease I酶切实验将构建的PX330-gRNA载体分别转染至95-D细胞,培养48h后,利用DNA提取试剂盒(天根)提取基因组DNA。利用引物362-seq-5.1和362-seq-3.1(表 1)扩增含sgRNA作用靶点的DNA片段。反应条件:95℃ 5min; (95℃ 30s,60℃ 30s,72℃ 30s)×30; 72℃ 10min。用PCR纯化试剂盒(Omega bio-tek)纯化PCR产物。取200ng纯化PCR产物在NEB Buffer 2中变性、复性,反应条件变性95℃,5min,退火95~85℃ (-2℃/s),85~25℃(-0.1℃/s)后,用T7内切核酸酶(NEB,M0302L) 37℃孵育15min,然后用2%的琼脂糖凝胶电泳分离,凝胶成像。
1.2.3 细胞转染和筛选将95-D细胞以5×105/孔接种至6孔板中培养至细胞汇合度达到60%~70%,用lipofectamine2000将PX330-gRNA和DONOR-362共转入细胞中。培养一周后用0.5μg/ml嘌呤霉素进行加压筛选。筛选两周后,利用流式细胞仪将依据GFP信号将单个细胞分选至96孔板继续培养2~3周。分选获得的细胞经扩大培养后冻存。
1.2.4 用Cre酶去除重组LoxP位点利用HiScribe T7 ARCA mRNA Kit (with tailing) (NEB公司)按照说明书操作合成Cre mRNA。简要描述如下,利用引物Cre-5.1-HindIII和Cre-3.1-XhoI通过PCR扩增获得含有T7启动子区的Cre编码区。以纯化后的PCR片段产物为模板合成Cre mRNA。将前述筛选得到的95-D-KD-GFP的细胞种于6孔板中过夜培养后,将3μg Cre mRNA转染至细胞。转染3天后扩大培养。利用流式细胞仪分选不含绿色荧光的细胞。
1.2.5 RNA提取及实时定量PCR用Trizol(Thermo Fisher scientific)分别提取野生型和敲除型95-D细胞的总RNA,利用miScript Ⅱ RT kit(QIAGEN)逆转录成cDNA,并利用miScript SYBR® Green PCR Kit(QIAGEN)。检测miR-362在野生型和敲除型95-D细胞中的表达水平,实验以U6为内参。
1.2.6 Transwell细胞迁移实验将95-D和95-D-KD细胞用胰酶消化,PBS洗1~2遍,用无血清培养基重悬,调整细胞密度至5×105个/ml,取细胞悬液100μl加入Transwell小室,24孔板下室加入500μl含10%胎牛血清的培养基,置于37℃孵箱常规培养20h后,取出小室,经淋洗、固定、染色后于显微镜下计数,每样本计数5个视野取平均值,实验重复3次,统计学分析并绘制柱状图。
1.2.7 Transwell细胞侵袭实验用50mg/L Matrigel(1:5)稀释后,包被Transwell小室上室底部并风干,将饥饿24h的95-D和95-D-KD细胞常规消化离心后用无血清培养基重悬细胞,将细胞浓度调整为1×106个/ml,取细胞悬液100μl加入Transwell小室上室,24孔板下室加入500μl含血清培基,置于37℃孵箱常规培养20h后,取出小室,经淋洗、固定、染色后于显微镜下计数,每样本计数5个视野取平均值,实验重复3次,统计学分析并绘制柱状图。
1.2.8 数据处理及统计学分析实验数据用GraphPad Prism 5.0软件分析, 各组数据的统计学差异采用方差分析(Two-Way ANOVA)或t检验,P < 0.01为有显著统计学差异。
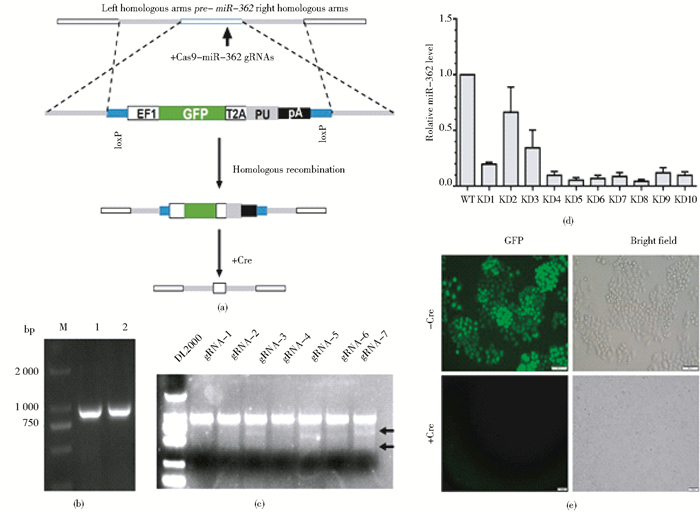
2 实验结果 2.1 sgRNA的设计和载体构建依据pre-miR-362基因序列,利用在线sgRNA设计软件,并分别在正义链的5′端添加CACC,反义链的5′端添加AAAC。设计合成了7条针对miR-362基因的sgRNA寡核苷酸链(表 1)。经退火后形成BbsI的黏性末端。将pX330-U6-Chimeric_BB-CBh-hSpCas9载体经BbsI酶切回收后分别与7条sgRNA序列连接,经测序验证完成PX330-sgRNA载体初步构建。
为提高敲除细胞的筛选效率,我们构建了donor载体,拟通过同源重组将靶序列替换为标志序列(GFP和PU, the puromycin resistance gene)(图 1a)。该载体中GFP有助于利用流式分选阳性细胞,PU基因有利于筛选稳定发生同源重组的细胞,该载体中含有的LoxP位点有助于在Cre酶的作用下将筛选获得的稳定细胞中的标志序列切除。我们利用引物362-F-Xbal-F与362-F-BamHI-R和362-B-EcoR1-F与362-B-XhoI-R分别从95-D细胞的基因组中通过PCR技术扩增了pre-miR-362上下游各约800bp的基因序列(图 1b),并将其分别连接至donor vector的XbaI和BamHI,以及EcoRI和XhoI酶切位点。转化至DH5α中,挑取单克隆,经测序验证完成Donor-362载体构建。
|
| 图 1 利用CRISPR/Cas9系统构建miR-362敲除细胞系 Figure 1 Construction of miR-362 knockdown cell line by CRISPR/Cas9 technology (a) Schematic diagram of homologous recombined donor vector targeting miR-362 (b) Gel electrophoresis results of miR-362 after PCR amplification of pre-and post-arm of miR-362 (c) Detection of plasmids transfected with different CRISPR systems by T7E1 assay (d) MiR-362 expression levels in different clones (e) Fluorescence microscopy photographes of marker genes deleted by Cre techonology in 95-D-KD cells |
利用脂质体将构建的不同的px330-sgRNA载体转染至95-D细胞,72h后提取细胞基因组DNA,利用PCR方法扩增相应片段,通过T7E1 assay评介CRIPSR系统的剪切能力。结果显示:经T7 endonuclease I处理后,在sgRNA-3、sgRNA-5和sgRNA-7组中出现了两条带(图 1c),提示sgRNA-3、sgRNA-5和sgRNA-7可引起靶点基因发生突变。选择sgRNA-7作为最终的打靶sgRNA。
2.3 建立稳定敲除pre-mir-362的95-D细胞株将PX330-MIR362-sgRNA7质粒和DONOR-362质粒共转染95-D细胞。为了减少DONOR-362载体随机整合至基因组的可能性,我们在转染1周后,用嘌呤霉素(0.5μg/ml)进行加压筛选。筛选1周后,利用流式细胞仪,根据GFP荧光信号将经嘌呤霉素加压筛选获得的阳性细胞分选至96孔板中(1细胞/孔)。经扩大培养后,我们在95-D敲除细胞中随机选取了10个克隆。分别提取细胞Total RNA,经miScript reverse kit反转录,通过实时定量PCR方法比较野生型95-D细胞和敲除型细胞中miR-362表达水平的差异。qRT-PCR结果显示(图 1d),与野生型95-D细胞相比,在95-D-KO-GFP细胞的10个克隆中,除克隆2和克隆3以外,其余克隆株中miR-362的表达水平均显著低于对照细胞(P<0.001),表达水平降低到原来的1.29%~1.95%。表明通过CRISPR/Cas9系统成功获得了稳定敲除miR-362的95-D肺癌细胞株。因为所构建的DONOR载体中含有2个LoxP位点可用于去除发生同源重组的标志序列(GFP和PU),我们通过体外转录获得了Cre重组酶mRNA,并将其转染到95-D-KD-GFP细胞的克隆8细胞中,以去除标志序列,最终获得了不含有GFP荧光的95-D-KD细胞(图 1e)。
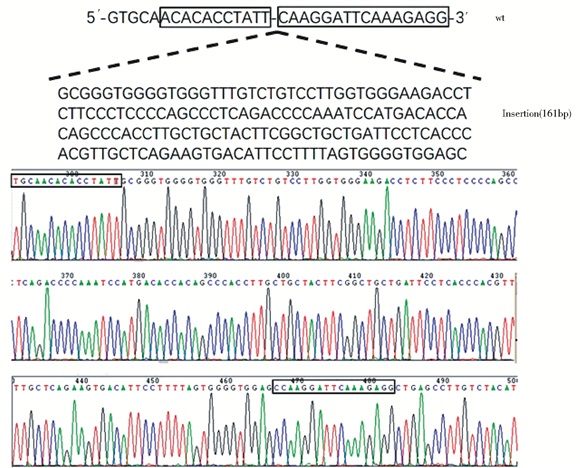
2.4 95-D-KD细胞的基因序列特点分析为明确95-D-KD细胞的基因序列的特点,我们利用引物D362-F和引物D362-R通过PCR扩增了95-D-KD基因组中的相应片段,通过测序分析发现,与野生型pre-miR-362序列相比,95-D-KD细胞在第53位(T)和第54位(C)碱基间插入了长度为161bp的序列(图 2)。
|
| 图 2 MiR-362敲除细胞系基因突变分析 Figure 2 Analysis of gene mutation in miR-362 knockdown cell line |
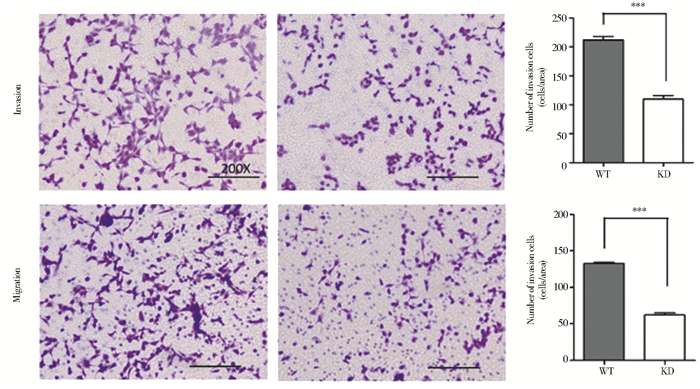
为了探讨miR-362的生物学功能,我们用transwell方法评价了野生型95-D和95-D-KD细胞在细胞运动方面的差异。如图 3所示,野生型95-D和敲除型95-D-KD细胞的迁移细胞数分别为132.7±3.2和62.3±6.1,侵袭细胞数分别为212.0±12.0和109.6±14.1。与野生型95-D细胞相比,95-D-KD细胞的迁移和侵袭能力分别下降了53.1%(P=0.000 6)和48.3% (P=0.000 2)。以上结果提示,敲除miR-362显著抑制了95-D细胞的迁移和侵袭能力,miR-362与细胞的运动能力密切相关。
|
| 图 3 Transwell迁移和侵袭实验 Figure 3 Transwell migration and invasion experiments |
非编码RNA占据了人转录组的绝大部分,然而,其生物学功能尚不十分清楚。有证据表明非编码RNA在多种生物学活动中发挥着作用,如细胞周期、分化、死亡等,并且在肿瘤的发生发展中也起着重要的作用。目前对非编码RNA生物学功能的研究多采用过表达和RNAi等手段,但是,RNAi方法具有特异性差、效率不高、范围受限、作用时间有限等不足,影响了对分子功能的深入研究。而CRISPR/Cas9系统则可以从基因水平将靶基因彻底敲除,相比之下更具优势。
CRISPR/Cas9系统是目前常用的基因操作工具之一,该系统包括CRISPR相关核酸酶和单链向导RNA(single guide RNA,gRNA)[8]。CRISPR是一些相互间隔的短回文重复序列,常产生发卡结构,长度为21~48bp[9],每个重复序列间存在外源性DNA靶点中与之结构相似的短重复序列,这决定了SRISPR系统的种类及靶基因的识别位点[10]。在DNA靶点内,每个典型间隔区与典型间隔相邻基序(protospacer adjacent motif,PAM)相邻[11],常用的PAM包括NGG、NAG和NNGG[12]。Cas9核酸酶含有两个核酸酶结构域,HNH结构域和RuvC样结构域,可分别切割目标DNA链的一条单链[13]。CRISPR/Cas9发挥作用时,通过PAM序列使gRNA与靶位点识别,介导Cas9核酸酶与基因组稳定结合,引起靶位点切割,从而产生双链断裂,引起非同源末端连接(non-homologous end joining, NHEJ)或同源性定向修复(homology-directed repair, HDR)。CRISPR/Cas9系统可以同时对靶基因进行高效的基因敲除(knock-out)和基因敲入(knock-in)编辑。
成熟的microRNAs长度仅为约22nt,在实验中,我们以pre-miR-362作为模板设计建立CRISPR/Cas9系统,DNA测序结果表明CRISPR/Cas9系统作用后,在pre-miR-362第53位和第54位核苷酸间插入了长为161bp的碱基(图 2),这可能影响了pre-miRNA的二级结构,进而影响了miR-362成熟的生物加工过程,最终导致miR-362表达的显著下调(图 1d)。
我们的实验结果进一步证明,利用携带有靶基因上下游同源臂基因的Donor载体,可有效的通过同源重组的方式提高基因敲除的效率。一方面,Donor载体中含有GFP和PU基因,GFP有助于利用流式细胞仪分选那些发生同源重组的单克隆细胞,有利于降低实验成本和细胞所受的压力;另一方面,由于Donor载体中携带有靶基因的同源臂基因,使得donor载体不易于结合到基因组中存在有脱靶效应(off-target)的位点,从而降低了CRISPR/Cas9的脱靶效应[14-15],那些未发生同源重组的细胞在PU的压力筛选下得以清除,提高筛选效率。此外,在我们所采用的Donor载体中还含有两个LoxP位点,可在Cre酶的作用下切除miR-362敲除细胞中的标志序列(GFP和PU),使细胞不再表达GFP,并对PU重新敏感,有助于再次细胞分选和筛选。
本研究利用CRISPR/Cas9基因编辑技术,成功构建了95-D-miR-362敲除细胞系,并用实时定量PCR方法检测了敲除效率。研究结果表明,本研究获得的敲除细胞系具有敲除效率高、效果稳定的优点,且敲除片段插入位点明确,最大限度的减少了对于肿瘤细胞其他重要生物学功能的影响,是用于研究miR-362功能的理想体外细胞模型。通过侵袭及迁移实验,证明了miR-362在促进肺癌细胞恶性行为中的作用,与其在其他多种肿瘤细胞中的研究结果一致。但miR-362促进肺癌侵袭转移的具体分子机制仍需深入研究。本研究构建的体外模型为研究miR-362在肿瘤发生发展中的作用机制提供了重要的细胞模型。
致谢 感谢第四军医大学细胞工程研究中心的全体科研人员。| [1] |
Liu Y, Hu X, Xia D, et al. MicroRNA-181b is downregulated in non-small cell lung cancer and inhibits cell motility by directly targeting HMGB1. Oncol Lett, 2016, 12(5): 4181-4186. |
| [2] |
Wang H, Sun T, Hu J, et al. miR-33a promotes glioma-initiating cell self-renewal via PKA and NOTCH pathways. J Clin Invest, 2014, 124(10): 4489-4502. DOI:10.1172/JCI75284 |
| [3] |
Ni F, Zhao H, Cui H, et al. MicroRNA-362-5p promotes tumor growth and metastasis by targeting CYLD in hepatocellular carcinoma. Cancer Lett, 2015, 356(2): 809-818. DOI:10.1016/j.canlet.2014.10.041 |
| [4] |
Jung C J, Iyengar S, Blahnik K R, et al. Epigenetic modulation of miR-122 facilitates human embryonic stem cell self-renewal and hepatocellular carcinoma proliferation. PLoS One, 2011, 6(11): e27740. DOI:10.1371/journal.pone.0027740 |
| [5] |
Nicoloso M S, Spizzo R, Shimizu M, et al. MicroRNAs——the micro steering wheel of tumour metastases. Nat Rev Cancer, 2009, 9(4): 293-302. DOI:10.1038/nrc2619 |
| [6] |
Xia J T, Chen L Z, Jian W H, et al. MicroRNA-362 induces cell proliferation and apoptosis resistance in gastric cancer by activation of NF-kappaB signaling. J Transl Med, 2014, 12(1): 33. DOI:10.1186/1479-5876-12-33 |
| [7] |
Wu K, Yang L, Chen J, et al. miR-362-5p inhibits proliferation and migration of neuroblastoma cells by targeting phosphatidylinositol 3-kinase-C2beta. FEBS Lett, 2015, 589(15): 1911-1919. DOI:10.1016/j.febslet.2015.05.056 |
| [8] |
Pelletier S, Gingras S, Green D R. Mouse genome engineering via CRISPR-Cas9 for study of immune function. Immunity, 2015, 42(1): 18-27. DOI:10.1016/j.immuni.2015.01.004 |
| [9] |
Grissa I, Vergnaud G, Pourcel C. The CRISPRdb database and tools to display CRISPRs and to generate dictionaries of spacers and repeats. BMC Bioinformatics, 2007, 8(1): 172. DOI:10.1186/1471-2105-8-172 |
| [10] |
Feng Y, Liu J, Kang Y, et al. miR-19a acts as an oncogenic microRNA and is up-regulated in bladder cancer. J Exp Clin Cancer Res, 2014, 33(1): 67. DOI:10.1186/s13046-014-0067-8 |
| [11] |
Ran F A, Hsu P D, Wright J, et al. Genome engineering using the CRISPR-Cas9 system. Nat Protoc, 2013, 8(11): 2281-2308. DOI:10.1038/nprot.2013.143 |
| [12] |
Xiao A, Cheng Z, Kong L, et al. CasOT:a genome-wide Cas9/gRNA off-target searching tool. Bioinformatics, 2014, 30(8): 1180-1182. DOI:10.1093/bioinformatics/btt764 |
| [13] |
Cong L, Ran F A, Cox D, et al. Multiplex genome engineering using CRISPR/Cas systems. Science, 2013, 339(6121): 819-823. DOI:10.1126/science.1231143 |
| [14] |
Lin Y, Cradick T J, Brown M T, et al. CRISPR/Cas9 systems have off-target activity with insertions or deletions between target DNA and guide RNA sequences. Nucleic Acids Res, 2014, 42(11): 7473-7485. DOI:10.1093/nar/gku402 |
| [15] |
Shen B, Zhang W, Zhang J, et al. Efficient genome modification by CRISPR-Cas9 nickase with minimal off-target effects. Nat Methods, 2014, 11(4): 399-402. DOI:10.1038/nmeth.2857 |
 2017, Vol. 37
2017, Vol. 37




