文章信息
- 李金蔓, 裴广倩, 范航, 黄勇, 童贻刚.
- LI Jin-man, PEI Guang-qian, FAN Hang, HUANG Yong, TONG Yi-gang.
- 肠道菌群核酸提取自动化流程的优化
- Optimization of Automatic Flow Chart for Nucleic Acid Extraction from Intestinal Microflora
- 中国生物工程杂志, 2017, 37(11): 6-11
- China Biotechnology, 2017, 37(11): 6-11
- http://dx.doi.org/DOI:10.13523/j.cb.20171102
-
文章历史
- 收稿日期: 2017-09-20
2. 中国人民解放军军事科学院微生物与流行病研究所 北京 100071
2. Institute of Microbiology and Epidemiology, the Academy of Military Sciences PLA China, Beijing 100071, China
人体的各个器官、皮肤表面和开放性的腔道内(食道、呼吸道、消化道、肠道、生殖道)都分布着大量的细菌[1],它们与人体细胞的比例高达10:1[2]。人体肠黏膜面积约200m2,肠道同时可以提供微生物生长所需的营养物质,包含有超过100万亿个细菌,是微生物定植最多的器官[3]。近年来,研究者已经证实:痤疮、系统性红斑狼疮、类风湿性关节炎等自身免疫系统疾病;糖尿病、高血压、高血脂等营养相关性疾病[4];抑郁症、阿尔茨海默病等神经精神系统疾病[5-6];溃疡性结肠炎、克罗恩病、结肠癌等消化系统疾病;妊娠期高血压、孕期妇女的免疫等女性妊娠期疾病的发生发展均与肠道菌群的变化密切相关。肠道菌群作为一个复杂而变化的生态系统,与宿主健康状态息息相关[7]。随着越来越多的研究者加入菌群研究的队伍,为了准确阐明肠道菌群与人体健康的关系研究所包含的样本量也从最开始的几个到现在的成百上千个[8-9]。随着实验样本量的增加,人工方法操作不稳定、速度慢、工作量大、效率低等问题逐渐显现出来。高通量测序技术每个步骤对样本的处理都会影响最终的测序质量。
1 材料与方法 1.1 材料 1.1.1 样品来源粪便样品来自志愿者,粪便样品按照标准采样方法采集后迅速放入-80℃进行冻存,运输过程中用冰块冷藏运输,2h内到达实验室。
1.1.2 主要试剂和仪器MagMAXTM Viral RNA Isolation Kit(Invitrogen, 美国),QIAamp® Fast DNA Stool Mini kit(QIAGEN, 德国),TissueLyserII(QIAGEN,德国),H2O3-PRO金属浴(金银杏生物科技有限公司,中国),MagMAXTM Express 96(Invitrogen, 美国),2×EasyTaq® PCR SuperMix(北京全式金公司),PCR仪(Gene Amp PCR System 9700),innuPREPPCRpure Lite Kit(Analytikjena, 德国),NEBNext® UltraTM II DNA Library Prep Kit for Illumina® (NEB,美国),Qubit2.0 Fluorometer(Life Technologies, 美国),CYBi® -Felix(Analytikjena, 德国), Miseq测序仪(Illumina, 美国)。
1.2 粪便样品匀浆处理将样品从-70℃冰箱取出后,每份取约200mg, 加QIAamp® Fast DNA Stool Mini Kit裂解液或PBS缓冲液,放入匀浆仪13 000Hz, 匀浆3min,使其变为匀质均一的悬混液。
1.3 粪便样品细菌DNA的提取选取MagMAXTM Viral RNA Isolation Kit(自动核酸提取仪配套试剂盒)和QIAamp® Fast DNA Stool Mini Kit两种试剂盒,比较手工提取和机器自动提取方法的差异。
1.3.1 MagMAX-96试剂盒机器提取200mg样品加入500μl的PBS缓冲液,用匀浆仪匀浆处理后,14 000r/min离心3min,取上清。按照MagMAXTM Viral RNA Isolation Kit说明书配制裂解液、磁珠、洗涤液和洗脱液并将其加入到相应的6个配套96孔板中[1个Sample Plate(包括20μl磁珠、50μl样品和130μl裂解液),2个Wash solution1, 2个Wash solution2, 1个Elution Buffer, 1个Tip comb plate], 然后将96孔板放到机器中对应的位置,提取样品中的细菌DNA。
1.3.2 QIAGEN试剂盒裂解后机器提取按照QIAamp® Fast DNA Stool Mini Kit办法对样品进行裂解。200mg样品加入1ml buffer(每种方法设置3个重复)用匀浆仪匀浆处理后,80℃温浴5min。14 000r/min离心3min,取上清。另取一新EP管加入200μl buffer AL,取400μl上述上清液,加入15μl Protein K涡旋混匀(Protein K和buffer AL不可提前混合),70℃温浴10min。然后取400μl裂解液加入样品板,加入等体积的100%异丙醇和20μl beads。按照MagMAXTM Viral RNA Isolation Kit说明书将洗涤和洗脱试剂加入到配套的96孔板,放到机器中对应的位置。程序选择AM1836(DW-100 High Volume), 提取样品中细菌DNA。
1.3.3 试剂盒直接手动提取按照QIAamp® Fast DNA Stool Mini Kit中办法提取细菌DNA(每种方法设置2个重复),于-20℃保存。
1.4 扩增细菌16S rDNA的V3-V4高变区本实验选取的引物为微生物组学研究中广泛使用的16S rDNA基因的V3-V4高变区通用引物:F(5′-CCTACGGGNGGCWGCAG-3′)、R(5′-GGACTACHVGGGTATCTAATCC-3′)。为了能实现多个样本同时测序,在通用引物前端添加4个标签碱基(最多可实现将24个样品混为一个样品进行建库)。PCR体系为50μl体系:2×EasyTaq® PCR SuperMix 25μl,上下游引物(所用引物均在5′端带有4bp碱基标签,引物浓度为10pmol)各1μl,模板2μl,Nuclease-free Water 21μl。PCR反应条件:预变性95℃ 5min;变性95℃ 30s,退火55℃ 30s,延伸72℃ 30s,30个循环;72℃延伸7min。PCR产物用innuPREPPCRpure Lite Kit纯化试剂盒进行纯化。
1.5 测序文库的构建及高通量测序纯化产物等量混合为一个样品,用水将体积补足至50μl。参照NEBNext® UltraTM II DNA Library Prep Kit for Illumina® 说明书,构建高通量测序文库。文库构建完成后,稀释文库至1ng/μl,随后使用Agilent 2100对文库的insert size进行检测,以保证文库的质量。检测合格的文库,用qPCR进行准确定量,之后进行Miseq测序。
1.6 生物信息学分析测序得到的数据,需要经过去除嵌合体、低质量碱基、NS、接头污染序列等过程完成对测序数据的过滤,以得到可信的目标序列,用于后续分析。我们将过滤后的序列称为Clean Reads。首先,将双端测序数据进行拼接,把fastq格式的序列转换为fasta格式;然后,对拼接完成的序列利用QIIME 1.5.0版本进行分析,包括OTUs的提取、聚类分析、Alpha多样性分析、Beta多样性分析。
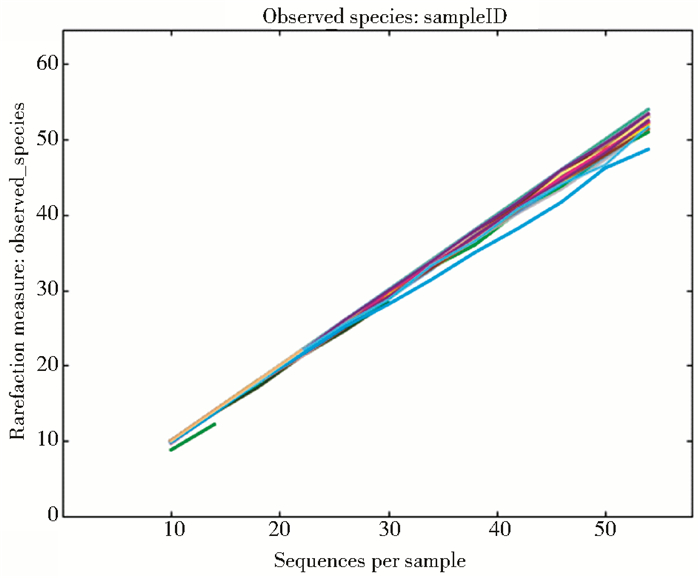
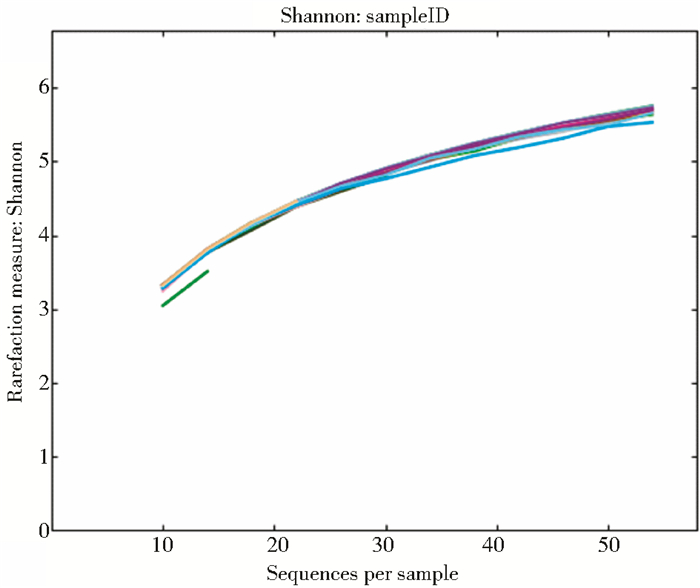
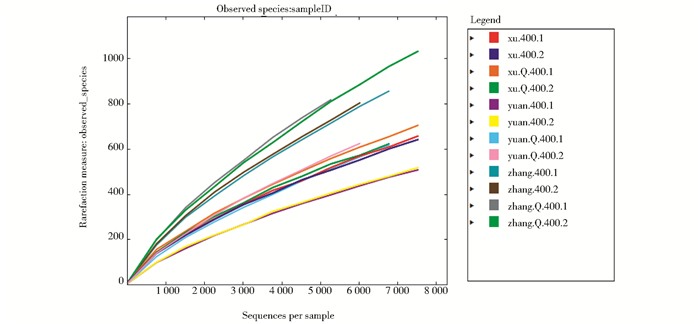
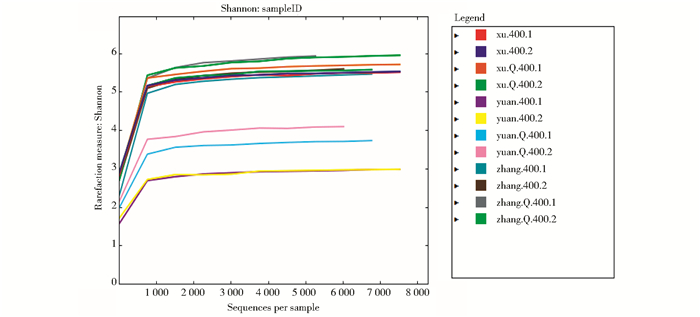
2 结果 2.1 样品α多样性分析测序结果使用QIIME 1.5.0软件进行分析。选择长度大于100bp的序列,根据97%的序列相似性进行OUT划分。结果显示:按照MagMAXTM Viral RNA Isolation Kit说明书(样品投入50μl)用MagMAX-96机器提取的改良前核酸浓度与改良提取方法(用QIAamp® Fast DNA Stool Mini Kit试剂盒裂解液裂解后,用MagMAXTM Express 96机器提取核酸)后和手工提取的核酸浓度相比数值低(表 1),测序得到的序列数均在60以内,稀释曲线呈上升趋势(图 1),Shannon曲线没有饱和(图 2),样本测序数量太少不能满足分析要求。改良方法后(用QIAamp® Fast DNA Stool Mini Kit试剂盒裂解液裂解后,用MagMAXTM Express 96机器提取核酸)结果显示:随着测序深度(横坐标代表每个样本包含的序列数)的增加,样品中检测到的OUT数目(纵坐标)呈正相关增加趋势(如图 3)。在序列数小于1 000条时,随着测序深度的增加,样品中可检测到的OUT数目呈迅速增加的趋势,但当测序数目在1 000~2 000时,检测到的OUT数目增加缓慢。测序数目达到2 000之后达到平台期,Shannon多样性曲线已饱和(图 4),测序结果可以满足分析要求,且改良后的方法和手工提取的方法相比,测序得到的序列数基本一致。
| 样品名称 | 改良前核酸浓度 (ng/μl) |
改良后核酸浓度 (ng/μl) |
手工核酸浓度 (ng/μl) |
| 1 | 1.1 | 11.7 | 20.1 |
| 2 | 3.2 | 33.5 | 50.2 |
| 3 | 2.3 | 27.8 | 33.5 |
| 4 | 1.3 | 20.0 | 27.8 |
| 5 | 4.3 | 55.2 | 55.0 |
| 6 | 2.7 | 33.2 | 37.6 |
| 7 | 3.4 | 32.7 | 40.7 |
| 8 | 3.6 | 40.2 | 50.7 |
| 9 | 2.8 | 30.3 | 37.5 |
|
| 图 1 改良前稀释曲线 Figure 1 Modified dilution curve |
|
| 图 2 改良前Shannon指数 Figure 2 Modified Shannon index |
|
| 图 3 优化核酸提取方法后和手工提取方法的稀释曲线 Figure 3 The dilution curve ofoptimized method of nucleic acid extraction andmanual extraction method |
|
| 图 4 优化核酸提取方法后和手工提取方法的Shannon指数 Figure 4 The Shannon index ofoptimized method of nucleic acid extraction andmanual extraction method |
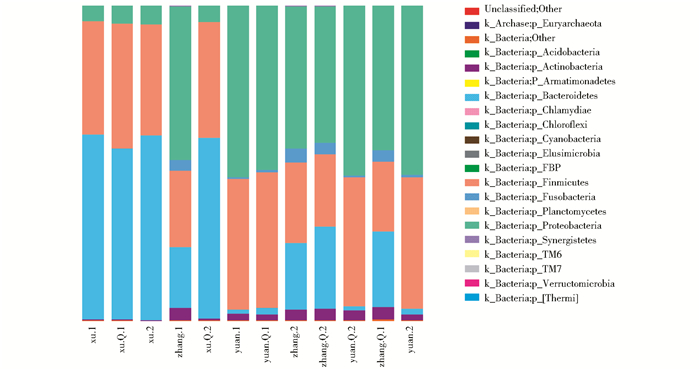
将测序质量合格的样品(改良后核酸提取方法和手工提取方法)中的OUT进行分类统计(样本分组方法详见表 2),其中所占比例较高的3门分别是Bacteroidetes、Firmicutes及Proteobacteria,且同一样品(每个样本每种方法做2个重复,在样品名称后用数字1和2区分)两种方法在组成上无明显差别(图 5)
| 样品名称 | 核酸提取方法 |
| xu.1 xu.2 |
手工提取 手工提取 |
| Zhang.1 | 手工提取 |
| Zhang.2 | 手工提取 |
| Yuan.1 | 手工提取 |
| Yuan.2 | 手工提取 |
| Xu.Q.1 | 改良后方法提取 |
| Xu.Q.2 | 改良后方法提取 |
| Zhang.Q.1 | 改良后方法提取 |
| Zhang.Q.2 | 改良后方法提取 |
| Yuan.Q.1 | 改良后方法提取 |
| Yuan.Q.2 | 改良后方法提取 |
|
| 图 5 样品在门水平的相对丰度 Figure 5 Relative abundance of samples at the phylum level |
根据Unifrac分析得到样品间距离矩阵PCoA分析图(图 6),直观显示样品中的微生物的相似性和差异性。结果显示:同一样品用两种方法(改良后的核酸方法与手工提取方法)结果可以聚在一起, 在微生物主成分无明显差别。而不同样品则分别聚类,距离较远。说明用QIAamp® Fast DNA Stool Mini Kit试剂盒裂解液处理后用MagMAXTM Express 96机器提取核酸对后续的实验及测序没有影响,可以替代用QIAamp® Fast DNA Stool Mini Kit试剂盒手工提取核酸的步骤。

|
| 图 6 PCoA分析图 Figure 6 PCoA analysis chart |
近年来,基于16S rDNA测序的宏基因组学迅速发展。由于该技术在探索各种环境样本内的微生物多样性方面具有无可比拟的优势,因此越来越多的科学家开始相关领域内的探索研究。目前,在样本的收集和保存、核酸的提取方法、扩增引物的选择等方面都取得了结论性的研究成果[10]。但是随着宏基因组学的热度持续升高,动辄成百上千的大样本研究已经成为主流趋势。随着样本数量的增加,人工操作工作量大、速度慢、存在人为误差的缺点渐渐显现出来。一套可以替代人工实验方法的自动化实验流程建立的需求越来越急切。本实验将改良前后的自动化核酸提取方法与手工提取方法进行对比。一种是选用与核酸自动提取仪配套的MagMAXTM Viral RNA Isolation Kit试剂盒,用核酸自动提取仪提取核酸(改良前方法);另一种是选用粪便提取试剂盒QIAamp® Fast DNA Stool Mini Kit中的裂解液对粪便样品进行裂解,然后用核酸自动提取仪提取样品核酸(改良后方法);将这两种方法与用粪便提取试剂盒QIAamp® Fast DNA Stool Mini Kit手工提取方法做比较。结果显示:改良前的方法每个样本得到的核酸浓度低,PCR扩增不成功,有效序列数太少(少于60条,样本OUT没有达到饱和),不能满足后期分析的要求,数据不可用,该方法不可行;将核酸提取方法改良后(用QIAamp® Fast DNA Stool Mini Kit试剂盒裂解样品后用MagMAXTM Express 96核酸自动提取仪提取核酸),测序质量和深度(样本序列数5 000~8 000,单个样本的OUT饱和)可以达到分析的要求。PCoA分析图结果显示:不同样品距离较远,分别聚类,说明主成分有较大差异,而相同样本用改良后的方法提取和手工方法提取结果明显聚集成堆(主成分相同),且在门水平下相同样本的同一物种所占的比例也基本一致,说明改良后的方法与手工方法相比,对测序结果没有影响,可以替代手工方法提取核酸,然后用自动化建库平台进行文库构建,实现从核酸提取到上机之前实验流程的自动化。
| [1] |
Sender R, Fuchs S, Milo R. Are we really vastly outnumbered? revisiting the ratio of bacterial to host cells in humans. Cell, 2016, 164(3): 337. DOI:10.1016/j.cell.2016.01.013 |
| [2] |
Dorrestein P C, Mazmanian S K, Knight R, et al. Finding the missing links among metabolites, microbes, and the host. Immunity, 2014, 40(6): 824-832. DOI:10.1016/j.immuni.2014.05.015 |
| [3] |
Santisteban M M, Kim S, Pepine C J, et al. Brain-gut-bone marrow axis:implications for hypertension and related therapeutics. Circulation Research, 2016, 118(8): 1327. DOI:10.1161/CIRCRESAHA.116.307709 |
| [4] |
Wang Z, Elizabeth K, Bennett B J, et al. Gut flora metabolism of phosphatidylcholine promotes cardiovascular disease. Nature, 2011, 472(7341): 57-63. DOI:10.1038/nature09922 |
| [5] |
Buffington S A, Prisco G V D, Auchtung T A, et al. Microbial reconstitution reverses maternal diet-induced social andsynaptic deficits in offspring. Cell, 2016, 165(7): 1762. DOI:10.1016/j.cell.2016.06.001 |
| [6] |
Hsiao E Y, Mcbride S W, Hsien S, et al. Microbiota modulate behavioral and physiological abnormalities associated with neurodevelopmental disorders. Cell, 2013, 155(7): 1451-1463. DOI:10.1016/j.cell.2013.11.024 |
| [7] |
Ma Y. Human papillomavirus community in healthy persons, defined by metagenomics analysis of human microbiome project shotgun sequencing data sets. Journal of Virology, 2014, 88(9): 4786-4797. DOI:10.1128/JVI.00093-14 |
| [8] |
Schirmer A, Gadkari R, Reeves C D, et al. Metagenomic analysis reveals diverse polyketide synthase gene clusters in microorganisms associated with the marine sponge discodermia dissoluta. Appl Environ Microbiol, 2005, 71(8): 4840-4849. DOI:10.1128/AEM.71.8.4840-4849.2005 |
| [9] |
Bobrova O, Kristoffersen J B, Oulas A, et al. Metagenomic 16S rRNA investigation of microbial communities in the Black Sea estuaries in South-West of Ukraine. Acta Biochimica Polonica, 2016, 63(2): 315. DOI:10.18388/abp.2015_1145 |
| [10] |
Shaw A G, Sim K, Powell E, et al. Latitude in sample handling and storage for infant faecal microbiota studies:the elephant in the room?. Microbiome, 2016, 4(1): 40. DOI:10.1186/s40168-016-0186-x |
 2017, Vol. 37
2017, Vol. 37




