文章信息
- 张丽丽, 徐碧玉, 刘菊华, 贾彩红, 张建斌, 金志强.
- ZHANG Li-li, XU Bi-yu, LIU Ju-hua, JIA Cai-hong, ZHANG Jian-bin, JIN Zhi-qiang.
- 转香蕉MaASR1基因的拟南芥株系在干旱胁迫条件下的表达谱分析
- Analysis of Banana MaASR1 Gene Expression Profiles in Arabidopsis Under Drought Stress
- 中国生物工程杂志, 2017, 37(11): 59-73
- China Biotechnology, 2017, 37(11): 59-73
- http://dx.doi.org/DOI:10.13523/j.cb.20171109
-
文章历史
- 收稿日期: 2017-08-14
- 修回日期: 2017-10-31
2. 中国热带农业科学院海口实验站/海南省香蕉遗传育种改良重点实验室 海口 570102
2. Haikou Experimental Station, Chinese Academy of Tropical Agricultural Sciences/Hainan Provincial Key Laboratory for Genetics and Breeding of Banana, Haikou 570102, China
由于各种生物和非生物胁迫的影响造成粮食产量下降,因此需要最大限度地减少这些损失,根据气候变化,确保粮食安全。环境的非生物胁迫,如干旱、极端温度、寒冷、重金属或高盐,严重损害植物生长和生产力。干旱是最重要的环境胁迫,超过任何其他环境因素,严重损害植物的生长和发育,限制植物生产和作物的性能[1]。
基因芯片是生物芯片的一种,又可以叫做DNA芯片、DNA微阵列(DNA microarray)或寡核苷酸微阵列(oligonucleotide microarray)。它的工作原理就是杂交测序的方法[2]。基因芯片技术是沟通基因序列信息与功能基因组间的纽带,目前,在植物研究中,基因芯片技术主要广泛应用于基因检测、基因表达水平检测、基因突变及多态性分析、功能基因组研究等。近年来基因组芯片技术开始应用于对干旱胁迫诱导下基因的变化及表达模式的研究[3-10]。在植物中对非生物胁迫的研究,是研究植物如何感知外界胁迫和适应非生物胁迫条件,然后利用这些知识来提高植物和作物对非生物胁迫的耐受性。高通量的筛选技术,芯片分析在模式植物和重要农业作物品种中已被用于高效剖析非生物胁迫包括干旱胁迫应答基因[11-16]。基因芯片技术推动了在非生物胁迫过程中对基因调控网络的研究,全基因组测序和染色质免疫沉淀技术推动了在胁迫条件下对基因表达的表观遗传控制的研究[17]。
我们从香蕉果实的cDNA文库中获得的一个ASR基因(MaASR1,登录号为AY628102),本实验室前期的研究表明香蕉植株在干旱胁迫条件下,MaASR1基因在根部和叶片中的表达量大幅上升,说明MaASR1基因主要参与植物在面临干旱胁迫时的应答反应[18]。为了深入研究MaASR1基因在香蕉干旱胁迫中发挥作用的抗性机制,将MaASR1基因转入模式植物拟南芥中,获得了转基因纯合株系L14 [18]。L14在干旱胁迫条件下的存活率优于野生型拟南芥植株[18]。转基因株系中,MaASR1基因的表达量越大,则该株系表现出来的抗旱能力则越强[18-19]。这些证据都能说明MaASR1基因的转入能够提高拟南芥的抗旱性[18]。为了深入研究MaASR1基因的过表达使拟南芥抗旱的分子机制,运用芯片技术(DNA microarray)来广谱的筛选MaASR1基因转入后自然条件下及干旱处理条件下差异基因的表达情况,以期能进一步的解析MaASR1基因发挥作用的调控机制。
1 材料与方法 1.1 材料野生型拟南芥(Arabidopsis thaliana. Columbia ecotype)购自Arabidopsis Biological Resource Center (Ohio university, Ohio State, USA)。转MaASR1基因拟南芥L14株系由本实验室前期转化所得[13]。
1.2 方法 1.2.1 对野生型和MaASR1转基因籽苗的干旱处理将野生型拟南芥和MaASR1转基因株系L14的种子播种于MS固体培养基中,然后在4℃条件下黑暗处理3d进行春化处理,之后将其转移到人工气候培养箱中培养。培养温度条件为21~23℃,每日光照时间为8h,黑暗时间为16h(短日照状态)。光照强度为2 000lx,培养箱相对湿度为70%。在籽苗生长15d后,选择生长出2~4片叶片的籽苗,将根部的培养基洗净。将各株系籽苗表面的水珠擦干,分别放在3张干燥的滤纸上,这样可以让滤纸自然吸收籽苗的水分,取两个培养皿盖在滤纸上方,防止蒸发带来的水分流失,然后把滤纸放置在人工气候培养箱中培养。培养条件与上面的相同,进行模拟干旱条件处理。分别在处理0h、2h和6h时各取0.2g材料,迅速的放置于液氮中冷冻,在-70℃存放[18]。
1.2.2 野生型和MaASR1转基因拟南芥株系的荧光定量PCR取1.2.1中干旱处理各阶段的野生型和转MaASR1基因拟南芥植株,用以提取RNA。各株系拟南芥材料总RNA的提取采用QIAGEN plant RNA Kit,具体方法参照该试剂盒说明书。然后用Fermentas的RevertAidTM First Strand cDNA Synthesis Kit试剂盒以上面提取的RNA为模板合成cDNA的第1条链。DREB2A荧光定量PCR的引物序列为F:5′-ATAACGAGACCGTAGAAGA-3′;R:5′-GCCAAAGGACCATACATAG-3′。以拟南芥看家基因Atactin(基因登录号AK318637)为内参,荧光定量PCR的引物序列为F:5′-CCAACAGAGAGAAGATGA-3′; R:5′-ATGTCTCTTACAATTTCCCG-3′。
1.2.3 表达谱芯片的制作和分析提取转MaASR1基因的L14与野生型拟南芥植株的RNA,通过博奥生物有限公司制作MaASR1转基因拟南芥L14与野生型在未作任何处理的表达谱芯片,命名为14 vs WT,其中14代表转基因株系L14,WT代表野生型拟南芥;干旱处理2h的MaASR1转基因拟南芥L14与野生型表达谱芯片,命名为Y vs W,其中Y代表转基因株系L14,W代表野生型拟南芥。每个芯片重复3次,以减少不同株系之间的差异所造成的误差。并对两种株系得到的芯片结果进行初步的基因筛选和分析。
实验中所采用的基因芯片为29k Arabidopsis Genome Array,共有29 110条70mer长度的Oligo DNA,来自于Operon公司的The Arabidopsis thaliana Genome Oligo Set Version 3.0,共代表了约26 173个基因,28 964个转录本。然后把此Oligo库用SmartTM Array (CapitalBio Corp., Beijing, China)点制在一张75mm×25mm、经过化学修饰的载玻片上。点制在芯片上的样品还包括拟南芥的11个看家基因作为阳性对照,Hex作为点样阳性对照,点样溶液50% DMSO作为阴性对照,以及酵母的8个基因间序列作为外标。整个点阵分成48个亚阵,每个亚阵有25列,26行,点间距为165μm,点的直径约为100μm。
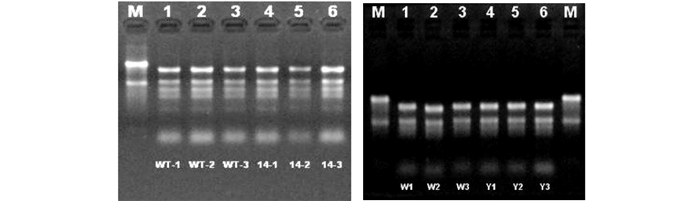
2 结果与分析 2.1 表达谱芯片RNA质量检测对未经任何处理的野生型和转基因(MaASR1)拟南芥幼苗,用Trizol方法提取RNA,RNA完整性如图 1所示,对经过2h干旱处理的野生型和转基因(MaASR1)拟南芥幼苗,用Trizol方法提取RNA,RNA完整性如图 1所示。
|
| 图 1 拟南芥株系RNA完整性检测 Figure 1 The RNA integrity testing of Arabidopsis (a) The RNA integrity testing of untreated Arabidopsis wild-type and transgenic lines (b) The RNA integrity testing of drought treated Arabidopsis wild-type and transgenic lines |
RNA完整性:RNA样品电泳条带清晰,26S:18S rRNA条带亮度接近1:1。符合表达谱芯片实验要求。
2.2 表达谱芯片RNA浓度和总量的检测对未经任何处理的野生型和转基因(MaASR1)拟南芥幼苗,用Trizol方法提取RNA,RNA纯度和总量如表 1所示。对经过2h干旱处理的野生型和转基因(MaASR1)拟南芥幼苗,用Trizol方法提取RNA,RNA纯度和总量如表 2所示。RNA纯度:RNA样品A260/280>1.80。RNA总量:RNA样品总量>8μg。符合表达谱芯片实验要求。
| 序号 | 样品编号 | A260/280 | A260/230 | 浓度 (μg/μl) |
总量 (μg) |
| 1 | WT-1 | 2.04 | 1.37 | 0.924 | 92.5 |
| 2 | WT-2 | 2.03 | 1.52 | 1.116 | 111.6 |
| 3 | WT-3 | 2.01 | 1.44 | 0.656 | 65.6 |
| 4 | 14-1 | 2.02 | 1.49 | 0.574 | 57.4 |
| 5 | 14-2 | 2.03 | 1.41 | 0.562 | 56.2 |
| 6 | 14-3 | 1.98 | 1.22 | 0.453 | 45.3 |
| 序号 | 样品编号 | A260/280 | A260/230 | 浓度 (μg/μl) |
总量 (μg) |
| 1 | WT1 | 2.01 | 1.04 | 1.302 | 84.62 |
| 2 | WT2 | 2.03 | 1.48 | 1.051 | 73.59 |
| 3 | WT3 | 2.02 | 1.59 | 1.491 | 96.90 |
| 4 | Y1 | 2.03 | 1.38 | 0.995 | 64.70 |
| 5 | Y2 | 2.00 | 1.42 | 0.911 | 59.21 |
| 6 | Y3 | 2.01 | 1.36 | 1.044 | 67.86 |
2.3 表达谱芯片差异基因聚类分析
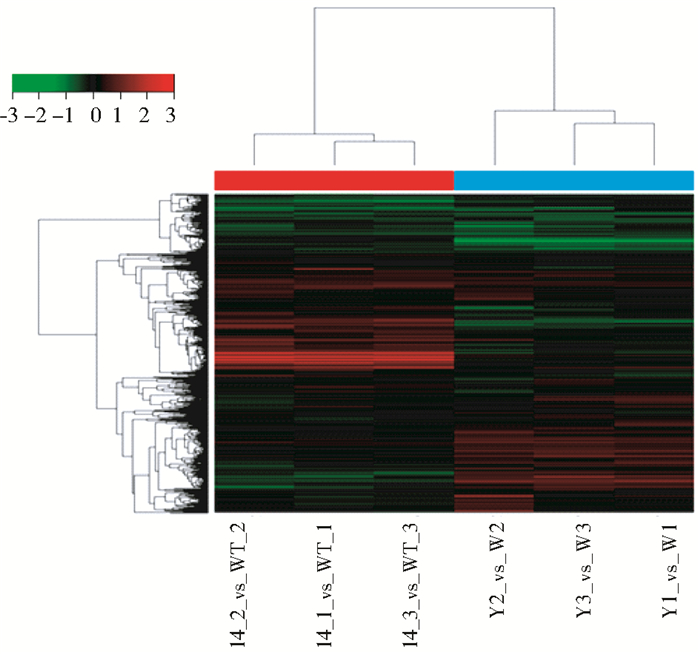
如图 2所示为6张表达谱芯片差异基因聚类图,从左到右依次为14_2_vs_WT_2、14_1_vs_WT_1、14_3_vs_WT_3、Y2_vs_W2、Y3_vs_W3、Y1_vs_W1。前3张芯片为自然条件下野生型拟南芥与转基因(MaASR1)拟南芥14号株系的基因表达谱的变化图,后3张芯片为经2h干旱胁迫处理的野生型拟南芥与转基因(MaASR1)拟南芥14号株系的基因表达谱的变化图。
|
| 图 2 表达谱芯片差异基因聚类图 Figure 2 The cluster data of differential gene expression profile |
从图 2中可以看出不同生物学重复间基因表达差异不大,芯片的重复情况良好。未经任何处理的野生型拟南芥与转基因拟南芥基因表达变化与经过干旱处理的差异十分明显。红色代表表达上调大于2倍的基因,筛选标准为:Ratio≥ 2;绿色代表表达下调大于2倍的基因筛选标准为:Ratio≤ 0.5。14 vs WT一共得到747个差异基因,其中上调基因559个、下调基因188个;Y vs W一共得到653个差异基因,其中上调基因256个,下调基因397个。
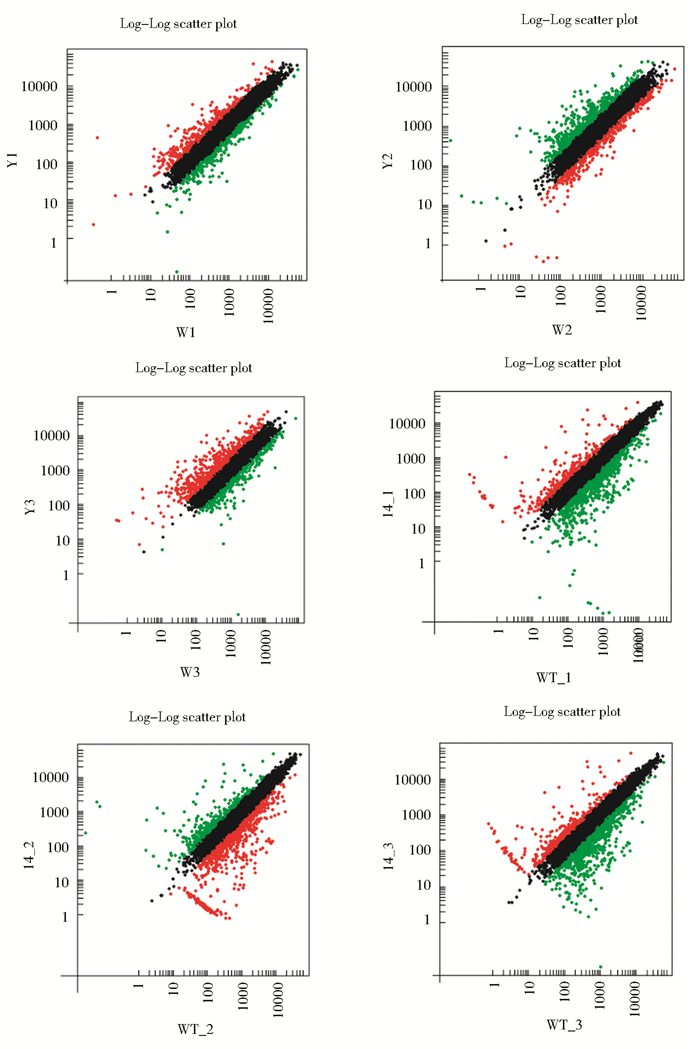
2.4 表达谱芯片样品比较散点分析如图 3所示,表示在同一张芯片中两个样品的比较分析散点图;可以比较直观地看出在野生型和MaASR1转基因型之间基因表达的差异情况。其中X轴和Y轴分别以野生型和转基因型的荧光信号强度值为坐标,图中每一个数据点代表所制作芯片上的一个基因点的杂交信号,红色标记表示Ratio值≥2的基因情况,绿色标记的数据点表示Ratio值≤0.5的基因,代表在选择的Ratio值范围内属于表达有显著差异的基因,黑色标记表示Ratio值在0.5~2,代表表达基因基本没有显著差异。
|
| 图 3 芯片样品比较分析散点图 Figure 3 The Scatter Plot of gene expression profile |
AraNet数据库采用修正的贝叶斯法预测拟南芥基因间的相互作用。数据库中总共收录了19 674个基因,涉及1 062 222个基因间的互作关系,将差异基因使用AraNet数据库映射,得到网络图结果,其中表达谱芯片14 vs WT差异基因集网络图如图 4所示,表达谱芯片Y vs W差异基因集网络图如图 5所示。由差异基因网络图中可以看出,图中红色节点代表上调基因,黄色节点代表下调基因Y vs W与14 vs WT相比,上调基因明显减少,下调基因明显增多。
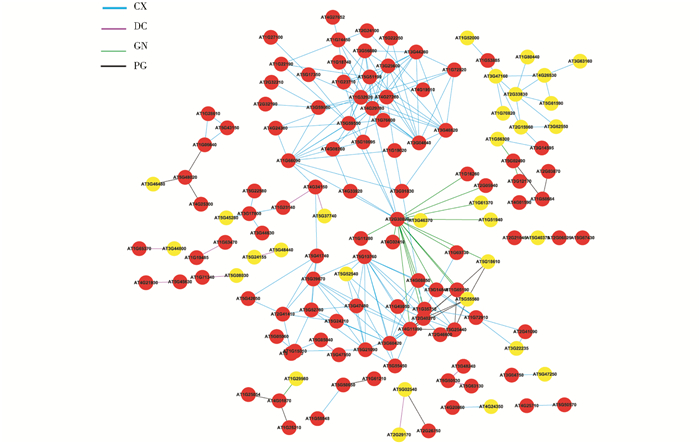
|
| 图 4 14 vs WT差异基因集网络图 Figure 4 The Network Diagram of differential gene in 14 vs WT CX: mRNA co-expression between Arabidopsis genes; DC: Domain co-occurrence between Arabidopsis proteins; GN: Gene neighborhoods between Arabidopsis orthologs in bacterial genomes; LC: Literature-curated Arabidopsis protein interactions; PG: Phylogenetic profile similarity between Arabidopsis homologs The red nodes represent the up-regulated genes, and the yellow nodes represent the downregulated genes |
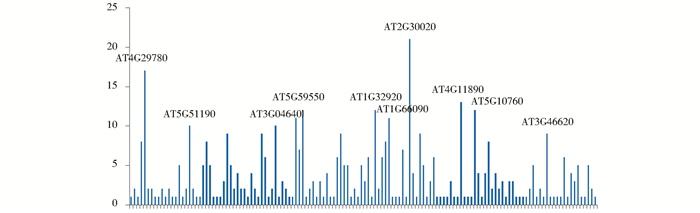
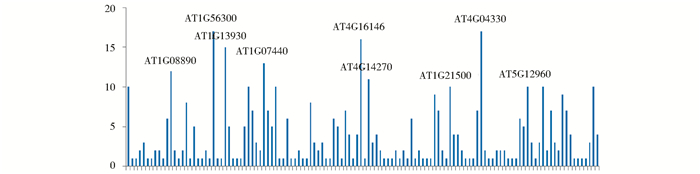
由图 6中可以看出,其中AT2G30020、AT4G29780、AT4G11890网络连接度较高。在14 vs WT芯片中,把连接度最高的基因整理汇总见表 3。
|
| 图 6 14 vs WT网络连接度分析结果 Figure 6 The result analysis of network connectivity in 14 vs WT |
| 基因登录号 | 基因生物学功能 |
| At4g29780 | similar to unknown protein [Arabidopsis thaliana] |
| At5g51190 | AP2 domain-containing transcription factor, putative |
| At3g04640 | glycine-rich protein |
| At5g59550 | zinc finger (C3HC4-type RING finger) family protein |
| At1g32920 | similar to unknown protein [Arabidopsis thaliana] |
| At1g66090 | "disease resistance protein (TIR-NBS class), putative" |
| At2g30020 | "protein phosphatase 2C, putative / PP2C, putative" |
| At4g11890 | protein kinase family protein |
| At5g10760 | aspartyl protease family protein |
| At3g46620 | zinc finger (C3HC4-type RING finger) family protein |
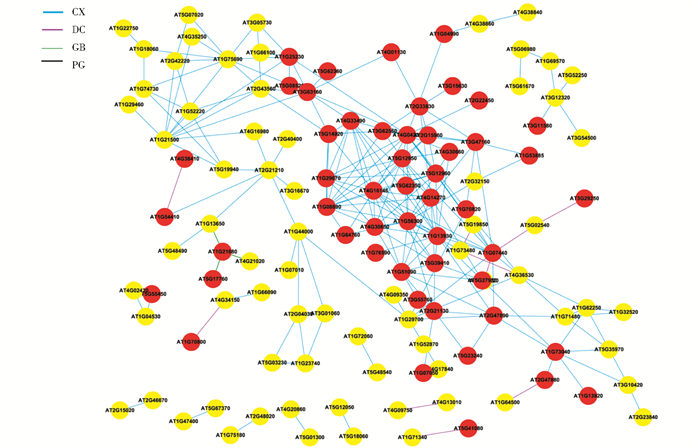
由图 7中可以看出,其中AT1G56300、AT4G16146、AT4G04330等基因连接度较高。在Y vs W芯片中,把连接度最高的基因整理汇总见表 4。
|
| 图 7 Y vs W网络连接度分析结果 Figure 7 The result analysis of network connectivity in Y vs W |
| 基因登录号 | 基因生物学功能 |
| At1g08890 | Sugar transporter family protein |
| At1g56300 | DNAJ heat shock N-terminal domain-containing protein |
| AT1G13930 | Similar to nodulin-related [Arabidopsis thaliana]; similar to drought-induced protein SDi-6 -common sunflower (fragment) |
| AT1G07440 | Oxidoreductase |
| At4g16146 | "Similar to unknown protein [Arabidopsis thaliana]; similar to negatively light-regulated protein, putative, expressed [Oryza sativa (japonica cultivar-group)]; contains InterPro domain Lg106-like; " |
| At4g14270 | Protein containing PAM2 motif which mediates interaction with the PABC domain of polyadenyl binding proteins. |
| At1g21500 | Similar to conserved hypothetical protein [Medicago truncatula] "1-phosphatidylinositol-4-phosphate kinase, class IA complex|GO:0005943" |
| AT4G04330 | Similar to unnamed protein product [Ostreococcus tauri] |
| At5g12960 | Similar to unknown protein [Arabidopsis thaliana] |
2.5.2 Gene ontology(GO)分析
在GO数据库中包含了基因参与的生物过程,基因所处的细胞位置和基因发挥的分子功能三方面的功能信息,并将概念粗细不同的功能概念组织成DAG(有向无环图)的结构。在基因表达谱分析中,GO常用于提供基因功能分类标签和基因功能研究的背景知识。
(1) 14 vs WT依据参与的生物过程分类(图 8)。
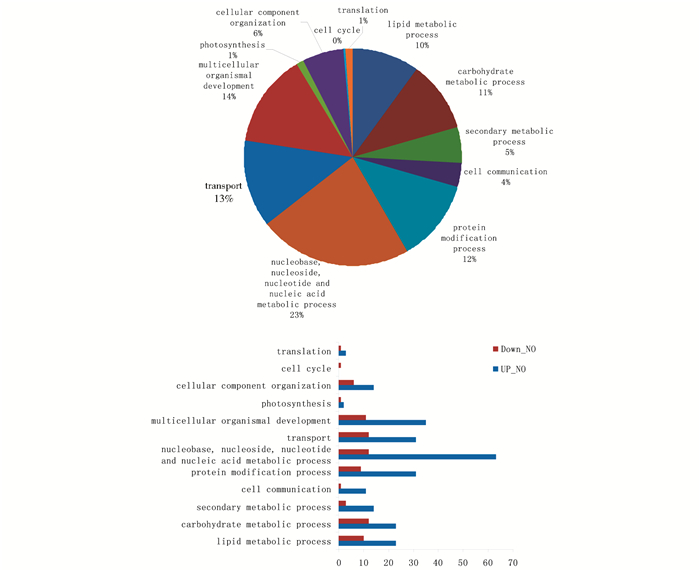
|
| 图 8 依据参与的生物过程分类结果图(14 vs WT) Figure 8 The result of 14 vs WT according to classification of the participation in the biological process (14 vs WT) |
(2) 14 vs WT依据定位分类(图 9)。
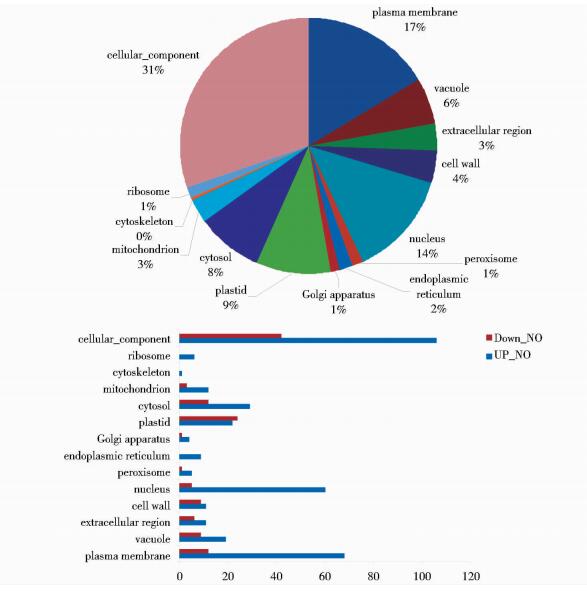
|
| 图 9 依据在细胞组分中的定位分类结果图(14 vs WT) Figure 9 The result of 14 vs WT according to the classification of the position in the cellular component (14 vs WT) |
(3) 14 vs WT依据生物学功能分类(图 10)。
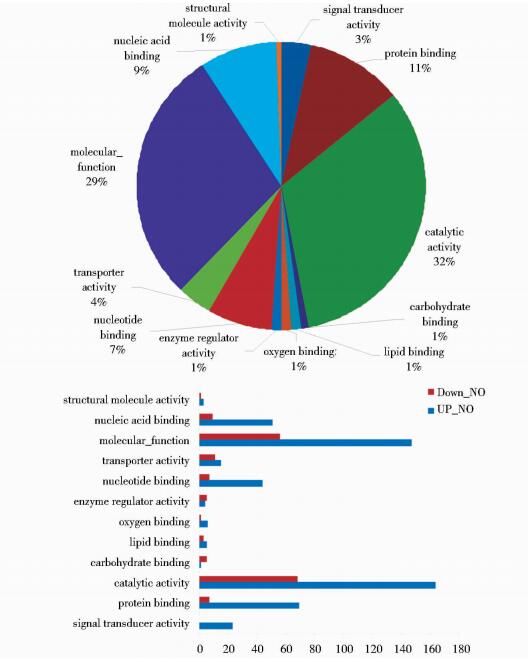
|
| 图 10 依据生物学功能分类结果图(14 vs WT) Figure 10 The result of 14 vs WT according to the classification of its molecular biological function (14 vs WT) |
(4) Y vs W依据参与的生物过程分类(图 11)。
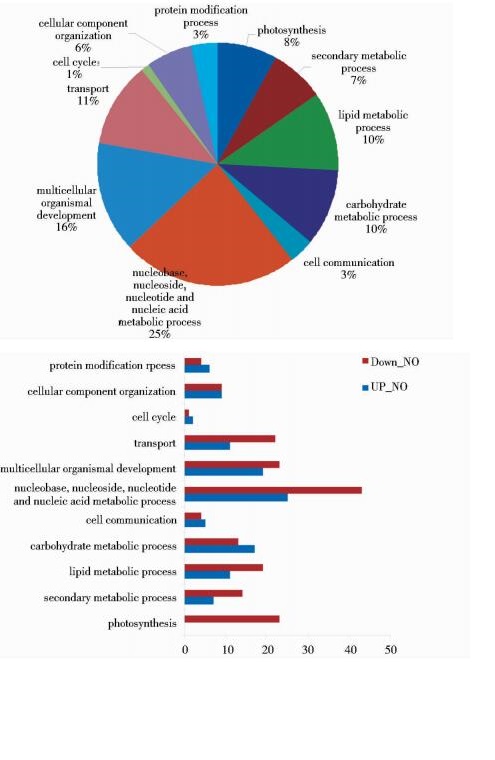
|
| 图 11 依据参与的生物过程分类结果图(Y vs W) Figure 11 The result of Y vs W according to classification of the participation in the biological process (Y vs W) |
(5) Y vs W依据定位分类(图 12)。
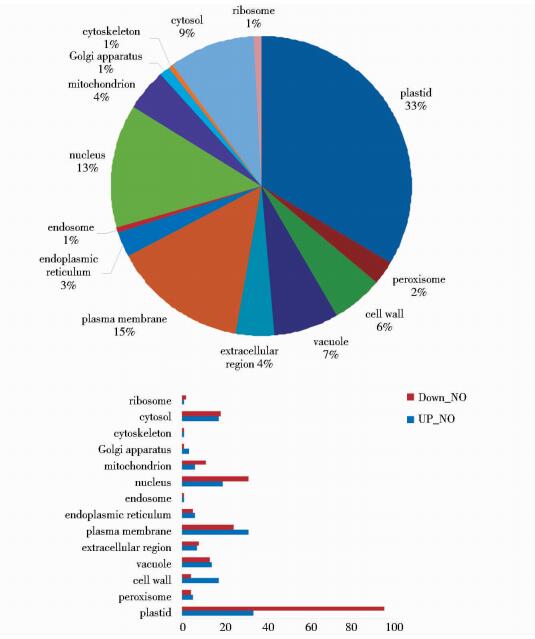
|
| 图 12 依据在细胞组分中的定位分类结果图(Y vs W) Figure 12 The result of Y vs W according to the classification of position in the cellular component (Y vs W) |
(6) Y vs W依据生物学功能分类(图 13)。
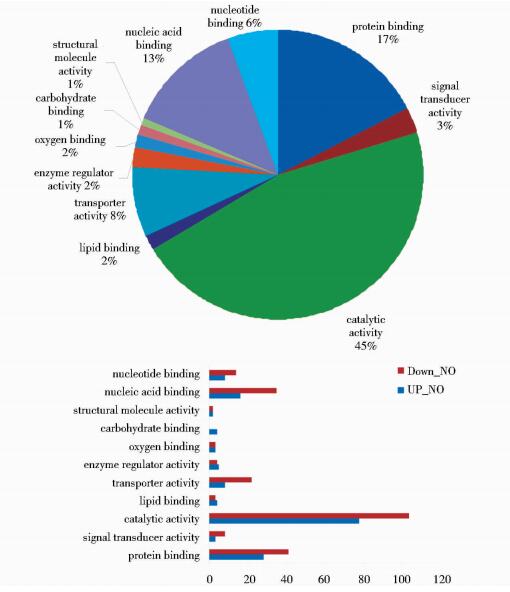
|
| 图 13 依据生物学功能分类结果图(Y vs W) Figure 13 The result of Y vs W according to the classification of its molecular biological function (Y vs W) |
依据参与的生物过程分类,Y vs W与14 vs WT相比如图 8, 图 11所示,参与蛋白质修饰(protein modification progress)的差异基因百分比由12%降到3%,而参与光合作用(photosynthesis)的差异基因百分比由1%升到8%。其他生物过程基因表达所占的比例变化不明显。与之相对应的上调及下调差异基因的表达情况为:参与蛋白质修饰的上调差异基因由31个变为6,下调差异基因由9个变为4个;参与光合作用的上调差异基因由2个变为0个,下调差异基因由1个变为23个。
依据定位分类,Y vs W与14 vs WT相比如图 9, 图 12所示,定位在质体(plastid)的差异基因百分比由9%升到33%,质体包含叶绿体、有色体和白色体。差异基因在其他器官的定位变化不明显。与之相对应的上调及下调差异基因的表达情况为:定位在质体(plastid)的上调差异基因由22个变为33,下调差异基因由24个变为94个。
依据生物学功能分类,Y vs W与14 vs WT相比如图 10, 图 13所示,有转运活性(transporter activity)的差异基因百分比由4%升到8%,有催化活性(catalytic activity)的差异基因百分比由32%升到45%,有蛋白质结合活性(protein binding)的差异基因百分比由11%升到17%。其他生物学功能差异基因表达所占的百分比变化不明显。与之相对应的上调及下调差异基因的表达情况为:有转运活性的上调差异基因由15个变为8,下调差异基因由11个变为22个;有催化活性的上调差异基因由164个变为78个,下调差异基因由68个变为104个。有蛋白质结合活性上调差异基因由69个变为28个,下调差异基因由7个变为41个。
2.5.3 Pathway分析14 vs WT差异基因集:与GO分析类似,我们同样将差异基因使用KEGG API向KEGG pathway数据库映射。共找到61个相关的pathway,主要涉及淀粉和蔗糖的代谢、磷酸戊糖途径、糖酵解及糖异生、植物激素信号转导(9个基因)等途径。
Y vs W差异基因集:与GO分析类似,我们同样将差异基因使用KEGG API向KEGG pathway数据库映射。共找到58个相关的pathway,主要涉及植物激素信号转导(共20个基因)、光合作用(12个基因)、叶绿素代谢、植物生长节律等代谢途径。
2.6 表达谱芯片中不依赖ABA途径基因验证DREB2A(DRE-BINDING PROTEIN 2A)为特异识别DRE/CRT响应元件的转录因子,它受干旱和盐胁迫诱导表达,但不对ABA应答,代表一条不依赖于ABA的胁迫应答途径[29]。从芯片中得到该胁迫应答转录因子DREB2A,利用RT-PCR检测野生型及MaASR1转基因拟南芥干旱处理不同时间内及在不做任何处理的条件下DREB2A的表达变化,来进行芯片结果的验证。
结果如图 14所示,在未受干旱胁迫时,DREB2A在MaASR1转基因拟南芥的基因表达量高于野生型,表达量是野生型的1.82倍;在干旱胁迫2h时,DREB2A在MaASR1转基因拟南芥的基因表达量也高于野生型,表达量是野生型的2.99倍;而在干旱胁迫6h时,DREB2A在MaASR1转基因拟南芥中的基因表达量则低于野生型,表达量是转基因型的1.93倍。
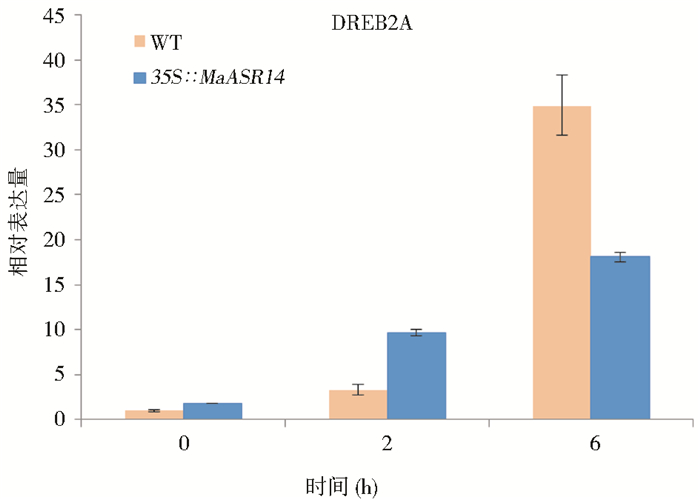
|
| 图 14 表达谱芯片中野生型及MaASR1转基因拟南芥DREB2A的表达 Figure 14 The expression of DREB2A of expression profile microarray in wild-type and MaASR1 transgenic lines |
在受到干旱胁迫时,野生型和转基因拟南芥中DREB2A的表达量随着胁迫程度的增加而大幅提高,DREB2A在野生型和转基因拟南芥中的基因表达量变化趋势一致。在干旱胁迫2h和6h时,DREB2A在野生型拟南芥中的表达量比未受胁迫时分别提高了3.23倍和34.88倍;在转基因型中分别提高了5.32倍和9.95倍。
3 讨论干旱可以引发植物大范围的响应,从细胞的新陈代谢、生长率到作物产量的变化。了解植物对干旱胁迫的生化和分子响应对研究在缺水条件下植物的抗逆机制至关重要。植物在分子、细胞和整个植株水平上响应和适应干旱是通过激活一系列被网络基因控制的生理生化反应。植物在干旱胁迫下主要的分子响应是基因表达的改变,这些基因根据推测的功能模式分为两大类[20]。第一组包括在信号级联反应和转录控制方面的基因,如植物激素、蛋白激酶、磷酸酶和转录因子(TF);第二组包含的是保护植物细胞免受外界胁迫的基因,如脱水蛋白、热激蛋白(HSPs)、衰老相关基因,以及渗透保护剂,膜保护剂,转运蛋白和抗氧化剂。
对表达谱芯片的生物信息学分析中,依据参与的生物过程分类(图 8, 图 11)发现,Y vs W与14 vs WT相比,参与光合作用的相关基因明显增多,主要是下调基因的数量明显增多。植物光合作用可以将无机物质二氧化碳和水转化为有机物, 同时将太阳光能转化为化学能, 是地球上最重要的化学反应, 也是绿色植物最为敏感的生理过程之一。水分是植物通过光合作用来制造有机物质的重要原料之一,作物内营养物质的吸收和转运都离不开水分,同时水分是植物本身最大的组成部分, 它给植物的蒸腾作用用以调节植株的整个生理过程。水分胁迫是抑制植物光合作用的最主要的环境因素之一。水分胁迫对植物生长和代谢的影响是多方面的,但对光合作用的影响则尤为突出和重要。植物对水分亏缺的最初反应是通过减少气孔孔径来调节气孔的开度,使气孔开度减小,气孔的阻力就会相应的增大,限制了植物对CO2的吸收,致使光合作用减弱。这样就可以降低植物的蒸腾作用,从而减少水分散失以达到保水的目的。随干旱程度的增加,非气孔性限制就会发挥主要作用[21],非气孔性限制是指水分胁迫造成光合酶的活性降低,光系统Ⅰ和光系统Ⅱ的活力下降,叶绿体的结构改变,叶绿素合成的速率减慢,最终表现为光合作用的下降[22]。
表达谱芯片所得到的光合作用相关基因的表达情况表明,在干旱胁迫条件下,MaASR1转基因拟南芥与野生型相比,与光合作用相关的光系统,光系统Ⅰ(LHCA1、LHCA4、LHCA6、PSAN、PSAK、PSAD2等)和光系统Ⅱ(LHB1B1、LHB1B2、LHCB3、LHCB6等)基因的表达量明显下降,质体(主要是叶绿体)(图 9, 图 12)相关基因表达量显著下降,还有RUBISCO活化酶基因的表达量也明显下降,这样就会影响最初的光能转换,电子传递速率下降,CO2同化效率降低,最终导致同化产物的减少。光合速率的降低可以降低蒸腾作用以保留水分,在一定程度上提高了植物的抗旱性,同时造成同化产物的减少,也就相应的造成了作物产量与野生型相比有一定的下降。有研究表明[23] OsbZIP23基因的过量表达会提高水稻对干旱和高盐的耐受能力,在正常情况下转基因的结实率与野生型相比却降低了20%,但在受到干旱胁迫时,野生型水稻的结实率只有36%左右,而转基因型水稻的结实率却保持在60%以上,差异极显著。这说明植物抗逆性的获得通常会伴随产量的少量下降,但是野生型在受到胁迫时产量与转基因型相比却大幅下降。
依据生物信息学的Pathway分析可以看出,MaASR1基因可以通过调节植物激素主要包括ABA、乙烯和生长素等相关基因的表达来赋予植物抗旱性。具体如何通过调节激素相关基因的表达来提高植物的抗旱性,还需要下一步更深入的研究。
在14 vs WT芯片链接度最高的基因中就有2个锌指蛋白家族成员zinc finger (C3HC4-type RING finger) family protein如图 5、表 3所示,在14 vs WT芯片结果中有8个C3HC4型锌指蛋白基因表达上调,有1个C3HC4型锌指蛋白基因下调。这表明香蕉MaASR1基因的转入大大提高了C3HC4型锌指蛋白基因的表达水平。说明其可以通过调控C3HC4型锌指蛋白基因的表达水平来赋予植物较强的耐旱能力。具体的调控方式还有待进一步的研究。研究表明一些ASR蛋白同C3HC4型锌指蛋白一样包含一个锌指DNA结合域[24],可以以单体或者同源二聚体的形式与DNA结合来发挥其调控作用。MaASR1和C3HC4型锌指蛋白都是作为转录因子来发挥作用的,推测它们可能通过蛋白质之间的互作来发挥其调控作用。
锌是植物必需的营养元素,锌指蛋白是因为其具有指状结构特征并且能够结合锌离子而得名。锌的存在可以用来维持锌指结构的稳定性,它提供了一个由不同肽序列折叠形成的完美的螺旋构象。锌指蛋白在真核生物的基因组中是含量最为丰富的一类转录因子, 根据转录因子中两种氨基酸即半胱氨酸(C)和组氨酸(H)残基的数目和位置, 可以将含锌指结构域的转录因子划分为C2H2、C2C2、C3H、C3HC4 (RING finger)和C3HC5(LIM finger) 5个亚类[25]。真核生物中基因表达的调控是分子生物学研究的热点与前沿,其基因调控主要是在转录水平上来进行。锌指蛋白作为转录因子来调控基因的转录,锌指蛋白通过与生物大分子如DNA、RNA结合来实现其对基因的转录调控,或者通过蛋白质之间的互作来直接调控基因转录。越来越多的研究表明, 锌指蛋白在调节植物防卫基因表达和抗性反应上起着很关键的作用,Sakamoto等[26]发现锌指蛋白AZF2和STZ在干旱、高盐、冷和ABA的诱导下表达显著提高,转STZ基因的拟南芥与野生型相比表现出较强的抗干旱能力。棉花锌指蛋白GhZFP1基因的过量表达显著提高了转基因烟草的耐盐性和抗病性[27]。吴学闯等[28]研究发现大豆C3HC4型锌指蛋白基因GmRZFP1除受ABA、高盐、干旱及低温胁迫诱导表达外, 还受乙烯、高温的诱导。
DREB代表一条不依赖ABA的胁迫应答途径,转录因子DREB2A(DEHYDRATION-RESPONSIVE ELEMENT BINDING PROTEIN2A)可以与顺式作用元件dehydration-responsive element/C-repeat(DRE/CRT)结合参与调控拟南芥干旱胁迫应答基因的表达,增强植物的耐旱性[29]。在培养皿中生长15天的拟南芥幼苗在干旱胁迫2h时,拟南芥的幼苗叶片还未发生卷曲,而在干旱胁迫6h时,拟南芥的幼苗叶片已经完全卷曲萎蔫,根部也已经干燥。在干旱胁迫2h条件下,DREB2A在MaASR1转基因拟南芥株系中的表达量明显高于野生型。而在在干旱胁迫6h条件下,DREB2A在MaASR1转基因拟南芥株系中的表达量却低于野生型。这一结果表明,MaASR1的转入不只提高了干旱条件下DREB2A的表达水平,并且在拟南芥还未出现明显的干旱胁迫症状时促使DREB2A对干旱胁迫的应答时间提前,说明MaASR1可能是通过调控不依赖ABA信号通路的胁迫信号转导途径来调控植物的抗旱应答,进而赋予植物抗旱能力。
综上所述,MaASR1基因的转入可以通过影响激素、光合作用、锌指蛋白及不依赖ABA途径的DREB2A等相关基因的表达来提高拟南芥的抗旱性。这为进一步解析MaASR1基因作为转录因子提高植物抗旱能力的分子机制奠定基础。
| [1] |
Shao H B, Chu L Y, Jaleel C A, et al. Understanding water deficit stress-induced changes in the basic metabolism of higher plants-biotechnologically and sustainably improving agriculture and the ecoenvironment in arid regions of the globe. Crit Rev Biotechnol, 2009, 29(2): 131-151. DOI:10.1080/07388550902869792 |
| [2] |
Ramsay G. DNA chips:State-of-the-art. Nat Biotechnol, 1998, 16(1): 40-44. DOI:10.1038/nbt0198-40 |
| [3] |
Seki M, Narusaka M, Ishida J, et al. Monitoring the expression profiles of 7000 Arabidopsis genes under drought, cold and high-salinity stresses using a full-length cDNA microarray. Plant J, 2002, 31(3): 279-292. DOI:10.1046/j.1365-313X.2002.01359.x |
| [4] |
Kreps J A, Wu Y J, Chang H S, et al. Transcriptome changes for Arabidopsis in response to salt, osmotic, and cold stress. Plant Physiol, 2002, 130(4): 2129-2141. DOI:10.1104/pp.008532 |
| [5] |
Brosche M, Vinocur B, Alatalo E R, et al. Gene expression and metabolite profiling of Populus euphratica growing in the Negev desert. Genome Biol, 2005, 6(12): R101. DOI:10.1186/gb-2005-6-12-r101 |
| [6] |
Street N R, Skogstrom O, Sjodin A, et al. The genetics and genomics of the drought response in Populus. Plant J, 2006, 48(3): 321-341. DOI:10.1111/tpj.2006.48.issue-3 |
| [7] |
Wang H G, Zhang H L, Gao F H, et al. Comparison of gene expression between upland and lowland rice cultivars under water stress using cDNA microarray. Theor Appl Genet, 2007, 115(8): 1109-1126. DOI:10.1007/s00122-007-0637-7 |
| [8] |
Hayano-Kanashiro C, Calderon-Vazquez C, Ibarra-Laclette E, et al. Analysis of gene expression and physiological responses in three mexican maize landraces under drought stress and recovery irrigation. PLoS One, 2009, 4(10): e7531. DOI:10.1371/journal.pone.0007531 |
| [9] |
Wilkins O, Waldron L, Nahal H, et al. Genotype and time of day shape the Populus drought response. Plant J, 2009, 60(4): 703-715. DOI:10.1111/tpj.2009.60.issue-4 |
| [10] |
Berta M, Giovannelli A, Sebastiani F, et al. Transcriptome changes in the cambial region of poplar (Populus alba L.) in response to water deficit. Plant Biology, 2010, 12(2): 341-354. DOI:10.1111/j.1438-8677.2009.00320.x |
| [11] |
Ji S J, Lu Y C, Feng J X, et al. Isolation and analyses of genes preferentially expressed during early cotton fiber development by subtractive PCR and cDNA array. Nucleic Acids Res, 2003, 31(10): 2534-2543. DOI:10.1093/nar/gkg358 |
| [12] |
Kawaguchi R, Girke T, Bray E A, et al. Differential mRNA translation contributes to gene regulation under non-stress and dehydration stress conditions in Arabidopsis thaliana. Plant J, 2004, 38(5): 823-839. DOI:10.1111/tpj.2004.38.issue-5 |
| [13] |
Zhou J L, Wang X F, Jiao Y L, et al. Global genome expression analysis of rice in response to drought and high-salinity stresses in shoot, flag leaf, and panicle. Plant Mol Biol, 2007, 63(5): 591-608. DOI:10.1007/s11103-006-9111-1 |
| [14] |
Aprile A, Mastrangelo A M, De Leonardis A M, et al. Transcriptional profiling in response to terminal drought stress reveals differential responses along the wheat genome. Bmc Genomics, 2009, 10: 279. DOI:10.1186/1471-2164-10-279 |
| [15] |
Cohen D, Bogeat-Triboulot M B, Tisserant E, et al. Comparative transcriptomics of drought responses in Populus:a meta-analysis of genome-wide expression profiling in mature leaves and root apices across two genotypes. Bmc Genomics, 2010, 11: 630. DOI:10.1186/1471-2164-11-630 |
| [16] |
Gong P J, Zhang J H, Li H X, et al. Transcriptional profiles of drought-responsive genes in modulating transcription signal transduction, and biochemical pathways in tomato. J Exp Bot, 2010, 61(13): 3563-3575. DOI:10.1093/jxb/erq167 |
| [17] |
Mittler R, Blumwald E. Genetic engineering for modern agriculture:challenges and perspectives. Annual Review of Plant Biology, 2010, 61: 443-462. DOI:10.1146/annurev-arplant-042809-112116 |
| [18] |
王园. 香蕉ASR基因抗逆功能的研究. 海南: 海南大学, 2010. Wang Y. Study of Function of MaASR1 Tolerance to Drought and Salt Resistance. Hainan:Hainan University, 2010. |
| [19] |
苗红霞, 王园, 徐碧玉, 等. 香蕉MaASR1基因的抗干旱作用. 植物学报, 2014, 49(5): 548-559. Miao H X, Wang Y, Xu B Y, et al. The role of banana MaASR1 in drought stress tolerance. Chinese Bulletin of Botany, 2014, 49(5): 548-559. |
| [20] |
Shinozaki K, Yamaguchi-Shinozaki K. Gene networks involved in drought stress response and tolerance. J Exp Bot, 2007, 58(2): 221-227. |
| [21] |
姚庆群, 谢贵水. 干旱胁迫下光合作用的气孔与非气孔限制. 热带农业科学, 2005, 25(4): 80-85. Yao Q Q, Xie G S. The photosynthetic stomatal and nonstomatal limitation under drought stress. Chinese Journal of Tropical Agriculture, 2005, 25(4): 80-85. |
| [22] |
Earl H J. Stomatal and non-stomatal restrictions to carbon assimilation in soybean (Glycine max) lines differing in water use efficiency. Environ Exp Bot, 2002, 48(3): 237-246. DOI:10.1016/S0098-8472(02)00041-2 |
| [23] |
向勇. 水稻抗逆境相关基因的分离和功能分析. 武汉: 华中农业大学, 2008. Xiang Y. Isolation and Functional Characterization of Rice Stress-related Genes. Wuhan:Huazhong Agricultural University, 2008. |
| [24] |
Kalifa Y, Gilad A, Konrad Z, et al. The water-and salt-stress-regulated Asr1(abscisic acid stress ripening) gene encodes a zinc-dependent DNA-binding protein. Biochem J, 2004, 381: 373-378. DOI:10.1042/BJ20031800 |
| [25] |
刘强, 张贵友, 陈受宜. 植物转录因子的结构与调控作用. 科学通报, 2000, 45(14): 1465-1474. Liu Q, Zhang G Y, Chen S Y. Structure and regulation of plant transcription factors. Chinese Science Bulletin, 2000, 45(14): 1465-1474. DOI:10.3321/j.issn:0023-074X.2000.14.002 |
| [26] |
Sakamoto H, Maruyama K, Sakuma Y, et al. Arabidopsis Cys2/His2-type zinc-finger proteins function as transcription repressors under drought, cold, and high-salinity stress conditions. Plant Physiol, 2004, 136(1): 2734-2746. DOI:10.1104/pp.104.046599 |
| [27] |
郭英慧. 棉花CCCH型锌指蛋白基因GhZEP1的分离、功能鉴定及其作用机制的研究. 泰安: 山东农业大学, 2007. Guo Y H. Isolation and Function Identification of a Novel CCCH-type Zinc Finger Protein Gene GhZFP1 in Cotton. Taian:Shandong Agricultural University, 2007. |
| [28] |
吴学闯, 曹新有, 陈明, 等. 大豆C3HC4型RING锌指蛋白基因GmRZFP1克隆与表达分析. 植物遗传资源学报, 2010, 11(3): 343-348. Wu X C, 2, Cao X Y, Chen M, et al. Isolation and expression pattern assay of a C3HC4-type RING zinc finger protein gene GmRZFP1 in Glycinemax (L.). Journal of Plant Genetic Resources, 2010, 11(3):343-348. |
| [29] |
Sakuma Y, Maruyama K, Osakabe Y, et al. Functional analysis of an Arabidopsis transcription factor, DREB2A, involved in drought-responsive gene expression. Plant Cell, 2006, 18(5): 1292-1309. DOI:10.1105/tpc.105.035881 |
 2017, Vol. 37
2017, Vol. 37